Ablation to Allergen-epitopes of Cow's Milk Proteins by Proteases from Pitcher Fluid of Nepenthes×Mirabilis (Lour.) Druce
-
摘要: 目的:利用废弃猪笼液中的蛋白酶来消减牛乳主要致敏原蛋白的致敏性。方法:首先采集了米兰达猪笼草(Nepenthes×Miranda)的猪笼液,经过滤、5 kDa膜超滤、真空冷冻干燥和复溶获得酶活为126.276 U/mL的猪笼液蛋白酶液。然后用此酶对牛乳蛋白进行酶解,通过液相色谱-二级质谱法(LC-MS/MS)鉴定酶解后肽段产物,对比致敏原表位数据库以判断蛋白酶对表位的酶切情况。结果:牛乳主要致敏原蛋白中P1位的K、V、F、R、Y、I、S和L以及P1’位的D、A、V、I、L、F、E、R、Y、N、T、Q、S、W和P的肽键被有效酶解,酶切位点位于相对应的表位内,且位于蛋白表面和内部的表位均有被酶解。其中,来自α-LA的表位Eα-LA1中的K-V以及表位Eα-LA2中的K-V和K-A被酶解频率较高,来自β-LG的被酶解频率高的表位处的肽键为Eβ-LG1和Eβ-LG2中的L-D、V-Y和Y-V,来自αs1-CN的表位Eαs1-CN1中大部分的R-F和F-F被高频酶解,来自αs2-CN的表位Eαs2-CN1中的L-N、R-Y、K-F和K-T和表位Eαs2-CN2中的K-T、K-L和K-I被酶解频率较高,β-CN中被酶解频率较高的表位为Eβ-CN1和Eβ-CN2中的L-Q、S-W、D-V、L-T、T-D和E-N。结论:猪笼液蛋白酶水解对牛乳蛋白致敏原蛋白表位表现出不同程度的破坏作用,猪笼液有望成为新型蛋白酶开发的资源,实现农业废弃物再利用。Abstract: Objective: The aim of this study was to abate the allergenicity of the major allergenic proteins in cow's milk by utilizing the proteases in pitcher fluids of Nepenthes mirabilis (Lour.) Druce. Methods: The pitcher fluid of Nepenthes×Miranda was firstly collected, and the protease solution with the proteolytic activity of 126.276 U/mL was obtained by filtration, ultrafiltration with a 5 kDa membrane, vacuum freeze-drying and re-solubilization. Next, these enzymes were used to digest proteins of cow's milk, and the products were identified by liquid chromatography-tandem mass spectrometry (LC-MS/MS). Then the peptide products were compared with database of allergen-epitopes, so as to determine the cleavage of the allergen-epitopes. Results: The peptide bonds of K, V, F, R, Y, I, S as well as L in the P1 positions and D, A, V, I, L, F, E, R, Y, N, T, Q, S, W as well as P in the P1' positions of the major allergenic proteins in cow's milk were effectively digested, and the cleavage sites were located in the linear epitopes. What's more, both epitopes located on the surface of the proteins and those located in the interior were digested. Specifically, K-V in epitope Eα-LA1 from α-LA and K-V as well as K-A in epitope Eα-LA2 were cleaved at a high frequency. Peptide bonds at epitopes from β-LG which were cleaved at a high frequency were L-D, V-Y, and Y-V in Eβ-LG1 and Eβ-LG2. The most of R-F and F-F in epitope Eαs1-CN1 from αs1-CN were cleaved at high frequencies. What's more, L-N, R-Y, K-F as well as K-T in epitope Eαs2-CN1 from αs2-CN and K-T, K-L as well as K-I in epitope Eαs2-CN2 were digested with high frequency. And L-Q, S-W, D-V, L-T, T-D as well as E-N in epitopes Eβ-CN1 and Eβ-CN2 from β-CN were digested with high frequency. Conclusion: The proteases from pitcher fluid exhibited different destructive effects on the allergen-epitopes of cow's milk proteins. Besides, the pitcher liquid of Nepenthes×Miranda is expected to be the resource for the development of novel proteases and the reuse of agricultural wastes will be achieved.
-
食物过敏已成为全球性的重大食品安全问题之一[1]。牛乳蛋白过敏是在人类生命早期最容易出现的过敏反应,已受到越来越多的关注[2]。牛乳中的蛋白质高达20多种,主要包括大约80%的酪蛋白(casein,CN)、20%的乳清蛋白(whey protein,WP)以及少量的牛血清白蛋白(bovine serum albumin,BSA)、免疫球蛋白(immunoglobulin,Igs)、乳铁蛋白(lactoferrin,Lf)和乳过氧化物酶(lactoperoxidase,Lp)等。其中酪蛋白、α-乳白蛋白(α-lactalbumin,α-LA)和β-乳球蛋白(β-lactoglobulin,β-LG)是牛乳蛋白中的主要致敏原蛋白[3−4]。在牛乳蛋白过敏患者中,65%~100%对酪蛋白过敏,0~67%对α-LA过敏,13%~62%对β-LG过敏[5]。牛乳蛋白过敏会使患者产生湿疹、腹泻和胃肠出血等症状,严重者甚至导致休克和死亡[6]。因此消减牛乳主要蛋白致敏性尤为重要。酶水解技术工艺简单,条件温和,安全性高,技术成熟,是目前使用最为普遍的方法[7],而在现有蛋白酶的乳蛋白水解产物中仍然会检测到残留的致敏原表位[8−10],探寻新型蛋白酶成为解决这一问题的新思路。
植物源蛋白酶来源丰富,易于获取,水解特异性广泛,但仅有少数商业化产品[11−12],有待于进一步探索。猪笼草具有体型较大、颜色诱人和生长期长的特点,它的捕虫笼分泌和储存了多种酶,猪笼液蛋白酶(Nepenthesins)是猪笼液中的主要蛋白酶[13],可用于氢/氘交换质谱的消解工具、谷蛋白消化和蛋白质组学分析等方面[14]。作为一大园艺品种,Nepenthes×Miranda的猪笼液相对不容易受外界污染,产量也比较可观,并且在出售前猪笼液往往会被作为废物丢弃,不仅造成资源浪费也会造成环境负担,有待进一步利用。在前期猪笼液蛋白酶的酶学性质表征时发现,Nepenthes×Miranda的猪笼液中含有的蛋白酶属于天冬氨酸蛋白酶的A1B亚家族,该亚家族也被称为猪笼草素型天冬氨酸蛋白酶(NEP),具有对化学试剂出色的稳定性[11,14]。其氨基酸序列不仅含有更多的半胱氨酸残基,还另外含有特异性插入片段(NAP),有助于维持其独特的稳定性[15],所以将Nepenthes×Miranda的猪笼液进行蛋白酶的进一步利用是非常必要的。
牛乳主要致敏原蛋白被有效酶解通常被认为是其致敏性降低的前提条件[16]。有研究报导,胃蛋白酶在pH1.5~3.5的条件下可将CN迅速酶解,pH4.0时酶解变缓慢;α-LA和β-LG在pH1.5~2.5时被胃蛋白酶水解,当pH>3.0时消化率较低[17]。虽然在合适的pH条件下胃蛋白酶可水解乳蛋白,但其水解产物仍会残留致敏性[18]。在前期研究中本团队发现猪笼液蛋白酶的最适pH为2.05~2.50,不仅此pH范围满足牛乳主要致敏原蛋白被消化的条件,而且猪笼液蛋白酶拥有比胃蛋白酶更广泛的水解特异性[19],有利于乳蛋白被高效地水解,所以将猪笼液蛋白酶应用在酶解牛乳蛋白并消减其致敏性上是一个切实可行的方案。
因此,本文从Nepenthes×Miranda分泌的猪笼液中制备新型植物源蛋白酶液,以此为研究对象探索其对牛乳主要蛋白的酶解,通过致敏原的残留肽段与表位系统分析,开展酶切位点与表位消减的关联性研究,为消减牛乳主要致敏原蛋白的致敏性提供科学依据。
1. 材料与方法
1.1 材料与仪器
米兰达猪笼草的猪笼液 2022年6月中下旬于广州菲朗园艺有限公司将米兰达猪笼草捕虫笼中的猪笼液收集起来所获得;浓缩乳清蛋白(WPC 80) 澳优乳业(中国)有限公司提供;酪蛋白 美国Sigma公司;4×还原型上样缓冲液 中国索莱宝公司;福林酚、三氯乙酸、乳酸、乳酸钠等化学试剂 均为国产分析纯。
UV-8000紫外可见分光光度计 配有氘灯和钨灯光源,中国上海元析公司;微量石英比色皿 中国伟鑫公司;Mini垂直蛋白电泳仪、ChemiDoc成像仪(配有Image Lab™图像采集和分析系统) 美国BIO-RAD公司;FDU-2110冷冻干燥机 日本EYELA公司;RO 15水浴锅 配有磁力搅拌系统,德国IKA公司;DK-8D三温三控恒温水浴锅 中国上海博讯实业公司;ST 16R高速冷冻离心机、RSLCnano纳升液相-Q Exactive HF质谱联用仪 美国Thermo公司;Milli-Q超纯水系统 美国Milipore公司;PB-10型pH计 德国Sartorius公司。
1.2 实验方法
1.2.1 猪笼液蛋白酶液制备
将收集的猪笼液用64层纱布过滤以去除大杂质,透过液再经5 kDa的膜超滤去除小分子杂质和大部分水分,获得猪笼液蛋白酶液,再经真空冷冻干燥获得猪笼液蛋白酶粉。取猪笼液蛋白酶液及其冻干粉,二者于4 ℃过夜充分溶解混匀,在5000×g,5 min,4 ℃的条件下离心取上清,以在不引入外界其他杂质或溶剂的情况下获得的具有较高蛋白水解活性的猪笼液蛋白酶液,用于后续牛乳蛋白的酶解。
1.2.2 猪笼液蛋白酶蛋白水解活性测定
将上述猪笼液蛋白酶液稀释80倍,于pH2.05±0.05的乳酸-乳酸钠缓冲液中配制底物酪蛋白(1 mg/mL),之后按照GB 1886.174-2016《食品添加剂 食品工业用酶制剂》[20]中的福林酚法测定蛋白水解活性,整个体系试剂添加量缩小至国标福林酚法试剂添加量的1/10,用10000×g,10 min,4 ℃的离心来代替溶液体系的过滤。
1.2.3 猪笼液蛋白酶酶解牛乳蛋白
本课题的前期研究已经探索出了猪笼液蛋白酶的最适pH为2.05~2.50,最适温度为50~55 ℃,基于此最佳酶解条件分别对牛乳乳清蛋白和酪蛋白进行酶解。
1.2.3.1 酶解乳清蛋白
参考Peslerbes等[21]和Oliveira等[22]的方法并稍做改进。称取10.00 g乳清蛋白粉(WPC),溶于200 mL超纯水,室温搅拌5 h,4 ℃搅拌过夜,用0.5 mol/L HCl调pH至2.50±0.05后,加超纯水将WPC溶液定容至250 mL,配成质量分数为4%的WPC溶液。85 ℃水浴加热30 min,冷却至室温。取5 mL WPC溶液,于50 ℃水浴锅伴随磁力搅拌预热5 min,预热结束后加入0.5 mL猪笼液蛋白酶涡旋混匀,于50 ℃和磁力搅拌的条件下酶解2.5 h。酶解结束后进行95 ℃,15 min的酶灭活。冷却至室温后,于3000×g,30 min,4 ℃的条件下离心,以分离蛋白溶液中存在的少量乳脂肪等杂质,取上清获得酶解乳清蛋白产物(HWPC)。
1.2.3.2 酶解酪蛋白
称取10.00 g酪蛋白粉(CN),溶于200 mL超纯水,100 ℃加热搅拌,期间加入适量0.5 mol/L的HCl以改变溶液pH并保证溶解充分,继续在此温度下加热30 min直至完全溶解,冷却至室温后,用0.5 mol/L HCl调pH至2.05±0.05,加超纯水将CN溶液定容至250 mL,配成质量分数为4%的CN溶液。取5 mL CN溶液,于50 ℃水浴锅伴随磁力搅拌预热5 min,预热结束后加入0.5 mL猪笼液蛋白酶涡旋混匀,于50 ℃和磁力搅拌的条件下酶解2.5 h。酶解结束后进行95 ℃,15 min的酶灭活。冷却至室温后,于3000×g,30 min,4 ℃的条件下离心,以分离蛋白溶液中存在的少量乳脂肪等杂质,取上清获得酶解酪蛋白产物(HCN)。
1.2.4 酶解后肽段分子量分布鉴定
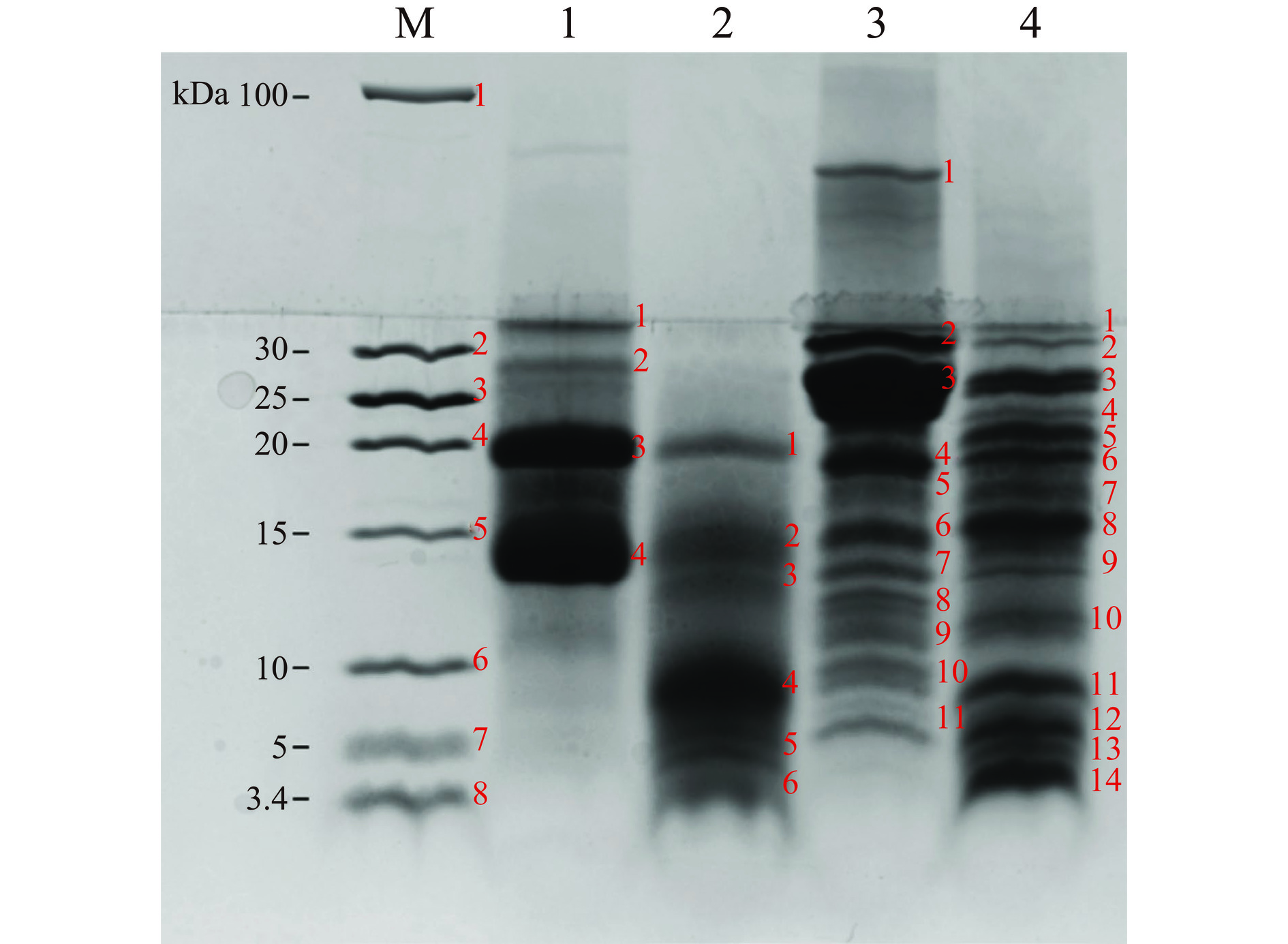
参考王靖舒[23]的Tricine-SDS-PAGE方法对牛乳蛋白酶解产物的分子量分布进行鉴定,并稍做改进,将未酶解的WPC和CN样品稀释至与酶解后的HWPC和HCN同一稀释度,用电泳专用的含溴酚蓝、变性剂和还原剂的还原型上样缓冲液处理样品(V4×还原型上样缓冲液:V样品=1:3),于99 ℃下加热5 min,离心30 s,取上清上样,低分子量蛋白Marker与样品均上样7 μL,凝胶脱色完成后用化学发光成像仪采集凝胶图像。
1.2.5 致敏原表位酶解鉴定
采用液相色谱串联二级质谱技术(LC-MS/MS)分别对HWPC和HCN进行序列鉴定,根据质谱鉴定到的肽段来判断酶的水解位点,被水解的肽键处的第一个氨基酸残基分别为P1位(底物蛋白N端)和P1’位(底物蛋白C端),第二个氨基酸残基分别为P2位和P2’位,以此类推。并将获得的肽段序列与过敏原数据库(IEDB:http://www.iedb.org/)进行对比以判断致敏原蛋白线性表位是否被有效断裂并查找残留表位的肽段。
1.2.5.1 HCN和HWPC前处理
样品于12000×g离心5 min,取上清,用截留量为10 kDa超滤管将上清进行超滤,滤过液使用C18脱盐柱脱盐,真空抽干后-20 ℃冻存。此部分由谱度众合(武汉)生命科技有限公司完成。
1.2.5.2 色谱条件
色谱柱:C18色谱柱(75 µm×25 cm,粒度1.9 µm,孔径100 Å);流动相A:0.1%甲酸/3%二甲基亚砜/97%超纯水,流动相B:0.1%甲酸/3%二甲基亚砜/97%乙腈,梯度洗脱,流速300 nL/min,柱温50 ℃,柱压30 MPa。此部分由谱度众合(武汉)生命科技有限公司完成。
1.2.5.3 质谱条件
采用数据依赖采集模式(DDA)进行数据采集。一级质谱(MS1)检测:分辨率60K@200 m/z,扫描范围350~1500 m/z,AGC target设置为3E6,最大注入时间30 ms,前体母离子选择1.4 Da,选取top 20的母离子进行碎裂,HCD碰撞能量设置为28%;二级质谱(MS2)检测:分辨率15K@200 m/z,扫描范围350~1500 m/z,AGC target设置为1E5,最大注入时间50 ms。动态排除时间30 s。此部分由谱度众合(武汉)生命科技有限公司完成。
1.3 数据处理
电泳图由Image Lab™(Version 6.1)图像采集和分析系统处理后展示;质谱数据由谱度众合(武汉)生命科技有限公司根据MaxQuant(Version 1.6.6)软件检索,采用了Andromeda算法,参考了UniProt中Bovine的蛋白质组数据库;蛋白结构模式图由PyMOL™ Molecular Graphics System(Version 2.4.0)处理后展示。
2. 结果与分析
2.1 酶解后肽段分子量分布鉴定
用于酶解牛乳蛋白的猪笼液蛋白酶的酶活为126.276 U/mL。酶解前后蛋白分子量分布及占比由图1和表1所示,WPC中分子量为14.2 kDa的α-LA和18.3 kDa的β-LG在其中相对占比分别为55.026%和26.625%,合计为81.651%,这与天然牛乳中二者的比例不对应,推测因商业化的婴配粉需在蛋白组成上接近母乳中不同蛋白质的比率[24],而提高了α-LA的比率。经酶解后,分子量在20 kDa左右的WPC酶解产物相对含量降低至9.315%。同时分子量在15 kDa附近的酶解产物相对含量分别降低至20.496%和5.500%。从图1泳道1的条带分布可知,WPC几乎不含3.4~10 kDa的肽段,而HWPC所含的此分子量范围内的肽段总含量高达64.689%。因此WPC中的α-LA和β-LG被显著水解。CN中分子量为23.6 kDa的αs1-CN、25.2 kDa的αs2-CN和24 kDa的β-CN的总相对含量如图1中泳道3的条带3所示,约为43.396%。κ-CN的分子量为19 kDa,相对含量约为10.571%。酶解后泳道4的条带6在20 kDa附近,相对含量为4.279%,此部分可能是κ-CN的残留、与κ-CN相似分子量且未被酶解的杂蛋白或高分子量杂蛋白被酶解后的部分产物,具体来源需要结合质谱鉴定进一步分析。HCN中分子量范围为3.4~10 kDa的肽段含量也显著增加。因此,CN中的αs1-CN、αs2-CN、β-CN和κ-CN有被显著水解。
表 1 不同泳道肽段相对含量百分比(%)Table 1. Relative percentages of peptide content in different lanes (%)条带编号 泳道1 泳道2 泳道3 泳道4 1 12.561 9.315 7.104 1.581 2 5.789 20.496 16.784 1.841 3 26.625 5.500 43.396 11.433 4 55.026 33.166 10.571 1.867 5 − 11.085 0.968 5.946 6 − 20.438 4.121 4.279 7 − − 2.604 1.792 8 − − 1.247 10.591 9 − − 4.120 1.535 10 − − 4.813 7.252 11 − − 4.271 14.194 12 − − − 16.007 13 − − − 8.238 14 − − − 13.442 注:−表示此位置无可见的蛋白条带;条带编号(1~14)对应图1中每条电泳条带右边的编号。 2.2 致敏原表位酶解鉴定
2.2.1 WPC中主要致敏原蛋白α-LA的线性表位的酶解
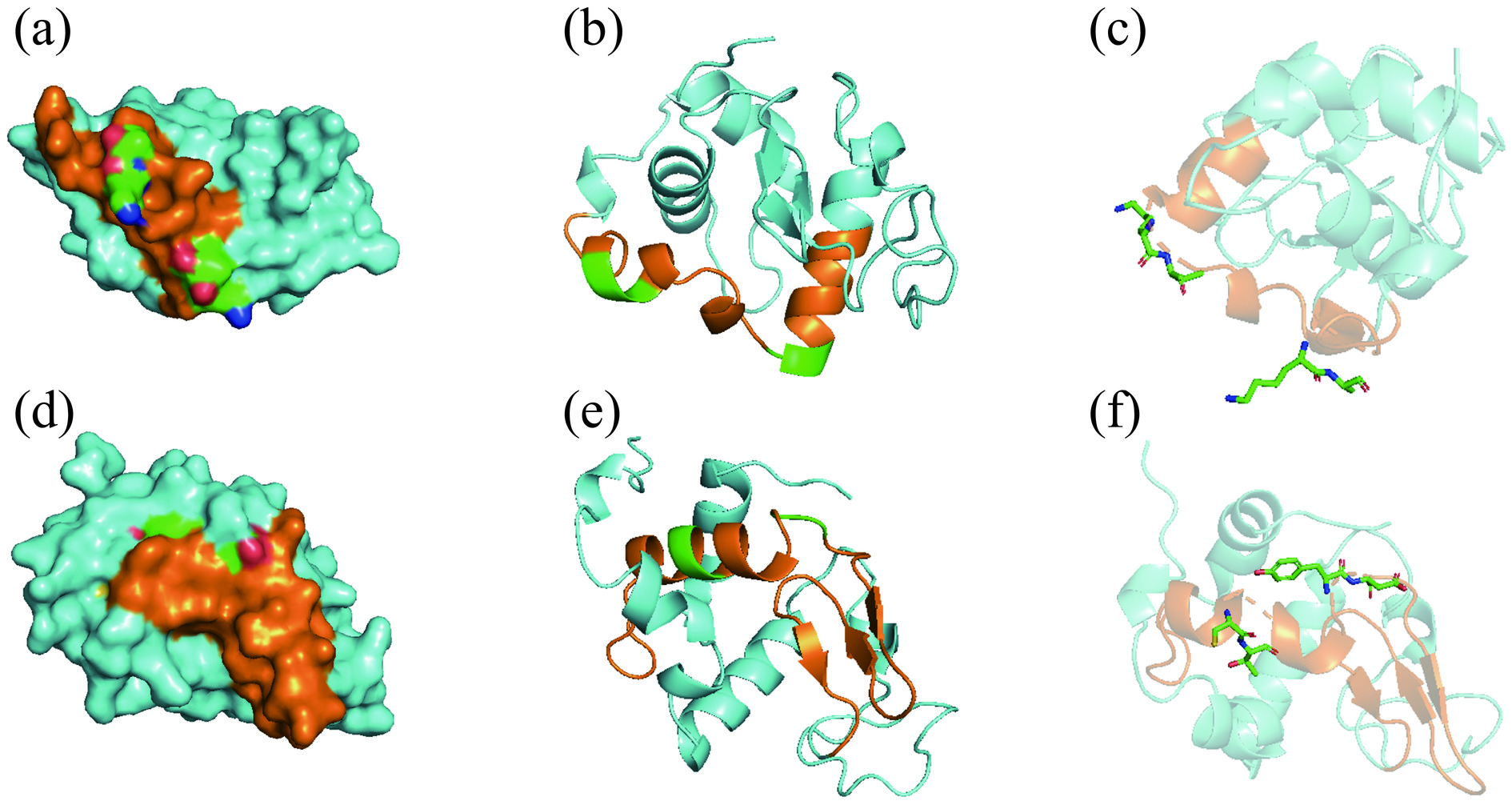
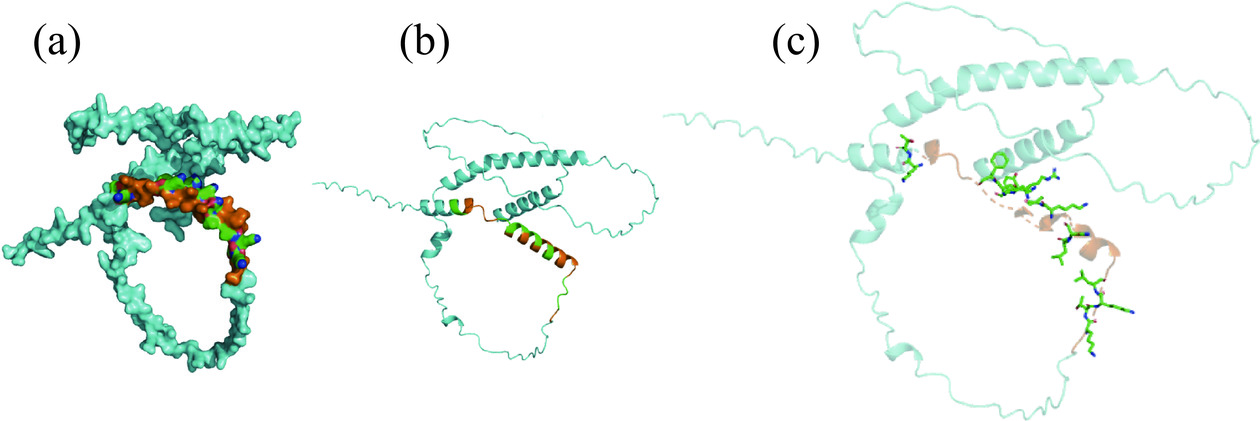
通过质谱鉴定获得来自α-LA的肽段共154条,剔除掉重复肽段,获得96个特异性氨基酸序列。对比所获肽段与数据库里已收录的α-LA的9个B细胞表位和1个T细胞表位,发现所有肽段均能对应到表位序列的一部分,如表2所示,说明猪笼液蛋白酶在α-LA线性表位内发生水解,并有效断裂了表位。所获数目较多的肽段如表3所示,通过肽段两端的氨基酸残基可以看出,猪笼液蛋白酶对P1位为K和P1’位为D的肽键有着较高的酶解效率。酶解所得数目最多的肽段Pα-LA1含有B细胞表位CVKKILDKVGINYWL(Eα-LA1)和T细胞表位DKVGINYWLAHKALCSEK(Eα-LA2)的部分序列,经对比发现猪笼液蛋白酶的裂解发生在Eα-LA1中的K-V以及Eα-LA2中的K-V和K-A,对表位产生了一定的破坏。由图2(a)~(c)所示,Eα-LA1和Eα-LA2位于α-LA的表面,且附近没有较大基团的空间位阻,有利于蛋白酶接近。此外,B细胞表位GYGGVSLPEWVCTTFHTSGYDTQAIVQNNDSTEYGLFQINNK(Eα-LA3)中包含种类最多的酶解肽段,尤其以N端为TTFH和DTQA的肽段种类和数量最多,并且Eα-LA3序列较长,在α-LA的表面和内部都有分布,如图2(d)所示,发生酶解频率较高的肽键分布在内部,说明猪笼液蛋白酶对P1位为C和Y以及P1’位为T的肽键也有着较高的酶解效率并且能有效破坏α-LA分子内部的表位。
表 2 质谱鉴定后肽段与α-LA过敏原表位数据库线性表位对比Table 2. Comparison of linear epitopes of peptides identified by mass spectrometry with the α-LA allergen epitope database表位
类型数据库中的α-LA
线性表位表位在α-LA中的
位置表位所含氨基酸残基数 HWPC中含有该表位部分序列的肽段种类数 B细胞表位 CEVFRELKDLKGYGG 6~20 15 5 ELKDLKGYGGVSLPE 11~25 15 14 LKGYGGVSLPEW 15~26 12 1 KGYGGVSLPEWVCTT 16~30 15 2 GYGGVSLPEWVCTTFHTSGYDTQAIVQNNDSTEYGLFQINNK
(Eα-LA3)17~58 42 42 DSTEYGLFQINNKIW 46~60 15 5 SCDKFLDDDLTDDIM 76~90 15 3 FLDDDLTDDIMCVK 80~93 14 7 CVKKILDKVGINYWL
(Eα-LA1)91~105 15 6 T细胞表位 DKVGINYWLAHKALCSEK(Eα-LA2) 97~114 18 11 表 3 酶解α-LA后获得的数目较多的肽段Table 3. Higher number of peptides obtained after enzymatic cleavage of α-LA肽段序列 肽段编号 数目 VGINYWLAHK Pα-LA1 23 STEYGLFQINNK Pα-LA2 4 FLDDDLTDDIM Pα-LA3 4 DTQAIVQNND Pα-LA4 4 DLKGYGGVSL Pα-LA5 4 DKVGINYWLAHK Pα-LA6 4 ![]() 图 2 α-LA表位分布、结构和酶切位点分布注:(a)~(c)Eα-LA1和Eα-LA2在α-LA上的分布位置及主要酶切位点的分布;(d)~(f)Eα-LA3在α-LA上的分布位置及主要酶切位点的分布;(a)~(f)中橙色表示表位;(a)、(c)、(d)和(f)中绿色代表组成被酶解肽键氨基酸残基的碳原子,红色代表组成被酶解肽键氨基酸残基的氧原子,深蓝色代表组成被酶解肽键氨基酸残基的氮原子;(b)和(e)中绿色代表酶水解位置。Figure 2. Distribution of α-LA epitopes, structure and distribution of enzymatic sites
图 2 α-LA表位分布、结构和酶切位点分布注:(a)~(c)Eα-LA1和Eα-LA2在α-LA上的分布位置及主要酶切位点的分布;(d)~(f)Eα-LA3在α-LA上的分布位置及主要酶切位点的分布;(a)~(f)中橙色表示表位;(a)、(c)、(d)和(f)中绿色代表组成被酶解肽键氨基酸残基的碳原子,红色代表组成被酶解肽键氨基酸残基的氧原子,深蓝色代表组成被酶解肽键氨基酸残基的氮原子;(b)和(e)中绿色代表酶水解位置。Figure 2. Distribution of α-LA epitopes, structure and distribution of enzymatic sites2.2.2 WPC中主要致敏原蛋白β-LG的线性表位的酶解
猪笼液蛋白酶对β-LG的水解较为广泛,通过质谱鉴定到了430条产物肽段,剔除掉重复肽段后有184个特异性氨基酸序列。对比所获肽段与数据库里已收录的β-LG的14个B细胞表位和7个T细胞表位,如表4所示,发现猪笼液蛋白酶也有效断裂了β-LG的线性表位,未见完整表位残存。所获数目较多的肽段如表5所示,通过肽段两端的氨基酸残基可以看出,猪笼液蛋白酶对P1位为V、R、Y、I、S、K和L以及P1’位为D、A、V、I和L的肽键有着较高的酶解效率,与酶解α-LA频率较高的位点一致。B细胞表位LAMAASDISLLDAQSAPLRV(Eβ-LG1)和DISLLDAQSAPLRVYVEELK(Eβ-LG2)中包含酶解肽段Pβ-LG1和Pβ-LG3,对比发现猪笼液蛋白酶水解了表位中的L-D、V-Y和Y-V;由图3(a)~(c)所示,Eβ-LG1和Eβ-LG2在β-LG的表面和内部都有分布,发生酶解频率较高的肽键分布在内部,说明猪笼液蛋白酶能够破坏β-LG的内部表位。值得注意的是,虽然在T细胞表位LIVTQTMKGLDIQKVAGTWYS(Eβ-LG3)中发现了种类最多的酶解肽段,尤其以N端为LIVT的肽段种类和数量最多,但对比UniProt数据库发现,此序列为成熟β-LG的开头序列,并不能以此衡量猪笼液蛋白酶对P1’位为L的肽键的水解能力。
表 4 质谱鉴定后肽段与β-LG过敏原表位数据库线性表位对比Table 4. Comparison of linear epitopes of peptides identified by mass spectrometry with the β-LG allergen epitope database表位类型 数据库中的β-LG线性表位 表位在β-LG中的位置 表位所含氨基酸残基数 HWPC中含有该表位部分序列的肽段种类数 B细胞表位 LAMAASDISLLDAQSAPLRV(Eβ-LG1) 22~41 20 14 DISLLDAQSAPLRVYVEELK(Eβ-LG2) 28~47 20 20 VYVEELKPTPEGDLEILLQK 41~60 20 6 VEELKPTPEGDLEILLQKWE 43~62 20 12 AQKKIIAEKTKIPAVFKIDA 67~86 20 1 KIIAEKTKIPAVFKIDALNE 70~89 20 3 TKIPAVFKIDALNENKVLVL 76~95 20 1 VLDTDYKKYLLFCMENSAEP 94~113 20 2 YLLFCMENSAEPEQSLACQCLVR 102~124 23 2 CMENSAEPEQSLACQCLVRT 106~125 20 1 ACQCLVRTPEVDDEALEKFD 118~137 20 6 DEALEKFDKALKALPMHIRL 130~149 20 9 LEKFDKALKALPMHIRLSFN 133~152 20 10 KALPMHIRLSFNPTQ 141~155 15 1 T细胞表位 LIVTQTMKGLDIQKVAGTWYS(Eβ-LG3) 1~21 21 32 TMKGLDIQKVAGTWYSLAMAASDISLLDAQ 6~35 30 20 KPTPEGDLEILLQKWENGECA 47~67 21 9 KIPAVFKIDALNENKVLVLDT 77~97 21 15 DALNENKVLVLDTDYKKY 85~102 18 10 LKALPMHIRLSFNPT 140~154 15 2 ALPMHIRLSFNPTQLEEQCHI 142~162 21 8 表 5 酶解β-LG后获得的数目较多的肽段Table 5. Higher number of peptides obtained after enzymatic cleavage of β-LG肽段序列 肽段编号 数目 DAQSAPLRV Pβ-LG1 42 ALKALPMHIR Pβ-LG2 38 DAQSAPLRVY Pβ-LG3 18 VEELKPTPEGDLEI Pβ-LG4 16 IQKVAGTWYS Pβ-LG5 11 LIVTQTMKGLDIQK Pβ-LG6 11 DKALKALPMHIR Pβ-LG7 9 LIVTQTMKGL Pβ-LG8 8 LSFNPTQL Pβ-LG9 6 2.2.3 CN中主要致敏原蛋白αs1-CN的线性表位的酶解
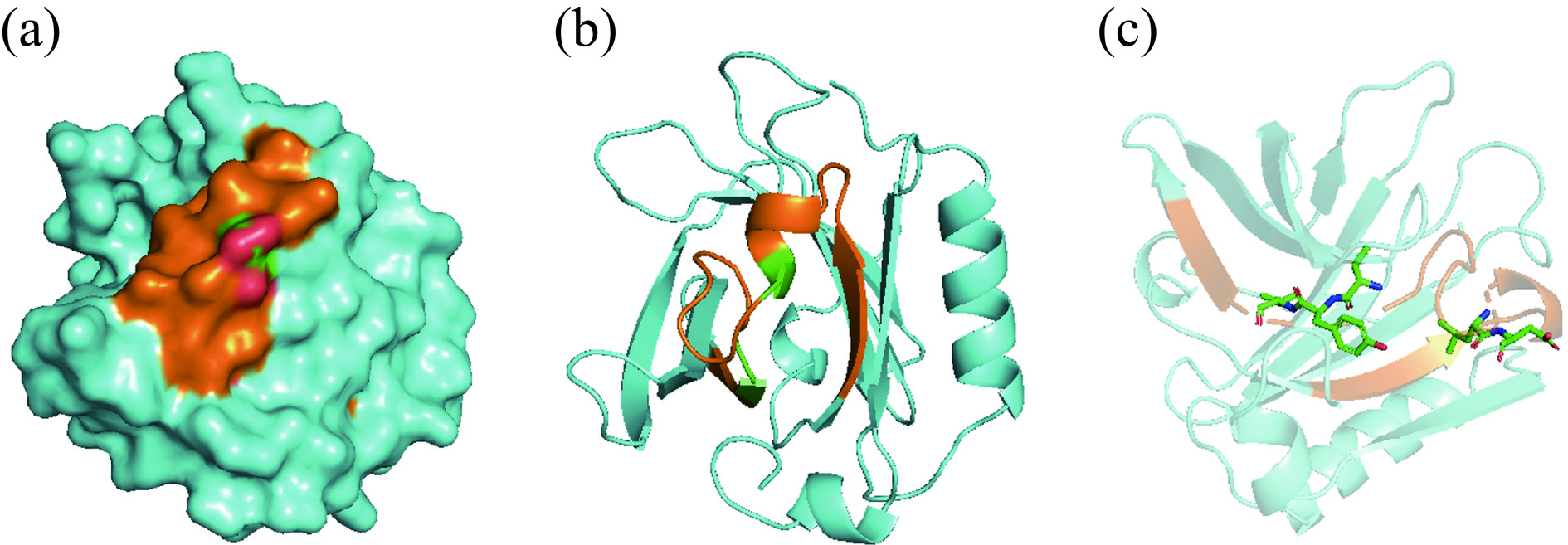
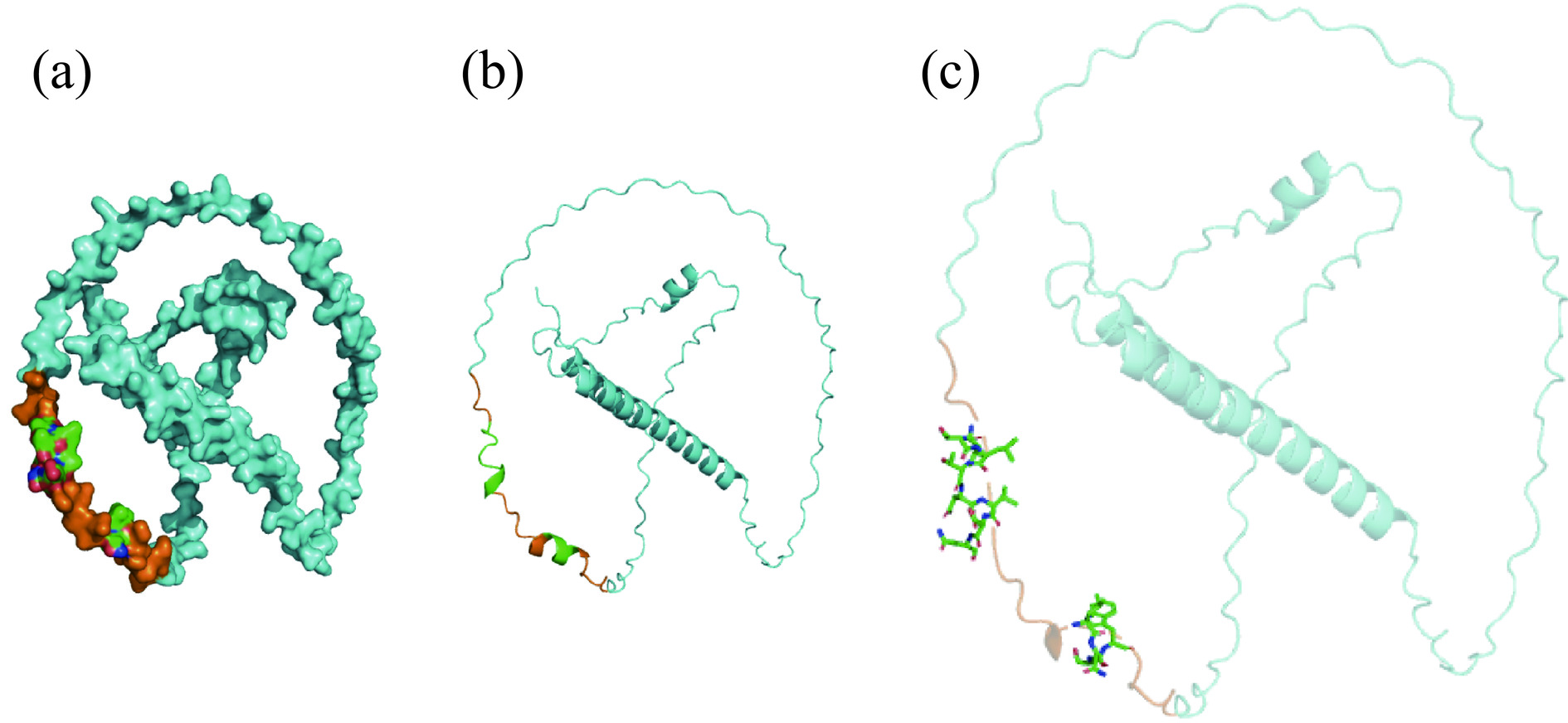
αs1-CN也被猪笼液蛋白酶有效酶解,鉴定到肽段共416条,特异性氨基酸序列共214个。对比所获肽段与数据库里已收录的αs1-CN的11个B细胞表位,如表6所示,发现猪笼液蛋白酶也有效水解了αs1-CN线性表位中的肽键。所获数目较多的肽段如表7所示,虽然N端为RPKH的肽段数目最多,但对比UniProt数据库发现,此序列为成熟αs1-CN的开头序列,并不能以此衡量猪笼液蛋白酶对P1’位为R的肽键的水解能力,但对比此类肽段C端的氨基酸残基,可以判定猪笼液蛋白酶对P1位为F和R的肽键有着较高的酶解效率,并且也对产生了数量较多的N端为FVAP和FFVA的肽段有一个较好的印证。同时由其它数量较多的肽段可以发现猪笼液蛋白酶能很好地酶解P1’位为F、A、E、I和R的肽键。B细胞表位RPKHPIKHQGLPQEVLNENLLRFFVAPFPEV(Eαs1-CN1)中包含酶解肽段Pαs1-CN1、Pαs1-CN2和Pαs1-CN3,对比发现猪笼液蛋白酶水解了该表位中大部分的R-F和F-F,由图4(a)~(c)所示,αs1-CN结构简单,由α-螺旋和无规卷曲构成,并没有形成球形结构,Eαs1-CN1位于αs1-CN的α-螺旋上,几乎不存在构象方面的阻碍,有利于酶解反应的发生。
表 6 质谱鉴定后肽段与αs1-CN过敏原表位数据库线性表位对比Table 6. Comparison of linear epitopes of peptides identified by mass spectrometry with the αs1-CN allergen epitope database表位类型 数据库中的αs1-CN线性表位 表位在αs1-CN
中的位置表位所含氨基
酸残基数HCN中含有该表位部分
序列的肽段种类数B细胞表位 RPKHPIKHQGLPQEVLNENLLRFFVAPFPEV(Eαs1-CN1) 1~31 31 44 VLNENLLRFFVAPFPEVFGKEKVNELSKDIGS 15~46 32 27 FGKEKVNELSKDIGSESTEDQAMEDIKQMEAES 32~64 33 9 ISSSEEIVPNSVEQKHIQKEDVPSERYLGYLEQLLR 65~100 36 10 KEDVPSERYLGYLEQLLRLK 83~102 20 31 DVPSERYLGYLEQLLRLKKY 85~104 20 9 VPSERYLGYLEQLLRLKKYKVPQLEIVPNS 86~115 30 4 RLHSMKEGIHAQQKEPMIGVNQELAYFYPE 119~148 30 25 PMIGVNQELAYFYPELFRQFYQLDAYPSGAWYYV 134~167 34 15 FYQLDAYPSGAWYYVPLGTQYTDAPSFSDI 153~182 30 12 PLGTQYTDAPSFSDIPNPIGSENSEKTTMPLW 168~199 32 28 表 7 酶解αs1-CN后获得的数目较多的肽段Table 7. Higher number of peptides obtained after enzymatic cleavage of αs1-CN肽段序列 肽段编号 数目 RPKHPIKHQGLPQEVLNENLLRF Pαs1-CN1 41 RPKHPIKHQGLPQEVLNENLLR Pαs1-CN2 12 除Pαs1-CN1和Pαs1-CN2外N端为RPKH的其它肽段 Pαs1-CN3 16 N端为FVAP的肽段 Pαs1-CN4 39 N端为FFVA的肽段 Pαs1-CN5 20 N端为APFP的肽段 Pαs1-CN6 18 N端为EGIH的肽段 Pαs1-CN7 17 N端为IPNP的肽段 Pαs1-CN8 16 2.2.4 CN中主要致敏原蛋白αs2-CN的线性表位的酶解
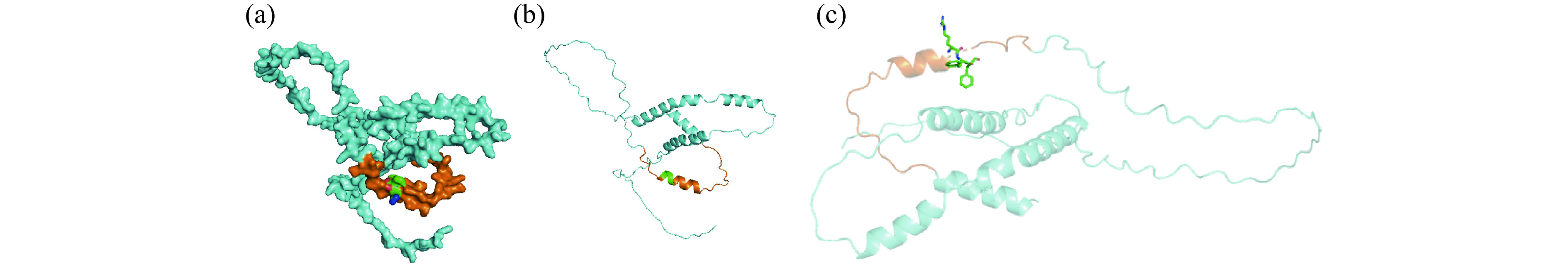
虽然αs2-CN被猪笼液蛋白酶水解后只产生了188条肽段,剔除掉重复肽段后剩108个特异性氨基酸序列,无论在数量上还是在种类上αs2-CN都较αs1-CN的产物肽段少。但对比所获肽段与数据库里已收录的αs2-CN的12个B细胞表位和2个T细胞表位,如表8所示,发现猪笼液蛋白酶对αs2-CN线性表位中的肽键是有效水解的。所获数目较多的肽段如表9所示,根据表9中的肽段并同时对比Pαs2-CN4、Pαs2-CN5和UniProt数据库中αs2-CN的完整序列,可以发现猪笼液蛋白酶对P1位为K以及P1’位为F、Y、N、T和L的肽键水解能力较强,值得注意的是,当P1位和P1’位同时为K时,该处肽键并没有被酶切,P1和P2位同时为K时,则可发生酶水解反应,可能的原因是相邻K相互竞争蛋白酶,但P2位K具体对P1位K起到一定的抑制作用还是促进作用还不好判定,需要进一步研究。B细胞表位EKNRLNFLKKISQRYQKFALPQYLKT(Eαs2-CN1)与酶解肽段Pαs2-CN1、Pαs2-CN2和Pαs2-CN3有一定的重合序列,且对比UniProt数据库,发现酶解肽段Pαs2-CN4 N端第一个氨基酸残基对应Eαs2-CN1最后一个氨基酸残基,说明猪笼液蛋白酶主要水解了该表位中的L-N、R-Y、K-F和K-T,B细胞表位TKKTKLTEEEKNRLNFLKKI(Eαs2-CN2)中包含酶解肽段Pαs1-CN5中的部分序列,对比发现该表位的K-T、K-L和K-I被酶解。如图5所示,虽然表位Eαs2-CN1和Eαs2-CN2相互连接,但含有多处酶裂解频率较高的位点,且αs2-CN结构简单也为酶的接近提供了分子基础,所以两个表位被有效破坏。
表 8 质谱鉴定后肽段与αs2-CN过敏原表位数据库线性表位对比Table 8. Comparison of linear epitopes of peptides identified by mass spectrometry with the αs2-CN allergen epitope database表位类型 数据库中的αs2-CN线性表位 表位在αs2-CN
中的位置表位所含氨基
酸残基数HCN中含有该表位部分
序列的肽段种类数B细胞表位 CKEVVRNANEEEYSIGSSSE 40~59 20 2 KITVDDKHYQKALNEINQFY 70~89 20 5 KHYQKALNEINQFYQKFPQY 76~95 20 11 QKALNEINQFYQKFPQYLQY 79~98 20 4 FYQKFPQYLQYLYQGPIVLN 88~107 20 14 QYLYQGPIVLNPWDQVKRNA 97~116 20 6 PIVLNPWDQVKRNAVPITPT 103~122 20 5 NAVPITPTLNREQLSTSEEN 115~134 20 2 EVFTKKTKLT 145~154 10 2 TKKTKLTEEEKNRLNFLKKI(Eαs2-CN2) 148~167 20 10 EKNRLNFLKKISQRYQKFALPQYLKT(Eαs2-CN1) 157~182 26 16 KTVYQHQKAMKPWIQPKTKVIPYVRYL 181~207 27 16 T细胞表位 EEKNRLNFLKKISQR 156~170 15 2 YQKFALPQYLKTVYQ 171~185 15 13 表 9 酶解αs2-CN后获得的数目较多的肽段Table 9. Higher number of peptides obtained after enzymatic cleavage of αs2-CN肽段序列 肽段编号 数目 N端为FALP的肽段 Pαs2-CN1 19 N端为YQKF的肽段 Pαs2-CN2 14 NFLKKISQRYQK Pαs2-CN3 10 N端为TVYQ的肽段 Pαs2-CN4 8 由Pαs2-CN4产生的N端为TKKT、TKLT和LTEE以及C端为KKLF的肽段 Pαs2-CN5 若干 2.2.5 CN中主要致敏原蛋白β-CN的线性表位的酶解
被酶解后的β-CN产生了共367条肽段,剔除掉重复肽段后获得了231个特异性氨基酸序列。对比所获肽段与数据库里已收录的β-CN的20个B细胞表位,如表10所示,发现β-CN的线性表位也被有效破坏。所获数目较多的肽段如表11所示,通过产物肽段N端的氨基酸残基可以看出,猪笼液蛋白酶对P1’位为Q、S、Y、T、W、V、D、N和P的肽键水解能力较强。表位ESQSLTLTDVENLHLPLPLL(Eβ-CN1)和VENLHLPLPLLQSWMHQPHQ(Eβ-CN2)被猪笼液蛋白酶水解后产生了数量和种类较多的酶解肽段,这些肽段与Pβ-CN1、5、6、7、8、9和10有一定的重合序列,对比发现猪笼液蛋白酶有效水解了表位的L-Q、S-W、D-V、L-T、T-D和E-N。如图6所示,β-CN结构更为松散和简单,有利于蛋白酶的接近,为β-CN被酶解产生数目和种类较多的肽段提供了分子层面的依据。但这种可产生较多数目和种类片段的广泛水解却是相对的,其分子内较长的α-螺旋虽然含有裂解频率较高的氨基酸位点,在β-CN中也处于一个较为开阔的位置,但被水解产生的肽段却较少,可能会使β-CN残留一定的致敏性。但显而易见的是,Eβ-CN1和Eβ-CN2相互连接,含有多处裂解频率较高的位点,被有效破坏。
表 10 质谱鉴定后肽段与β-CN过敏原表位数据库线性表位对比Table 10. Comparison of linear epitopes of peptides identified by mass spectrometry with the β-CN allergen epitope database表位类型 数据库中的β-CN线性表位 表位在β-CN中的位置 表位所含氨基酸残基数 HCN中含有该表位部分
序列的肽段种类数B细胞表位 RELEELNVPGEIVESLSSSE 1~20 20 4 RINKKIEKFQSEEQQQTEDELQDKIH 25~50 26 2 QSEEQQQTEDELQDKIHPFA 34~53 20 1 DELQDKIHPFAQTQSLVYPF 43~62 20 10 TQSLVYPFPGPIPNSLPQ 55~72 18 1 PLTQTPVVVPPFLQPEVMGV 76~95 20 7 QTPVVVPPFLQPEVMGVSKV 79~98 20 2 VVVPPFLQPEVMGVSKVKEA 82~101 20 12 EAMAPKHKEMPFPKYPVEPF 100~119 20 21 APKHKEMPFPKYPVEPFTES 103~122 20 1 PFPKYPVEPFTESQSLTLTD 110~129 20 24 ESQSLTLTDVENLHLPLPLL (Eβ-CN1) 121~140 20 33 SLTLTDVENLHLPLPLLQSW 124~143 20 9 VENLHLPLPLLQSWMHQPHQ(Eβ-CN2) 130~149 20 34 TVMFPPQSVLSLSQSKVLPV 154~173 20 10 SQSKVLPVPQKAVPYPQRDM 166~185 20 16 QKAVPYPQRDMPIQAFLLYQ 175~194 20 11 PQRDMPIQAFLLYQEPVLGP 181~200 20 2 IQAFLLYQEPVLGPVRGPFP 187~206 20 1 FLLYQEPVLGPVRGPFPIIV 190~209 20 30 表 11 酶解β-CN后获得的数目较多的肽段Table 11. Higher number of peptides obtained after enzymatic cleavage of β-CN肽段序列 肽段编号 数目 N端为QSWM的肽段 Pβ-CN1 22 N端为SLSQ的肽段 Pβ-CN2 20 N端为YQEP的肽段 Pβ-CN3 20 N端为SQSK的肽段 Pβ-CN4 18 N端为WMHQ的肽段 Pβ-CN5 17 N端为VENL的肽段 Pβ-CN6 15 N端为TDVE的肽段 Pβ-CN7 13 N端为TLTD的肽段 Pβ-CN8 11 N端为DVEN的肽段 Pβ-CN9 9 N端为NLHL的肽段 Pβ-CN10 9 N端为PVLG的肽段 Pβ-CN11 9 3. 讨论与结论
本研究利用猪笼液蛋白酶水解牛乳主要致敏原蛋白。首先通过Tricine-SDS-PAGE发现WPC和CN均被显著酶解。再通过LC-MS/MS鉴定酶解过程中产生的肽段,进一步确定酶切位点和主要致敏原蛋白的表位破坏情况。发现酶解频率很高的位点为P1位的K、V、F、R、Y、I、S以及L和P1’位的D、A、V、I、L、F、E、R、Y、N、T、Q、S、W和P。对比UniProt数据库中蛋白的完整氨基酸序列,可以发现,α-LA中的K98-V99和K108-A109,β-LG中的K138-A139、R148-L149、L32-D33、V41-Y42、Y42-V43和S21-L22,αs1-CN中的F23-F24、R22-F23、D85-V86、K124-E125和D175-A176,αs2-CN中的K173-F174、K150-T151、K152-L153、K166-I167、L161-N162和R170-Y171,β-CN中的L163-S164、L165-S166、L140-Q141、L125-T126和L127-T128等肽键被高效水解。酶切位点位于相对应的表位内,且位于蛋白表面和内部的表位均有被酶解。表明猪笼液蛋白酶拥有广泛的水解特异性,对牛乳蛋白致敏原蛋白表位表现出不同程度的破坏作用,猪笼液有望成为新型蛋白酶的开发的资源,消减牛乳主要致敏原蛋白致敏性的同时实现农业废弃物的再利用。
值得注意的是,对于质谱鉴定到的肽段,通过对比致敏原表位数据库可以发现α-LA、β-LG、αs1-CN、αs2-CN和β-CN的表位被有效酶解。虽然致敏原蛋白的线性表位被酶解,但产物肽段仍有一部分与表位是重合的,这部分是否还能致敏仍需要进一步体内实验来验证。同时也可以发现,质谱并未鉴定到来自κ-CN的肽段,可能是由于κ-CN产物肽段低于5个氨基酸残基或高于30个氨基酸残基甚至是κ-CN完全没被酶解。结合电泳结果来看,19 kDa的κ-CN酶解程度较高,但泳道4中仍存在20 kDa左右的条带6;一个均一体系内的酶和底物反应,同一底物出现一部分被反应完全而另一部分完全未发生反应是几乎不可能的事情;并且猪笼液蛋白酶具有广泛的水解特异性,所以κ-CN很大概率被完全酶解;同时由κ-CN氨基酸序列可知,其包含多个猪笼液蛋白高频率酶裂解位点,且分子结构简单,有被完全酶解的分子基础;因此,可以判定电泳结果中泳道4的条带6不属于κ-CN的残留,猪笼液蛋白酶对κ-CN具有很高的亲和力,酶解彻底,产生的肽段低于5个氨基酸残基。
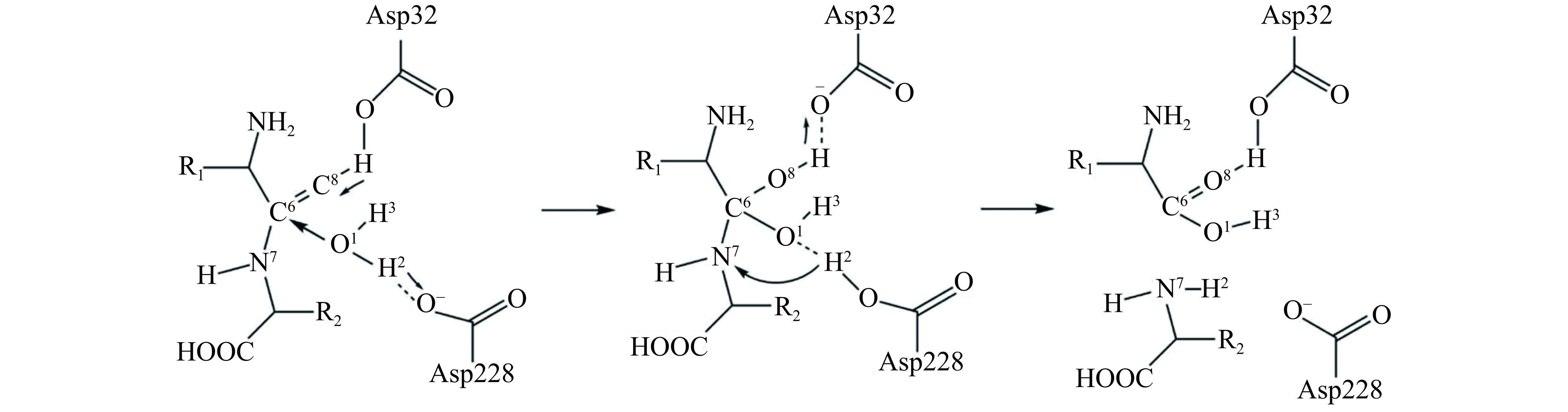
虽然本研究中猪笼液蛋白酶与底物的反应机制还需进一步探索,但目前学术界普遍认为,天冬氨酸蛋白酶家族遵循类似的催化机制[25]。如图7所示,水解肽键时,催化二元体中的两个天冬氨酸残基一个被质子化作为酸,一个没有被质子化作为碱并从相邻的保守水分子中获取一个质子,此过程产生的羟基作为亲核试剂,进攻底物肽键的羰基碳,酶分子中被酸化的天冬氨酸将其质子提供给底物肽键的羰基氧原子,形成四面体中间体结构;下一步酶分子中两个关键天冬氨酸残基作用互换,在上一步失去质子后的天冬氨酸残基从四面体中间体的羟基中得到一个质子,在上一步得到质子的天冬氨酸残基将它的质子提供给底物肽键的酰胺氮原子,肽键断裂[26]。在此过程中,底物以其一部分接触酶的活性位点,然后酶与底物相互调节,以使彼此有更多的部分相适应,更好地发生反应,符合Koshland等[27]的诱导契合学说。同时另一项研究也发现,与其它植物天冬氨酸蛋白酶不同,nepenthesin Ⅱ活性中心的天冬氨酸形成的结构能够容纳更多的独特侧链种类及其空间结构[28],所以这也解释了为什么猪笼液蛋白酶能够表现出广泛的水解特异性并水解产生更短的多肽。
大部分酸性蛋白酶的最适作用pH范围为3.8~5.6[29],在调味料生产[30]、蛋白水解[31]、明胶生产[32]、凝乳[33]及肉类嫩化[34]等方面应用广泛。本研究中猪笼液蛋白酶的最适作用pH范围为2.05~2.50,虽然上述食物基质几乎不会或很少达到此范围,猪笼液蛋白酶的应用范围会因此受限,但猪笼液蛋白酶特殊的作用pH范围也为其提供了应用在特殊领域的可能性,比如作为胃蛋白酶的替代,满足氢/氘交换质谱在较低pH条件下完成更高的底物催化效率的需求;还能够以片剂或其他制剂的形式用作乳糜泻患者治疗的助消化剂[14,35]。本研究也为猪笼液蛋白酶应用在消减牛乳蛋白致敏性上提供了一个良好的开端。
综上所述,Nepenthes×Miranda的猪笼液蛋白酶具有广泛的水解特异性,有效酶解了乳清蛋白和酪蛋白,并且断裂了其中主要致敏原蛋白表位上的肽键,有效破坏了表位。虽然乳蛋白酶解后肽段与致敏原表位有重叠的序列部分,其体内致敏性还需进一步研究,但本研究为消减乳蛋白致敏性提供了理论依据,也为解决猪笼液中蛋白酶资源的浪费的问题提供了一个行之有效的技术方案。
-
图 2 α-LA表位分布、结构和酶切位点分布
注:(a)~(c)Eα-LA1和Eα-LA2在α-LA上的分布位置及主要酶切位点的分布;(d)~(f)Eα-LA3在α-LA上的分布位置及主要酶切位点的分布;(a)~(f)中橙色表示表位;(a)、(c)、(d)和(f)中绿色代表组成被酶解肽键氨基酸残基的碳原子,红色代表组成被酶解肽键氨基酸残基的氧原子,深蓝色代表组成被酶解肽键氨基酸残基的氮原子;(b)和(e)中绿色代表酶水解位置。
Figure 2. Distribution of α-LA epitopes, structure and distribution of enzymatic sites
表 1 不同泳道肽段相对含量百分比(%)
Table 1 Relative percentages of peptide content in different lanes (%)
条带编号 泳道1 泳道2 泳道3 泳道4 1 12.561 9.315 7.104 1.581 2 5.789 20.496 16.784 1.841 3 26.625 5.500 43.396 11.433 4 55.026 33.166 10.571 1.867 5 − 11.085 0.968 5.946 6 − 20.438 4.121 4.279 7 − − 2.604 1.792 8 − − 1.247 10.591 9 − − 4.120 1.535 10 − − 4.813 7.252 11 − − 4.271 14.194 12 − − − 16.007 13 − − − 8.238 14 − − − 13.442 注:−表示此位置无可见的蛋白条带;条带编号(1~14)对应图1中每条电泳条带右边的编号。 表 2 质谱鉴定后肽段与α-LA过敏原表位数据库线性表位对比
Table 2 Comparison of linear epitopes of peptides identified by mass spectrometry with the α-LA allergen epitope database
表位
类型数据库中的α-LA
线性表位表位在α-LA中的
位置表位所含氨基酸残基数 HWPC中含有该表位部分序列的肽段种类数 B细胞表位 CEVFRELKDLKGYGG 6~20 15 5 ELKDLKGYGGVSLPE 11~25 15 14 LKGYGGVSLPEW 15~26 12 1 KGYGGVSLPEWVCTT 16~30 15 2 GYGGVSLPEWVCTTFHTSGYDTQAIVQNNDSTEYGLFQINNK
(Eα-LA3)17~58 42 42 DSTEYGLFQINNKIW 46~60 15 5 SCDKFLDDDLTDDIM 76~90 15 3 FLDDDLTDDIMCVK 80~93 14 7 CVKKILDKVGINYWL
(Eα-LA1)91~105 15 6 T细胞表位 DKVGINYWLAHKALCSEK(Eα-LA2) 97~114 18 11 表 3 酶解α-LA后获得的数目较多的肽段
Table 3 Higher number of peptides obtained after enzymatic cleavage of α-LA
肽段序列 肽段编号 数目 VGINYWLAHK Pα-LA1 23 STEYGLFQINNK Pα-LA2 4 FLDDDLTDDIM Pα-LA3 4 DTQAIVQNND Pα-LA4 4 DLKGYGGVSL Pα-LA5 4 DKVGINYWLAHK Pα-LA6 4 表 4 质谱鉴定后肽段与β-LG过敏原表位数据库线性表位对比
Table 4 Comparison of linear epitopes of peptides identified by mass spectrometry with the β-LG allergen epitope database
表位类型 数据库中的β-LG线性表位 表位在β-LG中的位置 表位所含氨基酸残基数 HWPC中含有该表位部分序列的肽段种类数 B细胞表位 LAMAASDISLLDAQSAPLRV(Eβ-LG1) 22~41 20 14 DISLLDAQSAPLRVYVEELK(Eβ-LG2) 28~47 20 20 VYVEELKPTPEGDLEILLQK 41~60 20 6 VEELKPTPEGDLEILLQKWE 43~62 20 12 AQKKIIAEKTKIPAVFKIDA 67~86 20 1 KIIAEKTKIPAVFKIDALNE 70~89 20 3 TKIPAVFKIDALNENKVLVL 76~95 20 1 VLDTDYKKYLLFCMENSAEP 94~113 20 2 YLLFCMENSAEPEQSLACQCLVR 102~124 23 2 CMENSAEPEQSLACQCLVRT 106~125 20 1 ACQCLVRTPEVDDEALEKFD 118~137 20 6 DEALEKFDKALKALPMHIRL 130~149 20 9 LEKFDKALKALPMHIRLSFN 133~152 20 10 KALPMHIRLSFNPTQ 141~155 15 1 T细胞表位 LIVTQTMKGLDIQKVAGTWYS(Eβ-LG3) 1~21 21 32 TMKGLDIQKVAGTWYSLAMAASDISLLDAQ 6~35 30 20 KPTPEGDLEILLQKWENGECA 47~67 21 9 KIPAVFKIDALNENKVLVLDT 77~97 21 15 DALNENKVLVLDTDYKKY 85~102 18 10 LKALPMHIRLSFNPT 140~154 15 2 ALPMHIRLSFNPTQLEEQCHI 142~162 21 8 表 5 酶解β-LG后获得的数目较多的肽段
Table 5 Higher number of peptides obtained after enzymatic cleavage of β-LG
肽段序列 肽段编号 数目 DAQSAPLRV Pβ-LG1 42 ALKALPMHIR Pβ-LG2 38 DAQSAPLRVY Pβ-LG3 18 VEELKPTPEGDLEI Pβ-LG4 16 IQKVAGTWYS Pβ-LG5 11 LIVTQTMKGLDIQK Pβ-LG6 11 DKALKALPMHIR Pβ-LG7 9 LIVTQTMKGL Pβ-LG8 8 LSFNPTQL Pβ-LG9 6 表 6 质谱鉴定后肽段与αs1-CN过敏原表位数据库线性表位对比
Table 6 Comparison of linear epitopes of peptides identified by mass spectrometry with the αs1-CN allergen epitope database
表位类型 数据库中的αs1-CN线性表位 表位在αs1-CN
中的位置表位所含氨基
酸残基数HCN中含有该表位部分
序列的肽段种类数B细胞表位 RPKHPIKHQGLPQEVLNENLLRFFVAPFPEV(Eαs1-CN1) 1~31 31 44 VLNENLLRFFVAPFPEVFGKEKVNELSKDIGS 15~46 32 27 FGKEKVNELSKDIGSESTEDQAMEDIKQMEAES 32~64 33 9 ISSSEEIVPNSVEQKHIQKEDVPSERYLGYLEQLLR 65~100 36 10 KEDVPSERYLGYLEQLLRLK 83~102 20 31 DVPSERYLGYLEQLLRLKKY 85~104 20 9 VPSERYLGYLEQLLRLKKYKVPQLEIVPNS 86~115 30 4 RLHSMKEGIHAQQKEPMIGVNQELAYFYPE 119~148 30 25 PMIGVNQELAYFYPELFRQFYQLDAYPSGAWYYV 134~167 34 15 FYQLDAYPSGAWYYVPLGTQYTDAPSFSDI 153~182 30 12 PLGTQYTDAPSFSDIPNPIGSENSEKTTMPLW 168~199 32 28 表 7 酶解αs1-CN后获得的数目较多的肽段
Table 7 Higher number of peptides obtained after enzymatic cleavage of αs1-CN
肽段序列 肽段编号 数目 RPKHPIKHQGLPQEVLNENLLRF Pαs1-CN1 41 RPKHPIKHQGLPQEVLNENLLR Pαs1-CN2 12 除Pαs1-CN1和Pαs1-CN2外N端为RPKH的其它肽段 Pαs1-CN3 16 N端为FVAP的肽段 Pαs1-CN4 39 N端为FFVA的肽段 Pαs1-CN5 20 N端为APFP的肽段 Pαs1-CN6 18 N端为EGIH的肽段 Pαs1-CN7 17 N端为IPNP的肽段 Pαs1-CN8 16 表 8 质谱鉴定后肽段与αs2-CN过敏原表位数据库线性表位对比
Table 8 Comparison of linear epitopes of peptides identified by mass spectrometry with the αs2-CN allergen epitope database
表位类型 数据库中的αs2-CN线性表位 表位在αs2-CN
中的位置表位所含氨基
酸残基数HCN中含有该表位部分
序列的肽段种类数B细胞表位 CKEVVRNANEEEYSIGSSSE 40~59 20 2 KITVDDKHYQKALNEINQFY 70~89 20 5 KHYQKALNEINQFYQKFPQY 76~95 20 11 QKALNEINQFYQKFPQYLQY 79~98 20 4 FYQKFPQYLQYLYQGPIVLN 88~107 20 14 QYLYQGPIVLNPWDQVKRNA 97~116 20 6 PIVLNPWDQVKRNAVPITPT 103~122 20 5 NAVPITPTLNREQLSTSEEN 115~134 20 2 EVFTKKTKLT 145~154 10 2 TKKTKLTEEEKNRLNFLKKI(Eαs2-CN2) 148~167 20 10 EKNRLNFLKKISQRYQKFALPQYLKT(Eαs2-CN1) 157~182 26 16 KTVYQHQKAMKPWIQPKTKVIPYVRYL 181~207 27 16 T细胞表位 EEKNRLNFLKKISQR 156~170 15 2 YQKFALPQYLKTVYQ 171~185 15 13 表 9 酶解αs2-CN后获得的数目较多的肽段
Table 9 Higher number of peptides obtained after enzymatic cleavage of αs2-CN
肽段序列 肽段编号 数目 N端为FALP的肽段 Pαs2-CN1 19 N端为YQKF的肽段 Pαs2-CN2 14 NFLKKISQRYQK Pαs2-CN3 10 N端为TVYQ的肽段 Pαs2-CN4 8 由Pαs2-CN4产生的N端为TKKT、TKLT和LTEE以及C端为KKLF的肽段 Pαs2-CN5 若干 表 10 质谱鉴定后肽段与β-CN过敏原表位数据库线性表位对比
Table 10 Comparison of linear epitopes of peptides identified by mass spectrometry with the β-CN allergen epitope database
表位类型 数据库中的β-CN线性表位 表位在β-CN中的位置 表位所含氨基酸残基数 HCN中含有该表位部分
序列的肽段种类数B细胞表位 RELEELNVPGEIVESLSSSE 1~20 20 4 RINKKIEKFQSEEQQQTEDELQDKIH 25~50 26 2 QSEEQQQTEDELQDKIHPFA 34~53 20 1 DELQDKIHPFAQTQSLVYPF 43~62 20 10 TQSLVYPFPGPIPNSLPQ 55~72 18 1 PLTQTPVVVPPFLQPEVMGV 76~95 20 7 QTPVVVPPFLQPEVMGVSKV 79~98 20 2 VVVPPFLQPEVMGVSKVKEA 82~101 20 12 EAMAPKHKEMPFPKYPVEPF 100~119 20 21 APKHKEMPFPKYPVEPFTES 103~122 20 1 PFPKYPVEPFTESQSLTLTD 110~129 20 24 ESQSLTLTDVENLHLPLPLL (Eβ-CN1) 121~140 20 33 SLTLTDVENLHLPLPLLQSW 124~143 20 9 VENLHLPLPLLQSWMHQPHQ(Eβ-CN2) 130~149 20 34 TVMFPPQSVLSLSQSKVLPV 154~173 20 10 SQSKVLPVPQKAVPYPQRDM 166~185 20 16 QKAVPYPQRDMPIQAFLLYQ 175~194 20 11 PQRDMPIQAFLLYQEPVLGP 181~200 20 2 IQAFLLYQEPVLGPVRGPFP 187~206 20 1 FLLYQEPVLGPVRGPFPIIV 190~209 20 30 表 11 酶解β-CN后获得的数目较多的肽段
Table 11 Higher number of peptides obtained after enzymatic cleavage of β-CN
肽段序列 肽段编号 数目 N端为QSWM的肽段 Pβ-CN1 22 N端为SLSQ的肽段 Pβ-CN2 20 N端为YQEP的肽段 Pβ-CN3 20 N端为SQSK的肽段 Pβ-CN4 18 N端为WMHQ的肽段 Pβ-CN5 17 N端为VENL的肽段 Pβ-CN6 15 N端为TDVE的肽段 Pβ-CN7 13 N端为TLTD的肽段 Pβ-CN8 11 N端为DVEN的肽段 Pβ-CN9 9 N端为NLHL的肽段 Pβ-CN10 9 N端为PVLG的肽段 Pβ-CN11 9 -
[1] LEE N A, LOPATA A L, COLGRAVE M L. Analytical methods for allergen control in food processing[J]. Foods,2023,12(7):1439. doi: 10.3390/foods12071439
[2] MATTHAI J, SATHIASEKHARAN M, PODDAR U, et al. Guidelines on diagnosis and management of cow's milk protein allergy[J]. Indian Pediatrics,2020,57(8):723−729. doi: 10.1007/s13312-020-1916-5
[3] BLANCHARD E, ZHU P, SCHUCK P. Infant formula powders[M]. Handbook of Food Powders, 2013:465-483.
[4] BØGH K L, LARSEN J M. Reducing allergenicity by proteolysis[M]. Agents of Change, 2021:499-523.
[5] HOCHWALLNER H, SCHULMEISTER U, SWOBODA I, et al. Cow's milk allergy:From allergens to new forms of diagnosis, therapy and prevention[J]. Methods,2014,66(1):22−33. doi: 10.1016/j.ymeth.2013.08.005
[6] D’AURIA E, SALVATORE S, POZZI E, et al. Cow's milk allergy:Immunomodulation by dietary intervention[J]. Nutrients,2019,11(6):1399. doi: 10.3390/nu11061399
[7] GOLKAR A, MILANI J M, VASILJEVIC T. Altering allergenicity of cow's milk by food processing for applications in infant formula[J]. Critical Reviews in Food Science and Nutrition,2019,59(1):159−172. doi: 10.1080/10408398.2017.1363156
[8] ZHANG Q, CHEN Q H, HE G Q. Effect of ultrasonic-ionic liquid pretreatment on the hydrolysis degree and antigenicity of enzymatic hydrolysates from whey protein[J]. Ultrasonics Sonochemistry,2020,63:104926. doi: 10.1016/j.ultsonch.2019.104926
[9] QUINTIERI L, MONACI L, BARUZZI F, et al. Reduction of whey protein concentrate antigenicity by using a combined enzymatic digestion and ultrafiltration approach[J]. Journal of Food Science and Technology,2017,54(7):1910−1916. doi: 10.1007/s13197-017-2625-5
[10] YE M, XU Z H, TAN H K, et al. Allergenicity reduction of cow milk treated by alkaline protease combined with Lactobacillus plantarum and Lactobacillus helveticus based on epitopes[J]. Food Chemistry,2023,421:136180. doi: 10.1016/j.foodchem.2023.136180
[11] RAVEE R, MOHD SALLEH F I, GOH H H. Discovery of digestive enzymes in carnivorous plants with focus on proteases[J]. PeerJ,2018,6:e4914. doi: 10.7717/peerj.4914
[12] FERNÁNDEZ-FERNÁNDEZ Á D, STAEL S, VAN BREUSEGEM F. Mechanisms controlling plant proteases and their substrates[J]. Cell Death and Differentiation,2023,30(4):1047−1058. doi: 10.1038/s41418-023-01120-5
[13] BUCH F, KAMAN W, BIKKER F, et al. Nepenthes in protease activity indicates digestive fluid dynamics in carnivorous Nepenthes plants[J]. PLoS One,2015,10(3):e0118853. doi: 10.1371/journal.pone.0118853
[14] BEKALU Z E, DIONISIO G, BRINCH P, et al. Molecular properties and new potentials of plant nepenthesins[J]. Plants,2020,9(5):570. doi: 10.3390/plants9050570
[15] ATHAUDA S B P, MATSUMOTO K, RAJAPAKSHE S, et al. Enzymic and structural characterization of nepenthesin, a unique member of a novel subfamily of aspartic proteinases[J]. Biochemical Journal,2004,381(1):295−306. doi: 10.1042/BJ20031575
[16] PANG L D, LIU M, LI X, et al. Effect of enzymatic hydrolysis combined with processing on allergenicity of food allergens[J]. Trends in Food Science & Technology,2023,143:104248.
[17] SAKAI K, YOSHINO K, SATTER M A, et al. Effects of pH variation and NaCl on in vitro digestibility of cow's milk proteins in commercially available infant formulas[J]. Journal of Nutritional Science and Vitaminology,2000,46(6):325−328. doi: 10.3177/jnsv.46.325
[18] JAKOPOVIĆ K L, CHEISON S C, KULOZIK U, et al. Comparison of selective hydrolysis of α-lactalbumin by acid protease A and protease M as alternative to pepsin:Potential for β-lactoglobulin purification in whey proteins[J]. Journal of Dairy Research,2019,86(1):114−119. doi: 10.1017/S0022029919000086
[19] REY M, YANG M, BURNS K M, et al. Nepenthesin from monkey cups for hydrogen/deuterium exchange mass spectrometry[J]. Molecular & Cellular Proteomics,2013,12(2):464−472.
[20] 国家卫生和计划生育委员会. GB 1886.174-2016 食品安全国家标准 食品添加剂 食品工业用酶制剂[S]. 北京:中国标准出版社, 2016. [National Health and Family Planning Commission. GB 1886.174-2016 National standard for food safety food additives enzyme preparation for food industry[S]. Beijing:Standards Press of China, 2016.] National Health and Family Planning Commission. GB 1886.174-2016 National standard for food safety food additives enzyme preparation for food industry[S]. Beijing: Standards Press of China, 2016.
[21] PESLERBES M, FELLENBERG A, JARDIN J, et al. Manufacture of whey protein hydrolysates using plant enzymes:Effect of processing conditions and simulated gastrointestinal digestion on angiotensin-Ⅰ-converting enzyme (ACE) inhibitory activity[J]. Foods,2022,11(16):2429. doi: 10.3390/foods11162429
[22] OLIVEIRA J P B, CANDREVA A M, RIZZO G, et al. Allergenicity reduction of cow's milk proteins using latex peptidases[J]. Food Chemistry,2019,284:245−253. doi: 10.1016/j.foodchem.2019.01.123
[23] 王靖舒. 鸡蛋卵转铁蛋白模拟胃肠消化产物的致敏性[D]. 南昌:南昌大学, 2022. [WANG J S. Allergenicity of the digestied egg ovotransferrin in the simulated gastrointestinal fluids in vitro[D]. Nanchang:Nanchang University, 2022.] WANG J S. Allergenicity of the digestied egg ovotransferrin in the simulated gastrointestinal fluids in vitro[D]. Nanchang: Nanchang University, 2022.
[24] 黄佳露. 强化β-酪蛋白和α-乳白蛋白对乳蛋白配方的消化性和抗炎活性的研究[D]. 无锡:江南大学, 2022. [HUANG J L. Digestibility and anti-inflammatory activity of β-casein and α-lactalbumin fortified milk protein formula[D]. Wuxi:Jiangnan University, 2022.] HUANG J L. Digestibility and anti-inflammatory activity of β-casein and α-lactalbumin fortified milk protein formula[D]. Wuxi: Jiangnan University, 2022.
[25] 赵玥. 胃蛋白酶活力和热稳定性理性设计研究[D]. 上海:华东师范大学, 2021. [ZHAO Y. Studies on rational design of activity and thermostability improvements of pepsin[D]. Shanghai:East China Normal University, 2021.] ZHAO Y. Studies on rational design of activity and thermostability improvements of pepsin[D]. Shanghai: East China Normal University, 2021.
[26] PAUL T J, BARMAN A, OZBIL M, et al. Mechanisms of peptide hydrolysis by aspartyl and metalloproteases[J]. Physical Chemistry Chemical Physics,2016,18(36):24790−24801. doi: 10.1039/C6CP02097F
[27] KOSHLAND D E J R, RAY W J J R, ERWIN M J. Protein structure and enzyme action[J]. Federation Proceedings, 1958, 17(4):1145−1150.
[28] ZHENG J, STRUTZENBERG T S, REICH A, et al. Comparative analysis of cleavage specificities of immobilized porcine pepsin and nepenthesin Ⅱ under hydrogen/deuterium exchange conditions[J]. Analytical Chemistry,2020,92(16):11018−11028. doi: 10.1021/acs.analchem.9b05694
[29] RAZZAQ A, SHAMSI S, ALI A, et al. Microbial proteases applications[J]. Frontiers in Bioengineering and Biotechnology,2019,7:110. doi: 10.3389/fbioe.2019.00110
[30] ALI B, KHAN K Y, MAJEED H, et al. Production of ingredient type flavoured white enzyme modified cheese[J]. Journal of Food Science and Technology,2019,56(4):1683−1695. doi: 10.1007/s13197-018-3526-y
[31] WANG L L, YAO Q, YUE J, et al. Application of acid protease in the industrial production of corn ethanol[J]. Systems Microbiology and Biomanufacturing,2022,2:361−368. doi: 10.1007/s43393-021-00075-3
[32] BKHAIRIA I, MHAMDI S, JRIDI M, et al. New acidic proteases from Liza aurata viscera:Characterization and application in gelatin production[J]. International Journal of Biological Macromolecules,2016,92:533−542. doi: 10.1016/j.ijbiomac.2016.07.063
[33] ARTIFON S E S, SUMNY E H, FABRIS T, et al. Biosynthesis of the acid protease produced by Lacticaseibacillus casei LBC 237 and Limosilactobacillus fermentum LBF 433 and their potential application in the bovine milk clotting[J]. Food Bioscience,2023,54:102879. doi: 10.1016/j.fbio.2023.102879
[34] MOHD AZMI S I, KUMAR P, SHARMA N, et al. Application of plant proteases in meat tenderization:Recent trends and future prospects[J]. Foods,2023,12(6):1336. doi: 10.3390/foods12061336
[35] REY M, YANG M, LEE L, et al. Addressing proteolytic efficiency in enzymatic degradation therapy for celiac disease[J]. Scientific Reports,2016,6:30980. doi: 10.1038/srep30980
-
期刊类型引用(10)
1. 胡业慧,段凯露,梅显贵,徐凌峰,范圣此. 山药采收加工过程中非药用部位及副产物的资源化利用研究进展. 中国现代中药. 2025(02): 374-380 .  百度学术
百度学术
2. 刘盈,张欣,刘会平,王兵,马笑笑,张慧慧,李灿. 西瓜皮中3种多糖的初步表征及抗氧化活性对比. 食品研究与开发. 2024(04): 110-117 .  百度学术
百度学术
3. 袁高阳,秦心睿,聂晓兵,金文芳,杨玉玉,刘诗菡,范宝磊,苗潇磊. 基于熵权TOPSIS模型对白芨多糖脱蛋白体系的评价研究. 食品工业科技. 2024(07): 76-85 .  本站查看
本站查看
4. 杨欣宇,高赛,刘寅初,高晓炜,冯随,高晓光. 响应面法优化山楂多糖和山药多糖的提取工艺及其抗氧化活性研究. 中国调味品. 2024(07): 38-44+59 .  百度学术
百度学术
5. 黄子芬,李晨月,徐柠檬,刘晨,赵海婷,陈勇杏,华燕,郭磊. 真空干燥对美味牛肝菌生物活性成分及其抗氧化活性的影响. 中国食品添加剂. 2024(07): 55-61 .  百度学术
百度学术
6. 马传贵,张志秀,沈亮,贺宗毅. 山药多糖药理作用及提取技术研究进展. 蔬菜. 2024(07): 22-27 .  百度学术
百度学术
7. 刘权,刘雨婷,霍滢,肖驰骋,李欣. 食品与生物样品糖类及其衍生物检测方法研究进展. 食品安全质量检测学报. 2024(14): 115-124 .  百度学术
百度学术
8. 张喜贺,李淑娅,宋新玲,陈伟. 茯苓多糖组分对复合乳酸菌的冻干保护作用及机理. 食品与发酵工业. 2024(18): 134-140 .  百度学术
百度学术
9. 阚金涛,皮正林,杨锴莉,赵津好,刘笑焱,张玉锋. 硫酸化修饰对椰子吸器多糖结构和抗氧化活性的影响. 食品工业科技. 2024(20): 114-123 .  本站查看
本站查看
10. 曾婷,杨然,李庆军,宫亚楠,王书珍,刘谦. 基于PI3K/AKT/Gsk-3β通路研究山药多糖对运动疲劳小鼠的影响. 山东科学. 2024(06): 51-58 .  百度学术
百度学术
其他类型引用(3)





 下载:
下载:
 下载:
下载:
