Screening of A β-Galactosidase-producing Pediococcus acidilactici Strain from Cheese and Its Characteristics for Galactooligosaccharides Synthesis
-
摘要: 为了给益生性低聚半乳糖(GOS)的合成提供新的乳酸菌酶源,本研究从新疆传统奶酪中筛选具有高转糖苷活性的β-半乳糖苷酶产酶菌株。在以乳糖为单一碳源的MRS固体培养基中加入显色剂5-溴-4-氯-3-吲哚-β-D-半乳糖苷(X-gal),以菌落生长颜色进行初筛,以粗酶液酶活性以及转糖苷反应混合物的薄层色谱(TLC)进行复筛,结合其形态学、生理生化特征及16S rRNA序列同源性分析对产转糖苷活性的β-半乳糖苷酶菌株进行鉴定。单因素实验确定菌株的产酶条件和粗酶液的转糖苷反应条件,高效液相色谱分析转糖苷反应产物的组分及含量。筛选获得产转糖苷活性β-半乳糖苷酶的菌株6株,其中Pediococcus acidilactici Y1的产酶水平和转糖苷活性最高。单因素实验结果表明,P. acidilactici Y1在20 mg/mL乳糖,37 ℃培养18 h时,产酶水平最高可达(15.52±0.34)U/g。在初始乳糖浓度300 mg/mL,加酶量4.24 U/mL,50 ℃反应28 h时,GOS得率最高为38.4%±0.56%(w/w),乳糖残留量为30.9%±0.44%(w/w)。其中转移二糖、转移三糖以及转移四糖的质量分数分别为15.6%±0.21%(w/w)、18.3%±0.15%(w/w)和4.5%±0.01%(w/w)。以上结果表明,P. acidilactici Y1是一株产转糖苷活性β-半乳糖苷酶的新菌株,在益生性GOS的合成领域具有应用前景。Abstract: This study aimed to provide a new lactic acid bacteria (LAB) enzyme source for synthesizing probiotic galactooligosaccharides (GOS). It screened β-galactosidase producing strains with high transglycosidase activity from traditional cheese in Xinjiang, China. A modified MRS medium with lactose as the single carbon source supplemented with 5-bromo-4-chloro-3-indole-β-D-galactoside was used for the first-round screening of LAB strains producing β-galactosidases. Thin layer chromatography (TLC) analysis of the reaction products catalyzed by the crude enzyme from selected strains was used for the second-round screening. The isolate with the highest transglycosylation activity was identified based on the physiological and biochemical characteristics, along with the sequence analysis of the 16S rRNA gene. The conditions for enzyme production and transglycosylation reaction were determined by one-way experiment. TLC combined with high performance liquid chromatography was conducted to analyze the content of each GOS component. Six strains producing β-galactosidase with transglycosidase activity were screened, among which Pediococcus acidilactici Y1 exhibited the highest enzyme production level and transglycosidase activity. After optimization, P. acidilactici Y1 produced β-galactosidase at a level of up to 15.52±0.34 U/g (with 20 mg/mL of initial lactose after 18 h at 37 ℃). The highest GOS yield of 38.4%±0.56% (w/w) was achieved with an initial lactose concentration of 300 mg/mL, an enzyme amount of 4.24 U/mL, and a reaction temperature of 50 ℃ for 28 h. Also, the residual rate of lactose decreased to 30.9%±0.44% (w/w). The content of transfer disaccharide, transfer trisaccharide, and transfer trisaccharide was 15.6%±0.21% (w/w), 18.3%±0.15% (w/w), and 4.5%±0.01% (w/w), respectively. The results of this study showed that P. acidilactici Y1 was a new β-galactosidase-producing strain, with application prospects in the synthesis of probiotic GOS.
-
低聚半乳糖(galacto-oligosaccharides,GOS)是由一个末端葡萄糖单元与2个或多个半乳糖单元组成的一种益生元[1−2]。GOS能够促进肠道双歧杆菌和乳杆菌等益生菌在肠道内的定殖,在婴儿配方奶粉中的功能特性已得到广泛的认可[3−4]。GOS还具有抑制致病菌在结肠上皮的附着,降低血清胆固醇和血压,预防结肠癌和增强免疫力等益生特性[5]。现有商业化GOS合成主要通过β-半乳糖苷酶转糖苷活性催化乳糖制得[6]。β-半乳糖苷酶可以以乳糖为底物转移半乳糖基合成不同聚合度和糖苷键类型的GOS[7]。由于以β-半乳糖苷酶催化合成的GOS得率、产物聚合度以及糖苷键类型很大程度上取决于酶的来源[8]。因此,筛选不同微生物来源的高转糖苷活性β-半乳糖苷酶对于制备具有特定聚合度和糖苷键的GOS具有重要意义。
目前商业化的β-半乳糖苷酶主要来源于Klyuveromyces lactis (Lactozym 3000L HPG,Novo Nordisk),Klyuveromyces fragilis (Maxilact LGI 5000,DSM Food Specialities),Aspergillus aculeatus (Pectinex Ultra SP-L,Novozymes),Aspergillus oryzae (Enzeco™ Fungal Lactase,Enzyme Development Corporation), Bifidobacterium bifidum (Saphera® 2600L,Novozyme),Bacillus circulans (Biolactasa® NTL*2,Biocon)。这些商业化β-半乳糖苷酶的GOS转化率多在20%~50%之间[9]。但乳酸菌来源的β-半乳糖苷酶因其安全性好(Generally Recognized as Safe,GRAS),由其制备的酶制剂无需进行高度纯化即可应用于食品行业,显著降低了下游处理成本[10−11]。此外,已有研究表明来源乳酸菌的β-半乳糖苷酶对β-(1-6)和β-(1-3)糖苷键具有较高的偏好性,因而以其催化乳糖制备的GOS对肠道益生菌具有更强的增殖效应[12−15]。乳酸菌广泛存在于不同的发酵乳制品中,如奶酪和发酵乳。而新疆传统奶酪是由未经巴氏杀菌的牛乳与空气中的微生物、原料乳自然发酵制成,在发酵过程中通常不使用发酵剂而仅由存在于牛乳和环境中的原生微生物群产生[16−17]。因此,从新疆传统奶酪中筛选乳酸菌具有极大的优势。目前,以来源于Bifidobacterium和Lactobacillaceae属的β-半乳糖苷酶以乳糖为底物合成GOS的催化特性和产物组成研究最为全面深入[18−23],这些乳酸菌来源的β-半乳糖苷酶合成GOS的得率多在30%~40%[24]。然而对Pediococcus属来源β-半乳糖苷酶的酶学特性,特别是对其转糖苷活性的研究较少[25−26]。目前,仅见一株P. acidilactici来源的β-半乳糖苷酶合成GOS条件的初步研究,但未明确报道该β-半乳糖苷酶合成GOS的得率和组成[27]。因此,从P. acidilactici属中挖掘具有转糖苷活性的新型β-半乳糖苷酶在工业化应用中具有重要意义。
本研究筛选并鉴定了一株具有转糖苷活性的β-半乳糖苷酶Pediococcus acidilactici Y1产生菌株,该菌株来源于新疆伊犁少数民族牧民家中的奶酪样品。对其产β-半乳糖苷酶的不同培养基组成以及催化乳糖水解反应过程中的酶促转糖苷条件进行测定,并对合成的GOS组成成分进行分析,该菌株产β-半乳糖苷酶水平高于已报道的P. acidilactici其他菌株且具有较强的转糖苷活性,为安全高效合成GOS提供了新的β-半乳糖苷酶产酶菌株。
1. 材料与方法
1.1 材料与仪器
奶酪样品 从新疆伊犁地区少数民族牧民家中采集,4 ℃条件下保存备用;MRS培养基(1 L):分别称取乳糖(20 g)、牛肉膏(10 g)、蛋白胨(10 g)、酵母浸粉(5 g)、K2HPO4(5 g)、柠檬酸氢二铵(2 g)、乙酸钠(5 g)、MgSO4·7H2O(0.58 g)、MnSO4 (0.25 g),吐温-80 1 mL,pH(6.2~6.4),121 ℃灭菌15 min;(固体培养基:再加入20 g琼脂粉);优化培养基:将所有碳源单独灭菌然后将每种碳源加入高压灭菌的MRS培养基中(其他成分及含量保持不变)以达到最终所需浓度;5-溴-4-氯-3-吲哚-β-D-半乳糖(X-Gal)、邻硝基苯-β-D-半乳糖苷(o-nitrophenyl β-D-galactopyranoside,o-NPG) 生工生物工程(上海)股份有限公司;Silica gel 60 No.553薄层色谱(Thin Layer Chromatography, TLC)硅胶板 默克公司;细菌基因组提取试剂盒 南京诺唯赞生物科技股份有限公司;其他所用化学试剂均为分析纯,所有用于光谱学研究的溶剂均为色谱级。
LC-10A高效液相色谱(配有RID-10A示差检测器) 岛津(上海)实验器材有限公司;氨基柱APS-2 HYPERSIL(4.6 mm×250 mm)、Multiskan FC型酶标仪、Applied Biosystems PCR热循环仪 赛默飞世尔科技公司;色谱柱Hi-Plex Na Column(300 mm×7.7 mm) 美国Agilent(中国)有限公司;CF1524R高速冷冻型微量离心机 美国赛洛捷克公司;超声波细胞粉碎机 宁波新芝生物科技股份有限公司;倒置荧光显微镜 重庆奥特光学仪器有限责任公司。
1.2 实验方法
1.2.1 奶酪样品处理与菌株分离纯化
在超净工作台内取出保存好的奶酪样品,使用无菌小刀将奶酪切至粉末状,称取5 g置于45 mL已经灭过菌的生理盐水中,混合均匀之后在含有X-gal的以乳糖为碳源的MRS固体培养基中进行稀释涂布,将平板倒置于37 ℃恒温培养24~48 h。挑取能够水解X-gal且颜色为蓝色的单菌落在MRS固体平板上划线,并重复平板划线的操作直至得到纯培养物。使用MRS培养基制成的斜面以及40%甘油对纯化的菌株进行保藏,分别放置于4 ℃与−80 ℃保藏,以便后续实验的进行。
1.2.2 β-半乳糖苷酶产生菌株粗酶液的获取及酶活力测定
参考韩明明等[23]方法对收集的湿菌体进行破碎以及粗酶液提取,方法稍有修改。挑取蓝色单菌落于MRS培养基中37 ℃,200 r/min培养18 h。之后在4 ℃条件下冷冻离心(8000 r/min,10 min)发酵液,收集菌体。使用pH7.0,0.1 mol/L的磷酸盐缓冲液洗涤3次,按照1 mL/1 g ww(wet weight)的比例加入磷酸盐缓冲液重悬菌体,在重悬液中加入终浓度为1 mg/mL的溶菌酶37 ℃孵育2.5 h,冰浴条件下超声破碎(工作3 s停6 s、功率125 W、时间15 min),12000 r/min冷冻离心20 min,上清液即为粗酶液。取20 μL粗酶液与80 μL 0.1 mol/L磷酸缓冲液(pH7.0)配制的o-NPG溶液在适宜温度下反应10 min后于酶标孔中加入终止液(100 μL 1 mol/L碳酸钠溶液),并在420 nm处使用酶标仪测定吸光度,计算酶活力,筛选出高产β-半乳糖苷酶活力的菌株。对照组采用20 μL已灭活的粗酶液在相同条件下参与反应。酶活单位定义为:1 min催化水解1 μmol o-NPG所需的β-半乳糖苷酶的量。酶活力(E)计算公式如式(1)所示:
(1) 式中:OD420:反应结束时酶反应液420 nm处吸光值;ODblank:反应结束时空白对照420 nm处吸光值;N:酶液稀释倍数;t:酶液与底物反应时间,min;0.2:将 200 µL 稀释液折算为 1 mL;50:将 20 µL 稀释液折算为1 mL。
1.2.3 β-半乳糖苷酶产生菌株的转糖苷活性分析
参考王丽军等[28]方法对β-半乳糖苷酶产生菌株的转糖苷活性进行TLC分析。取超声破碎获得的粗酶液50 μL,与150 μL 300 mg/mL的乳糖溶液反应,于40 ℃反应12 h,100 ℃处理10 min终止反应,反应混合物以无水乙醇稀释至合适浓度,12000 r/min离心1 min,取上清液点样至加热活化过的薄层硅胶板,以正丁醇-乙醇-水(5:3:2,V/V/V)的混合溶剂为展开剂进行展开,显色剂(3,5二羟基甲苯0.5%(w/v)溶解在20%的硫酸中)喷雾于TLC板后于烘箱中110 ℃烘烤10 min显色。
1.2.4 菌落形态及生理生化分析
取培养一定时间的菌液,在相同的培养基中进行稀释,将其在含X-gal和不含X-gal的以乳糖为碳源的MRS固体平板中37 ℃恒温培养并对菌落的生长形态进行观察记录。用移液器吸取少量菌液于载玻片上进行革兰氏染色,显微镜观察菌体形态。通过一系列糖、醇类的发酵实验,淀粉水解实验以及氨基酸和蛋白质的代谢试验对菌株的生理生化现象进行分析[29]。
1.2.5 16S rRNA基因的克隆及其序列分析
细菌DNA提取试剂盒对产β-半乳糖苷酶的菌株提取其基因组DNA,并使用细菌特异性引物27F与1492R进行PCR扩增,将扩增产物送至上海派森诺生物技术有限公司测序。测定的16S rRNA序列,通过NCBI-BLAST进行数据库搜索,使用软件MEGA 7.0进行多序列比对以及系统发育树的构建,确定菌株的种属分布。
1.2.6 β-半乳糖苷酶产生菌株的培养基配方选择
1.2.6.1 碳源选择
参考Chanalia等[27]方法对最优碳源进行优化,略有修改。保持MRS液体培养基中的其他成分及含量不变,在MRS培养基中分别加入不同的碳源:20 mg/mL乳糖、20 mg/mL蔗糖、20 mg/mL葡萄糖、20 mg/mL麦芽糖、20 mg/mL果糖,以不加任何碳源为对照,在37 ℃,200 r/min培养18 h,以菌株的产酶水平为筛选依据,测定粗酶液酶活力。
1.2.6.2 碳源质量浓度选择
参考Bhowmik等[26]方法探究碳源浓度对菌株产酶水平的影响。将培养基中的碳源浓度分别调整为:5、10、20、30 mg/mL,在37 ℃,200 r/min培养18 h,以菌株的产酶水平为筛选依据,测定粗酶液酶活力。
1.2.6.3 生长曲线与产酶曲线的测定
参考韩明明等[23]方法考察发酵时间对菌株产酶水平的影响。将培养好的种子液(2%接种量)接种至β-半乳糖苷酶改良的MRS培养基中,每隔4 h测一次菌体浓度(OD600 nm)以及对应的粗酶液酶活力,在37 ℃,200 r/min的条件下培养至24 h。
1.2.7 β-半乳糖苷酶转糖苷及反应条件的选择
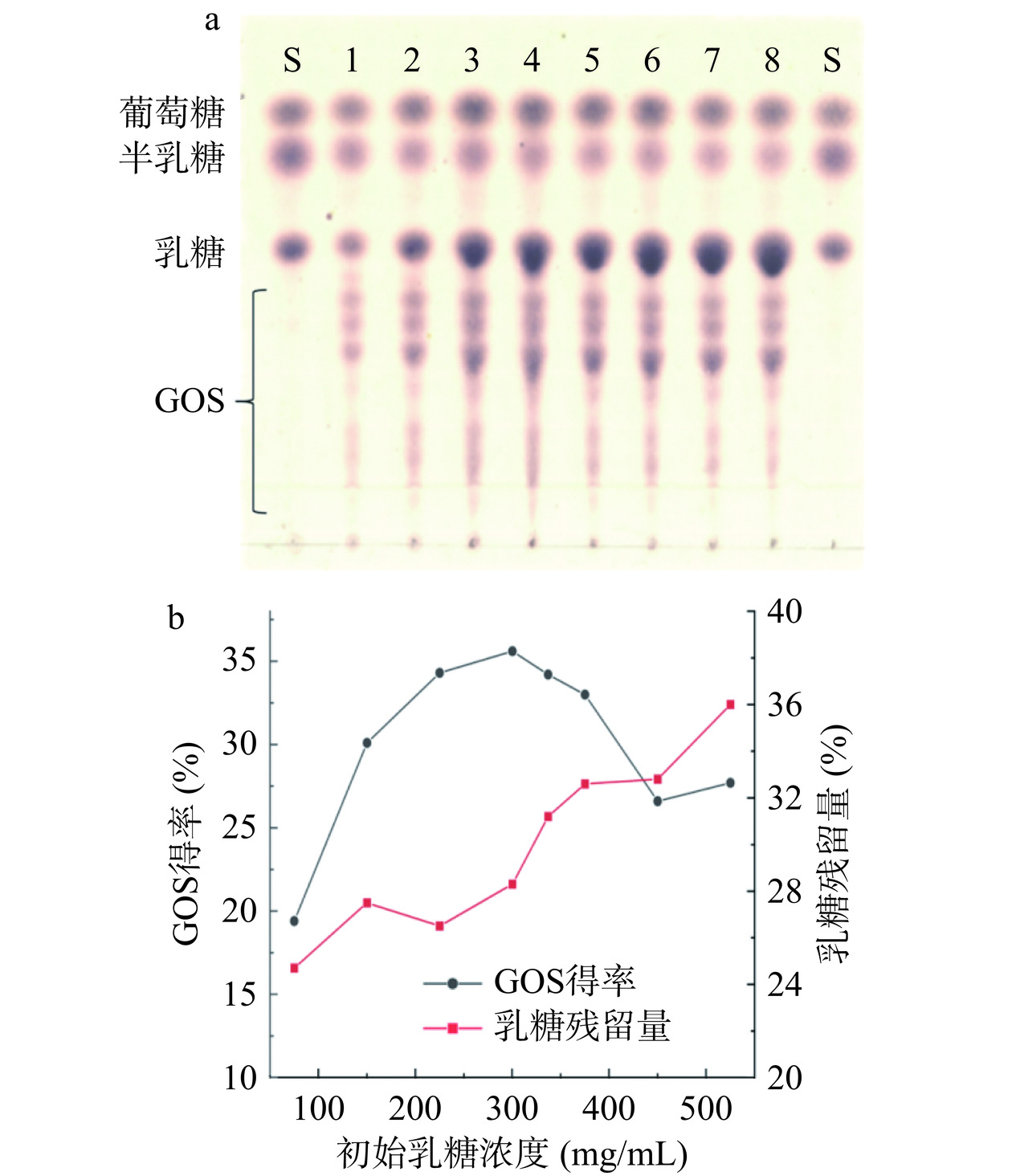
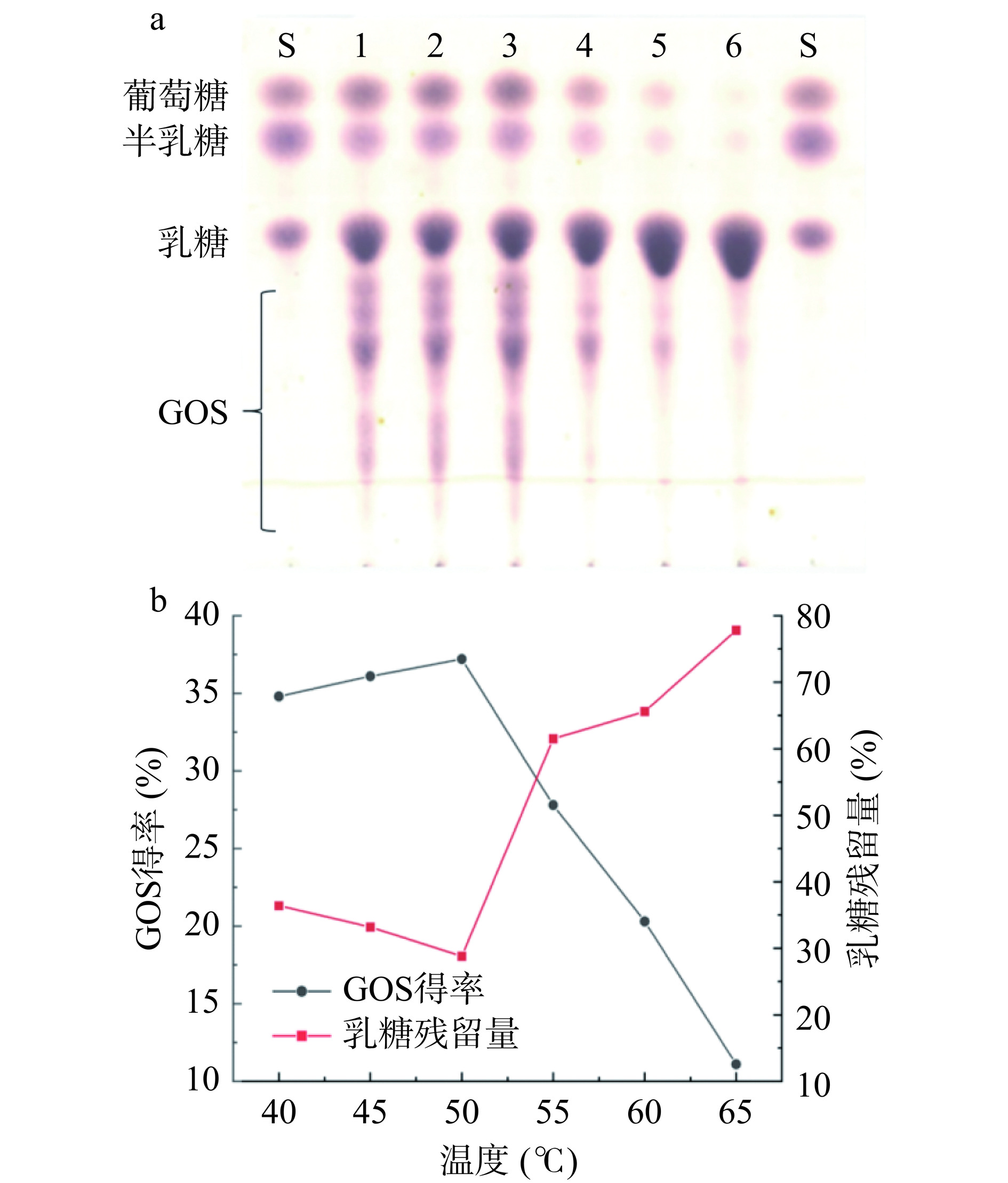
参考韩明明等[23]方法对不同初始质量浓度的乳糖溶液(75、150、225、300、337、375、450、525 mg/mL,pH7.0)、不同反应温度(40、45、50、55、50、60、65 ℃)和不同反应时间(2、4、6、8、12、16、20、24、28、32、40、48 h)条件下的转糖苷反应产物进行TLC与高效液相色谱(HPLC)分析。其中对探究不同初始质量浓度的乳糖溶液以及不同反应温度的TLC板进行扫描并使用图像软件ImageJ对转糖苷产物进行半定量测定。
1.2.8 高效液相色谱(HPLC)分析
参考许俊勇[9]的方法对转糖苷反应产物成分进行HPLC分析,方法略有修改。使用屈臣氏水(三蒸水)对转糖苷反应产物进行稀释并经0.22 μm滤膜后进样分析。反应时间对GOS得率的测定使用Aps-2HYPERSIL (4.6 mm×250 mm) 色谱柱和RID-10A示差检测器进行分析,流动相为70%(v/v)乙腈/水溶液,流速和柱温分别设置为1 mL/min和40 ℃,进样量10 μL。参考高鑫[30]的方法,采用Hi-Plex Na (300 mm×7.7 mm)色谱柱和示差检测器RID-10A对最优转苷条件下合成的GOS的组成成分进行分析,流动相为屈臣氏水(三蒸水),流速和柱温分别设置为0.2 mL/min和80 ℃,进样量20 μL。参考韩明明等[23]方法对转糖苷产物进行定性分析并采用岛津LabSolutions色谱数据处理系统进行峰面积积分,用归一化法计算低聚半乳糖得率以及乳糖残留量,计算方法如式(2)、式(3)所示:
(2) (3) 1.3 数据处理
所有实验测定至少进行三次且均设置空白对照,数据结果使用SPSS软件进行处理,P<0.05表示具有显著差异。结果显示为平均值±标准差,使用Origin Pro 8软件对实验图谱进行绘制。
2. 结果与分析
2.1 β-半乳糖苷酶产生菌株的筛选与鉴定
2.1.1 β-半乳糖苷酶产生菌株的筛选
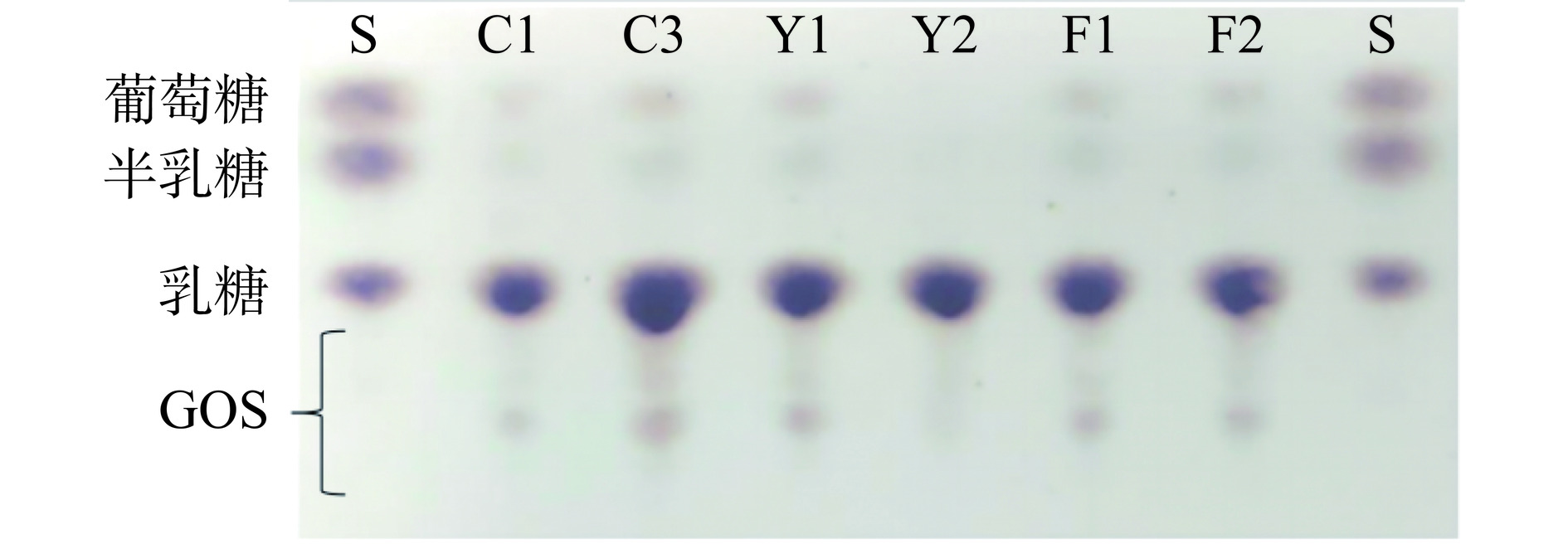
初筛以乳糖为唯一碳源从奶酪样品中分离出能够水解X-gal的菌株。挑取蓝色菌落的菌株进行摇瓶发酵获得粗酶液,不同初筛菌株的产酶水平如表1所示。初筛获得的6株菌株的β-半乳糖苷酶活力最高为Y1菌株(14.66±0.69)U/g。
表 1 不同菌株的产酶水平Table 1. β-Galactosidase production level in different strains菌株编号 酶活力(U/g) C1 9.57±0.65 C3 8.60±0.13 F1 6.08±0.08 F2 12.34±0.55 Y1 14.66±0.69 Y2 9.72±0.95 进一步,以粗酶液(0.1 U/mL)与300 mg/mL乳糖在40 ℃条件下反应12 h,通过TLC对转苷反应产物进行分析。结果如图1所示,6株菌均有转半乳糖苷产物生成。结合酶活测定和TLC分析结果,选择Y1菌株开展进一步的研究。
2.1.2 β-半乳糖苷酶产生菌株的鉴定
2.1.2.1 形态学与生理生化鉴定
将Y1菌株分别在含X-gal的MRS和不含X-gal的MRS固体培养基中进行恒温培养,并对平板上的菌落形态进行观察,结果如图2所示,菌落颜色为乳白色,边缘整齐,表面湿润且有光泽,形态为圆形(图2a),在含有X-gal的MRS固体培养基中呈蓝色(图2b),革兰氏染色为阳性(图2c)。
菌株Y1的生理生化特征如表2所示,在进行乳糖、半乳糖、葡萄糖发酵时产酸但不产气,鉴定为阳性;在进行蔗糖、麦芽糖、木糖、棉子糖、甘露醇、山梨醇发酵时既不产酸也不产气,鉴定为阴性;淀粉水解结果为阴性,明胶液化结果为阴性,过氧化氢酶实验结果为阴性,硫化氢生成实验结果为阴性,基于以上的生理生化特征,可以推测出菌株Y1属于片球菌属(Pediococcus sp.)。
表 2 生理生化结果分析Table 2. Identification of physiological and biochemical results检测项目 结果 检测项目 结果 乳糖 + 棉子糖 − 半乳糖 + 木糖 − 麦芽糖 − 淀粉水解 − 葡萄糖 + 过氧化氢酶 − 蔗糖 − 硫化氢生成 − 山梨醇 − 明胶液化 − 甘露醇 − 注:+:阳性反应;−:阴性反应。 2.1.2.2 分子生物学分析
对菌株Y1的16S rRNA基因序列进行PCR扩增,获得1572 bp大小的扩增片段。通过NCBI-BLAST进行数据库搜索并构建系统发育树。结果如图3所示,菌株Y1的16S rRNA基因序列与多株Pediococcus acidilactici的16S rRNA基因序列相似性最高。结合形态学、生理生化及分子生物学鉴定结果,将菌株Y1暂定为Pediococcus acidilactici Y1,并提交至中国典型培养物保藏中心保藏(CCTCC M 20232025)。
2.2 β-半乳糖苷酶产生菌株的培养基组成条件测定
2.2.1 碳源对菌株生长及产酶能力的影响
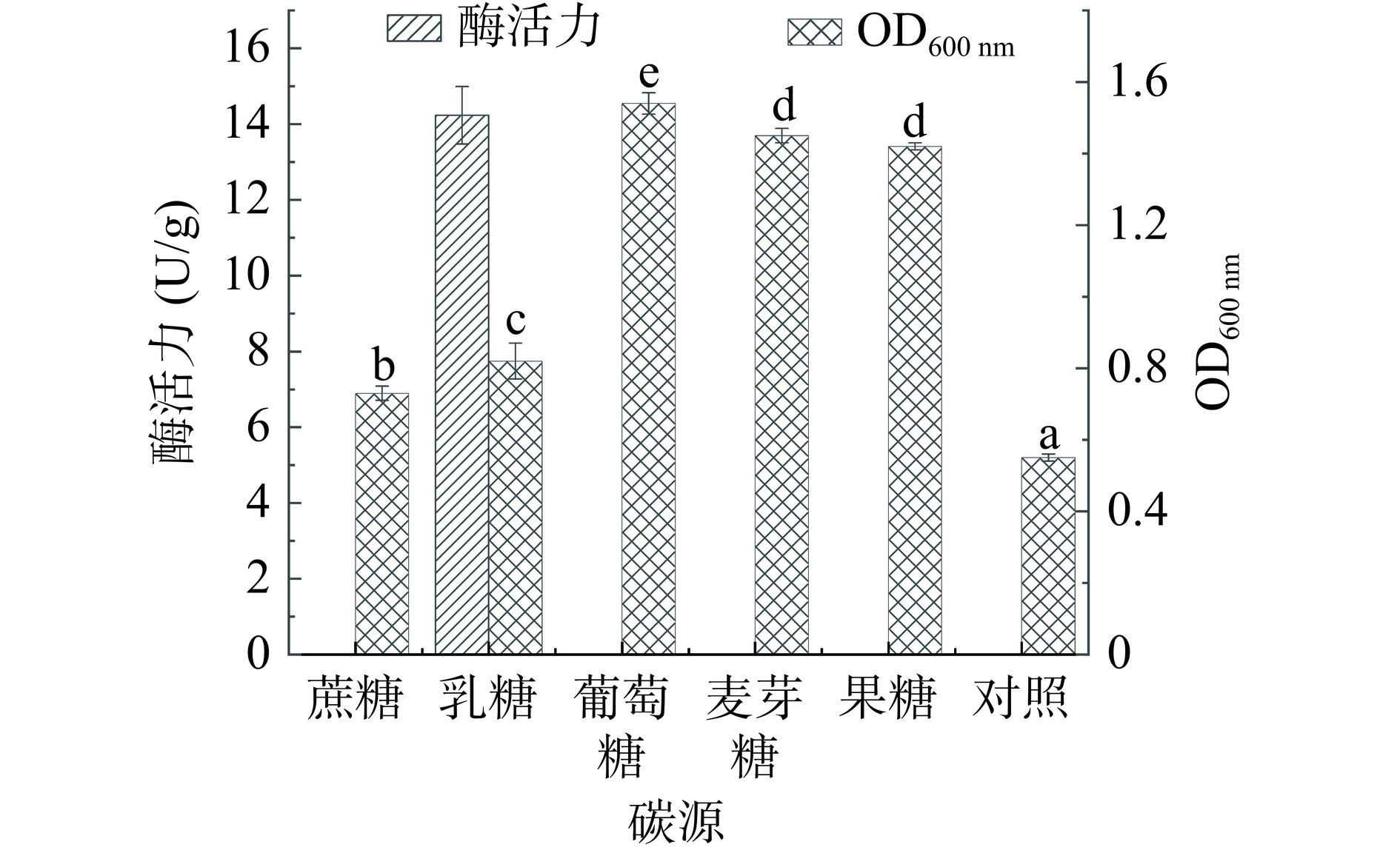
有研究表明,碳源在β-半乳糖苷酶生物合成过程中的作用因微生物而异[31]。在含有乳糖、葡萄糖、蔗糖、麦芽糖、以及果糖作为唯一碳源的培养基中检测到菌株的OD600 nm和β-半乳糖苷酶的酶活力如图4所示。结果显示仅在以乳糖为单一碳源时,P. acidilactici Y1能够产生β-半乳糖苷酶,其酶活力为(14.23±0.76)U/g,OD600 nm为(0.82±0.04),在葡萄糖、蔗糖、麦芽糖、果糖为碳源时均不能产β-半乳糖苷酶,这与已报道的来源于P. pentosaceus ATCC 25745的β-半乳糖苷酶严格受乳糖诱导产酶的特性一致[26]。与本研究中分离的产β-半乳糖苷酶菌株P. acidilactici Y1不同,印度学者Chanalia等[27]发现一株P. acidilactici 菌株在以葡萄糖、半乳糖、乳糖、麦芽糖及蔗糖为单一碳源的培养基中均可检测出β-半乳糖苷酶的活性,乳糖对该菌株表达β-半乳糖苷酶没有明显的诱导效应。而本研究筛选的P. acidilactici Y1所产β-半乳糖苷酶为乳糖诱导型。
2.2.2 乳糖浓度对菌株生长以及β-半乳糖苷酶表达能力的影响
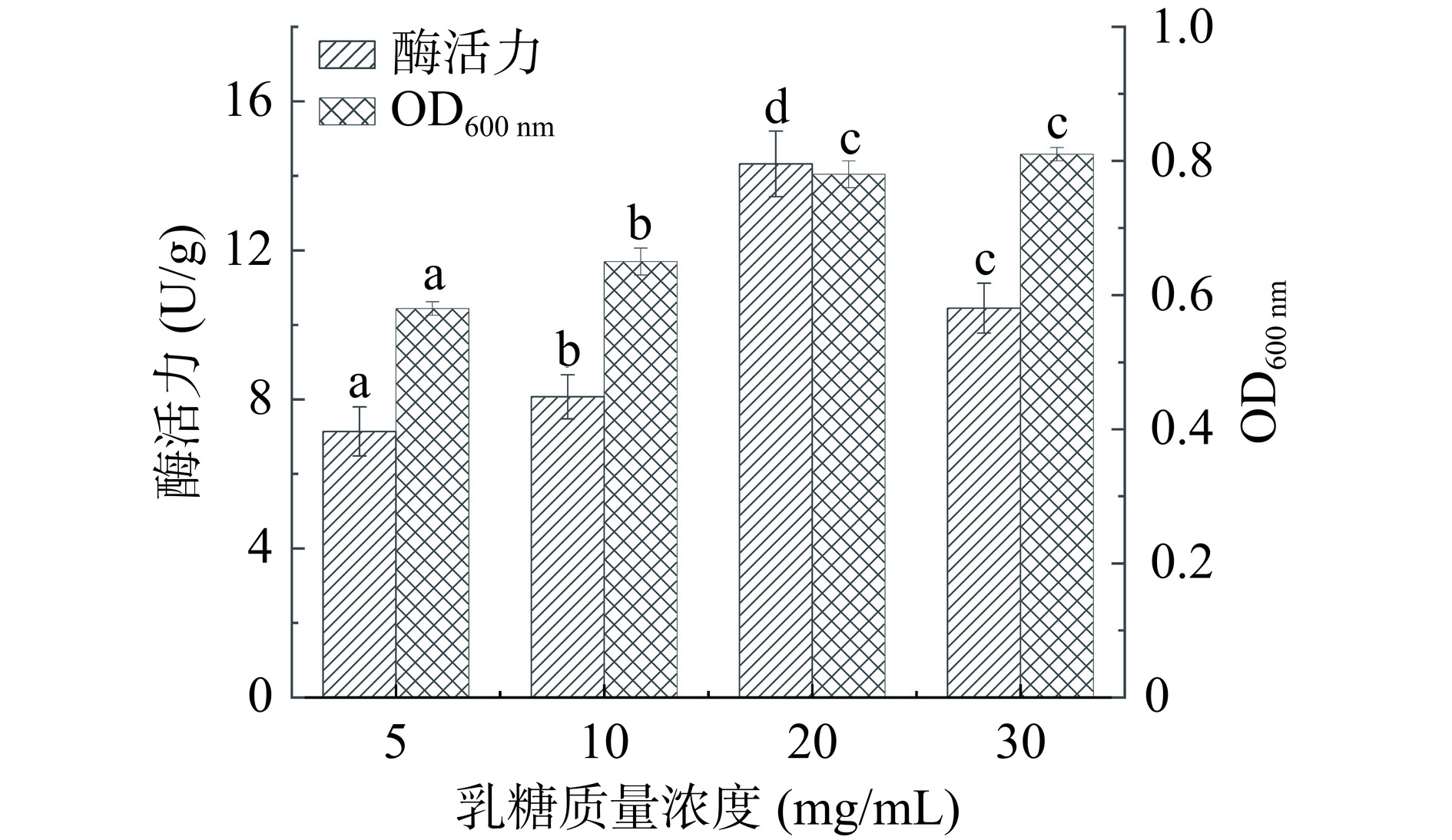
乳糖浓度对不同Pediococcus属菌株的β-半乳糖苷酶表达影响也不同。结果如图5所示,在乳糖浓度为5~30 mg/mL的培养基中观察到P. acidilactici Y1菌体OD600 nm范围为0.58~0.81,且菌株的产酶水平随着乳糖浓度的升高而增加,直至乳糖浓度为20 mg/mL时观察到β-半乳糖苷酶最高活性为(14.32±0.13)U/g,进一步增加乳糖浓度会导致菌株的产酶水平降低。已有研究表明P. pentosaceus ATCC 25745和P. acidilactici ATCC 25740分别在3%和0.5%的乳糖质量浓度中检测到最高β-半乳糖糖苷酶的活性[26]。这些结果表明,Pediococcus属来源β-半乳糖苷酶的表达调控在菌株水平存在显著差异。
2.2.3 发酵时间对菌株生长以及产酶能力的影响
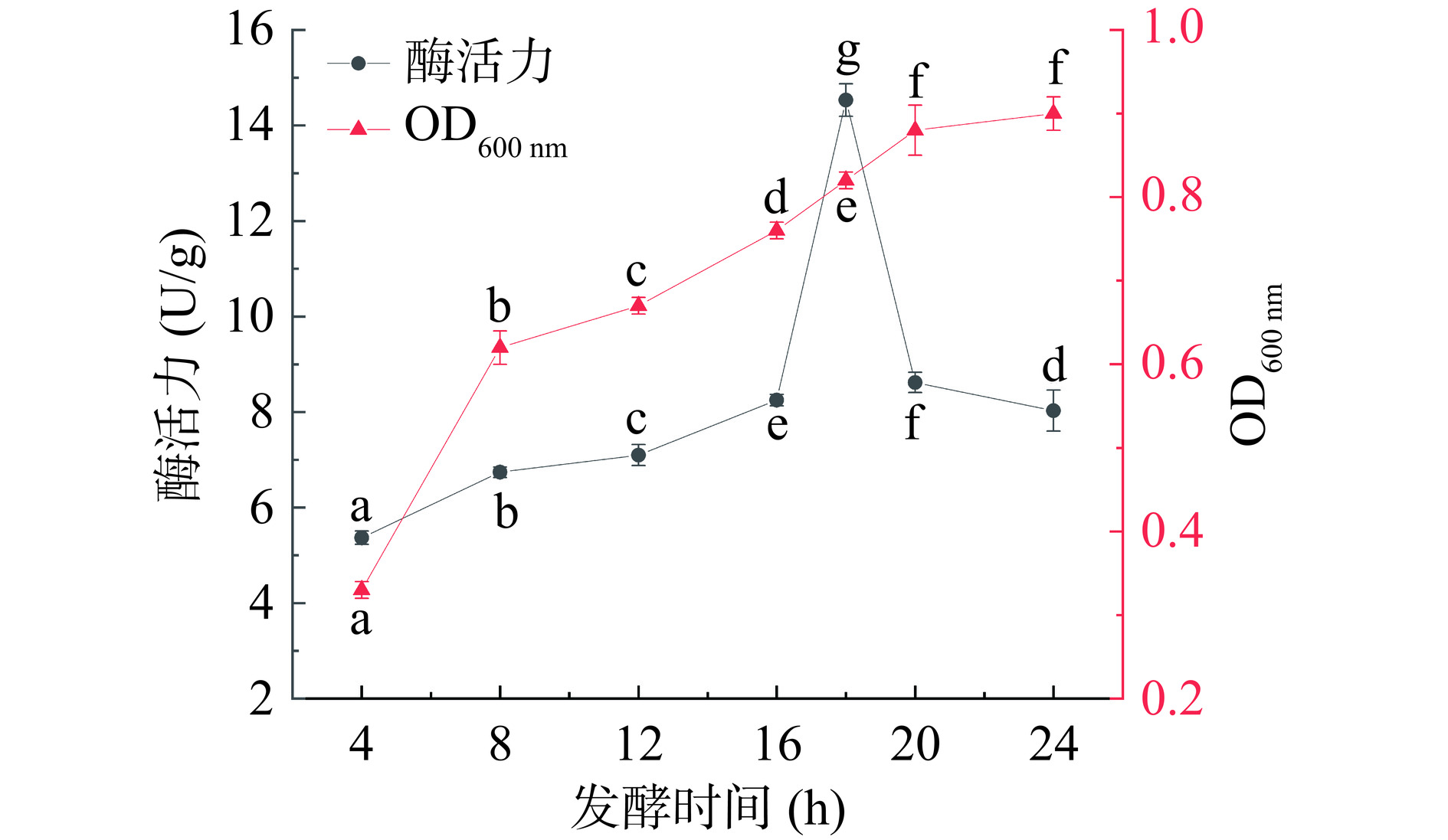
在改良的MRS液体培养中考察发酵时间对菌株产酶水平的影响。结果如图6所示,P. acidilactici Y1的菌体浓度随着发酵的开始而逐渐增加,并在培养约20 h后达到最大值,此后,菌体浓度未发生显著性变化。另一方面,P. acidilactici Y1的产酶水平从发酵开始显著增加(P<0.05),在发酵16 h后菌株的产酶水平最高达到最大值(15.52±0.34)U/g,OD600 nm为(0.82±0.05)。随着发酵时间的增加,产酶水平开始降低,发酵20 h时,菌株的酶活力降至(8.62±0.13)U/g。
2.3 以乳糖为底物合成GOS条件的测定
2.3.1 初始乳糖质量浓度
β-半乳糖苷酶催化乳糖合成GOS是水解反应和转半乳糖苷反应竞争的平衡[31]。有文献报道,在合成GOS的过程中,初始乳糖浓度越高,GOS的得率也就越高[32],但是由于溶解度的限制,初始乳糖浓度不是越高越好。基于此,对乳糖浓度进行了优化。结果如图7所示,GOS得率随着乳糖浓度的增加而增加,当乳糖质量浓度增至300 mg/mL时,GOS得率达到最大值约为35.6%(w/w),乳糖残留量约为28.3%(w/w),进一步增加乳糖浓度,会导致GOS的得率下降。因此,本研究选择300 mg/mL为最佳的初始乳糖浓度。
2.3.2 反应温度对酶促转糖苷反应的影响
反应温度是影响GOS得率的另一个重要因素,随着反应温度的升高,乳糖的溶解度及反应速率均会提高,但酶活性和稳定性也随之降低[32]。不同反应温度对P. acidilactici Y1来源β-半乳糖苷酶催化乳糖制备GOS得率的影响如图8所示。随着反应温度升高,GOS得率呈先升高后降低的趋势,在50 ℃的反应温度下,GOS的得率最高可达约37.2%(w/w);当反应温度升高至65 ℃,GOS得率急剧下降,仅11.1%(w/w),此时乳糖残留量增至77.8%(w/w)。因此,选择50 ℃为最佳反应温度。
2.3.3 反应时间对酶促转糖苷反应的影响
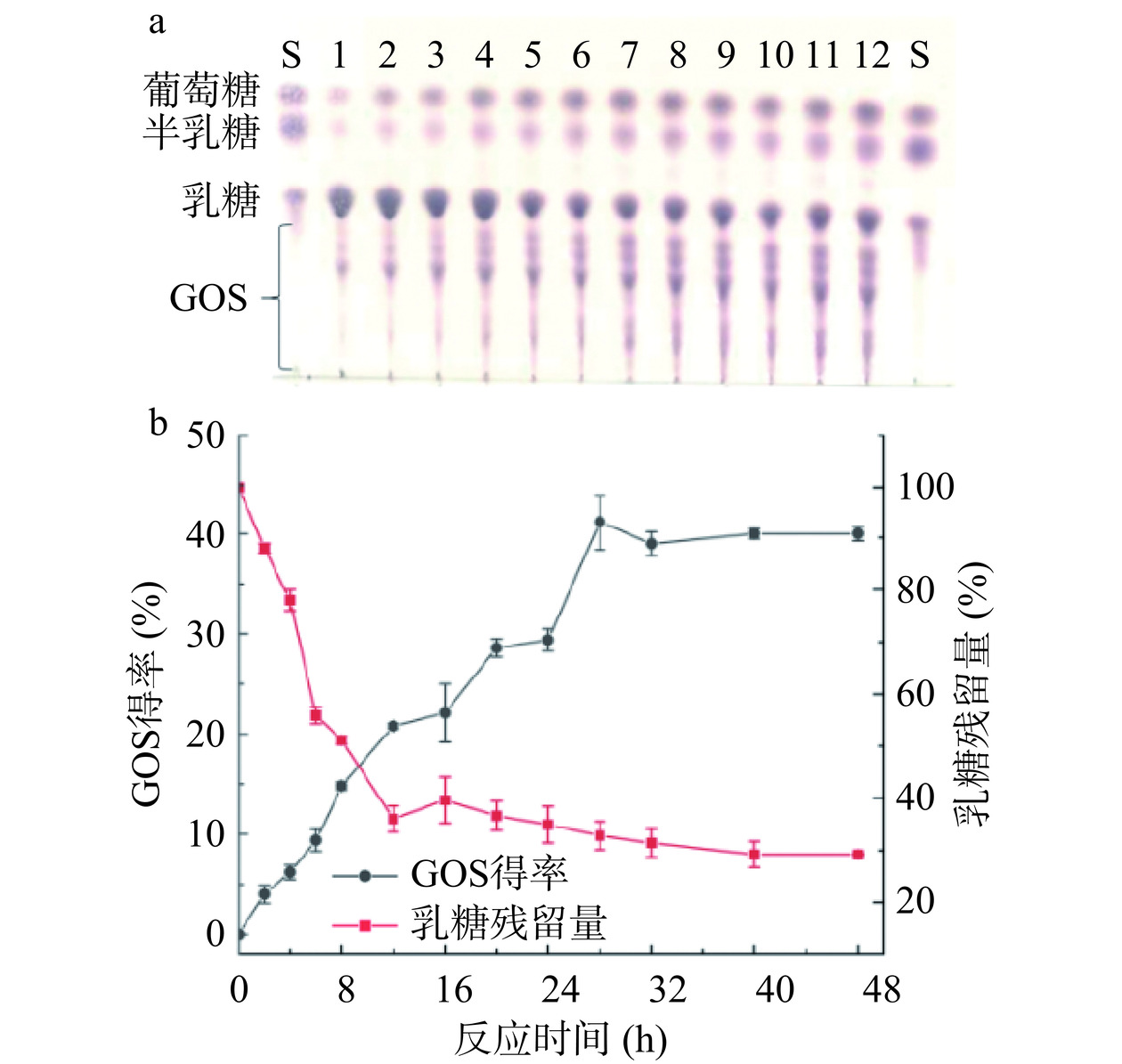
β-半乳糖苷酶以乳糖为底物合成GOS时,GOS的得率受到反应时间的影响,反应后期水解反应占优势导致GOS得率下降[32]。因此,当GOS得率达到最大值时,必须及时停止反应。P. acidilactici Y1来源的β-半乳糖苷酶催化不同反应时间的GOS得率结果如图9所示,在反应开始2 h内就有GOS的生成,并在反应28 h时,GOS得率达到最大,约为41.2%±2.81%(w/w),随着反应的继续,GOS得率开始下降,反应48 h后,GOS的得率下降至40.0%±0.65%(w/w),乳糖残留量为29.2%±0.10%(w/w)。
2.4 以乳糖为底物合成GOS产物的组成分析
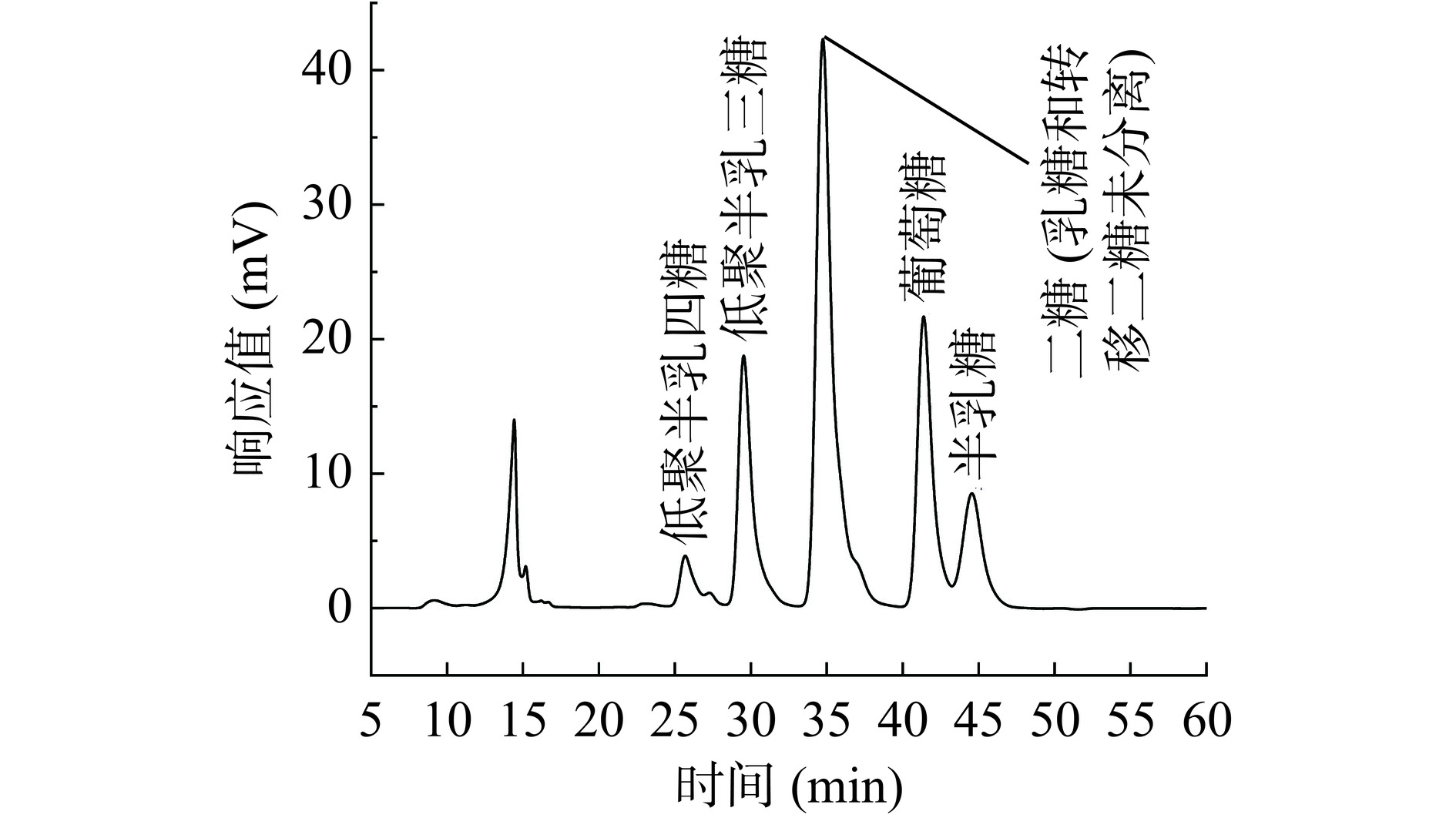
进一步对最优转糖苷条件下GOS的组成成分进行了HPLC分析(图10),通过峰面积积分法对不同GOS组分的得率进行计算,并结合氨基柱的分析结果,可以确定转糖苷产物的组成成分如表3所示,P. acidilactici Y1来源的β-半乳糖苷酶合成GOS得率的最高可达38.4%±0.56%(w/w),由15.6%±0.21%(w/w)的转移二糖,18.3%±0.15%(w/w)的三糖以及4.5%±0.01%(w/w)的低聚半乳四糖组成,残留乳糖质量分数为30.9%±0.44%(w/w)。
目前,已报道的乳酸菌来源的β-半乳糖苷酶合成GOS的得率多在30%~40%[14]。本研究首次对Pediococcus属来源β-半乳糖苷酶合成GOS的得率与组成进行了详细的报道,其合成的GOS的得率(38.4%±0.56%)高于L. plantarum CICC 22186(30.63%)[33]、L. pentosus KUB-ST10-1[34](31%)、B. breve DSM 20213(33%)以及来源于L. bulgaricus 43的β-半乳糖苷酶GOS得率(34%)[35,19]。与来源于L. acidophilus R22的β-半乳糖苷酶的GOS得率(38.5%)以及来源L. fermentum K4的β-半乳糖苷酶的GOS得率(37%)接近[36,22],低于已报道的来源于L. delbrueckii subsp. bulgaricus CRL450[5]的β-半乳糖苷酶的GOS得率(41.3%)以及L. plantarum WCFS1的β-半乳糖苷酶的GOS得率(41%)[21,37]。有研究表明,相比于益生效果不明的GOS-2,聚合度≥3的GOS的益生效果更加显著[30]。P. acidilactici Y1来源的β-半乳糖苷酶合成GOS中三糖以及三糖以上的得率分别为18.3%±0.15%、4.5%±0.01%,聚合度≥3的GOS高于本课题组前期分离的L. kefiri L6来源β-半乳糖苷酶合成的GOS(转移三糖和转移三糖以上的质量分数分别为13.85%,4.15%)[23]、L. reuteri L103[38]的GOS(GOS-3和GOS-4分别17.3%,4.8%)以及L. plantarum MCC2156[39]合成的GOS(17%的转移三糖和4%转移三糖以上的低聚半乳糖)。因此,P. acidilactici Y1来源β-半乳糖苷酶在合成制备高聚合度GOS方面可能更具优势。
表 3 转糖苷产物的组成成分分析Table 3. Component analysis of transglucoside products来源 得率(%) 单糖 二糖 三糖 四糖 总GOS 葡萄糖 半乳糖 乳糖 转移二糖 P.acidilactici Y1 20.4±0.16 10.7±0.08 30.9±0.44 15.6±0.21 18.3±0.15 4.5±0.01 38.4±0.56 3. 结论
本研究从奶酪样品中分离鉴定了一株能够产转糖苷活性β-半乳糖苷酶的P. acidilactici Y1新菌株,并首次对该菌株来源的β-半乳糖苷酶以乳糖为底物合成GOS的反应条件及最优反应条件下GOS的得率与组成进行探究,该酶催化的GOS得率(38.4%±0.56%)较高,与其他乳酸菌来源β-半乳糖苷酶相比在合成益生效果更好的高聚合度的GOS上更具优势。由于乳酸菌的GRAS性质,使得P. acidilactici Y1所产β-半乳糖苷酶在富含GOS发酵乳制品的制备中具有较高的应用价值。本课题组正在对该菌株来源的LacLM型β-半乳糖苷酶进行表达纯化和酶学特性的研究,以期为该酶在发酵乳制品中原位合成制备GOS提供理论依据。
-
表 1 不同菌株的产酶水平
Table 1 β-Galactosidase production level in different strains
菌株编号 酶活力(U/g) C1 9.57±0.65 C3 8.60±0.13 F1 6.08±0.08 F2 12.34±0.55 Y1 14.66±0.69 Y2 9.72±0.95 表 2 生理生化结果分析
Table 2 Identification of physiological and biochemical results
检测项目 结果 检测项目 结果 乳糖 + 棉子糖 − 半乳糖 + 木糖 − 麦芽糖 − 淀粉水解 − 葡萄糖 + 过氧化氢酶 − 蔗糖 − 硫化氢生成 − 山梨醇 − 明胶液化 − 甘露醇 − 注:+:阳性反应;−:阴性反应。 表 3 转糖苷产物的组成成分分析
Table 3 Component analysis of transglucoside products
来源 得率(%) 单糖 二糖 三糖 四糖 总GOS 葡萄糖 半乳糖 乳糖 转移二糖 P.acidilactici Y1 20.4±0.16 10.7±0.08 30.9±0.44 15.6±0.21 18.3±0.15 4.5±0.01 38.4±0.56 -
[1] TORRES D P M, GONCALVES M D P F, TEIXEIRA J A, et al. Galacto-oligosaccharides:production, properties, applications, and significance as prebiotics[J]. Comprehensive Reviews in Food Science and Food Safety,2010,9(5):438−454. doi: 10.1111/j.1541-4337.2010.00119.x
[2] YU L, O'SULLIVAN D J. Production of galactooligosaccharides using a hyperthermophilic β-galactosidase in permeabilized whole cells of Lactococcus lactis[J]. Journal of Dairy Science,2014,97(2):694−703. doi: 10.3168/jds.2013-7492
[3] SHI X, WU D, XU Y, et al. Engineering the optimum pH of β-galactosidase from Aspergillus oryzae for efficient hydrolysis of lactose[J]. Journal of Dairy Science,2022,105(6):4772−4782. doi: 10.3168/jds.2021-21760
[4] 潘玉宁, 刘成志, 颜春荣, 等. 低聚半乳糖的生理功能研究进展[J]. 食品安全质量检测学报,2019,10(10):2849−2855. [PAN Y N, LIU C Z, YAN C R, et al. Research progress of physiological function of galacto-oligosaccharides[J]. Journal of Food Safety & Quality,2019,10(10):2849−2855.] doi: 10.3969/j.issn.2095-0381.2019.10.005 PAN Y N, LIU C Z, YAN C R, et al. Research progress of physiological function of galacto-oligosaccharides[J]. Journal of Food Safety & Quality, 2019, 10(10): 2849−2855. doi: 10.3969/j.issn.2095-0381.2019.10.005
[5] FARA A, SABATER C, PALACIOS J, et al. Prebiotic galactooligosaccharides production from lactose and lactulose by Lactobacillus delbrueckii subsp. bulgaricus CRL450[J]. Food & Function,2020,11(7):5875−5886.
[6] ISHIKAWA K, KATAOKA M, YANAMOTO T, et al. Crystal structure of β-galactosidase from Bacillus circulans ATCC 31382 (BgaD) and the construction of the thermophilic mutants[J]. FEBS Journal,2015,282(13):2540−2552. doi: 10.1111/febs.13298
[7] XU Y P, WU Q, BAI L, et al. Cloning, expression, and bioinformatics analysis and characterization of a β-galactosidase from Bacillus coagulans T242[J]. Journal of Dairy Science,2021,104(3):2735−2747. doi: 10.3168/jds.2020-18942
[8] WU Y F, YUAN S G, CHEN S, et al. Enhancing the production of galacto-oligosaccharides by mutagenesis of Sulfolobus solfataricus β-galactosidase[J]. Food Chemistry,2013,138(2−3):1588−1895. doi: 10.1016/j.foodchem.2012.11.052
[9] 许俊勇. Bacillus circulans ATCC 31382 β-半乳糖苷酶转糖苷产物特异性分子改造及发酵优化[D]. 无锡:江南大学, 2022. [XU J Y. Engineering of transglycosylation products specificity and fermentation optimization of β-galactosidase from Bacillus circulans ATCC 31382[D]. Wuxi:Jiangnan University, 2022.] XU J Y. Engineering of transglycosylation products specificity and fermentation optimization of β-galactosidase from Bacillus circulans ATCC 31382[D]. Wuxi: Jiangnan University, 2022.
[10] CAREVIC M, VUKASINOVIC-SEKULIC M, COROVIC M, et al. Evaluation of β-galactosidase from Lactobacillus acidophilus as biocatalyst for galacto-oligosaccharides synthesis:Product structural characterization and enzyme immobilization[J]. Journal of Bioscience and Bioengineering,2018,126(6):697−704. doi: 10.1016/j.jbiosc.2018.06.003
[11] HE X, HAN N, WANG Y P. Cloning, purification, and characterization of a heterodimeric β-Galactosidase from Lactobacillus kefiranofaciens ZW3[J]. Journal of Microbiology and Biotechnology,2016,26(1):20−27. doi: 10.4014/jmb.1507.07013
[12] KITTIBUNCHAKUL S, PHAM M L, TRAN A M, et al. β-Galactosidase from Lactobacillus helveticus DSM 20075:Biochemical characterization and recombinant expression for applications in dairy industry[J]. International Journal of Molecular Sciences,2019,20(4):947. doi: 10.3390/ijms20040947
[13] DELGADO-FERNANDEZ P, PLAZA-VINUESA L, LIZASOAIN-SANCHEZ S, et al. Hydrolysis of lactose and transglycosylation of selected sugar alcohols by LacA β-Galactosidase from Lactobacillus plantarum WCFS1[J]. Journal of Agricultural and Food Chemistry,2020,68(26):7040−7050. doi: 10.1021/acs.jafc.0c02439
[14] RUIZ-RAMIREZ S, JIMENEZ-FLORES R. Properties of β-Galactosidases derived from Lactobacillaceae species and its capacity for galacto-oligosaccharides production[J]. Journal of Dairy Science,2023,106(12):8193−8206. doi: 10.3168/jds.2023-23392
[15] NAG D, KUMAR V, KUMAR V, et al. A new extracellular β-Galactosidase producing Kluyveromyces sp. PCH397 from yak milk and its applications for lactose hydrolysis and prebiotics synthesis[J]. Indian Journal of Microbiology,2021,61(3):391−395. doi: 10.1007/s12088-021-00955-1
[16] LI J, ZHENG Y, XU H Y, et al. Bacterial microbiota of Kazakhstan cheese revealed by single molecule real time (SMRT) sequencing and its comparison with Belgian, Kalmykian and Italian artisanal cheeses[J]. BMC Microbiology,2017,17(1):13. doi: 10.1186/s12866-016-0911-4
[17] LI S, LI Y D, DU Z, et al. Impact of NSLAB on Kazakh cheese flavor[J]. Food Research International,2021,144:110315. doi: 10.1016/j.foodres.2021.110315
[18] JI D, OEY I, AGYEI D. Purification, characterization and thermal inactivation kinetics of β-galactosidase from Lactobacillus leichmannii 313[J]. LWT-Food Science and Technology,2019,116:108545. doi: 10.1016/j.lwt.2019.108545
[19] ARREOLA S L, INTANON M, SULJIC J, et al. Two β-galactosidases from the human isolate Bifidobacterium breve DSM 20213:Molecular cloning and expression, biochemical characterization and synthesis of galacto-oligosaccharides[J]. PLoS One,2014,9(8):e104056. doi: 10.1371/journal.pone.0104056
[20] THONGARAM T, HOEFLINGER J L, CHOW J, et al. Prebiotic galactooligosaccharide metabolism by probiotic lactobacilli and Bifidobacteria[J]. Journal of Agricultural and Food Chemistry,2017,65(20):4184−4192. doi: 10.1021/acs.jafc.7b00851
[21] IQBAL S, NGUYEN T H, NGUYEN T T, et al. beta-Galactosidase from Lactobacillus plantarum WCFS1:Biochemical characterization and formation of prebiotic galacto-oligosaccharides[J]. Carbohydrate Research,2010,345(10):1408−1416. doi: 10.1016/j.carres.2010.03.028
[22] NGUYEN T H, SPLECHTNA B, KRASTEVA S, et al. Characterization and molecular cloning of a heterodimeric β-galactosidases from the probiotic strain Lactobacillus acidophilus R22[J]. FEMS Microbiology Letters,2007,269(1):136−144. doi: 10.1111/j.1574-6968.2006.00614.x
[23] 韩明明, 褚宇欣, 关波, 等. 驼乳中产转糖基活性β-半乳糖苷酶乳酸菌的筛选鉴定及其酶学特性分析[J]. 食品科学,2021,42(24):74−83. [HAN M M, CHU Y X, GUAN B, et al. Screening, identification and enzymatic characterization of lactic acid bacteria producing β-galactosidase with transgalactosylation activity from camel milk[J]. Food Science,2021,42(24):74−83.] doi: 10.7506/spkx1002-6630-20201013-103 HAN M M, CHU Y X, GUAN B, et al. Screening, identification and enzymatic characterization of lactic acid bacteria producing β-galactosidase with transgalactosylation activity from camel milk[J]. Food Science, 2021, 42(24): 74−83. doi: 10.7506/spkx1002-6630-20201013-103
[24] SPLECHTNA B, NGUYEN T H, STEINBOCK M, et al. Production of prebiotic galacto-oligosaccharides from lactose using beta-galactosidases from Lactobacillus reuteri[J]. Journal of Agricultural and Food Chemistry,2006,54(14):4999−5006. doi: 10.1021/jf053127m
[25] ATTRI P, JODHA D, GANDHI D, et al. In vitro evaluation of Pediococcus acidilactici NCDC 252 for its probiotic attributes[J]. International Journal of Dairy Technology,2015,68(4):533−542. doi: 10.1111/1471-0307.12194
[26] BHOWMIK T, MARTH E H. β-Galactosidase of Pediococcus species:Induction, purification and partial characterization[J]. Applied Microbiology and Biotechnology,1990,33(3):317−323. doi: 10.1007/BF00164529
[27] CHANALIA P, GANDHI D, ATTRI P, et al. Purification and characterization of β-galactosidase from probiotic Pediococcus acidilactici and its use in milk lactose hydrolysis and galactooligosaccharide synthesis[J]. Bioorganic Chemistry,2018,77:176−189. doi: 10.1016/j.bioorg.2018.01.006
[28] 王丽军, 牟元珍, 关波, 等. 传统奶酪中产转糖基活性β-半乳糖苷酶乳酸菌的筛选鉴定及其合成低聚半乳糖条件[J]. 食品科学,2019,40(22):88−95. [WANG L J, MOU Y Z, GUAN B, et al. Screening and identification of lactic acid bacteria producing β-galactosidases with transglycosylation activity from traditional cheese, and optimization of conditions for galacto-oligosaccharides synthesis[J]. Food Science,2019,40(22):88−95.] WANG L J, MOU Y Z, GUAN B, et al. Screening and identification of lactic acid bacteria producing β-galactosidases with transglycosylation activity from traditional cheese, and optimization of conditions for galacto-oligosaccharides synthesis[J]. Food Science, 2019, 40(22): 88−95.
[29] R E布坎南, N E吉本斯. 伯杰氏细菌鉴定手册[M]. 8版. 中国科学院微生物研究所《伯杰氏细菌鉴定手册》翻译组, 译. 北京:科学出版社, 1984:1038−1160. [R E Buchanan, N E Gibbons. Berger's bacteria identification manual[M]. 8th edition. Translation Group of the Handbook of Identification of Burkholderia cepacia, Institute of Microbiology, Chinese Academy of Sciences, China, Translation. Beijing:Science Press, 1984:1038−1160.] R E Buchanan, N E Gibbons. Berger's bacteria identification manual[M]. 8th edition. Translation Group of the Handbook of Identification of Burkholderia cepacia, Institute of Microbiology, Chinese Academy of Sciences, China, Translation. Beijing: Science Press, 1984: 1038−1160.
[30] 高鑫. 米曲霉来源β-半乳糖苷酶的分子改造及其制备低聚半乳糖的研究[D]. 无锡:江南大学, 2019. [GAO X. Molecular modification of the beta-galactosidase from Aspergillus oryzae and the study of its ability to prepare galactoligosaccharide[D]. Wuxi:Jiangnan University, 2019.] GAO X. Molecular modification of the beta-galactosidase from Aspergillus oryzae and the study of its ability to prepare galactoligosaccharide[D]. Wuxi: Jiangnan University, 2019.
[31] HSU C A, YU R C, CHOU C C. Production of β-galactosidase by Bifidobacteria as influenced by various culture conditions[J]. International Journal of Food Microbiology,2005,104(2):197−206. doi: 10.1016/j.ijfoodmicro.2005.02.010
[32] PANESAR P S, KAUR R, SINGH R S, et al. Biocatalytic strategies in the production of galacto-oligosaccharides and its global status[J]. International Journal of Biological Macromolecules,2018,111:667−679. doi: 10.1016/j.ijbiomac.2018.01.062
[33] ZHANG X, YAO C, WANG T, et al. Production of high-purity galacto-oligosaccharides (GOS) by Lactobacillus-derived β-galactosidase[J]. European Food Research and Technology,2021,247(6):1501−1510. doi: 10.1007/s00217-021-03727-9
[34] MAISCHBERGER T, LEITNER E, NITISINPRASERT S, et al. β-Galactosidase from Lactobacillus pentosus:Purification, characterization and formation of galacto-oligosaccharides[J]. Biotechnology Journal,2010,5(8):838−847. doi: 10.1002/biot.201000126
[35] ARSOV A, IVANOV I, TSIGORIYNA L, et al. In vitro production of galactooligosaccharides by a novel β-Galactosidase of Lactobacillus bulgaricus[J]. International Journal of Molecular Sciences,2022,23(22):14308. doi: 10.3390/ijms232214308
[36] LIU G X, KONG J, LU W W, et al. β-Galactosidase with transgalactosylation activity from Lactobacillus fermentum K4[J]. Journal of Dairy Science,2011,94(12):5811−5820. doi: 10.3168/jds.2011-4479
[37] NGUYEN T T, NGUYEN H A, ARREOLA S L, et al. Homodimeric β-Galactosidase from Lactobacillus delbrueckii subsp. bulgaricus DSM 20081:Expression in Lactobacillus plantarum and Biochemical characterization[J]. Journal of Agricultural and Food Chemistry,2012,60(7):1713−1721. doi: 10.1021/jf203909e
[38] NGUYEN T H, SPLECHTNA B, YAMABHAI M, et al. Cloning and expression of the beta-galactosidase genes from Lactobacillus reuteri in Escherichia coli[J]. J Biotechnol,2007,129(4):581−591. doi: 10.1016/j.jbiotec.2007.01.034
[39] GOBINATH D, PRAPULLA S G. Permeabilized probiotic Lactobacillus plantarum as a source of β-galactosidase for the synthesis of prebiotic galactooligosaccharides[J]. Biotechnology Letters,2014,36(1):153−157. doi: 10.1007/s10529-013-1345-9
-
期刊类型引用(4)
1. 任卫威,张宇航,郑玉成,陈文贤,林宏政,徐梦婷,陈谢勇,陈百文,叶乃兴. 不同香型安溪黄金桂风味品质解析. 食品科学. 2025(05): 263-271 .  百度学术
百度学术
2. 李元朝,黎勤吉,郭玉琼,郝志龙,金珊. 枸杞茶感官审评方法的建立及其主要呈香物质的探索. 食品工业科技. 2025(04): 30-41 .  本站查看
本站查看
3. 周晶晶,张雯萍,李光发,谢荣富,何吉杭,郝韵智,李秋明,余欣茹,林宏政,孙云. 九龙大白茶白茶风味品质的分析. 食品科学. 2025(06): 201-210 .  百度学术
百度学术
4. 刘学艳,杨文光,徐婷,罗正飞,王绍梅,龚正礼. 并堆工艺对云南白茶品质影响的研究. 中国茶叶. 2025(03): 25-33 .  百度学术
百度学术
其他类型引用(3)





 下载:
下载:


 下载:
下载:
