Adulteration Detection and Origin Identification of Yak Milk Powder Based on Near-infrared Spectroscopy Technology
-
摘要: 牦牛奶粉的掺假检测和产地识别有助于保障食品安全、维护消费者权益,是促进乳制品市场健康发展的重要举措。传统的DNA检测方法和稳定同位素分析技术的检测周期长,难以满足快速、低成本现场分析的需求。针对以上问题,本研究建立了一种基于近红外光谱技术(Near-infrared Spectroscopy,NIRS)快速辨别牦牛奶粉掺假及产地的方法。收集了来自四川、甘肃、云南及青海的9个品牌的牦牛奶粉。在制备掺假样品之前,采用聚合酶链式反应(Polymerase Chain Reaction,PCR)技术和DNA凝胶电泳验证所收集的牦牛奶粉中是否掺杂了牛奶粉。完成验证后,进行掺假样品的制备以及近红外光谱数据的采集。采用K最邻近法(K-Nearest Neighbors,KNN)建立分类模型,偏最小二乘回归(Partial Least Squares Regression,PLSR)建立定量预测模型。通过优化光谱预处理方法和变量筛选方法进一步提升定量预测模型的预测能力。结果表明,KNN对牦牛奶粉掺假检测(纯牛奶粉、纯牦牛奶粉、掺杂着牛奶粉的牦牛奶粉)及产地识别(四川、甘肃、云南、青海)实现了100%的正确分类。掺假定量预测模型的校正集相关系数(Rc)为0.9975,预测集相关系数(Rp)为0.9913,预测集均方根误差(Root Mean Square Error of Prediction,RMSEP)为1.9823%,性能偏差比(Ratio of Performance to Deviation,RPD)为7.2522。本方法可快速、准确地预测牦牛奶粉中牛奶粉的掺杂以及牦牛奶粉产地的辨别,为牦牛奶粉的质量控制提供技术支持。Abstract: Adulteration detection and origin identification of yak milk powder were essential to ensure food safety and safeguard consumer rights interests, thereby promoting the healthy development of the dairy product market. Traditional DNA detection methods and isotope analysis showed long detection time, which were inapplicable to rapid, low-cost on-site analysis. To address these issues, a rapid adulteration detection and identification of the origin of yak milk powder based on near-infrared Spectroscopy (NIRS) technology was established in this study. Yak milk powder samples from nine brands from Sichuan, Gansu, Yunnan, and Qinghai were collected. Before preparing adulterated samples, polymerase chain reaction (PCR) technology and DNA gel electrophoresis were used to verify whether the collected yak milk powder were adulterated with cow milk powder. Then adulterated samples were prepared and NIRS data were collected. The K-nearest neighbors (KNN) method was employed to establish a classification model. Partial least squares regression (PLSR) was used to establish a quantitative prediction model. The predictive ability of quantitative prediction model was improved by optimizing spectral preprocessing methods and variable selection methods. Results showed that KNN achieved 100% correct classification for adulteration detection (pure cow milk powder, pure yak milk powder, yak milk powder adulterated with cow milk powder) and origin identification (Sichuan, Gansu, Yunnan, Qinghai). The calibration set correlation coefficient (Rc), the prediction set correlation coefficient (Rp), the root mean square error of prediction (RMSEP), and the ratio of performance to deviation (RPD) of the adulteration quantitative prediction model were 0.9975, 0.9913, 1.9823%, and 7.2522, respectively. This method enables rapid and accurate prediction of cow milk powder adulteration in yak milk powder and the identification of the origin of yak milk powder, providing technical support for the quality control of yak milk powder.
-
在我国,牦牛主要生活于青藏高原地区,其生存能力很强,能够适应极端的气候条件,是当地重要畜牧资源[1]。与普通牛奶相比,牦牛奶的蛋白质、脂肪等营养成分的含量更高,因此,牦牛奶也被称为天然的浓缩奶[2−4]。由于牦牛的产奶期短、产奶量少,且产地偏远,通常牦牛奶被加工成牦牛奶粉使其具有更加稳定的形态,以方便运输和销售。牦牛奶粉营养价值高、价格昂贵的特点使其容易成为不法商贩的掺假对象。在牦牛奶粉中掺杂普通牛奶粉是常见的掺假方法。传统的DNA检测方法可以准确辨别牦牛奶粉的掺假问题,但该方法费时费力且需要消耗化学试剂,难以满足现场分析的需求。此外,不同产地的牦牛奶粉营养成分组成存在差异,影响其市场定位和定价。使用稳定同位素分析技术可以准确识别牦牛奶粉的产地,但该方法需要对样品进行较为复杂的前处理,且检测成本较高。建立快速、准确的牦牛奶粉产地识别方法对维护消费者权益和市场秩序具有重要意义。
近红外光谱作为一种快速检测技术,可通过物质的近红外吸收光谱获取样品的化学信息,在食品、医药、农业以及化工等领域应用广泛[5−7]。目前已有相关报道将近红外光谱技术用于乳制品掺假检测及产地识别,如Elainy等[8]通过偏最小二乘判别法(Partial Least Squares Discriminant Analysis,PLS-DA)以及PLSR结合近红外光谱技术准确辨别了山羊奶中牛奶的掺假;Mabood等[9]通过PLS-DA和PLSR结合近红外光谱技术实现了对骆驼奶中掺杂牛奶的快速检测;Alessandra等[10]将近红外光谱技术与最小二乘支持向量机(Least Squares Support Vector Machine,LS-SVM)技术相结合实现了对奶粉中的掺杂物(淀粉、乳清以及蔗糖)的快速检测;Zhang等[11]将模糊不相关判别法(Fuzzy uncorrelated Discriminant Transformation,FUDT)与近红外光谱技术结合,准确识别了牛奶的产地。以上研究证明了近红外光谱技术在乳制品掺假检测和产地识别方面的可行性。
鉴于此,本研究采用便携式近红外光谱仪,结合KNN建立了牦牛奶粉掺假及产地的分类模型,并进一步通过PLSR实现了对牦牛奶粉中牛奶粉含量的快速定量检测,通过优化光谱预处理方法以及变量筛选方法,进一步提升定量预测模型的预测能力,为牦牛奶粉掺假检测及产地识别提供一种快速、绿色、无损的检测方法。
1. 材料与方法
1.1 材料与仪器
牦牛奶粉 产自四川、甘肃、云南、青海(每个产地50个,共计200个),9种主流品牌,其中四川2种,甘肃3种,云南2种,青海2种;纯牛奶粉 产自内蒙古、新疆(200个),4种主流品牌。随机在1%~10%,11%~20%,21%~30%,31%~40%,41%~50%,51%~60%,61%~70%,71%~80%,81%~90%,91%~100%这10个掺假比例范围中,生成牛奶粉的掺假比例(7%、15%、23%、36%、48%、54%、62%、75%、83%、92%)制备掺假样品(每个范围掺假40个,共计400个);Ezup柱式深加工产品基因组DNA抽提试剂盒、4S Gelred核酸染料、2X SanTaq PCR Mix 预混液、琼脂糖、50X TAE 缓冲液、合成引物(BO/YA-FW、BO/YA-RV和BO-FW)、无水乙醇 分析纯,以上材料均来自生物工程(生工)上海股份有限公司。
EXPEC1330便携式近红外光谱分析仪 聚光科技(杭州)股份有限公司;HH-6水浴锅 常州润华电器有限公司;H1850离心机 湖南湘仪实验室器材开发股份有限公司;SimpliAmp热循环仪 美国赛默飞世尔科技公司;BG-Power600k凝胶电泳仪 上海贝晶生物技术有限公司;PTY-224电子天平 福建华志电子科技有限公司。
1.2 实验方法
1.2.1 聚合酶链式反应和DNA凝胶电泳
本研究通过PCR技术结合DNA凝胶电泳实验,验证所收集的牦牛奶粉样品的真实性[12]。根据文献[12]报道,引物BO/YA-FW(5′-GAAAAGGTCCAAATGTCGTAGGT-3′)和BO/YA-RV(5′-TCCGATTAGTGCGTATTTTGAGT-3′)可同时在奶牛和牦牛的ND1基因上扩增出293 bp的片段,而BO-FW(5′-CTCAATATTTATCCTAGCACCTATCA-3′)可在奶牛的ND1基因上扩增出190 bp的片段,在牦牛的ND1基因上无法扩增出190 bp的片段。本研究使用上述方法对9种牦牛奶粉以及4种牛奶粉进行PCR扩增及DNA凝胶电泳,判断收集的原始样本(牦牛奶粉)中是否掺杂了牛奶粉。
称取200 mg奶粉并使用Ezup试剂盒从9种牦牛奶粉样品以及4种牛奶粉提取DNA[13],提取后的DNA置于−20 ℃保存。在25 µL的反应体系(含有10 µL 2X SanTaq PCR Master Mix,10 µL蒸馏水,2 µL目标DNA模板,1 µL BO/ YA-FW,1 µL BO/YA-RV,1 µL BO-FW)中进行PCR反应。扩增程序为:初始变性在90 ℃持续12 min。循环30次:在90 ℃持续30 s,60 ℃持续30 s,72 ℃持续30 s。最终延伸在72 ℃持续5 min。扩增产物在4S Gelred的染色2%琼脂糖凝胶上以100 V的电压电泳40 min。
1.2.2 近红外光谱采集
本研究使用便携式近红外光谱分析仪对样品进行近红外光谱数据的采集。将每个样品放入100 mm×100 mm的扫描皿中并平铺均匀,通过仪器的集成软件收集近红外光谱数据,扫描次数32次,分辨率8 cm−1,波长1000~1800 nm,光谱采集均在室温下进行(24±1 ℃),每个样品扫描3次取平均光谱,用于构建化学计量学模型。光谱预处理、变量筛选以及模型的建立在软件ChemDataSolution 4.2.1中进行。
1.3 数据处理
1.3.1 近红外光谱预处理
由于多种因素的影响,近红外光谱数据可能会受到干扰,例如近红外光谱仪器产生的噪音;样本质地不均匀可能导致的近红外光谱基线漂移;光线在样本中散射和吸收可能导致的光谱形状和强度变化。对近红外光谱预处理,有助于提高数据质量、降低噪音、减弱干扰因素[14]。在进行变量筛选前,本研究使用多元散射校正(Multiple Scattering Correction,MSC)、标准正态变化(Standard Normal Variate,SNV)、去趋势化(Detrending)、SG平滑(Savitzky-Golay Smoothing,SG)、面积归一化(Area Normalized)、一阶求导(First Derivative,1D)、移动平均(Moving Average,MA)对近红外光谱进行预处理。
1.3.2 变量筛选
变量筛选可选择出与因变量相关的自变量,降低数据维度,消除冗余变量,提高模型的解释能力和泛化能力。本研究使用费舍尔判别法(Fisher's Ratio Method,FR)、变量投影法(Variable Importance in Projection,VIP)、蒙特卡洛非信息变量剔除法(Monte Carlo Uninformative Variable Elimination,MC-UVE)、竞争适应性重加权采样法(Competitive Adaptive Reweighted Sampling,CARS)和随机蛙跳法(Random Jumping Frog Algorithm,RF)这5种方法对样本的光谱数据进行降维处理。
1.3.3 建模方法
本研究对比了主成分分析结合马氏距离值(Principal Component Analysis combined with Mahalanobis Distance,PCA-MD)和KNN建立的分类模型的预测效果。PCA-MD可在多维数据集中识别和量化变量之间的模式和关系,通过对每个已知类别的样本构造有监督的PCA模型,然后计算未知新样本到各类的马氏距离,并以此确定样本类别[15−17]。KNN是一种常见的监督学习算法,主要用于分类和回归。KNN基于邻近性原理,即假设相似的样本在特征空间中的位置相近。如果一个样本的邻居中大多数属于某个类别,那么该样本很可能也属于这个类别。对于分类任务,KNN采用多数表决的原则,待分类样本的类别将由其K个最近邻中属于某一类别的样本决定,取占多数的类别[18−20]。KNN对于小型数据集和相对低维度的特征空间具有较好的性能。
为进一步确认掺假牦牛奶粉中牛奶粉的具体含量,采用PLSR建立定量预测模型,并使用交叉验证评估模型的泛化能力。PLSR是一种多元统计分析的方法,主要用于处理多重共线性和高维数据,适用于具有高度相关性和多共线性的自变量的回归问题。PLSR可通过分解解释变量矩阵和响应变量矩阵,产生一组新的主成分,这些主成分同时捕获了解释变量和响应变量之间的最大协方差。通过逐步建立主成分,并最大化解释变量和响应变量之间的协方差,使得在保留关键信息的同时降低维度,从而通过这些新的主成分建立目标的回归模型[21−23]。
1.3.4 评价参数
通过校正集相关系数(Correlation Coefficient of Calibration,Rc),预测集相关系数(Correlation Coefficient of Prediction,Rp),校正集均方根误差(Root Mean Square Error of Calibration,RMSEC),交叉验证均方根误差(Root Mean Square Error Cross Validation,RMSECV),预测集均方根误差(Root Mean Square Error of Prediction,RMSEP),以及性能偏差比(Ratio of Performance to Deviation,RPD)等模型参数来判断不同预处理方法下模型的预测性能。其中,相关系数反映的是模型对数据的拟合程度,均方根误差反映的是模型的预测精度,交叉验证均方根误差与性能偏差比反映的是模型的泛化能力,R越接近于1,RMSEC、RMSECV、RMSEP越接近于0,RPD越大则模型预测效果越好。
2. 结果与分析
2.1 PCR扩增与DNA凝胶电泳
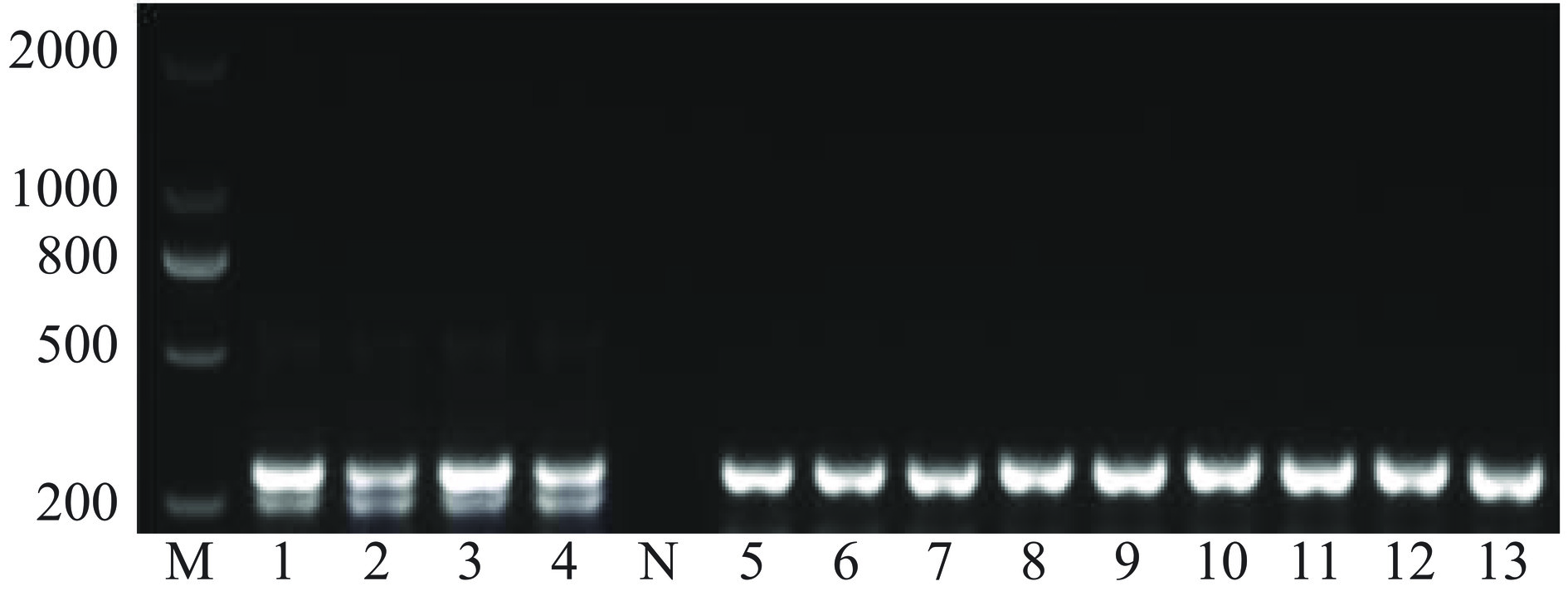
DNA凝胶电泳结果如图1所示,BO/YA-FW和BO/YA-RV可以同时在奶牛和牦牛的线粒体ND1基因上扩增出293 bp的片段;BO-FW引物可在奶牛的ND1基因上扩增出190 bp的片段,但在牦牛的ND1基因上无法扩增出相同长度的片段。因此,1到4号样本经PCR扩增后显示出了293 bp和190 bp两个片段;空白对照N未显示任何扩增片段,证明实验中没有污染或杂质;5到13号样本经PCR扩增后仅显示了293 bp的片段,而未观察到190 bp的片段。以上结果表明这9种牦牛奶粉中均不含有牛奶粉。
2.2 光谱分析
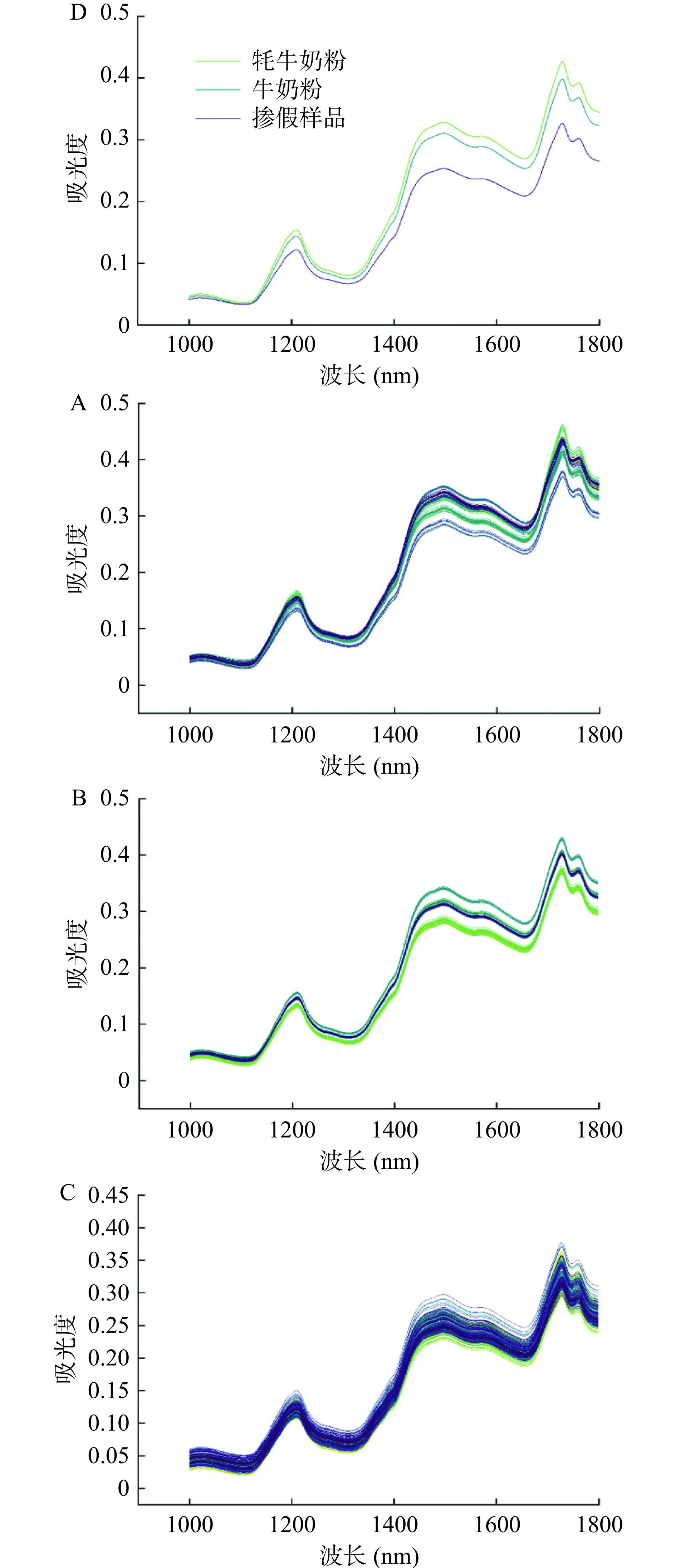
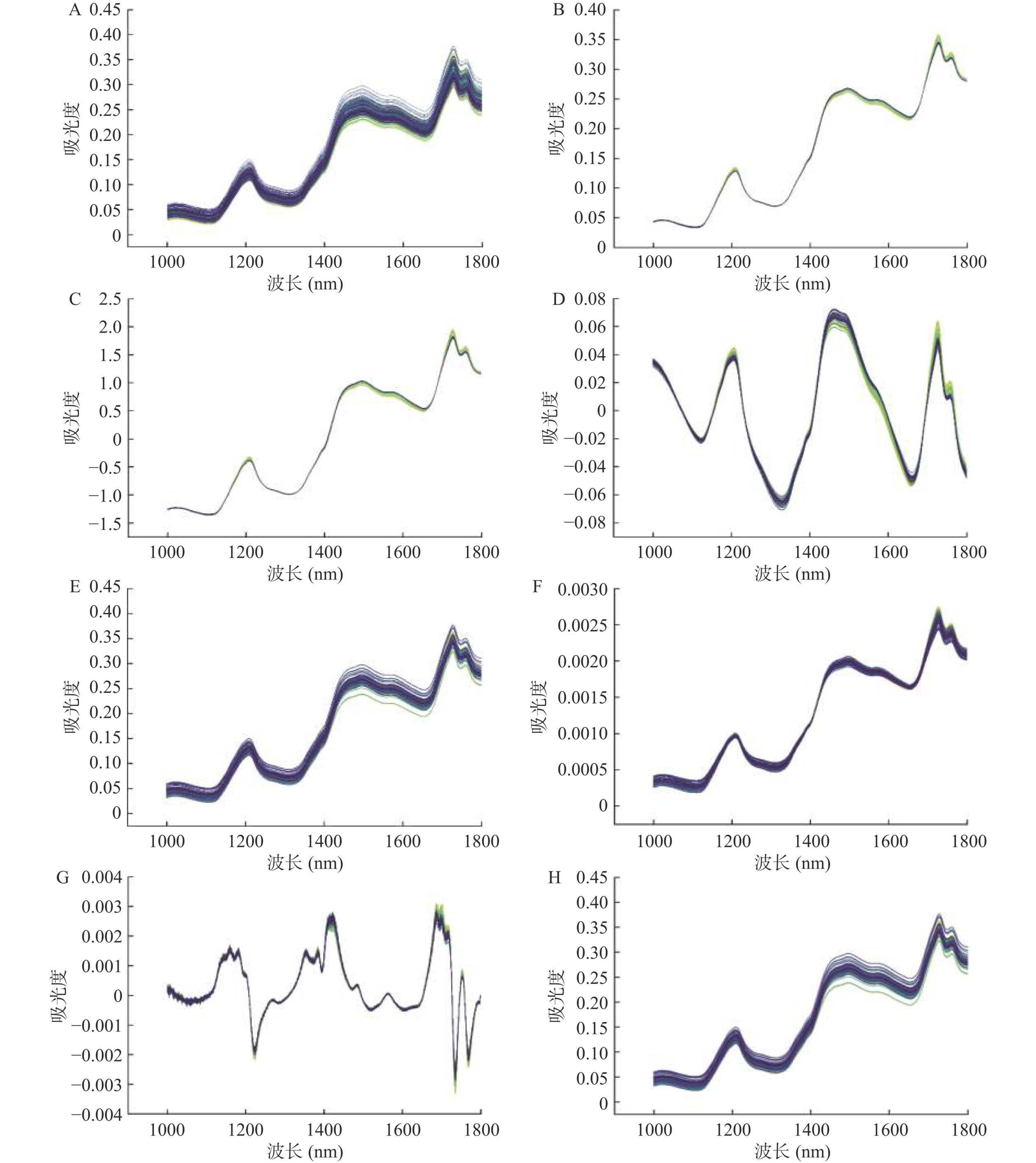
牛奶粉样品,牦牛奶粉样品以及掺假样品的原始近红外光谱如图2所示。牦牛奶粉、牛奶粉以及掺假样品的近红外光谱的整体趋势较为一致,证明样品之间的组成成分较为一致。而在同一个波长中,每个样品之间的吸光度不完全重叠,证明每个样品的组分含量可能存在差异。
2.3 牦牛奶粉掺假检测和产地识别的PCA-MD分类模型
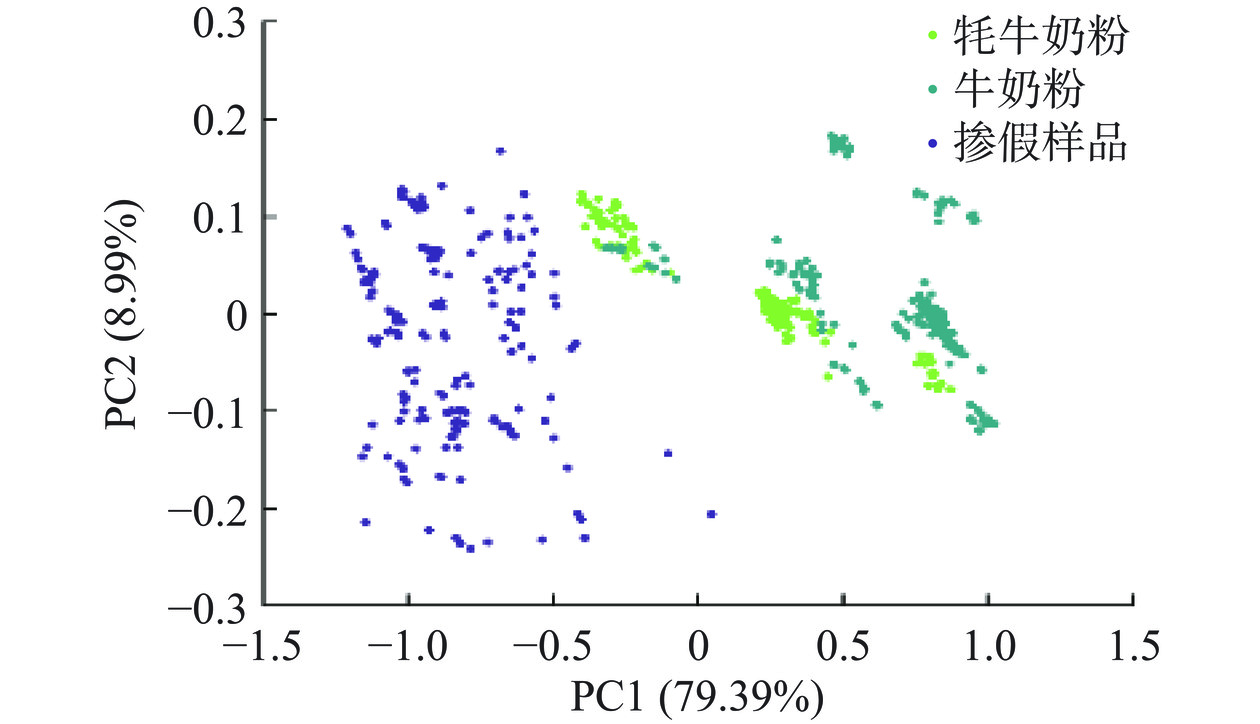
本研究首先考察了PCA-MD在牦牛奶粉掺假检测和产地识别中的预测效果。对于牦牛奶粉的掺假检测,本研究对200个牦牛奶粉样品、200个牛奶粉样品,以及200个掺假样品进行主成分分析,其得分图如图3所示。结果显示,第一主成分的解释方差为79.39%,第二主成分的解释方差为8.99%,掺假样品与其他两个样品在第一主成分上具有一定的分离趋势。但牦牛奶粉样品与牛奶粉样品之间存在部分重合,这可能表明在某些方面,牛奶粉和牦牛奶粉在主成分方向上具有一些相似性,导致它们在这个方向上不能完全分离。随后,采用随机划分的方法将70%的牦牛奶粉样品(420个)划分为校正集,30%的牦牛奶粉样品(180个)划分为预测集,并使用1D处理后的近红外光谱数据结合PCA-MD(1D-PCA-MD)建立牦牛奶粉掺假的分类模型。校正集及预测集的分类结果如表1、表2所示,校正集分类的准确率为92.62%,预测集分类的准确率为91.67%。校正集及预测集均把部分牛奶粉错误地识别为牦牛奶粉,这与得分图中样本的分布情况相对应。以上结果表明,1D-PCA-MD对牦牛奶粉的掺假不能实现很好的分类和识别。
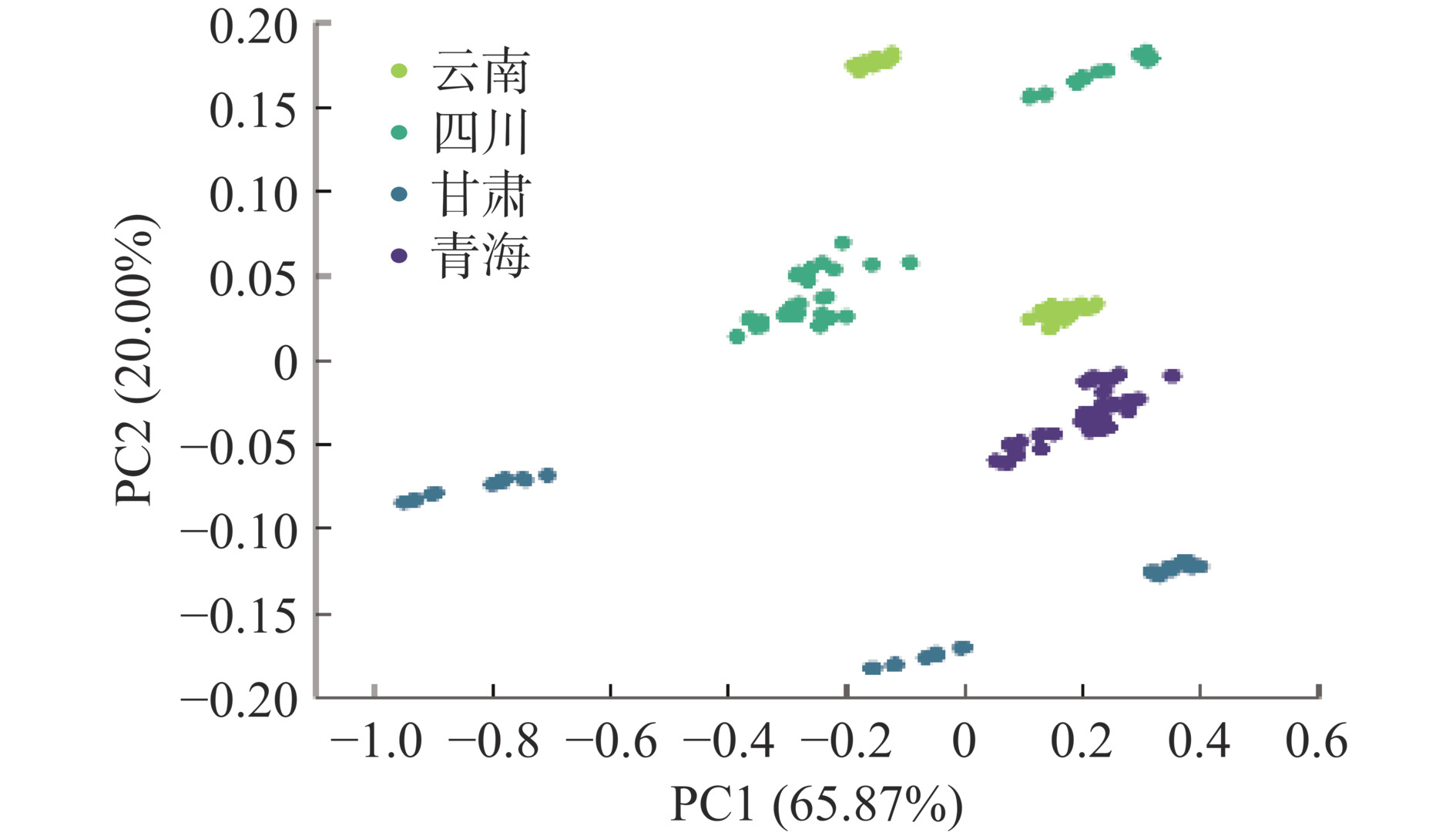
表 1 牦牛奶粉掺假校正集分类结果(1D-PCA-MD)Table 1. Classification of correction set for yak milk powder adulteration (1D-PCA-MD)样品 牦牛奶粉 掺假牦牛奶粉 牛奶粉 牦牛奶粉(预测) 134 0 31 掺假牦牛奶粉(预测) 0 156 0 牛奶粉(预测) 0 0 99 表 2 牦牛奶粉掺假预测集分类结果(1D-PCA-MD)Table 2. Classification of prediction sets for yak milk powder production adulteration (1D-PCA-MD)样品 牦牛奶粉 掺假牦牛奶粉 牛奶粉 牦牛奶粉(预测) 66 0 15 掺假牦牛奶粉(预测) 0 44 0 牛奶粉(预测) 0 0 55 对于牦牛奶粉的产地识别,本研究对四个产地的牦牛奶粉进行主成分分析,其得分图如图4所示。结果显示,第一主成分的解释方差为65.87%,第二主成分的解释方差为20.00%,可以看出各产地在得分图中均有较为明显的区分趋势。四个不同产地的牦牛奶粉样本在得分图上形成了各自独立的簇状结构,由于样本在主成分方向上的分布明显且没有重叠,说明主成分分析可有效区分不同产地的牦牛奶粉样本。随后,采用随机划分的方法将70%的牦牛奶粉样品(140个)划分为校正集,30%的牦牛奶粉样品(60个)划分为预测集,并使用1D-PCA-MD建立牦牛奶粉产地的分类模型。校正集及预测集的分类结果如表3、表4所示,分类准确率均达到了100%。以上结果表明该模型能够实现牦牛奶粉的产地的准确预测。
表 3 牦牛奶粉产地校正集分类结果(1D-PCA-MD)Table 3. Classification of correction sets for yak milk powder origin (1D-PCA-MD)产地 四川 甘肃 云南 青海 四川(预测) 38 0 0 0 甘肃(预测) 0 28 0 0 云南(预测) 0 0 42 0 青海(预测) 0 0 0 32 表 4 牦牛奶粉产地预测集分类结果(1D-PCA-MD)Table 4. Classification of prediction sets for yak milk powder origin (1D-PCA-MD)产地 四川 甘肃 云南 青海 四川(预测) 12 0 0 0 甘肃(预测) 0 22 0 0 云南(预测) 0 0 8 0 青海(预测) 0 0 0 28 2.4 牦牛奶粉掺假检测和产地识别的KNN分类模型
虽然1D-PCA-MD在牦牛奶粉产地识别中具有较好的预测能力,但1D-PCA-MD在牦牛奶粉掺假检测中预测结果较差,不能准确辨别牦牛奶粉的掺假。为了提高牦牛奶粉掺假检测和产地识别分类模型的预测能力和准确度,本研究进一步采用KNN建立牦牛奶粉掺假检测和产地识别的分类模型。在数据集规模较小的情况下,KNN通常能够给出比较准确的结果,且KNN不对数据的分布做出任何假设,可以很好地处理非线性关系和复杂的数据结构。
对于牦牛奶粉的掺假检测,本研究采用随机划分的方法将70%的牦牛奶粉样品(420个)划分为校正集,30%的牦牛奶粉样品(180个)划分为预测集,并使用1D-KNN建立牦牛奶粉掺假的分类模型。校正集及预测集的分类结果如表5、表6所示,分类准确率均达到了100%。以上结果表明该模型对于牦牛奶粉掺假具有优异的预测能力。
表 5 牦牛奶粉掺假校正集分类结果(1D-KNN)Table 5. Classification of correction set for yak milk powder adulteration (1D-KNN)样品 牦牛奶粉 掺假牦牛奶粉 牛奶粉 牦牛奶粉(预测) 134 0 0 掺假牦牛奶粉(预测) 0 156 0 牛奶粉(预测) 0 0 130 表 6 牦牛奶粉掺假预测集分类结果(1D-KNN)Table 6. Classification of prediction sets for yak milk powder production adulteration (1D-KNN)样品 牦牛奶粉 掺假牦牛奶粉 牛奶粉 牦牛奶粉(预测) 66 0 0 掺假牦牛奶粉(预测) 0 44 0 牛奶粉(预测) 0 0 70 对于牦牛奶粉的产地识别,本研究采用随机划分的方法将70%的牦牛奶粉样品(140个)划分为校正集,30%的牦牛奶粉样品(60个)划分为预测集,并使用1D-KNN建立牦牛奶粉产地的分类模型。校正集及预测集的分类结果如表7、表8所示,分类准确率均达到了100%。以上结果表明该模型对于牦牛奶粉的产地具有优异的预测能力。
表 7 牦牛奶粉产地校正集分类结果(1D-KNN)Table 7. Classification of correction sets for yak milk powder origin (1D-KNN)产地 四川 甘肃 云南 青海 四川(预测) 38 0 0 0 甘肃(预测) 0 28 0 0 云南(预测) 0 0 42 0 青海(预测) 0 0 0 32 表 8 牦牛奶粉产地预测集分类结果(1D-KNN)Table 8. Classification of prediction sets for yak milk powder origin (1D-KNN)产地 四川 甘肃 云南 青海 四川(预测) 12 0 0 0 甘肃(预测) 0 22 0 0 云南(预测) 0 0 8 0 青海(预测) 0 0 0 28 2.5 牦牛奶粉中掺杂牛奶粉的PLSR定量预测模型
为进一步确认牦牛奶粉中掺杂牛奶粉的具体含量,本研究建立了PLSR模型对牦牛奶粉中掺杂的牛奶粉进行定量预测。如图5所示,首先对400个掺假样品的原始近红外光谱(A)进行预处理, MSC和Detrending可减小光谱数据因散射而导致的多重共线性与基线漂移等影响;SNV和Area Normalized可减小样品之间的强度差异;SG平滑和MA可减小噪音影响,提高数据信噪比;1D可放大隐藏在原始光谱中的信息。随后,采用随机划分的方法将70%的掺假牦牛奶粉样品(280个)划分为校正集,30%的掺假牦牛奶粉样品(120个)划分为预测集,并通过Rc、Rp、RMSEC、RMSECV、 RMSEP以及RPD来确定最优的近红外光谱预处理方法。光谱预处理后PLSR的建模结果如表9所示,通过比对参数,1D处理后的近红外光谱数据建立的PLSR模型的预测能力优于其他的模型,其Rc为0.9953,Rp为0.9756,RMSEC为2.6632,RMSECV为6.0575,RMSEP为6.0406,RPD为4.5345。
表 9 不同光谱预处理方法下牦牛奶粉掺假定量预测模型的评价参数Table 9. Assessment parameters of quantitative prediction model for adulteration of yak milk powder after different spectral preprocessing预处理方法 Rc Rp RMSEC RMSECV RMSEP RPD Raw 0.9598 0.9545 7.7051 8.7069 8.1852 3.1548 MSC 0.9701 0.9658 6.6627 7.7512 7.1492 3.5437 Detrending 0.9673 0.9656 6.9635 8.2089 7.1064 3.3461 SNV 0.9701 0.9658 6.6638 7.7523 7.1498 3.5432 SG 0.9682 0.9679 6.8748 7.7957 7.1787 3.5234 Area Normalized 0.9703 0.9684 6.6481 7.6641 6.8875 3.5839 1D 0.9953 0.9756 2.6632 6.0575 6.0406 4.5345 MA 0.9580 0.9555 7.8730 8.8588 8.1074 3.1006 直接使用全近红外光谱数据进行建模可能会因为冗余信息导致模型的预测能力下降,因此,在近红外光谱建立模型时,常通过变量筛选来提取数据的关键信息并剔除冗余信息。本研究使用5种变量筛选方法(FR、VIP、MC-UVE、CARS、RF)对1D处理后的近红外光谱数据进行降维处理,进一步提高模型的预测能力。FR主要用于选择能够区分不同类别的特征。该方法的目标是使类间方差与类内方差的比值最大,使得选择的特征在不同类别之间具有显著差异[24−26]。VIP主要用于选择对模型性能有重要贡献的波长。该方法主要通过分析模型的主成分,评估各个变量对于解释与预测目标变量的重要性[27−29]。MC-UVE主要用于去除对模型性能贡献较小的特征,避免过度拟合,并提高模型的解释能力[30−32]。CARS是一种基于逐步回归和随机抽样的变量筛选方法,通过竞争机制逐步选择最相关和影响力最大的变量子集[33−35]。RF是一种基于随机搜索的变量筛选方法,通过模拟蛙跳,随机选择一些变量进行建模,并通过评估模型的性能来判断变量的重要性[36−38]。各变量筛选处理后的PLSR的建模结果如表10所示,通过比对参数,可以看出使用FR、MC-UVE、RF以及CARS处理后的模型预测能力均优于使用全光谱建立的模型,但通过VIP处理后的模型预测能力弱于全光谱建立的模型。该现象可能是因为VIP法可能会忽略变量之间的复杂关系,导致丢失重要信息。某些变量虽然在单独分析时不显著,但与其他变量相结合时却对模型性能有重要影响。即使某个变量本身的VIP值很高,但如果它与其他变量高度相关,那么选择它可能会引入冗余信息,影响模型的性能和解释能力。基于以上结果,本研究最终选择的变量筛选方法为CARS,其筛选出的变量有233个,在此基础上所建立的PLSR模型的预测能力优于基于其他变量筛选方法所建立的模型,该模型的Rc为0.9975,Rp为0.9913,RMSEC为1.9823,RMSECV为3.8781,RMSEP为3.4603,RPD为7.2522。
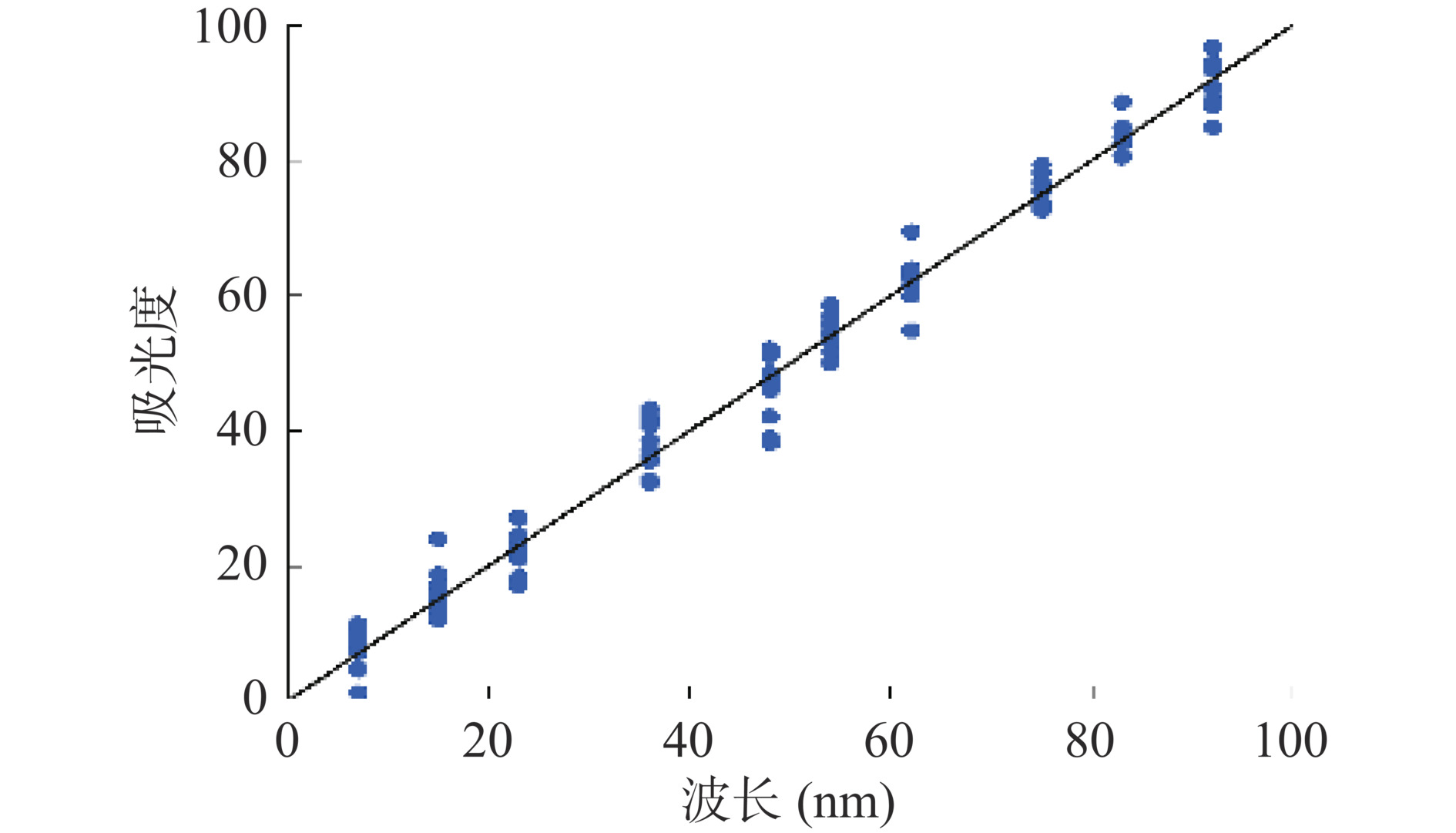
表 10 不同变量筛选方法下牦牛奶粉掺假定量预测模型的评价参数Table 10. Assessment parameters of quantitative prediction model for adulteration of yak milk powder after different variable screening变量筛选方法 变量数 Rc Rp RMSEC RMSECV RMSEP RPD 全光谱 800 0.9953 0.9756 2.6632 6.0575 6.0406 4.5345 FR 794 0.9958 0.9829 2.5250 7.4712 4.9493 3.7101 VIP 238 0.9827 0.9669 5.1601 8.3806 6.9541 3.3260 MC-UVE 651 0.9961 0.9832 2.4365 5.9992 5.0690 4.5878 RF 284 0.9968 0.9913 2.2163 4.3084 3.5760 6.4736 CARS 233 0.9975 0.9913 1.9823 3.8781 3.4603 7.2522 本研究得到的最优牦牛奶粉掺假定量预测模型为1D-CARS-PLSR,如图6所示,各个数据点均分布在y=x这条直线附近。以上结果表明该模型能够较为准确的预测出牦牛奶粉中掺杂的牛奶粉含量,为牦牛奶粉的掺假检测提供了一种快速有效的定量检测方法。
3. 结论
本研究采用1D处理的近红外光谱数据结合KNN建立了分类模型,对牦牛奶粉掺假检测与产地识别的准确率可达到100%。为进一步实现牦牛奶粉中掺杂牛奶粉的具体含量,本研究将近红外光谱数据与牦牛奶粉中的牛奶粉掺假比例进行关联,通过对比性能参数选择出最优的预处理方法和变量筛选方法并在此基础上建立了牦牛奶粉掺假定量预测模型,最佳定量预测模型为1D-CARS-PLSR,其Rc为0.9975,Rp为0.9913,RMSEC为1.9823,RMSECV为3.8781,RMSEP为3.4603,RPD为7.2522。结果表明,本研究的分类模型和定量预测模型准确可靠,与传统的DNA检测和稳定同位素分析技术相比,该方法耗时短、操作简单、不需要对样品进行复杂的前处理,且不需要使用任何化学试剂,为牦牛奶粉掺假检测和产地识别提供了可靠的现场快速检测方法,为乳制品市场监管提供了有力的技术支持。在后续的工作中可增加样本量,并对光谱预处理方法、变量筛选方法以及建模方法进行更加深入的研究,进一步提高模型的预测性能和泛化能力。
-
表 1 牦牛奶粉掺假校正集分类结果(1D-PCA-MD)
Table 1 Classification of correction set for yak milk powder adulteration (1D-PCA-MD)
样品 牦牛奶粉 掺假牦牛奶粉 牛奶粉 牦牛奶粉(预测) 134 0 31 掺假牦牛奶粉(预测) 0 156 0 牛奶粉(预测) 0 0 99 表 2 牦牛奶粉掺假预测集分类结果(1D-PCA-MD)
Table 2 Classification of prediction sets for yak milk powder production adulteration (1D-PCA-MD)
样品 牦牛奶粉 掺假牦牛奶粉 牛奶粉 牦牛奶粉(预测) 66 0 15 掺假牦牛奶粉(预测) 0 44 0 牛奶粉(预测) 0 0 55 表 3 牦牛奶粉产地校正集分类结果(1D-PCA-MD)
Table 3 Classification of correction sets for yak milk powder origin (1D-PCA-MD)
产地 四川 甘肃 云南 青海 四川(预测) 38 0 0 0 甘肃(预测) 0 28 0 0 云南(预测) 0 0 42 0 青海(预测) 0 0 0 32 表 4 牦牛奶粉产地预测集分类结果(1D-PCA-MD)
Table 4 Classification of prediction sets for yak milk powder origin (1D-PCA-MD)
产地 四川 甘肃 云南 青海 四川(预测) 12 0 0 0 甘肃(预测) 0 22 0 0 云南(预测) 0 0 8 0 青海(预测) 0 0 0 28 表 5 牦牛奶粉掺假校正集分类结果(1D-KNN)
Table 5 Classification of correction set for yak milk powder adulteration (1D-KNN)
样品 牦牛奶粉 掺假牦牛奶粉 牛奶粉 牦牛奶粉(预测) 134 0 0 掺假牦牛奶粉(预测) 0 156 0 牛奶粉(预测) 0 0 130 表 6 牦牛奶粉掺假预测集分类结果(1D-KNN)
Table 6 Classification of prediction sets for yak milk powder production adulteration (1D-KNN)
样品 牦牛奶粉 掺假牦牛奶粉 牛奶粉 牦牛奶粉(预测) 66 0 0 掺假牦牛奶粉(预测) 0 44 0 牛奶粉(预测) 0 0 70 表 7 牦牛奶粉产地校正集分类结果(1D-KNN)
Table 7 Classification of correction sets for yak milk powder origin (1D-KNN)
产地 四川 甘肃 云南 青海 四川(预测) 38 0 0 0 甘肃(预测) 0 28 0 0 云南(预测) 0 0 42 0 青海(预测) 0 0 0 32 表 8 牦牛奶粉产地预测集分类结果(1D-KNN)
Table 8 Classification of prediction sets for yak milk powder origin (1D-KNN)
产地 四川 甘肃 云南 青海 四川(预测) 12 0 0 0 甘肃(预测) 0 22 0 0 云南(预测) 0 0 8 0 青海(预测) 0 0 0 28 表 9 不同光谱预处理方法下牦牛奶粉掺假定量预测模型的评价参数
Table 9 Assessment parameters of quantitative prediction model for adulteration of yak milk powder after different spectral preprocessing
预处理方法 Rc Rp RMSEC RMSECV RMSEP RPD Raw 0.9598 0.9545 7.7051 8.7069 8.1852 3.1548 MSC 0.9701 0.9658 6.6627 7.7512 7.1492 3.5437 Detrending 0.9673 0.9656 6.9635 8.2089 7.1064 3.3461 SNV 0.9701 0.9658 6.6638 7.7523 7.1498 3.5432 SG 0.9682 0.9679 6.8748 7.7957 7.1787 3.5234 Area Normalized 0.9703 0.9684 6.6481 7.6641 6.8875 3.5839 1D 0.9953 0.9756 2.6632 6.0575 6.0406 4.5345 MA 0.9580 0.9555 7.8730 8.8588 8.1074 3.1006 表 10 不同变量筛选方法下牦牛奶粉掺假定量预测模型的评价参数
Table 10 Assessment parameters of quantitative prediction model for adulteration of yak milk powder after different variable screening
变量筛选方法 变量数 Rc Rp RMSEC RMSECV RMSEP RPD 全光谱 800 0.9953 0.9756 2.6632 6.0575 6.0406 4.5345 FR 794 0.9958 0.9829 2.5250 7.4712 4.9493 3.7101 VIP 238 0.9827 0.9669 5.1601 8.3806 6.9541 3.3260 MC-UVE 651 0.9961 0.9832 2.4365 5.9992 5.0690 4.5878 RF 284 0.9968 0.9913 2.2163 4.3084 3.5760 6.4736 CARS 233 0.9975 0.9913 1.9823 3.8781 3.4603 7.2522 -
[1] JING X P, DING L M, ZHOU J W, et al. The adaptive strategies of yaks to live in the Asian highlands[J]. Animal Nutrition,2022,9(2):249−258.
[2] MA X M, XI B, KOREJO R A, et al. Yak milk and its health benefits:a comprehensive review[J]. Frontiers in Veterinary Science,2023,10:1213039. doi: 10.3389/fvets.2023.1213039
[3] LI A L, LIU C, HAN X T, et al. Tibetan plateau yak milk:A comprehensive review of nutritional values, health benefits, and processing technology[J]. Food Chemistry:X,2023,20:100919.
[4] SINGH T P, ARORA S, SARKAR M. Yak milk and milk products:Functional, bioactive constituents and therapeutic potential[J]. International Dairy Journal,2023,142:105637. doi: 10.1016/j.idairyj.2023.105637
[5] JAMRÓGIEWICZ M. Application of the near-infrared spectroscopy in the pharmaceutical technology[J]. Journal of Pharmaceutical,2012,66:1−10.
[6] QU J H, LIU D, CHENG J H, et al. Applications of near-infrared spectroscopy in food safety evaluation and control:a review of recent research advances[J]. Critical Reviews in Food Science Nutrition,2015,55(13):1939−1954. doi: 10.1080/10408398.2013.871693
[7] ZHANG W W, KASUN L C, WANG Q J, et al. A review of machine learning for near-infrared spectroscopy[J]. Sensors,2022,22(24):9764. doi: 10.3390/s22249764
[8] ELAINY V D S P, DAVID D D S F, MÁRIO C U D A, et al. Simultaneous determination of goat milk adulteration with cow milk and their fat and protein contents using NIR spectroscopy and PLS algorithms[J]. LWT,2020,127:109427. doi: 10.1016/j.lwt.2020.109427
[9] MABOOD F, JABEEN F, HUSSAIN J, et al. FT-NIRS coupled with chemometric methods as a rapid alternative tool for the detection & quantification of cow milk adulteration in camel milk samples[J]. Vibrational Spectroscopy,2017,92:245−250. doi: 10.1016/j.vibspec.2017.07.004
[10] ALESSANDRA B, FERRAO M F, MELLO C, et al. Least-squares support vector machines and near infrared spectroscopy for quantification of common adulterants in powdered milk[J]. Analytica Chimica Acta,2006,579(1):25−32. doi: 10.1016/j.aca.2006.07.008
[11] ZHANG T F, WU X H, WU B, et al. Rapid authentication of the geographical origin of milk using portable near-infrared spectrometer and fuzzy uncorrelated discriminant transformation[J]. Journal of Food Process Engineering,2022,45(8):14040. doi: 10.1111/jfpe.14040
[12] BAI W L, YIN R H, ZHAO S J, et al. Rapid detection of bovine milk in yak milk using a polymerase chain reaction technique[J]. Journal of Dairy Science,2009,92(4):1354−1360. doi: 10.3168/jds.2008-1727
[13] WU X Y, NA Q, HAO S Q, et al. Detection of ovine or bovine milk components in commercial camel milk powder using a pcr-based method[J]. Molecules,2022,27(9):3017. doi: 10.3390/molecules27093017
[14] SCHOOT M, KAPPER C, VAN KOLLENBURG G H, et al. Investigating the need for preprocessing of near-infrared spectroscopic data as a function of sample size[J]. Chemometrics Intelligent Laboratory Systems,2020,204:104105. doi: 10.1016/j.chemolab.2020.104105
[15] MAĆKIEWICZ A, RATAJCZAK W, GEOSCIENCES. Principal components analysis (PCA)[J]. Computers,1993,19(3):303−342.
[16] KURITA T. Principal component analysis (PCA)[J]. Computer Vision:A Reference Guide, 2019:1−4.
[17] SALEM B. Principal component analysis (PCA)[J]. La Tunisie Medicale,2021,99(4):383−389.
[18] GUO G, WANG H, BELL D, et al. KNN model-based approach in classification. Proceedings of the on the move to meaningful internet systems[C]//Sicily:OTM Confederated International Conferences,2003.
[19] ZHANG S C, LI X L, ZONG M, et al. Efficient KNN classification with different numbers of nearest neighbors[J]. IEEE Transactions on Neural Networks Learning Systems,2017,29(5):1774−1785.
[20] ZHANG Z H. Introduction to machine learning:k-nearest neighbors[J]. Annals of Translational Medicine,2016,4(11):218. doi: 10.21037/atm.2016.03.37
[21] TOBIAS R D. An introduction to partial least squares regression[C]// Proceedings of the proceedings of the twentieth annual SAS users group international conference,1995.
[22] HAIR J F, SARSTEDT M, RINGLE C M. Rethinking some of the rethinking of partial least squares[J]. European Journal of Marketing,2019,53(4):566−584. doi: 10.1108/EJM-10-2018-0665
[23] FU P, MEACHAM-HENSOLD K, GUAN K Y, et al. Estimating photosynthetic traits from reflectance spectra:a synthesis of spectral indices, numerical inversion, and partial least square regression[J]. Plant, Cell & Environment, 2020, 43(5):1241-1258.
[24] LI X Y, GUAN C T, ZHANG H H, et al. A unified Fisher’s ratio learning method for spatial filter optimization[J]. IEEE transactions on neural networks learning systems,2016,28(11):2727−2737.
[25] DAI H Y, LEEDER J S, CUI Y H. A modified generalized Fisher method for combining probabilities from dependent tests[J]. Frontiers in Genetics,2014,5:74581.
[26] FERRANDI G, KRAVCHENKO I V, HOCHSTENBACH M E, et al. On the trace ratio method and Fisher's discriminant analysis for robust multigroup classification[J]. ARXIV,2022,22(11):08120.
[27] MEHMOOD T, LILAND K H, SNIPEN L, et al. A review of variable selection methods in partial least squares regression[J]. Chemometrics Intelligent Laboratory Systems,2012,118:62−69. doi: 10.1016/j.chemolab.2012.07.010
[28] FARRÉS M, PLATIKANOV S, TSAKOVSKI S, et al. Comparison of the variable importance in projection (VIP) and of the selectivity ratio (SR) methods for variable selection and interpretation[J]. Journal of Chemometrics,2015,29(10):528−536. doi: 10.1002/cem.2736
[29] GREENWELL B M, BOEHMKE B C, GRAY B. Variable importance plots-an introduction to the vip package[J]. Contributed Research Article,2020,12(1):343−366.
[30] FU J S, YU H D, CHEN Z, et al. A review on hybrid strategy-based wavelength selection methods in analysis of near-infrared spectral data[J]. Infrared Physics Technology,2022,125:104231. doi: 10.1016/j.infrared.2022.104231
[31] JIANG W W, LU C H, ZHANG Y J, et al. Moving-window-improved Monte carlo uninformative variable elimination combining successive projections algorithm for near-infrared spectroscopy (NIRS)[J]. Journal of Spectroscopy,2020,20:1−12.
[32] SONG X Z, HUANG Y, TIAN K D, et al. Near infrared spectral variable optimization by final complexity adapted models combined with uninformative variables elimination-a validation study[J]. Optik,2020,203:164019. doi: 10.1016/j.ijleo.2019.164019
[33] WANG Z X, HE Q P, WANG J. Comparison of variable selection methods for PLS-based soft sensor modeling[J]. Journal of Process Control,2015,26:56−72. doi: 10.1016/j.jprocont.2015.01.003
[34] ALHARAN A F, FATLAWI H K, ALI N S. A cluster-based feature selection method for image texture classification[J]. Indonesian Journal of Electrical Engineering Computer Science,2019,14(3):1433−1442. doi: 10.11591/ijeecs.v14.i3.pp1433-1442
[35] SEWAL P, SINGH H. Analyzing distributed Spark MLlib regression algorithms for accuracy, execution efficiency and scalability using best subset selection approach[J]. Multimedia Tools Applications,2023,23:1−20.
[36] MAAROOF B B, RASHID T A, ABDULLA J M, et al. Current studies and applications of shuffled frog leaping algorithm:a review[J]. Archives of Computational Methods in Engineering,2022,2(1):1−16.
[37] AL MAMOORI G, SOVA O, ZHUK O, et al. The development of solution search method using improved jumping frog algorithm[J]. Eastern-European Journal of Enterprise Technologies, 2023, 124(3).
[38] TANG D Y, ZHAO J, YANG J, et al. An evolutionary frog leaping algorithm for global optimization problems and applications[J]. Computational Intelligence Neuroscience,2021,21:8928182.
-
期刊类型引用(1)
1. 庞祥艺,张娟,王楠,李晓雨,陈嘉词,樊霞,郑文新,陈爱亮. 驼乳中牛乳掺假快速检测双重胶体金免疫层析试纸条的研制. 食品科技. 2024(12): 281-287 .  百度学术
百度学术
其他类型引用(0)





 下载:
下载:
 下载:
下载:



