Investigation on the Recognition of Split Aflatoxin M1 Aptamer
-
摘要: 本研究以黄曲霉毒素M1为研究对象,采用劈裂适配体的方式,克服适配体构象不稳定造成的灵敏度不高的问题,利用电化学方法研究劈裂适配体的识别能力及特异性,并通过圆二色谱初步探究结合机制。结果表明,将适配体核苷酸链按照1:1进行劈裂得到的识别效果最佳,并且对黄曲霉毒素M1具有较高的特异性。劈裂适配体后单独两个片段与AFM1作用后圆二色谱图没有改变,正峰强度明显增大,负峰移动并且强度增大,证明适配体构象发生改变。基于劈裂适配体可以改善方法的灵敏度,通过对完整适配体进行劈裂,得到了构建传感器的更佳识别元件。本研究为提高适配体快速检测方法的灵敏度提供了一种新思路。Abstract: In this work, AFM1 was chosen as the target, and the aptamer was split to solve the low sensitivity due to unstable structure of the aptamer. The recognition ability and specificity of the split aptamer were evaluated using electrochemical methods, and the binding mechanism was explored through the circular dichroism. The results showed that the split aptamer employing a 1:1 split mode exhibited the best performance in the recognition and specificity for AFM1. The circular dichroism spectrum remained unchanged. The positive peak intensity of the circular dichroism spectrum increased significantly and the negative peak moved and the intensity increased, which demonstrated that the conformation of the aptamer changed. The sensitivity was enhanced by employing split aptamers. Superior recognition elements for the construction of sensors was obtained by the fragmentation of intact aptamers. The investigation provides a new idea to enhance the sensitivity of the detection mehtods based on the apatamer.
-
Keywords:
- flatoxin M1 /
- split aptamer /
- recognition ability /
- electrochemistry
-
随着人们生活水平的提高,乳及乳制品已成为重要的消费品之一,其安全性受到社会广泛关注,而黄曲霉毒素M1(Aflatoxin M1,AFM1)是影响乳及乳制品安全的主要因素。AFM1具有致畸、致癌、致突变的作用,被国际癌症机构列为I类致癌物,严重危害人类健康。另外,AFM1热稳定性很强,不能通过巴氏灭菌、超高温杀菌等加工方式将其降解[1−3]。因此,构建快速、灵敏的AFM1检测方法对于保障人体健康具有重要意义。
由于适配体的高灵敏、高稳定性及低成本等特点,基于适配体的快速检测技术发展迅速。自发现AFM1适配体可通过SELEX法成功筛选后[4−6],科研人员已成功构建了多种基于AFM1适配体的快速检测传感器[7−10]。Sameiyan等[11]基于二价适配体-适配体互补链(Bivalent binding aptamer-cDNA,BBA-cDNA)结构及二硫化钼(MoS2)构建一种新型荧光适配体传感器检测AFM1。与之前研究不同[12−13],实验中BBA-cDNA结构未破坏,cDNA吸附在二价适配体上,然而此传感器仍然能实现对AFM1的快速响应。Ge等[14]开发了一种基于自组装双交叉DNA纳米机器,由于能量共振转移,荧光强度降低。加入AFM1和AFB1后,适配体识别黄曲霉毒素,双交叉DNA纳米结构被破坏,猝灭基团远离荧光染料,荧光强度恢复。Yadav等[15]构建了基于金属硫化物的适配体荧光传感器同时检测AFB1和AFM1,并比较了基于聚乙二醇改性前后的金属硫化物构建传感器的灵敏度,结果表明,与为改性材料相比,利用聚乙二醇改性后的纳米材料所构建传感器的检出限低248倍。Yang等[16]建立了基于分子印迹聚合物(Molecular imprinting,MIP)和适配体的双识别电化学传感器检测牛奶中AFM1。
尽管小分子的适配体筛选技术及应用研究不断发展[17−18],但由于小分子尺度较小,结合位点少[19−20],引起传感信号的变化也非常有限,开发的适配体存在灵敏度低、特异性差的问题[21−22]。另外,适配体因其柔性结构的问题,在环境中变现出结构不稳定的现象,而发生非特异性识别,造成假阳性信号的产生[23−25],进一步限制了适配体快速检测技术的发展。有研究发现将一段完整的适配体劈裂成两段或两段以上,当添加目标物后,劈裂的适配体仍然可以重新结合共同识别目标物[26−29]。与单适配体相比,具有合作识别的分裂适配体可能会增强靶标结合的亲和力[30−32]。基于劈裂适配体构建的检测方法可以有效减少假阳性结果,提高方法的准确性[33−35]。目前,基于劈裂适配体已成功用于ATP[36−38]、多巴胺[39]、凝血酶[40]等的检测。
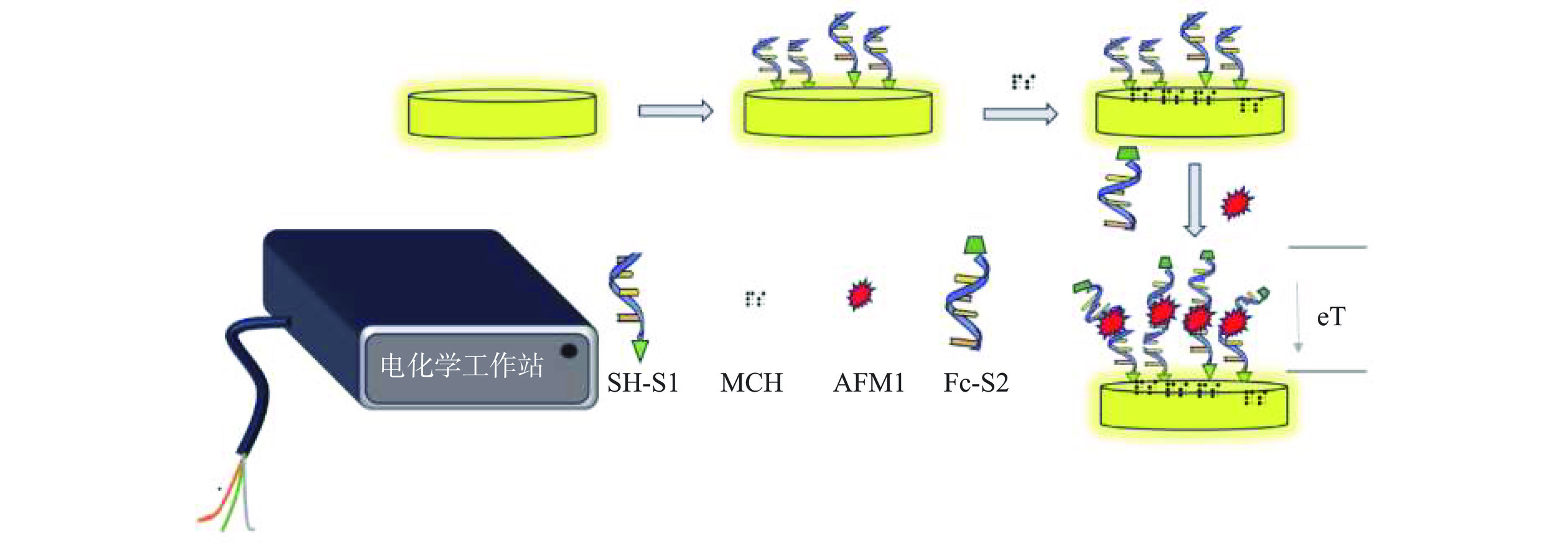
因此,本文以AFM1为研究对象,利用电化学方法。原理如图1所示,将AFM1适配体劈裂成两段,一段修饰巯基(SH-S1)通过Au-S固定在电极表面,另一段修饰二茂铁(Fc-S2)作为响应信号。将S1固定到电极表面后,采用6-巯基乙醇(MCH)封闭未结合位点,减小非特异性结合。在体系中添加AFM1,S1与S2形成一定构象共同识别AFM1,S2一端的响应信号Fc靠近工作电极,与电极进行电子传递,产生Fc信号。本文通过研究劈裂适配体的亲和力、特异性以及对AFM1的检测效果,利用圆二色谱法初步探究的结合机制,为后续建立传感检测方法打下基础。
1. 材料与方法
1.1 材料与仪器
铁氰化钾、过氧化氢、亚铁氰化钾、三羟甲基氨基甲烷(Tris) 分析纯,上海麦克林生化科技有限公司;氯化钾、磷酸三氯乙酯(TCEP)、氯化钠、三氯化六铵合钌、98% 6-巯基乙醇(MCH) 分析纯,上海阿拉丁生化科技有限公司;盐酸 分析纯,成都科龙化工试剂公司;98%硫酸、无水乙醇 分析纯,重庆市钛新化工有限公司;劈裂适配体 由生物工程(上海)股份有限公司合成,适配体序列见表1。
表 1 适配体序列Table 1. Aptamer sequence适配体 序列 AFM1适配体 5´-SH-ACTGCTAGAGATTTTCCACAT-Fc-3´[38] mode 1(3:1) 5´-SH-ACTGCTAGAGATTTTC-3 5´-CACAT-Fc-3´ mode 2(2:1) 5´-SH-ACTGCTAGAGATTT-3´ 5´-TCCACAT-Fc-3´ mode 3(1:1) 5´-SH-ACTGCTAGAGA-3´ 5´-TTTCCACAT-Fc-3´ mode 4(1:2) 5´-SH-ACTGCTA-3´ 5´-GAGATTTTCCACAT-Fc-3´ mode 5(1:3) 5´-SH-ACTGC-3´ 5´-TAGAGATTTTCCACAT-Fc-3´ 电化学工作站CHI660E、CHI111银/氯化银、CHI115铂丝电极、CHI101金电极 上海辰华仪器有限公司;HH-4J型磁力搅拌恒温水浴锅 江苏金坛白塔新宝仪器厂;KQ3200DB超声波清洗器 昆山市超声仪器有限公司;NK200-1B氮吹仪 常州朗越仪器制造有限公司;Multifuge X3R离心机 赛默飞世尔科技公司;Milli-Q超纯水仪 美国Millipore公司;J-81010400圆二色谱仪 日本分光株式会社。
1.2 实验方法
1.2.1 劈裂适配体识别效果
电极的预处理[41]:依次取直径为0.3、0.05 µm的抛光粉(氧化铝)按照“8”字手法将金电极打磨至表面光滑,然后将电极分别置于无水乙醇和超纯水中超声清洗。随后将电极置于食人鱼溶液(H2SO4/H2O2,3/1)中浸泡,10 min后超纯水清洗表面,氮吹至干燥。最后,在0.5 mol/L H2SO4溶液中,设置扫描速度和扫描点位分别为0.1 V/s和−0.2~+1.6 V,采用循环伏安法(Cyclic voltammetry,CV)进行扫描,待扫描曲线稳定后对电极进行电化学抛光处理。抛光完成后用超纯水洗净,氮吹干燥备用。
AFM1原始序列按照比例3:1、2:1、1:1、1:2、1:3进行劈裂(表1)。采用相同浓度(0.5 μg/L)AFM1,探究五种劈裂模式的适配体识别AFM1的效果。
1.2.2 劈裂适配体固定环境的研究
S1在电极表面的实际修饰密度可以准确反映适配体在电极表面的状态。本文基于Cottrell方程[42]来计算适配体的实际修饰密度见公式(1):
Q=(2nFAD01/2C0t1/2)π1/2+Qdl+nFAΓ0 (1) 式中,Q代表[Ru(NH3)6]3+扩散到电极表面产生的电荷(C);n为每摩尔中用于还原的电子数量;F代表法拉第常量,即每摩尔电子所携带的电荷量,其值为96485 C/mol;A为电极面积(cm2);D0为扩散系数(cm2/s);C0为体积浓度(mol/cm2);t代表时间(s);Qdl表示电层电容(F),可根据计时库伦法(Chronocoulometry,CC)测试曲线的截距差计算得出;nFAΓ0表示吸附[Ru(NH3)6]3+产生的电荷(mol/cm2);Γ0表示吸附到S1骨架上的[Ru(NH3)6]3+的密度,可以通过两条CC测试曲线的截距差计算得出。
S1在电极表面的密度Γ0可根据公式(1)、(2)[43]计算得出:
ΓDNA=Γ0×zm×NA (2) 式中,ΓDNA表示S1的电极表面修饰密度(mol/cm2);m表示S1的碱基数目;z表示氧化还原分子的电荷量;NA表示 Avogadro常数6.02×1023。
1.2.2.1 离子强度对劈裂适配体固定电极的影响
将10 µmol/L的SH-S1溶液和1 mmol/L的TCEP溶液各取10 μL混合并孵育1 h,分别加入Na+浓度为50、100、150、200、250 mmol/L的Tris-HCl溶液(pH7.4),至溶液的终体积为200 μL,SH-S1溶液终浓度为0.5 µmol/L。金电极与该溶液孵育16 h,通过Au-S键将SH-S1在电极表面固定,氮吹干燥。最后置于MCH(1 mmol/L)溶液中孵育1 h,用缓冲溶液洗涤,氮气吹干,传感器平台构建完成。随后将其与含0.5 µmol/L的Fc-S2及0.5 μg/L靶标物AFM1的混合溶液孵育1 h。置于电解支持液(5 mmol/L铁氰化钾、5 mmol/L亚铁氰化钾混合溶液与0.1 mol/L氯化钾水溶液等体积混合)中,根据CC计时测试结果按照公式计算电极表面S1的密度,再通过CV及电化学阻抗法(Electrochemical impedance spectroscopy,EIS),以电流峰值和奎斯特曲线半径来研究固定情况[44]。
1.2.2.2 pH对劈裂适配体固定电极的影响
将10 µmol/L的SH-S1溶液和1 mmol/L的TCEP溶液各取10 μL混合并孵育1 h,分别用不同pH(5.4、6.4、7.4、8.4、9.4)的Tris-HCl(150 mmol/L)溶液稀释至溶液的终体积为200 μL,SH-S1溶液终浓度为0.5 µmol/L。金电极与该溶液孵育16 h后,氮吹干燥。最后置于MCH(1 mmol/L)溶液中孵育1 h,用缓冲溶液洗涤,氮气吹干,传感器平台构建完成。检测内容同1.2.2.1。
1.2.3 劈裂适配体构建传感器检测条件的研究
1.2.3.1 SH-S1孵育浓度对检测的影响
将10 µmol/L的SH-S1溶液和1 mmol/L TCEP溶液各取10 μL混合并孵育1 h,用Tris-HCl溶液(pH7.4,200 µmol/L Na+)将溶液分别稀释至SH-S1终浓度分别为0.1、0.3、0.5、0.7、0.9 µmol/L。按上述步骤构建传感器平台。然后将其与含0.5 µmol/L的Fc-S2及0.5 μg/L靶标物AFM1的混合溶液孵育1 h。通过方波伏安法(Square wave voltammetry,SWV),以IFc信号值为指标,研究不同浓度适配体的检测效果[45]。
1.2.3.2 孵育时间对检测效果的影响
将10 µmol/L的 SH-S1溶液和1 mmol/L的TCEP溶液各取10 μL混合孵育1 h后,用Tris-HCl溶液(pH7.4,200 µmol/L Na+)稀释至SH-S1终浓度为0.5 µmol/L。按上述步骤构建传感器平台。然后与含0.5 µmol/L的Fc-S2及0.5 μg/L靶标物AFM1的混合溶液分别孵育30、60、90、120 min。通过SWV探究适配体在不同孵育时间下的检测效果。
1.2.3.3 离子强度对识别效果的影响
缓冲溶液Tris-HCl(pH7.4)中分别添加0、50、100、200、300、400、500、600 µmol/L的Na+,将SH-S1稀释至终浓度为0.5 µmol/L,按上述步骤构建传感器平台。然后与含0.5 µmol/L的Fc-S2及0.5 μg/L靶标物AFM1的混合溶液孵育1 h。通过SWV研究不同离子强度对适配体识别性能的影响。
1.2.3.4 pH对识别效果的影响
将缓冲溶液Tris-HCl(200 µmol/L Na+)pH分别调至5.4、6.4、7.4、8.4、9.4,将SH-S1稀释至终浓度为0.5 µmol/L,按上述步骤构建传感器平台。然后与含0.5 µmol/L的Fc-S2及0.5 μg/L靶标物AFM1的混合溶液孵育1 h。通过SWV研究pH对适配体识别性能的影响。
1.2.4 劈裂适配体识别能力和亲和力研究
将10 µmol/L的SH-S1溶液和1 mmol/L的TCEP溶液各取10 μL混合,孵育时间1 h,用Tris-HCl溶液(pH7.4)稀释至SH-S1终浓度为0.5 µmol/L。加入离心管内,将金电极浸入其中16 h,氮气吹干后。置于MCH(1 mmol/L)溶液中孵育1 h,将未结合的位点除掉。用缓冲溶液洗涤,氮气吹干,传感器平台构建完成。将电极完全沉浸在含有Fc-S2(0.5 µmol/L)与不同浓度AFM1溶液中孵育。利用CV和EIS研究电极修饰过程,并利用SWV研究劈裂适配体的识别效果和亲和力。
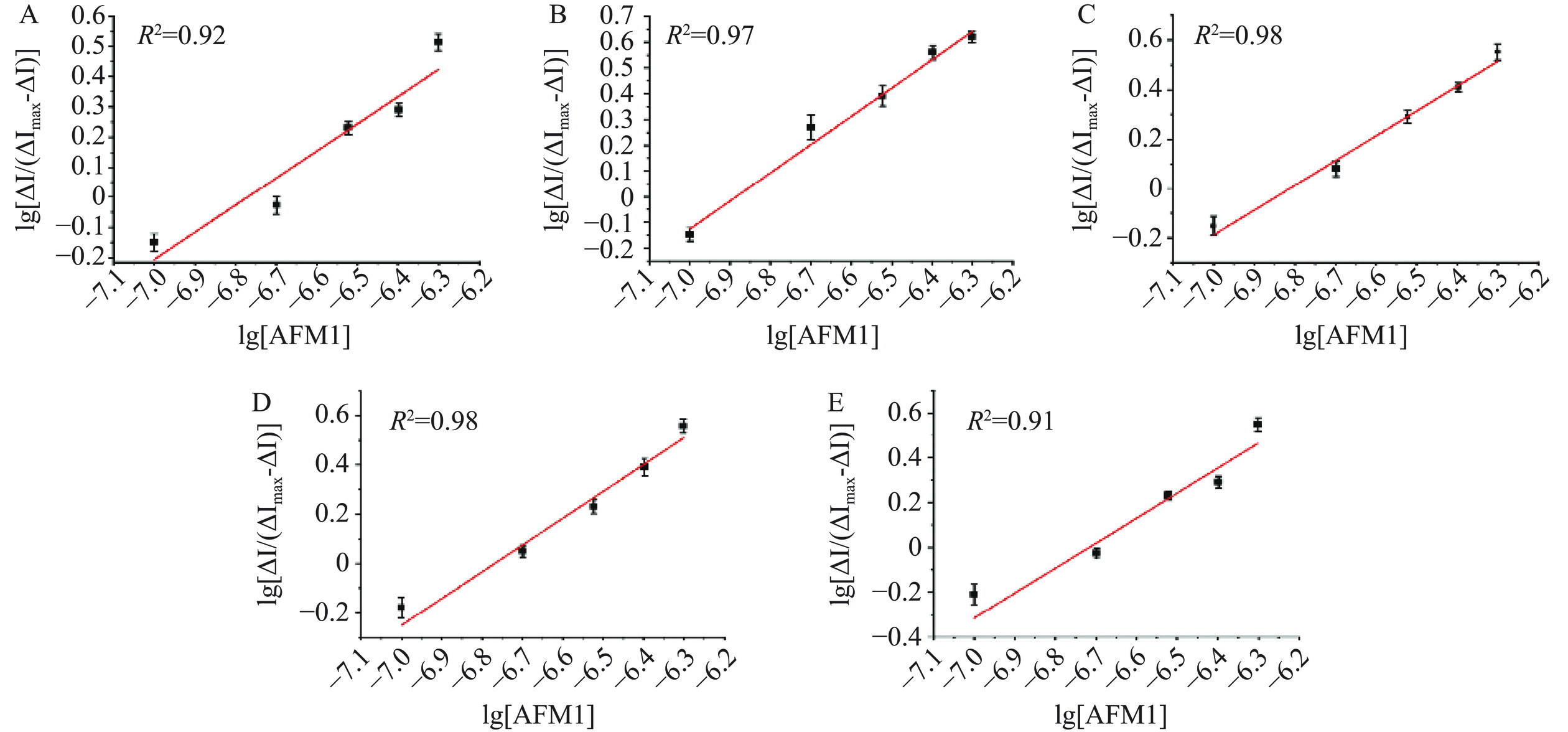
通过电化学方法研究劈裂适配体的亲和力。在一定浓度范围内,随着AFM1浓度的增加,电响应信号逐渐增加。基于电信号数据利用公式(3)[46−47]计算解离常数Kd,Kd值表征五种劈裂适配体的亲和力,Kd值越小,代表适配体和靶标物的结合能力越强。
log1[DNA]=logKd+logΔI(ΔImax−ΔI) (3) 式中,[DNA]代表适配体的浓度(nmol/L),Imax代表产生的最大电流信号值(nA),I代表不同AFM1浓度下产生的电流信号值(nA)。
线性范围和检测限:将亚甲基蓝(MB)-S2和Fc-S1与一定浓度的AFM1溶液混合,将建立的传感器平台浸入上述溶液中孵育一段时间,用SWV进行检测。以AFM1标准品浓度为横坐标,IMB/IFc为纵坐标,建立标准曲线,确定线性范围并计算检测限。
1.2.5 劈裂适配体特异性研究
配制与AFM1相同浓度(0.5 μg/L)的AFM2、AFB1、AFG1、AFG2作为干扰毒素研究劈裂适配体的特异性。将建立的传感器平台置于含有Fc-S2(0.5 µmol/L)和不同种类毒素的溶液中孵育1 h。用SWV进行检测。
1.2.6 利用圆二色谱研究劈裂适配体构象
将完整的AFM1适配体(10 mmol/L)和劈裂后的两段适配体S1(10 mmol/L)、S2(10 mmol/L)在95 ℃下变性处理10 min。取等量的S1和S2进行混合得到S1+S2,将完整AFM1适配体、S1、S2、S1+S2静置30 min,使其形成稳定空间构象。另取等量S1和S2与AFM1孵育60 min,使S1、S2共同识别AFM1,将上述溶液均进行圆二色谱测定[48]。
1.3 数据处理
亲和力数据处理:采用电化学方法得到相关数据,以适配体浓度的对数为横坐标,电信号变化量的对数为纵坐标,使用线性回归分析绘制曲线,得到劈裂适配体的解离常数。
2. 结果与分析
2.1 劈裂适配体识别效果
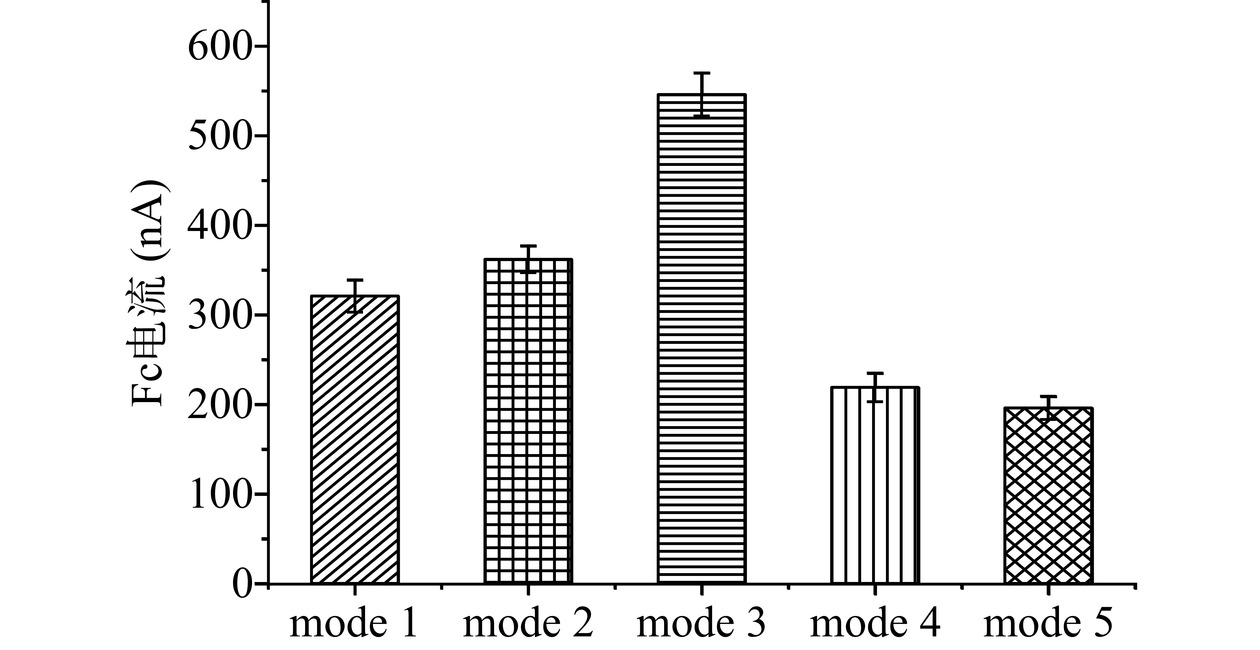
研究五种劈裂模式下的适配体对AFM1的识别效果,结果如图2,加入0.5 μg/L AFM1后,以mode 3(1:1)为识别元件时,Fc信号最大,为546±24 nA,表明在五种劈裂方式中,以mode 3(1:1)模式劈裂的适配体具有最好的识别效果。
2.2 劈裂适配体固定环境的研究
适配体构象可能会受到体系中离子强度和pH的影响,从而影响其在电极表面的固定。因此,本文以识别效果最好的mode 3(1:1)模式的劈裂适配体为研究对象,选择0.5 μmol/L SH-S1固定电极,采用CC研究电极表面S1的密度,利用CV和EIS研究S1在电极表面的固定情况。
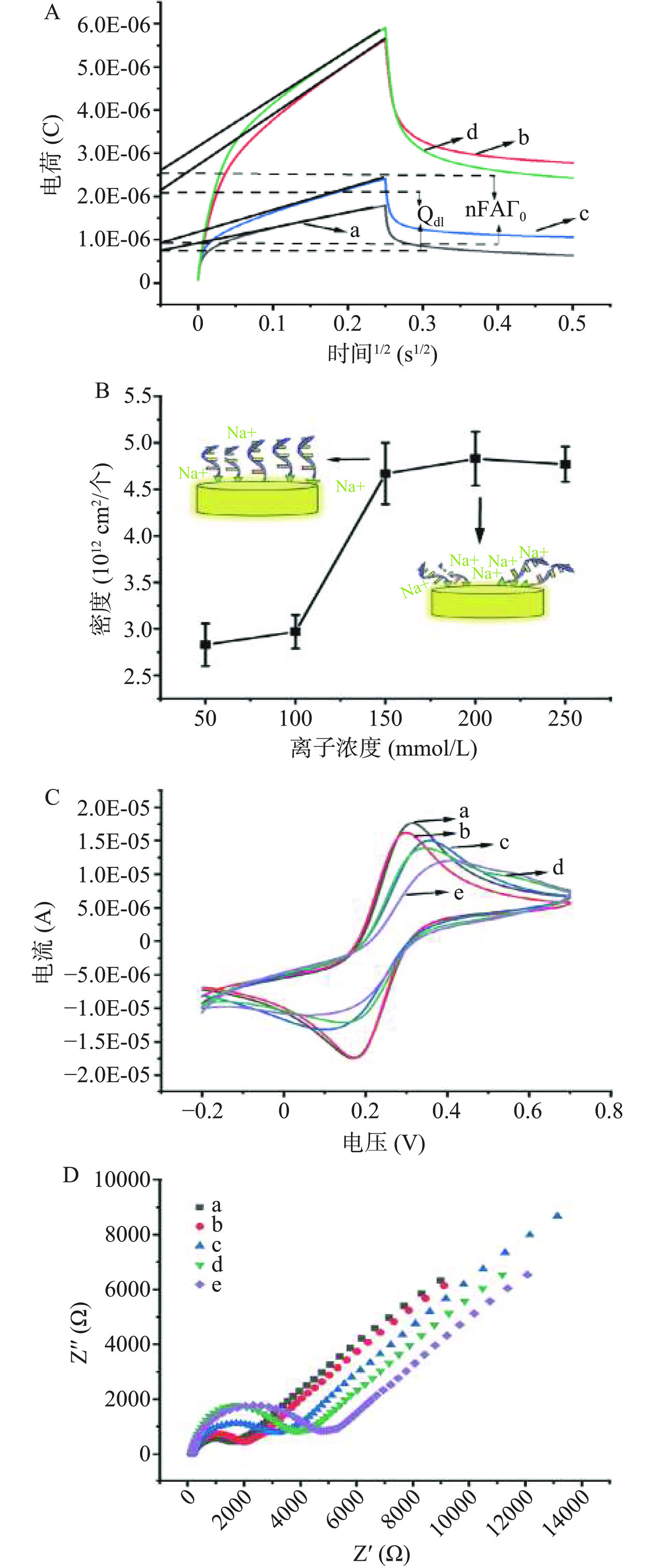
基于式(1)、式(2)和计时库伦测试结果(图3A)计算电极表面S1的密度。如图3B所示,随着Na+浓度增加,电极表面S1的密度逐渐增加,当Na+为150 mmol/L时,密度达到最大且不再随Na+浓度的增加而变化,说明0.5 μmol/L S1几乎都修饰在电极表面。
![]() 图 3 离子强度对适配体固定的影响注:A:计时库伦曲线,B:不同Na+浓度下电极表面适配体密度图,C:不同Na+浓度下循环伏安图,D:不同Na+浓度下阻抗图;图A中a~d表示:a:MCH修饰电极在不含[Ru(NH3)6]3+,b:MCH修饰电极在含[Ru(NH3)6]3+,c:MCH/S1修饰电极在不含[Ru(NH3)6]3+,d:MCH/S1修饰电极在含[Ru(NH3)6]3+;图C、D中a~e表示:a:250 mmol/L,b:200 mmol/L,c:150 mmol/L,d:100 mmol/L,e:50 mmol/L。Figure 3. Influence of ionic strength on aptamer immobilization
图 3 离子强度对适配体固定的影响注:A:计时库伦曲线,B:不同Na+浓度下电极表面适配体密度图,C:不同Na+浓度下循环伏安图,D:不同Na+浓度下阻抗图;图A中a~d表示:a:MCH修饰电极在不含[Ru(NH3)6]3+,b:MCH修饰电极在含[Ru(NH3)6]3+,c:MCH/S1修饰电极在不含[Ru(NH3)6]3+,d:MCH/S1修饰电极在含[Ru(NH3)6]3+;图C、D中a~e表示:a:250 mmol/L,b:200 mmol/L,c:150 mmol/L,d:100 mmol/L,e:50 mmol/L。Figure 3. Influence of ionic strength on aptamer immobilization图3C和D显示不同Na+浓度固定劈裂适配体S1的CV图及EIS图。如图所示,随着Na+浓度的增加,氧化还原的电流峰值逐渐增大后几乎不变,奈奎斯特曲线半径逐渐减小后几乎不变。说明随着Na+浓度增加,修饰S1后电极表面电子传递速度逐渐增加。结合电极表面S1密度及CV和EIS图发现,在低Na+浓度环境中,电极表面S1密度较小,而且电子传递速度较慢,这种现象的原因可能是S1的柔性结构,在电极表面呈平铺覆盖状,阻碍了电子传递速度。在高浓度Na+环境下,由于S1与足够Na+之间的静电相互作用,S1保持刚性结构,在电极表面可以固定更多的S1,S1密度逐渐增大(图3B)。因此,150 mmol/L为固定劈裂适配体的最佳Na+浓度。
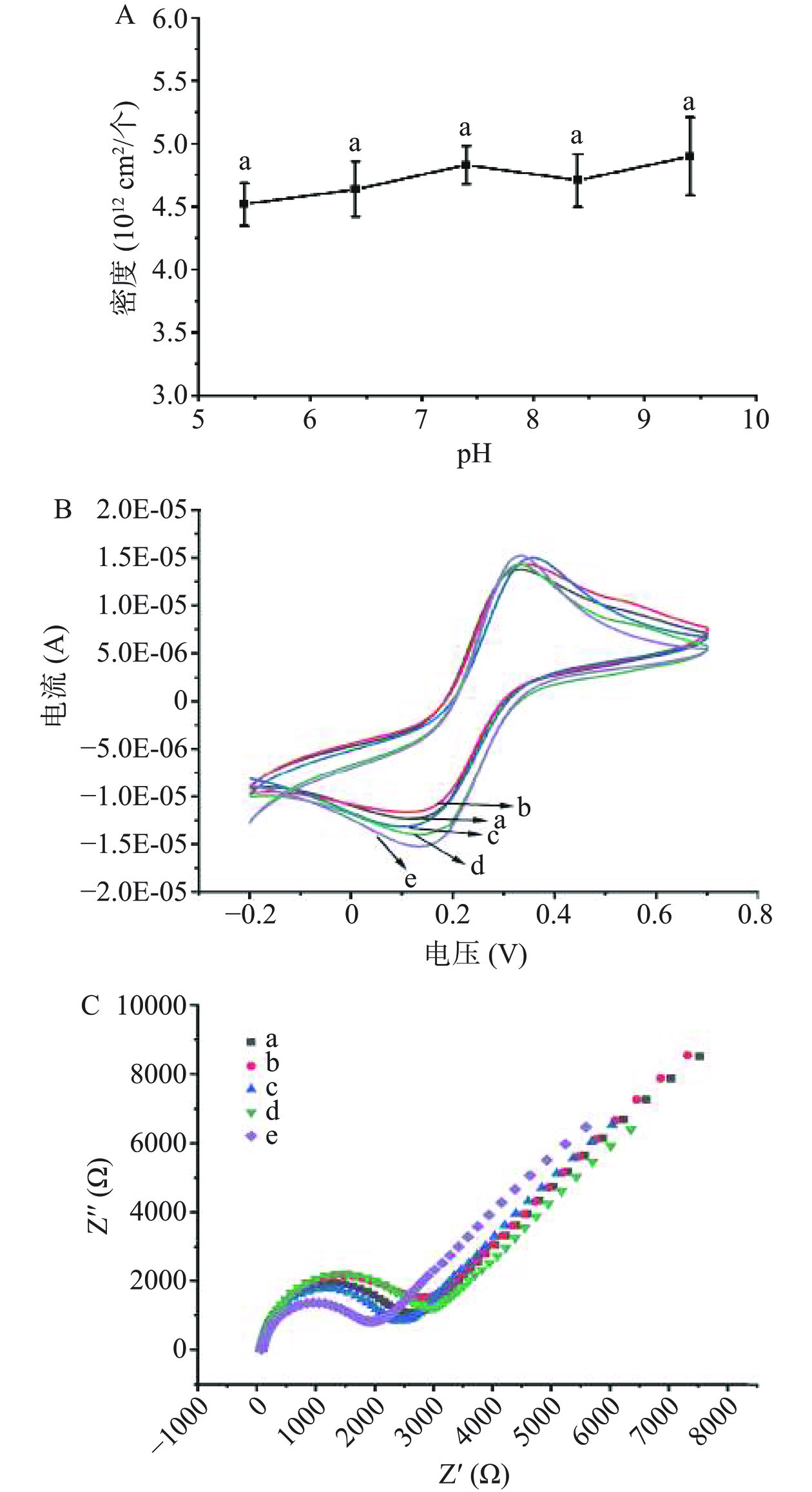
图4为pH对S1固定电极密度和固定情况的影响。如图4A所示,pH对电极表面S1的固定密度无明显影响。同时图4B和C的CV和EIS结果显示,不同pH下氧化还原电流峰和奈奎斯特曲线半径均无明显差异。说明在一定范围内pH对S1在电极表面的固定及S1的结构无明显影响,选择7.4为后续固定电极时溶液pH。
2.3 劈裂适配体构建传感器检测条件的研究
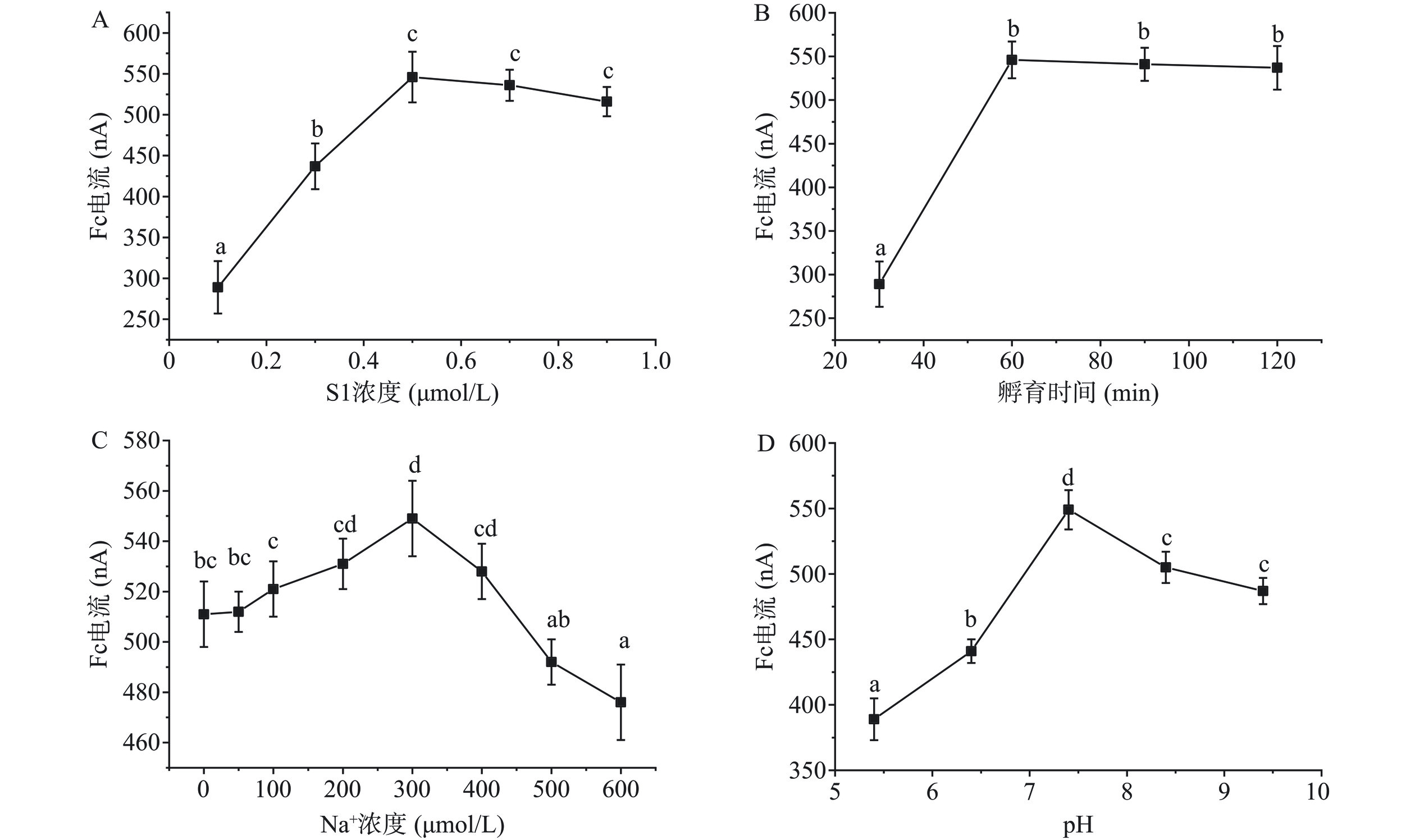
本文以二茂铁产生的信号值(IFc)研究S1浓度、孵育时间及识别环境对识别效果的影响(图5)。图5A显示S1浓度对识别效果的影响。从图可知,传感信号值(IFc)随着S1浓度的增加呈现快速增加后减小的趋势。因为S1浓度低时,固定在电极表面的S1数量较少,检测的灵敏度不高。而高浓度的S1固定在电极表面后,会产生空间位阻,灵敏度也会受到影响[49]。S1浓度到0.5 μmol/L时,传感电信号达到最大,说明此时识别效果最好。
图5B显示了劈裂适配体与靶标物AFM1的孵育时间对识别效果的影响。传感电信号(IFc)会随孵育时间增加而增大,因为孵育时间较短,劈裂适配体不能形成构象完全识别AFM1,影响响应电信号。当孵育60 min后,信号值(IFc)达到最高且趋于平衡。说明此时劈裂适配体已完成目标物AFM1的识别。因此最佳孵育时间确定为60 min。
识别环境的离子强度和pH对适配体构象的形成和稳定有一定的影响,进而影响适配体对目标物的识别。在一定范围内,IFc响应信号随着Na+浓度增加先增加后减小。因为离子浓度过低,劈裂适配体S1和S2自身发生卷曲,影响对目标物AFM1的识别效果。当检测环境中离子浓度过高时,S1和S2呈刚性结构,同样影响对目标物的识别。当Na+浓度为300 μmol/L时,IFc信号值最大。因此识别环境中的最适离子强度为300 μmol/L Na+。如图5D可知,在 pH 为 5.4 到 7.4 范围内,随着 pH 增加 IFc 信号值逐渐增加,当pH大于7.4时,IFc信号值随着 pH 的增加而逐渐减小。表明中性环境下(pH7.4)劈裂适配体结合靶标物的效果最佳。
2.4 劈裂适配体识别能力的研究
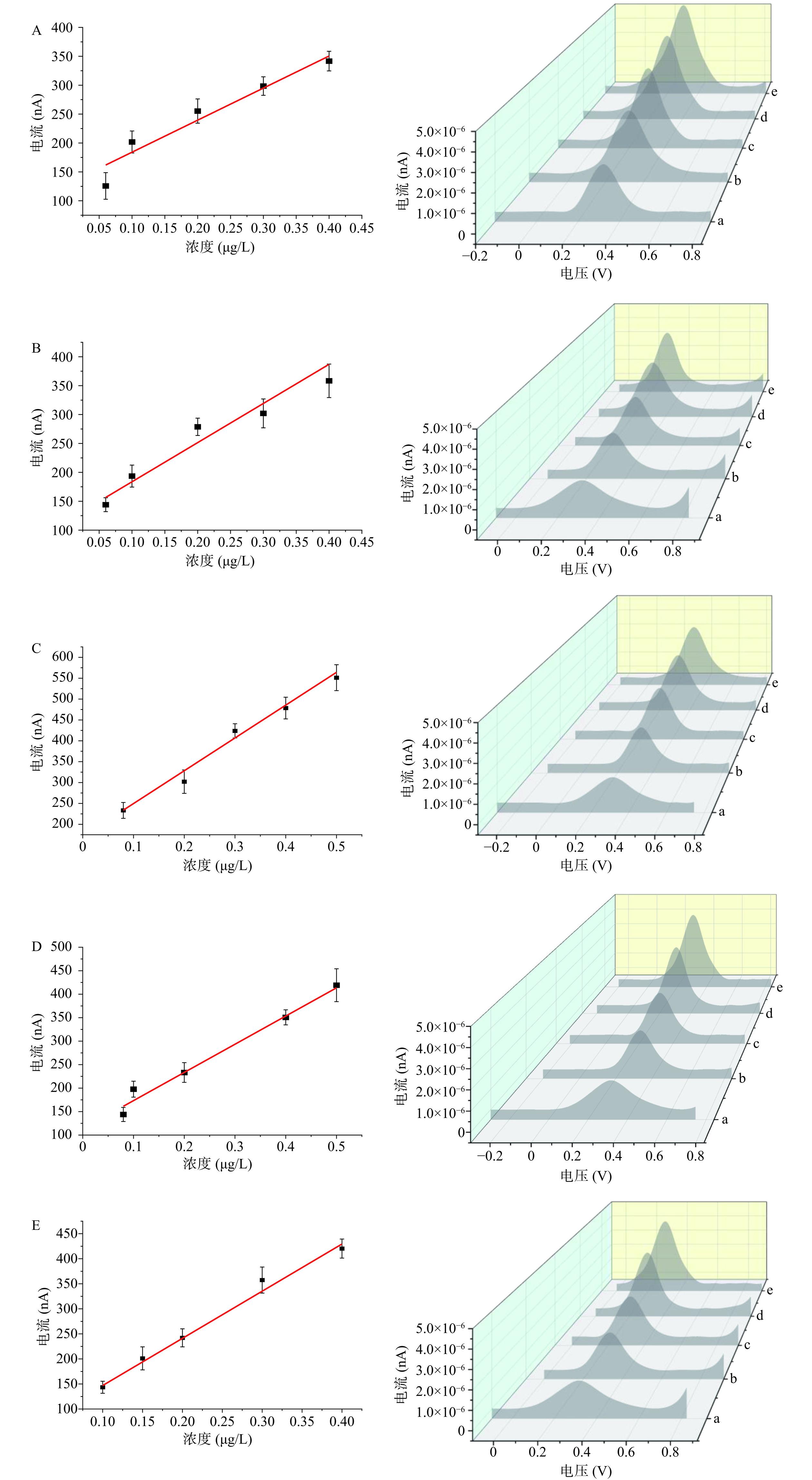
为了分析劈裂后的适配体识别能力,构建基于五种劈裂适配体的电化学传感器。根据传感器对目标物检测的线性范围和检出限,评价电化学传感器的传感性能。结果显示(图6), 随着AFM1浓度增加,基于不同劈裂适配体的传感器响应信号均逐渐增加。由于AFM1的加入,S1与游离S2形成一定构象共同识别AFM1,导致Fc响应电信号增加。通过实验考察了五种电化学传感器的线性范围和检出限。结果表明,mode 1~5的电化学传感器线性范围分别为0.06~0.4、0.06~0.4、0.08~0.5、0.08~0.5、0.1~0.4 μg/L。以mode 1、mode 2和mode 3构建的传感器检出限为0.02 μg/L,以mode 4和mode 5构建的传感器检出限为0.03 μg/L。与另外四种传感器相比,基于mode 3建立的传感器明显拥有更宽的线性范围和更高的灵敏度。证明了以mode 3(1:1)劈裂适配体得到劈裂适配体识别效果最好。
![]() 图 6 基于不同劈裂适配体的传感器与一定浓度AFM1作用后的SWV图和线性图注:A~E:mode 1~5;a~e对应浓度:0.06、0.1、0.2、0.3、0.4 μg/L(A);0.06、0.1、0.2、0.3、0.4 μg/L(B);0.08、0.2、0.3、0.4、0.5 μg/L(C);0.08、0.1、0.2、0.4、0.5 μg/L(D);0.1、0.15、0.2、0.3、0.4 μg/L(E)。Figure 6. SWV and linear plots of sensors based on different splitting adaptors interacting with a certain concentration of AFM1
图 6 基于不同劈裂适配体的传感器与一定浓度AFM1作用后的SWV图和线性图注:A~E:mode 1~5;a~e对应浓度:0.06、0.1、0.2、0.3、0.4 μg/L(A);0.06、0.1、0.2、0.3、0.4 μg/L(B);0.08、0.2、0.3、0.4、0.5 μg/L(C);0.08、0.1、0.2、0.4、0.5 μg/L(D);0.1、0.15、0.2、0.3、0.4 μg/L(E)。Figure 6. SWV and linear plots of sensors based on different splitting adaptors interacting with a certain concentration of AFM12.5 劈裂适配体亲和力的研究
如图7所示,五种劈裂适配体电信号结果拟合效果好,拟合的函数可靠度高,计算得到的Kd值分别是:128.77±41、262.80±53、87.12±29、339.78±101、454.19±112 nmol/L。由结果可知,以mode 3(1:1)得到的劈裂适配体与目标物的亲和力最强,解释了以mode 3模式劈裂适配体为识别元件的传感器灵敏度更高。
2.6 劈裂适配体特异性的研究
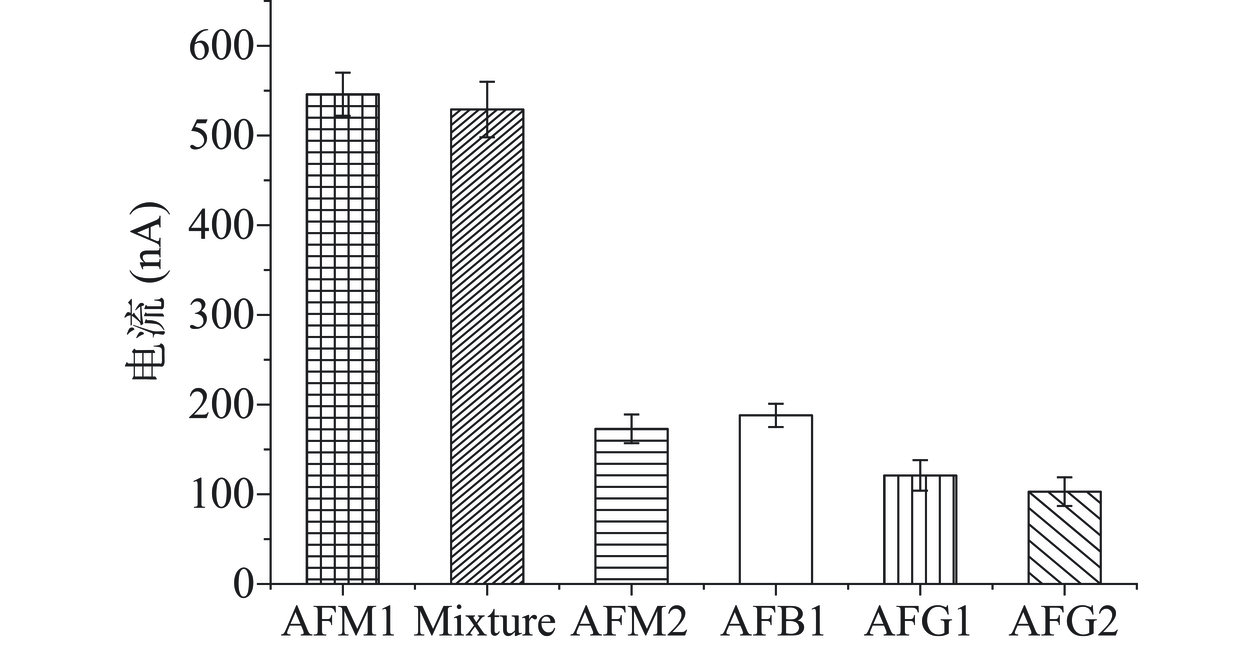
为了进一步说明劈裂适配体对目标物的识别能力,研究劈裂适配体对目标物结构类似物质的响应,分析劈裂适配体的特异性。以mode 3为例,选择相同浓度(0.5 μg/L)结构类似物AFM2、AFB1、AFG1和AFG2,以及包含AFM1(0.5 μg/L)的混合毒素研究劈裂适配体的特异性。如图8可知,与其他对照组毒素相比,AFM1引起的的IFc电信号值最大。而包含AFM1的混合毒素引起的IFc电信号与AFM1引起的信号无明显差异。说明劈裂适配体对AFM1的特异性较好。
2.7 劈裂适配体构象研究
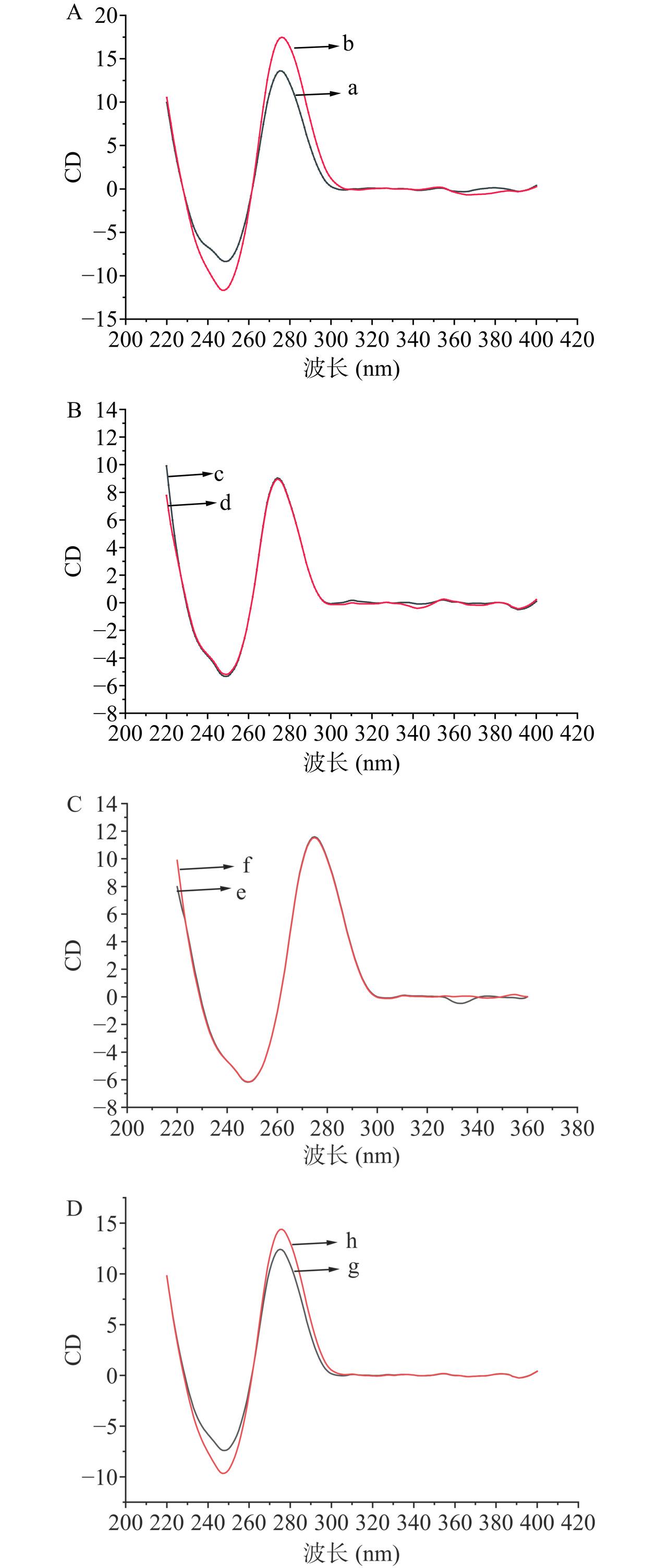
圆二色谱法(CD)是研究适配体构象变化的常见方法,通过适配体与目标物结合前后的构象变化来研究适配体和目标物之间的相互作用。图9分别表示完整的适配体、mode 3模式下单独的S1、S2及S1与S2协同识别目标物前后的圆二色谱图。从图9A可以看出,AFM1适配体的圆二色谱图(曲线a)在248 nm呈现负峰,在275 nm存在正峰,说明AFM1适配体是一种典型的B型DNA[50]。适配体与AFM1作用后(曲线b),圆二色谱图显示出275 nm处正峰强度明显增加,此正峰代表碱基堆叠作用,负峰由248 nm移至249 nm且强度增加,此处负峰代表DNA的螺旋性,说明AFM1与适配体结合会诱导适配体碱基堆积效应增强,同时螺旋增加,适配体构象发生变化。从图9B和9C可知,S1和S2分别与AFM1作用前后圆二色谱图并未发生变化,说明S1和S2与AFM1作用后构象没有发生变化,间接说明S1和S2不能识别AFM1。从图9D可知,S1、S2与AFM1作用后,275 nm处的正峰和248 nm的负峰强度均增加,且负峰发生红移,说明S1和S2与AFM1作用后构象发生变化。此现象与完整适配体结合AFM1后的圆二色谱图相一致,说明S1和S2可以共同识别AFM1。
3. 结论
适配体的构象不稳定会造成适配体传感器的低灵敏度及假阳性问题。本文按照不同比例将适配体进行劈裂,通过电化学方法研究劈裂适配体对目标物AFM1的识别能力及特异性。首先研究了适配体固定环境及识别环境的影响,结果表明,高浓度Na+有利于劈裂适配体在电极表面的固定,150 mmol/L Na+时固定效果最佳且趋于稳定,pH则对固定效果影响不大。识别环境中Na+为300 μmol/L,pH7.4条件下,劈裂适配体与AFM1孵育60 min,识别效果最佳。在此最佳条件下,基于五种劈裂适配体构建的电化学传感器中mode 3的线性范围为0.08~0.5 μg/L,检出限为0.02 μg/L,优于其他四种模式。通过计算五种模式的解离常数可以得知,与其他四种劈裂方式相比,按照1:1方式得到劈裂适配体的亲和力最强。另外,圆二色谱结果也证明了S1和S2可以共同识别AFM1。本研究旨在推动适配体传感器发展,可以对解决适配体传感器灵敏度不高和假阳性问题起到参考作用。
-
图 3 离子强度对适配体固定的影响
注:A:计时库伦曲线,B:不同Na+浓度下电极表面适配体密度图,C:不同Na+浓度下循环伏安图,D:不同Na+浓度下阻抗图;图A中a~d表示:a:MCH修饰电极在不含[Ru(NH3)6]3+,b:MCH修饰电极在含[Ru(NH3)6]3+,c:MCH/S1修饰电极在不含[Ru(NH3)6]3+,d:MCH/S1修饰电极在含[Ru(NH3)6]3+;图C、D中a~e表示:a:250 mmol/L,b:200 mmol/L,c:150 mmol/L,d:100 mmol/L,e:50 mmol/L。
Figure 3. Influence of ionic strength on aptamer immobilization
图 6 基于不同劈裂适配体的传感器与一定浓度AFM1作用后的SWV图和线性图
注:A~E:mode 1~5;a~e对应浓度:0.06、0.1、0.2、0.3、0.4 μg/L(A);0.06、0.1、0.2、0.3、0.4 μg/L(B);0.08、0.2、0.3、0.4、0.5 μg/L(C);0.08、0.1、0.2、0.4、0.5 μg/L(D);0.1、0.15、0.2、0.3、0.4 μg/L(E)。
Figure 6. SWV and linear plots of sensors based on different splitting adaptors interacting with a certain concentration of AFM1
表 1 适配体序列
Table 1 Aptamer sequence
适配体 序列 AFM1适配体 5´-SH-ACTGCTAGAGATTTTCCACAT-Fc-3´[38] mode 1(3:1) 5´-SH-ACTGCTAGAGATTTTC-3 5´-CACAT-Fc-3´ mode 2(2:1) 5´-SH-ACTGCTAGAGATTT-3´ 5´-TCCACAT-Fc-3´ mode 3(1:1) 5´-SH-ACTGCTAGAGA-3´ 5´-TTTCCACAT-Fc-3´ mode 4(1:2) 5´-SH-ACTGCTA-3´ 5´-GAGATTTTCCACAT-Fc-3´ mode 5(1:3) 5´-SH-ACTGC-3´ 5´-TAGAGATTTTCCACAT-Fc-3´ -
[1] CAMPAGNOLLO, FERNANDA BOVO, et al. The occurrence and effect of unit operations for dairy products processing on the fate of aflatoxin M1:A review[J]. Food Control,2016,68:310−329. doi: 10.1016/j.foodcont.2016.04.007
[2] LIU S G, ZHANG D, HE Y, et al. A split aptamer sensing platform for highly sensitive detection of theophylline based on dual-color fluorescence colocalization and single molecule photobleaching[J]. Biosensors & Bioelectronics,2020,166:112461.
[3] AISSA S B, MARS A, CATANANTE G, et al. Design of a redox-active surface for ultrasensitive redox capacitive aptasensing of aflatoxin M1 in milk[J]. Talanta,2019,195:525−532. doi: 10.1016/j.talanta.2018.11.026
[4] WEI X, MA P, IMRAN MAHMOOD K, et al. Screening of a high-affinity aptamer for aflatoxin M1 and development of its colorimetric aptasensor[J]. Journal of Agricultural and Food Chemistry,2023,71(19):7546−7556. doi: 10.1021/acs.jafc.3c01586
[5] LIU R, ZHANG F, SANG Y, et al. Selection and characterization of DNA aptamers for constructing aptamer-AuNPs colorimetric method for detection of AFM1[J]. Foods,2022,11(12):1802. doi: 10.3390/foods11121802
[6] PANDEY A K, RAJPUT Y S, SINGH D, et al. Prediction of shorter oligonucleotide sequences recognizing aflatoxin M1[J]. Biotechnology and Applied Biochemistry,2018,65(3):397−406. doi: 10.1002/bab.1586
[7] AHMADI S F, HOJIATOLESLAMY M, KIANI H, et al. Monitoring of aflatoxin M1 in milk using a novel electrochemical aptasensor based on reduced graphene oxide and gold nanoparticles[J]. Food Chemistry,2022,373:131321. doi: 10.1016/j.foodchem.2021.131321
[8] PANG Y H, GUO L L, SHEN X F, et al. Rolling circle amplified DNAzyme followed with covalent organic frameworks:Cascade signal amplification of electrochemical ELISA for alfatoxin M1 sensing[J]. Electrochimica Acta,2020,341:136055. doi: 10.1016/j.electacta.2020.136055
[9] HE L, SHEN Z, WANG J, et al. Simultaneously responsive microfluidic chip aptasensor for determination of kanamycin, aflatoxin M1, and 17β-estradiol based on magnetic tripartite DNA assembly nanostructure probes[J]. Microchimica Acta,2020,187:1−11. doi: 10.1007/s00604-019-3921-8
[10] JALALIAN S H, RAMEZANI M, DANESH N M, et al. A novel electrochemical aptasensor for detection of aflatoxin M1 based on target-induced immobilization of gold nanoparticles on the surface of electrode[J]. Biosensors and Bioelectronics,2018,117:487−492. doi: 10.1016/j.bios.2018.06.055
[11] SAMEIYAN E, KHOSHBIN Z, LAVAEE P, et al. A bivalent binding aptamer-cDNA on MoS2 nanosheets based fluorescent aptasensor for detection of aflatoxin M1[J]. Talanta,2021,235:122779. doi: 10.1016/j.talanta.2021.122779
[12] FAN Y Y, WEN J, LI J, et al. Structure-switching aptasensors for sensitive detection of ochratoxin A[J]. Luminescence,2023,38(9):1678−1685. doi: 10.1002/bio.4556
[13] YU H, ZHU J, SHEN G, et al. Improving aptamer performance:Key factors and strategies[J]. Microchimica Acta,2023,190(7):255. doi: 10.1007/s00604-023-05836-6
[14] GE G, WANG T, LLU Z, et al. A self-assembled DNA double-crossover-based fluorescent aptasensor for highly sensitivity and selectivity in the simultaneous detection of aflatoxin M1 and aflatoxin B1[J]. Talanta,2023,265:124908. doi: 10.1016/j.talanta.2023.124908
[15] YADAV K, MOOVENDARAN K, DHENADHAYALAN N, et al. From food toxins to biomarkers:Multiplexed detection of aflatoxin B1 and aflatoxin M1 in milk and human serum using PEGylated ternary transition metal sulfides[J]. Sensors and Actuators Reports,2023,5:100156. doi: 10.1016/j.snr.2023.100156
[16] YANG D, HUI Y, LIU Y, et al. Novel dual-recognition electrochemical biosensor for the sensitive detection of AFM1 in milk[J]. Food Chemistry, 2023:137362.
[17] DONG Z, XU X, NI J, et al. Cruciate DNA probes for amplified multiplexed imaging of microRNAs in living cells[J]. Journal of Materials Chemistry B,2023,11(1):204−210. doi: 10.1039/D2TB02027K
[18] BRINZA N D. Beyond the cycle:Investigating the sequencing, binding affinity, and utility of aptamers selected with CE-SELEX[D]. Minnesota:University of Minnesota, 2023.
[19] YANO-OZAWA Y, LOBSIGER N, MUTO Y, et al. Molecular detection using aptamer-modified gold nanoparticles with an immobilized DNA brush for the prevention of non-specific aggregation[J]. RSC advances,2021,11(20):11984−11991. doi: 10.1039/D0RA05149G
[20] SONG J, ZHENG Y, HUANG M, et al. A sequential multidimensional analysis algorithm for aptamer identification based on structure analysis and machine learning[J]. Analytical Chemistry,2019,92(4):3307−3314.
[21] HU J, CHEN X, XU J, et al. A signal-enhanced regenerative electrochemical aptasensor for amyloid-β oligomers based on triple-helix aptamer probe[J]. Journal of the Electrochemical Society,2023,170(1):017507. doi: 10.1149/1945-7111/acb5c5
[22] YE H, YANG Z, KHAN I M, et al. Split aptamer acquisition mechanisms and current application in antibiotics detection:A short review[J]. Critical Reviews in Food Science and Nutrition,2022:1−12.
[23] ALKHAMIS O, CANOURA J, LY P T, et al. Using exonucleases for aptamer characterization, engineering, and sensing[J]. Accounts of Chemical Research,2023:e81-9971.
[24] AHO A, VIRTA P. Assembly of split aptamers by dynamic pH-responsive covalent ligation[J]. Chemical Communications,2023,59(38):5689−5692. doi: 10.1039/D3CC01158E
[25] ZHANG X, DU Y, LIU X, et al. Enhanced anode electrochemiluminescence in split aptamer sensor for kanamycin trace monitoring[J]. Food Chemistry,2023,420:136083. doi: 10.1016/j.foodchem.2023.136083
[26] DEBIAIS M, LELIEVRE A, SMIETANA M, et al. Splitting aptamers and nucleic acid enzymes for the development of advanced biosensors[J]. Nucleic Acids Research,2020,48(7):3400−3422. doi: 10.1093/nar/gkaa132
[27] PARK H, KWON N, PARK G, et al. Fast-response electrochemical biosensor based on a truncated aptamer and MXene heterolayer for West Nile virus detection in human serum[J]. Bioelectrochemistry,2023,154:108540. doi: 10.1016/j.bioelechem.2023.108540
[28] MA Y, GENG F, WANG Y, et al. Novel strategy to improve the sensing performances of split ATP aptamer based fluorescent indicator displacement assay through enhanced molecular recognition[J]. Biosensors and Bioelectronics,2019,134:36−41. doi: 10.1016/j.bios.2019.03.047
[29] YU H, CANOURA J, GUNTUPALLI B, et al. A cooperative-binding split aptamer assay for rapid, specific and ultra-sensitive fluorescence detection of cocaine in saliva[J]. Chemical Science,2017,8(1):131−141. doi: 10.1039/C6SC01833E
[30] AFONIN K A, VIARD M, MARTINS A N, et al. Activation of different split functionalities on re-association of RNA–DNA hybrids[J]. Nature Nanotechnology,2013,8(4):296−304. doi: 10.1038/nnano.2013.44
[31] QI X, YAN X, ZHAO Y, et al. Highly sensitive and specific detection of small molecules using advanced aptasensors based on split aptamers:A review[J]. TrAC Trends in Analytical Chemistry,2020,133:116069. doi: 10.1016/j.trac.2020.116069
[32] CHEN A, YAN M, YANG S. Split aptamers and their applications in sandwich aptasensors[J]. TrAC Trends in Analytical Chemistry,2016,80:581−593. doi: 10.1016/j.trac.2016.04.006
[33] FENG L, LYU Z, OFFENHAUSSER A, et al. Multi-level logic gate operation based on amplified aptasensor performance[J]. Angewandte Chemie International Edition,2015,54(26):7693−7697. doi: 10.1002/anie.201502315
[34] WALTER H K, BAUER J, STEINMEYER J, et al. “DNA origami traffic lights” with a split aptamer sensor for a bicolor fluorescence readout[J]. Nano Letters,2017,17(4):2467−2472. doi: 10.1021/acs.nanolett.7b00159
[35] WEN Y, PEI H, WAN Y, et al. DNA nanostructure-decorated surfaces for enhanced aptamer-target binding and electrochemical cocaine sensors[J]. Analytical Chemistry,2011,83(19):7418−7423. doi: 10.1021/ac201491p
[36] BING T, MEI H, ZHANG N, et al. Exact tailoring of an ATP controlled streptavidin binding aptamer[J]. RSC Advances,2014,4(29):15111−15114. doi: 10.1039/c4ra00714j
[37] ZHANG H, LIU Y, ZHANG K, et al. Single molecule fluorescent colocalization of split aptamers for ultrasensitive detection of biomolecules[J]. Analytical Chemistry,2018,90(15):9315−9321. doi: 10.1021/acs.analchem.8b01916
[38] ZHENG X, PENG R, JIANG X, et al. Fluorescence resonance energy transfer-based DNA nanoprism with a split aptamer for adenosine triphosphate sensing in living cells[J]. Analytical Chemistry,2017,89(20):10941−10947. doi: 10.1021/acs.analchem.7b02763
[39] GUO T, WU C, OFFENHAUSSER A, et al. A novel ratiometric electrochemical biosensor based on a split aptamer for the detection of dopamine with logic gate operations[J]. Physica Status Solidi (a),2020,217(13):1900924. doi: 10.1002/pssa.201900924
[40] DUAN W, WANG X, WANG H, et al. Fluorescent and colorimetric dual-mode aptasensor for thrombin detection based on target-induced conjunction of split aptamer fragments[J]. Talanta,2018,180:76−80. doi: 10.1016/j.talanta.2017.12.033
[41] 曾程. 物理打磨对玻碳电极性能影响的研究[J]. 广州化工,2020(9):73−74. [ZENG C. Study on the effect of physical grinding on the performance of glassy carbon electrode[J]. Guangzhou Chemical Industry,2020(9):73−74.] doi: 10.3969/j.issn.1001-9677.2020.09.026 ZENG C. Study on the effect of physical grinding on the performance of glassy carbon electrode[J]. Guangzhou Chemical Industry, 2020(9): 73−74. doi: 10.3969/j.issn.1001-9677.2020.09.026
[42] STEEL A B, HERNE T M, TARLOV M J. Electrochemical quantitation of DNA immobilized on gold[J]. Analytical Chemistry,1998,70(22):4670−4677. doi: 10.1021/ac980037q
[43] ZHANG J, SONG S, ZHANG L, et al. Sequence-specific detection of femtomolar DNA via a chronocoulometric DNA sensor (CDS):Effects of nanoparticle-mediated amplification and nanoscale control of DNA assembly at electrodes[J]. Journal of the American Chemical Society,2006,128(26):8575−8580. doi: 10.1021/ja061521a
[44] YING G, WANG M, YIY, et al. Construction and application of an electrochemical biosensor based on an endotoxin aptamer[J]. Biotechnology and Applied Biochemistry,2018,65(3):323−327.
[45] HU Z, ZHU R, FIGUEROA-MIRANDA G, et al. Truncated electrochemical aptasensor with enhanced antifouling capability for highly sensitive serotonin detection[J]. Biosensors,2023,13(9):881.
[46] ZIA-UR-REHMAN, SHAH A, MUHAMMAD N, et al. Synthesis, characterization and DNA binding studies of penta- and hexa-coordinated diorganotin(IV) 4-(4-nitrophenyl)piperazine-1-carbodithioates[J]. J Organomet Chem,2009,694(13):1998−2004.
[47] FENG Q, LI N Q, JIANG Y Y. Electrochemical studies of porphyrin interacting with DNA and determination of DNA[J]. Anal Chim Acta,1997,344(1):97−104.
[48] YUE F, LI H, KONG Q, et al. Selection of broad-spectrum aptamer and its application in fabrication of aptasensor for detection of aminoglycoside antibiotics residues in milk[J]. Sensors and Actuators B:Chemical,2022,351:130959.
[49] DONG N, LI Y, MENG S, et al. Tetrahedral DNA nanostructure-based ratiometric electrochemical aptasensor for fumonisin B1:A unity of opposites in binding site and steric hindrance of large-sized DNA for signal amplification[J]. Sensors and Actuators B:Chemical,2023,394:134341.
[50] SUBASTRI A, RAMAMURTHY C H, SUYAVARAN A, et al. Spectroscopic and molecular docking studies on the interaction of troxerutin with DNA[J]. International Journal of Biological Macromolecules,2015,78:122−129. doi: 10.1016/j.ijbiomac.2015.03.036
-
期刊类型引用(5)
1. 王月蓉,赵广河,赵丰丽,覃云斌,陈静,张弘. 脐橙皮水溶性膳食纤维提取工艺优化及其体外益生活性评价. 广西师范大学学报(自然科学版). 2025(01): 101-109 .  百度学术
百度学术
2. 沈康,郭瑞成,徐天旭,王伟华. DEAE-52纤维素柱层析纯化处理对西梅可溶性膳食纤维的影响. 食品与发酵工业. 2024(17): 209-217 .  百度学术
百度学术
3. 卢翠文,欧萍,叶有明,何晓燕,杨东美. 微波辅助酶法提取茶酒糟中可溶性膳食纤维及其抗氧化性能研究. 饲料研究. 2024(20): 75-79 .  百度学术
百度学术
4. 池玉闽,董怡,何强,张文学,向燕,何培君,邓莎,何贵萍,贾利蓉. 油橄榄果肉和核壳中膳食纤维的功能特性分析. 现代食品科技. 2023(05): 157-163 .  百度学术
百度学术
5. 王虎玄,赵天添,王聪,张一凡,朱亚南,孙宏民. 陕北狗头红枣可溶性膳食纤维提取工艺优化及其理化特性与抗氧化活性研究. 陕西科技大学学报. 2022(05): 54-62+99 .  百度学术
百度学术
其他类型引用(2)





 下载:
下载:
 下载:
下载:



