Advance on Transcription Factors Regulating the Synthesis of Secondary Metabolites Mediated by NRPS in Filamentous Fungi
-
摘要: 丝状真菌(Filamentous fungi)产生的次级代谢产物(Secondary Metabolites,SM)广泛应用于生物医药、生物防治及食品等领域。大多数SM的生物合成与非核糖体肽合成酶(Nonribosomal peptide synthetase,NRPS)的催化密切相关,故NRPS的表达调控对SM的生物合成至关重要。为了深入了解丝状真菌NRPS基因簇的表达调控模式,本文就通路特异性和全局性两种转录因子调控丝状真菌NRPS介导SM生成的研究进展进行了综述,以期深化对丝状真菌中转录因子调控NRPS介导的SM生成机制的认识,为丝状真菌天然产物的开发提供参考依据。
-
关键词:
- 转录因子 /
- 丝状真菌 /
- 非核糖体肽合成酶(NRPS) /
- 次级代谢产物 /
- 生物合成
Abstract: Secondary metabolites (SM) produced by filamentous fungi have been extensively applied in biomedicine, biological control, food, and other fields. The biosynthesis of most SMs is closely associated with the catalysis of non-ribosome peptide synthetase (NRPS), therefore the expression regulation of NRPS is crucial for SM biosynthesis. In order to further understand the expression regulation patterns of NRPS gene clusters in filamentous fungi, herein, the research progresses on the transcription factors regulating NRPS-mediated SM production are reviewed, of which both pathway-specific transcription factors and global transcription factors are individually descripted. The summarization is hoped to deepen the comprehension of NRPS-mediated SMs production regulated by transcription factors and provide a reference for development of natural products in filamentous fungi in the future. -
丝状真菌如曲霉、青霉、镰刀菌等是一类产菌丝的非酵母态真菌[1]。自1928年人们从丝状真菌产黄曲霉(Penicillium chrysogenum)中分离到第一例抗生素——青霉素以来,丝状真菌及其合成的SMs便受到了人们的广泛关注,使之成为了重要的天然产物资源库[2]。迄今人们已从丝状真菌中分离到上千种SMs,主要分为聚酮类、非核糖体肽类(non-ribosomal peptide,NRP)、生物碱及萜类等,因其活性广泛多样,现被广泛应用于生物医药、生物防治及食品等领域[3]。
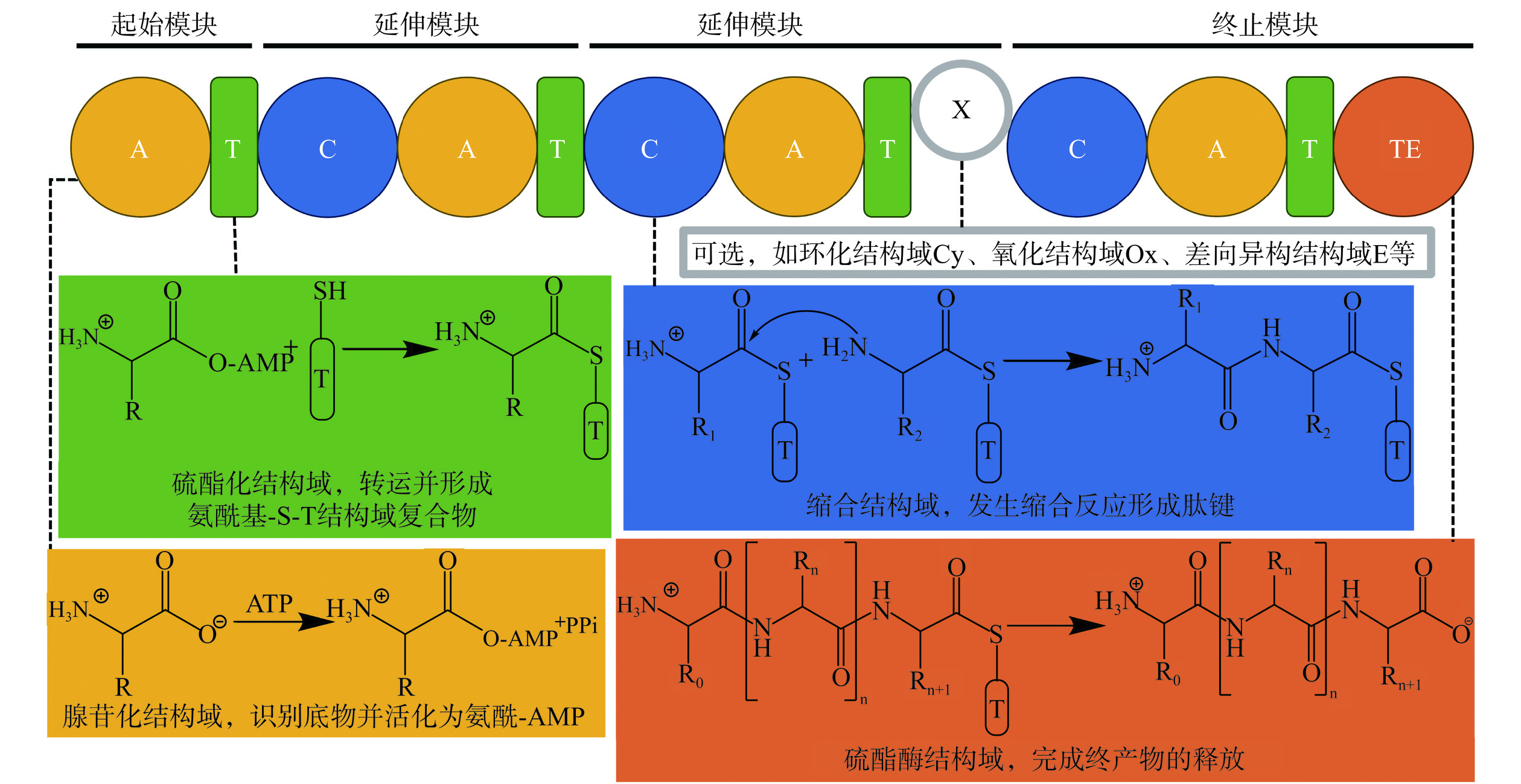
NRP和部分聚酮类等SMs的生物合成离不开NRPS的参与。NRPS是一类具有模块化结构的合酶,其模块由具有特定功能的结构域形成,如图1所示,常见的NRPS结构域包括腺苷化结构域(A)、硫酯化结构域(T)、缩合结构域(C)、硫酯酶结构域(TE),分别负责氨基酸的识别与活化、肽链的运载、肽键的形成以及终产物的释放,此外还有一些选择性的修饰结构域,如环化结构域(Cy)、氧化结构域(Ox)、差向异构结构域(E)等[4]。在功能上,NRPS不依赖核糖体与tRNA,而是通过各结构域协同互作直接催化氨基酸或其他短链羧酸聚合形成具有肽链结构的SM。然而,超过半数的NRPS及其所属的生物合成基因簇(biosynthetic gene cluster,BGC)在实验室条件下均处于沉默或低表达状态,如何激活或高效表达此类BGC成为了SM发掘与应用的研究热点[5−6]。除了NRP,一些NRPSs还可以与聚酮合成酶(polyketide biosynthase,PKS)整合形成PKS-NRPS杂合体,进而催化合成结构更加复杂多样的SMs[7],如抗生素红霉素、免疫抑制剂雷帕霉素等。
转录因子是生物体内调控基因表达的重要因素,它是一类能够调控基因转录效率的DNA结合蛋白,主要通过影响转录初始复合物的形成,进而干预靶基因的转录与表达[8]。参与调控丝状真菌次级代谢的转录因子依据其作用特点可分为两类:一类是通路特异性转录因子,其介导的转录调控具有高度选择性,通常仅影响其自身编码序列所在BGC的转录;另一类是全局转录因子,主要响应光、pH、温度以及营养等外界环境信号,直接或间接调控多个BGCs[9]。丝状真菌与次级代谢相关的转录因子依据结构域划分主要包括6类,分别是具有Zn(II)2Cys6结构的锌簇蛋白、具有Cys2His2结构的锌指蛋白、具有碱性亮氨酸拉链结构的bZIP型转录因子、具有锚蛋白重复序列的bANK型转录因子、具有碱性螺旋-环-螺旋结构的bHLH型、以及MYB型等[9]。本文综述了近年来在SM合成过程中影响NRPS表达的各类转录因子,为丝状真菌天然产物发掘的转录调节及转录因子激活沉默BGCs提供新视野。
1. 通路特异性转录因子调控NRPS相关SMs合成
丝状真菌的通路特异性转录因子介导的转录调控作用直接且针对性强,对其进行遗传操作被认为是发掘新型SM的重要手段[10]。它们可以通过调节NRPS基因簇的表达,来调节NRP及PKS-NRPS所介导的SM的合成。
1.1 通路特异性转录因子调控NRP的生物合成
NRP含有约500种不参与常规蛋白组成的稀有氨基酸[11],范围从三肽到二十五肽,有些NRP还会自身环化或脂化,或以其他通路的次级代谢产物为前体,这使得NRP具有多样的结构和生物活性[12]。表1列举了近年来已被证实的几种由通路特异性转录因子调控的NRP。
表 1 通路特异性转录因子调控NRPs生成Table 1. Pathway-specific transcription factors regulate the generation of NRPsNRP类产物 菌种 受转录因子调控的PKS/NRPS基因簇 转录因子 转录因子类型 参考文献 环孢素 膨大弯颈霉 NRPS:SimA
PKS:SimGSimL bZIP [13] Leucinostatin 淡紫拟青霉 NRPS:LcsA
PKS:LcsB、LcsCLcsF bZIP [14] Apicidin 半截镰刀菌 NRPS:APS1 APS2 bZIP [15] Apicidin F 藤仓镰刀菌 NRPS:APF1 APF2 bANK [16] 镰刀菌素A 禾谷镰刀菌 NRPS:NRPS5、NRPS9 Fgm4 bANK [17] Gramillins 禾谷镰刀菌 NRPS:NRPS8 GRA2 bHLH [18] 环孢素是一种由膨大弯颈霉(Tolypocladium inflatum)合成的环十一肽化合物,是临床上广泛使用的免疫抑制剂。Yang等[13]在研究环孢素生物合成机制时,发现环孢素的合成受到一个由十二个酶编码基因组成的BGC调控,其中,PKS酶SimG、细胞色素SimI和氨基转移酶SimJ共同参与合成环孢素前体化合物Bmt,后者经NRPS酶SimA装配环化生成环孢素。转录组测序和验证实验表明,该BGC受bZIP型通路特异性转录因子SimL控制,过表达SimL可促进该BGC转录,SimL缺失导致该BGC中部分基因表达失活。虽然NRPS酶SimA与PKS酶SimG共同参与环孢素的合成,但二者并未形成PKS-NRPS杂合体,而是分别作用于环孢素合成的不同阶段。
一些脂肽类生物防治剂具有与环孢素相似的合成及调控模式。Wang等[14]对生物防治菌淡紫拟青霉(Purpureocillium lilacinum)分离株PLBJ-1和PLFJ-1进行基因组测序,通过比对分析鉴定出了一个脂肽类抗生素基因簇lcs,其合成Leucinostatins A/B具有显著的抗真菌及抗寄生虫活性,可能成为重要的生物防治剂。系统发育分析显示,NRPS酶LcsA是亮氨酸抑制素A/B合成途径中的核心合成酶,相关合成酶还包括PKS酶LcsB、LcsC、酰基CoA连接酶LcsD等。该合成途径受bZIP型通路特异性转录因子LcsF调控,使用KSTNP-OElcsF质粒过表达LcsF后,LcsA、LcsB、LcsC等关键合成基因表达显著升高,培养基中Leucinostatins A/B滴度水平亦在72 h后升高超过50%。
Jin等[15]通过构建沉默突变体与基因缺失分析,在半截镰刀菌(Fusarium semitectum)中确定了Apicidin,一种具有抗疟原虫活性的环四肽SM,其生物合成途径由NRPS酶APS1在内的八个基因共同组成,并受bANK型通路特异性转录因子APS2调控。后续Niehaus等[16]还在藤仓镰刀菌(F. fujikuroi)基因组中发现了一种新型Apicidin衍生物,并将其命名为Apicidin.F,该化合物与Apicidin具有类似的结构和抗疟原虫活性,且二者的合成均受bANK型通路特异性转录因子APF2调控,该类转录因子的DNA结合区不含亮氨酸拉链结构,但在C末端拥有多个锚蛋白重复序列。
含有类似bANK结构的通路特异性转录因子还包括禾谷镰刀菌(F. graminearum)中fg3_54基因簇编码的Fgm4,其主要调控八肽化合物镰刀菌素A的合成,该化合物直接影响禾谷镰刀菌株对小麦的侵袭性与毒力,其生物合成途径由NRPS5和NRPS9共同参与完成。基因敲除突变株FG-Δfgm4、FG-Δnrps5、FG-Δnrps9与基因簇敲除突变株FG-Δfg3_54均丧失了针对小麦的侵袭力和毒性,各突变株致病性在添加外源性镰刀菌素A后恢复[17]。Bahadoor等[18]在研究禾谷镰刀菌毒性SM时发现了两种双环脂肽类真菌毒素gramillin A与gramillin B,二者均促进禾谷镰刀菌对玉米毒力。通过差异表达分析发现,NRPS8基因簇主导了gramillins类产物的合成,其中GRA1被鉴定为gramillins的生物合成基因簇NRPS8中的一组七模块化的NRPS,而GRA2则为该BGC的转录因子。
1.2 通路特异性转录因子调控PKS-NRPS杂合体介导的SM合成
除直接合成小分子NRPs类外,在丝状真菌中,NRPS的编码基因还可与PKS编码基因整合形成PKS-NRPS杂合体,后者能够催化结构更加复杂多样、功能与生物活性更加广泛的SM[7]。根据产物中含氮杂环的结构差异,PKS-NRPS杂合体催化合成的SM可简单分为含五元含氮杂环的吡咯酮类和含六元含氮杂环的吡啶酮类[19],此外还包括含六元含氧杂环的吡喃酮类物质等,而在此类产物的合成过程中起调控作用的通路特异性转录因子大多是具有Zn(II)2Cys6结构的锌簇蛋白。表2中以这些化合物的结构分类,列举了部分通路特异性转录因子调控PKS-NRPS杂合体介导的SM合成的案例。
表 2 通路特异性转录因子调控PKS-NRPS杂合体介导的SM生成Table 2. Pathway-specific transcription factors regulate the generation of SM mediated by PKS-NRPS化合物类型 SM 菌种 受转录因子调控的PKS/NRPS基因簇 转录因子 转录因子类型 参考文献 吡咯酮 哈茨酸 哈茨木霉 PKS-NRPS:hacA hacI Zn(II)2Cys6 [20] trichosetin 藤仓镰刀菌 PKS-NRPS:PKS-NRPS1(FFUJ_02219) TF22 Zn(II)2Cys6 [21] MCA17-1 熊果木聚孢菌 PKS-NRPS:mca17_A05163 TF17 Zn(II)2Cys6 [22] phomacin D/E Parastagonospora nodorum PKS-NRPS:phmA phmR Zn(II)2Cys6 [23] Chaetoglobosin A 球毛壳菌 PKS-NRPS:CgcheA CgcheR Zn(II)2Cys6 [24] 吡啶酮 leporin B 黄曲霉 PKS-NRPS:lepA(AFLA_066840) lepE Zn(II)2Cys6 [25] Tenellin等 白僵菌 PKS-NRPS:TenS TenR − [26] 吡喃酮 Ustilaginoidin等 罗伯茨绿僵菌 − UpmR Zn(II)2Cys6 [27] pyranterreones 土曲霉 PKS-NRPS:ATEG_00913 ATEG_00919 Zn(II)2Cys6 [28] azasperpyranone 土曲霉 NRPS:ATEG_03630、ATEG_07661
PKS:ATEG_03629、ATEG_07659ATEG_03638(A)、
ATEG_07666(B)、
ATEG_07667(B)MYB [29] 注:“−”表示文中未介绍。 1.2.1 通路特异性转录因子调控吡咯酮类物质合成
哈茨酸(harzianic acids,Has)是一种分离自木霉菌(Trichoderma. spp)的天然广谱抗菌素,具有典型的五元含氮杂环结构。Pang等[20]在一种Has高产的木棉菌突变株(T. guizhouense NJAU 4742,Tgau)的基因组中分离出了Has生物合成基因簇hac-BGC,其中包含PKS-NRPS杂合体基因hacA以及两个推定的通路特异性转录因子hacF与hacI。在过表达菌株TgOEhacF中,Has合成相关基因表达及培养基中Has滴度无明显改变,后二者的水平在过表达菌株TgOEhacI中显著升高。此外,在hacA敲除突变菌株TgauhacA中还观察到hacF与Has转运蛋白hacG表达激活,表明转录因子hacI主要调控Has生物合成,而hacF则是该合成途径的下游调控子并和终产物的转运有关。
Equisetin类化合物是在木贼镰刀菌(F. equiseti)培养物中发现的天然抗菌抗病毒化合物,其能有效抑制革兰阳性菌生长与人类免疫缺陷病毒1型整合酶的活性。Janevska等[21]通过生物信息学分析,在水稻恶苗病菌藤仓镰刀菌基因组中发现了一个Equisetin同源基因簇,该簇在自然状态下保持沉默且缺失N-甲基化酶编码基因eqxD,通过构巢曲霉启动子PoliC过表达推定的通路特异性转录因子TF22与N-甲基化酶eqxD后,成功获得Equisetin与其前体化合物trichosetin,并观察到PKS-NRPS1、DA、ER、TF23与MFS-T等BGC表达显著升高,表明TF22是调节Equisetin生物合成途径的主要转录因子。此外,另一推定转录因子TF23过表达后,仅有转运蛋白MFS-T编码基因表达升高,且TF23与MFS-T均可响应外源trichosetin刺激并上调表达,表明TF23可能与MFS-T协同参与trichosetin的转运与菌株自我保护。
Cheng等[22]在主产物金轮霉素合成抑制的熊果木聚孢菌(Calcarisporium arbuscula)突变株ΔaurA中,应用tef1p启动子过表达通路特异性锌指转录因子TF17成功激活沉默基因簇mca17并得到一种具有抗肝纤维化活性的新型化合物MCA17-1,该化合物由PKS-NRPS杂合体基因mca17_A05163合成,其可在体外抑制转化生长因子-β通路激活,从而拮抗LX-2肝细胞纤维化。
细胞松弛素(Cytochalasan)是一类重要的天然化合物,其可与细胞内肌动蛋白结合并影响其聚合,具有潜在的植物毒力和抗肿瘤活性。Li等[23]在小麦颖枯病病原菌Parastagonospora nodorum基因组中找到一个PKS-NRPS合成基因簇phm,对簇内转录因子phmR过表达后得到两种亮氨酸源性细胞松弛素phomacin D/E及其乙酰基加合物phomacin F,该合成途径主要由PKS-NRPS杂合体phmA主导,敲除phmA后菌株小麦毒力明显下降。Cheng等[24]在球毛壳菌(Chaetomium globosum)中通过对推定的通路特异性转录因子CgcheR进行遗传操作探讨Chaetoglobosin A生物合成的转录调控机制,发现过表达CgcheR使Chaetoglobosin A产量从52 mg/L提高至260 mg/L,而干扰CgcheR则显著抑制了Chaetoglobosin A生物合成基因的转录,其中包括PKS-NRPS杂合体CgcheA、烯酰还原酶CgcheB、P450细胞色素CgcheE等,同时Chaetoglobosin A产量下降。
1.2.2 通路特异性转录因子调控吡啶酮类物质合成
在六元含氮杂环吡啶酮类产物中,2-吡啶酮类产物的生物合成途径及转录调控机制广受关注,该类化合物是一种天然生物碱类物质,其作为药物中间体在抗感染、肿瘤治疗等领域拥有广阔的应用前景。Cary等[25]通过生信分析在丝状真菌黄曲霉(A. flavus)中发现了一个由14个编码基因构成的BGC,过表达推定的通路特异性转录因子lepE后得到已知的2-吡啶酮类物质leporin B与其脱氢前体leporin C。敲除lepE后观察到PKS-NRPS杂合体lepA,单羧酸转运蛋白lepC,短链脱氢酶lepF,P450细胞色素lepH及烯酰还原酶lepG表达下调。在过表达株OE::lepE的基础上构建基因突变株并对产物进行差异分析,结果显示lepA与lepG主要负责关键前体pre-leporin C的合成与释放,其他酶则促进产物进一步成熟。该生物合成途径的终产物为leporin B三聚-铁配合物,这种独特的结构系首次发现,其表现出了与已知化合物leporin A类似的抗昆虫与拒食特性。
Chen等[26]在将白僵菌(Beauveria bassiana)与竞争者绿僵菌共培养时发现,白僵菌基因组中沉默的PKS-NRPS杂合体TenS基因簇被激活,同时产生大量tenellin-2-吡啶酮类代谢产物。体外纯化后鉴定得到七种2-吡啶酮类化合物,其中包括三种已知化合物pyridovericin-N-O-(4-O-methyl-β-d-glucopyranoside)、15-hydroxytenellin和tenellin,以及四种新化合物pyridovericin、1-O-methyl-15-HT、(8Z)-1-O-methyl-15-HT和O-methyltenellin A。通路特异性转录因子TenR随后被鉴定为TenS基因簇的关键调控因子,将其过表达获得的突变株OE::tenR可产生2-吡啶酮类产物farinosone B。此外,甲基糖苷型tenellin衍生物的合成还需簇外基因编码的甲基化酶和糖基化酶参与。
1.2.3 通路特异性转录因子调控吡喃酮类物质合成
吡喃酮因其结构多样性和广谱生物活性而备受关注,Sun等[27]在关于罗伯茨绿僵菌(Metarhizium robertsii)抗细菌机制的研究表明,其基因组中存在一种“超级基因簇”,可以同时调控多种具有不同结构的SM合成,该基因簇又可分为mUstP、mPsoA、mFmaA三个亚基因簇,并同时接受通路特异性转录因子UpmR的调控,其合成产物包括吡喃酮类、萜类和聚酮类化合物,这些产物与绿僵菌的抗菌能力相关,在过表达转录因子UpmR的突变株OE-R中获得了一系列具有联合抗菌作用的天然小分子产物,其中便包括了具有聚酮-氨基酸结构的pseurotins类产物,该类产物在PKS-NRPS亚基因簇缺陷型突变株OE-RΔmPsoA中完全消失,表明其主要由PKS-NRPS杂合体mPsoA合成。
Tang等[28]通过组成型启动子PgpdAt在土曲霉中激活通路特异性转录因子pytR从而唤醒沉默的pyt基因簇,在获得的产物中鉴定出了十种吡喃酮类化合物(pyranterreones)。通过基因敲除构建突变株,并对代谢产物进行差异性分析,结果显示缺失PKS-NRPS杂合体基因pylA的突变株ΔpylA几乎无法检测到吡喃酮产物,表明吡喃酮类产物主链结构由PKS-NRPS杂合体pylA合成,硫酯酶基因缺失突变株ΔpylI代谢产物谱与ΔpylA突变株一致,表明pylA可能仅参与产物中间体形成,而产物中间体的释放则由硫酯酶pylI负责。
通常情况下,丝状真菌次级代谢产物的合成过程中各种酶的编码基因通常位于同一生物合成基因簇内。2020年,Huang等[29]在土曲霉(Aspergillus terreus)中发现,位于两个独立基因簇中的四个核心基因共同负责合成一类具有稠环结构的新型化合物azasperpyranones,而三个通路特异性转录因子ATEG_03638、ATEG_07666、ATEG_07667对这两个彼此之间存在一定空间距离的独立基因簇形成了多层级的转录调控,协同调控该类新型天然化合物的生物合成,揭示了一种全新的SM合成调控机制。这些发现表明,通路特异性转录因子与真菌次级代谢之间或存在更加多样的调控关系。然而,通路特异性转录因子功能多样性及其对真菌NRPS相关SM合成的影响和意义仍需要更多研究探讨与证据支持。
对已推定的通路特异性转录因子进行遗传操作仍是当下发掘并研究丝状真菌NRPS相关SM的重要模式。然而,对于目标产物的高效合成与应用,该方案仍然存在一些困难与问题:首先,此方法不适用于不含转录因子的序列或尚未完成测序的BGCs;其次,虽然通路特异性转录因子针对性较强,但生物合成过程仍难免存在副产物,对于目标产物的合成而言,多基因联合调控可能是进一步提高合成效率的有效手段[30];此外,部分SM具有自毒性,在产物合成水平升高后,菌株出现明显的生长缺陷,进一步降低了生产效率[21],结合生物工程手段调整培养条件,或通过药物及遗传改造诱导菌株产生自身保护机制或可改善这一现象。
2. 全局转录因子对丝状真菌NRPS相关SMs的影响
2.1 全局转录因子与丝状真菌次级代谢
丝状真菌SM的产生是一个复杂的过程,大致包括感受外界环境、调控基因表达、生物合成基因簇的转录和翻译、经过酶促反应使前体分子生成终产物、借助转运蛋白将SM分泌到培养基中等过程,其中的每一个过程都是在数个乃至数十个基因及编码蛋白的共同作用下完成的,众多转录因子的协同调控在丝状真菌次级代谢调节网络中发挥了核心作用[31]。绝大多数情况下,丝状真菌SM合成受到全局转录因子与通路特异性转录因子的协同作用(图2)。
在禾谷镰刀菌中,全局转录因子TRI6除了对多个次级代谢关键基因起到或正向或负向的影响外,还影响着34个转录因子的表达,这34个被影响的转录因子中包含具有Cys2His2锌指结构域的全局转录因子[32]。而在藤仓镰刀菌中,通路特异性转录因子Fum21对伏马菌素BGC的激活作用受到氮调节相关全局转录因子AreA和AreB的共同调控,后者不仅能调控Fum21的表达,还可直接影响伏马菌素合成关键基因Fum1、Fum6和Fum8的水平[33]。此外,在对藤仓镰刀菌产Apicidin F的研究中也证实,氮调节相关转录因子AreB和pH相关转录因子PacC在全氮和酸性pH条件下分别诱导通路特异性转录因子APF2对Apicidin F合成基因簇的激活作用,而APF2编码基因的过度表达在低氮和弱碱条件下也会显著提高产品产量[16]。由此可见,在真菌次级代谢合成调控网络中,通路特异性转录因子在整体上接受全局转录因子的调控并影响SM合成。
在调控模式上,全局转录因子通常涉及极其复杂的真菌代谢调节网络,大量证据表明(表3),其可广泛影响相应调节基因及BGCs的转录,使真菌发生适应性改变,其中便伴随着大量相关次级代谢改变。如pH相关全局转录因子PacC,可通过上调葡萄糖氧化酶编码基因表达促进葡萄糖酸生成,酸化环境,从而维持碳黑曲霉(A. carbonarius)的正常生长及其SM赭曲霉素A(ochratoxin A,OTA)的生物合成[34]。然而,全局转录因子及其同源基因可在多种真菌中发挥类似的调控作用,但其介导的具体效应与之对次级代谢的影响却不尽相同。例如,与碳黑曲霉相反,PacC对禾谷镰刀菌的次级代谢及毒力积累表现为负调控作用,酸性环境下,定向敲除PacC显著增强了其次级代谢与致病性[35]。在碳黑曲霉中,PacC表达能够诱导环境酸化,而在粉红单端孢菌(Trichothecium roseum)中,TrPacC则作为碱化调控因子,主要在酸性环境下表达[36]。此外,饥饿反应相关转录因子RimO、碳代谢相关转录因子CreA、氮代谢相关转录因子AreA、光反应相关转录因子MetR以及甲基转移酶LaeA等全局转录因子均参与调控丝状真菌次级代谢网络[37−41]。一些结构特殊的全局转录因子还可以通过影响表观遗传与转录后修饰等方式调控真菌次级代谢。如转录因子LaeA/LAE1在灰葡萄孢(Botrytis cinerea)及构巢曲霉(A. nidulans)中的次级代谢调节作用便可能与其介导的组蛋白甲基化修饰作用密切相关。LaeA编码蛋白具有类组蛋白甲基化转移酶结构,在敲除突变株Δbclea1与LaeAΔ中均发现抑制性组蛋白甲基化标记H3K9me3水平显著升高并伴随BGC表达抑制[42−43]。而转录因子XdWC2则主要参与类胡罗卜素类物质的转录后修饰,从而调控虾青素的合成[44]。
表 3 已被广泛研究的丝状真菌全局转录因子Table 3. Extensively studied global transcription factors of filamentous fungi对一些真菌病原菌而言,其侵袭性与致病能力与LaeA等全局转录因子介导下的毒性SM合成高度相关。在大麦蓝霉菌(Ramularia collo-cygni)感染大麦植株早期,全局转录因子LaeA以及两种推定合成细胞松弛素类物质的PKS-NRPS杂合体基因Pks1与Hps2在定殖的早期出现峰值表达,且峰值在叶片坏死表现出现之前回落,表明其或与菌株定殖及侵袭性有关[45]。烟曲霉(A. fumigatus)可感染免疫低下患者并导致侵袭性曲霉菌病。Perrin等[46]发现,LaeA缺陷突变株ΔLaeA毒力下降,且全基因组微阵列分析显示,ΔLaeA次级代谢基因表达受到全面抑制,包括推定的铁载体生物合成酶NRPS2/sidE在内的多个NRPS表达显著下调,表明LaeA可能是影响烟曲霉致病力的新靶点。Feng等[47]在研究苹果子囊菌(ascomycete Valsa mali)的毒力时发现,敲除VmLaeA后苹果子囊菌毒力显著下降,病斑长度减少48%。转录与蛋白组学分析表明,敲除VmLaeA后,共有31个BGC表达发生改变,其中13个PKS基因簇、3个NRPS基因簇和1个萜烯合成基因表达下调;另有7个PKS基因簇、5个NRPS基因簇和2个PKS-NRPS杂合基因簇表达上调。此外,Gressler等[53]在致病真菌土曲霉基因组中发现了一个与植物毒力高度相关的PKS-NRPS基因簇,其在碱性环境下激活,但在富葡萄糖环境中被完全抑制,该生物合成途径同时受到pH相关全局转录因子PacC与碳相关转录因子CreA调控。
此外,在真菌生物合成领域,全局转录因子也被用于一些特殊的代谢产物的合成。如在聚β-l-苹果酸(β-l-malic acid)(PMA)的生物合成中,具有类A、C、T结构域的NRPS酶PMA synthetase,后者通过催化唯一前体苹果酸之间的酯键合成,将游离的苹果酸聚合为PMA分子。该生物合成途径受到钙相关全局转录促进因子Crz1,氮代谢相关全局转录抑制因子Gat1与GATA型全局转录因子NsdD共同调节[49−50]。
2.2 全局转录因子在SM生产中的应用
近年来,随着对真菌次级代谢认识的进一步深入,基于全局转录因子的合成优化策略正在成为进一步提高产物合成效率、减少副产物、挖掘新型天然产物的潜在方案。全局转录因子McrA是在构巢曲霉中发现的一种Zn(II)2Cys6型负调控转录因子,在构巢曲霉中敲除mcrA后,与SMs相关的6个转录因子发生上调,表达量提高5倍以上的基因多达623个,其中AN1242作为生产nidulanin A所需的关键NRPS,其表达量上调了510倍,并产生了新的抗生素felinone A和多种SMs。对于多种SMs,mcrAΔ可以在转录促进因子laeA敲除的情况下重新激活真菌次级代谢合成,即mcrAΔ对次级代谢的促进作用会覆盖LaeAΔ的抑制作用[51]。该团队进一步发现,甲基转移酶llmG在mcrA缺失时明显上调,而在敲除mcrA基础上过表达llmG得到的突变株mcrAΔ,gpdA(p)llmG上,其合成活性升高幅度显著高于任意单一策略[52]。对棘白霉素B工业生产菌株A. pachycristatus的研究也进一步证实了ΔApc. mcrA产生大量未知的SM,可以为发现新型天然产物提供新的思路[53]。
赭曲霉素A(ochratoxin A,OTA)是常见的谷物污染物,污染范围广且具有极强的肝肾毒性和致畸作用。大量证据表明,OTA合成受到一个由五基因构成的保守基因簇控制[54],该合成途径受到多个全局转录因子的调控,如抑制LaeA/VeA/VelB蛋白三聚体、bZIP等可有效阻断OTA的合成[55−56]。此外,肉桂醛是一种源自肉桂植物的天然制剂,Wang等[57]在研究肉桂醛抗真菌作用时观察到0.4~1.6 mmol/L浓度的肉桂醛熏蒸处理可以显著抑制赭曲霉生长,同时,肉桂醛的存在还使LaeA、veA、velB等转录调节因子下调,继而导致OTA合成基因簇沉默与OTA合成抑制[57]。同样能够抑制食品内真菌生长,并抑制LaeA表达及相关OTA合成的化合物还包括紫檀烯等二苯乙烯类化合物[58]。最新研究显示,OTA合成依赖全局转录因子Azf1介导的氧化磷酸化及ROS积累,在AnAzf1缺失造成的低ROS水平与氧化磷酸化受损环境下,黑曲霉OTA合成被完全阻断[59],该研究为使用抗氧化剂对抗谷物贮藏过程中OTA污染提供了理论基础。
土曲霉的SM土霉素terrein已被证明具有广泛的生物学和药理学活性,如抗癌、抗炎和抗微生物等。Yao等[30]在研究中发现,TerR是土霉素BGC中的通路特异性转录因子,将其过表达得到的菌株OEterR-6可使土霉素产量提高两倍。全局转录因子StuA在多个物种中参与调控SM的表达,ΔstuA::OEterR菌株使土霉素产量提高至野生型的约30倍,同时能够完全抑制丁内酯类生物合成,后者是土霉素工业生产中的主要副产物之一。在此调节模式中,全局转录因子StuA既可以通过沉默通路特异性转录因子TerR直接抑制SM土霉素的生物合成,又能通过增加该合成途径主要副产物丁内酯类的合成,间接影响土霉素的合成效率。该研究通过对转录因子的改造,极大地提高了土霉素的产量,不仅有助于加深对次级代谢调控机制的理解,而且为丝状真菌SM的产量改良提供了一个范例。
尽管由于介导的调控效应缺乏特异性,且涉及复杂的代谢合成网络,由此带来的产物纯化问题使全局转录因子在既定产物合成领域缺乏实际价值,但探讨全局转录因子对丝状真菌SM的影响,对全面认识和探寻真菌天然产物的生物合成机制具有重要意义,为人们探寻新型天然产物提供了更多可能性,且随着对丝状真菌次级代谢调控网络认识的不断加深,基于全局转录因子的合成策略与生物防治正愈发得到重视。
3. 展望
通过研究转录因子对丝状真菌次级代谢的影响,不难看出,全局转录因子与通路特异性转录因子对丝状真菌SM的合成调控具有不同的特征,全局转录因子对丝状真菌次级代谢的影响宽泛,其在不同条件下的激活或敲除能够调控几十甚至数百个BGCs的表达,所介导的调控效应亦存在明显差异,而通路特异性转录因子介导的调控效应则往往具有更强的针对性,通常仅对编码基因簇产生影响。在丝状真菌次级代谢过程中,全局转录因子与通路特异性转录因子之间存在协同效应,且整体遵循全局转录因子响应外界刺激活化、并通过调控通路特异性转录因子表达影响SM合成的模式,但在二者之间并不存在严格的调控层级,全局转录因子还可通过表观遗传等途径直接影响SM合成。
对于丝状真菌NRPS相关SM这一类极具前景的天然化合物宝库而言,尽管制约因素仍然存在,但基于转录因子调控的遗传改造和理化干预依然取得了重要成果,在可预见的未来,将会有更多更有价值的天然产物以及他们独特的合成机制和精妙的调控模式走进人们的视线。在此策略中,产物合成效率、副产物的控制以及合成菌株的稳定是值得人们关注的问题。全面深入了解全局转录因子与通路特异性转录因子在SM合成中的关系,将有助于工业菌株的优化设计及真菌天然产物的大规模生产。因此,建立在进一步认识次级代谢调控机制之上的多轮遗传改造,是发掘出更多活性天然产物并推动其高效生产的有效策略。
-
表 1 通路特异性转录因子调控NRPs生成
Table 1 Pathway-specific transcription factors regulate the generation of NRPs
NRP类产物 菌种 受转录因子调控的PKS/NRPS基因簇 转录因子 转录因子类型 参考文献 环孢素 膨大弯颈霉 NRPS:SimA
PKS:SimGSimL bZIP [13] Leucinostatin 淡紫拟青霉 NRPS:LcsA
PKS:LcsB、LcsCLcsF bZIP [14] Apicidin 半截镰刀菌 NRPS:APS1 APS2 bZIP [15] Apicidin F 藤仓镰刀菌 NRPS:APF1 APF2 bANK [16] 镰刀菌素A 禾谷镰刀菌 NRPS:NRPS5、NRPS9 Fgm4 bANK [17] Gramillins 禾谷镰刀菌 NRPS:NRPS8 GRA2 bHLH [18] 表 2 通路特异性转录因子调控PKS-NRPS杂合体介导的SM生成
Table 2 Pathway-specific transcription factors regulate the generation of SM mediated by PKS-NRPS
化合物类型 SM 菌种 受转录因子调控的PKS/NRPS基因簇 转录因子 转录因子类型 参考文献 吡咯酮 哈茨酸 哈茨木霉 PKS-NRPS:hacA hacI Zn(II)2Cys6 [20] trichosetin 藤仓镰刀菌 PKS-NRPS:PKS-NRPS1(FFUJ_02219) TF22 Zn(II)2Cys6 [21] MCA17-1 熊果木聚孢菌 PKS-NRPS:mca17_A05163 TF17 Zn(II)2Cys6 [22] phomacin D/E Parastagonospora nodorum PKS-NRPS:phmA phmR Zn(II)2Cys6 [23] Chaetoglobosin A 球毛壳菌 PKS-NRPS:CgcheA CgcheR Zn(II)2Cys6 [24] 吡啶酮 leporin B 黄曲霉 PKS-NRPS:lepA(AFLA_066840) lepE Zn(II)2Cys6 [25] Tenellin等 白僵菌 PKS-NRPS:TenS TenR − [26] 吡喃酮 Ustilaginoidin等 罗伯茨绿僵菌 − UpmR Zn(II)2Cys6 [27] pyranterreones 土曲霉 PKS-NRPS:ATEG_00913 ATEG_00919 Zn(II)2Cys6 [28] azasperpyranone 土曲霉 NRPS:ATEG_03630、ATEG_07661
PKS:ATEG_03629、ATEG_07659ATEG_03638(A)、
ATEG_07666(B)、
ATEG_07667(B)MYB [29] 注:“−”表示文中未介绍。 表 3 已被广泛研究的丝状真菌全局转录因子
Table 3 Extensively studied global transcription factors of filamentous fungi
-
[1] NARANJO-ORTIZ M A, GABALDÓN T. Fungal evolution:Diversity, taxonomy and phylogeny of the Fungi[J]. Fungal Biology Reviews,2019,94:2101−2137.
[2] KELLER N P. Fungal secondary metabolism:regulation, function and drug discovery[J]. Nat Rev Microbiol,2019,17(3):167−180. doi: 10.1038/s41579-018-0121-1
[3] VASSAUX A, MEUNIER L, VANDENBOL M, et al. Nonribosomal peptides in fungal cell factories:From genome mining to optimized heterologous production[J]. Biotechnol Adv,2019,37(8):107449. doi: 10.1016/j.biotechadv.2019.107449
[4] PATEL K D, MACDONALD M R, AHMED S F, et al. Structural advances toward understanding the catalytic activity and conformational dynamics of modular nonribosomal peptide synthetases[J]. Nat Prod Rep,2023,51:1521.
[5] SCHERLACH K, HERTWECK C. Mining and unearthing hidden biosynthetic potential[J]. Nat Commun,2021,12:3864. doi: 10.1038/s41467-021-24133-5
[6] RUTLEDGE P J, CHALLIS G L. Discovery of microbial natural products by activation of silent biosynthetic gene clusters[J]. Nat Rev Microbiol,2015,13(8):509−523. doi: 10.1038/nrmicro3496
[7] MIYANAGA A, KUDO F, EGUCHI T. Protein-protein interactions in polyketide synthase-nonribosomal peptide synthetase hybrid assembly lines[J]. Nat Prod Rep,2018,35(11):1185−1209. doi: 10.1039/C8NP00022K
[8] BRAKHAGE A A. Regulation of fungal secondary metabolism[J]. Nat Rev Microbiol,2013,11(1):21−32. doi: 10.1038/nrmicro2916
[9] LYU H N, LIU H W, KELLER N P, et al. Harnessing diverse transcriptional regulators for natural product discovery in fungi[J]. Nat Prod Rep,2020,37(1):6−16. doi: 10.1039/C8NP00027A
[10] WANG W J, YU Y C, KELLER N P, et al. Presence, mode of action, and application of pathway specific transcription factors in Aspergillus biosynthetic gene clusters[J]. Int J Mol Sci,2021,22(16):8709. doi: 10.3390/ijms22168709
[11] WALSH C T, O'BRIEN R V, KHOSLA C. Nonproteinogenic amino acid building blocks for nonribosomal peptide and hybrid polyketide scaffolds[J]. Angew Chem Int Ed,2013,52:7098−7124. doi: 10.1002/anie.201208344
[12] SÜSSMUTH R D, MAINZ A. Nonribosomal peptide synthesis-principles and prospects[J]. Angew Chem Int Ed,2017,56(14):3770−3821. doi: 10.1002/anie.201609079
[13] YANG X Q, FENG P, YIN Y, et al. Cyclosporine biosynthesis in Tolypocladium inflatum benefits fungal adaptation to the environment[J]. mBio,2018,9(5):e01211−18.
[14] WANG G, LIU Z G, LIN R M, et al. Biosynthesis of antibiotic leucinostatins in bio-control fungus Purpureocillium lilacinum and their inhibition on phytophthora revealed by genome mining[J]. PLoS Pathog,2016,12(7):e1005685. doi: 10.1371/journal.ppat.1005685
[15] JIN J M, LEE S, LEE J, et al. Functional characterization and manipulation of the apicidin biosynthetic pathway in Fusarium semitectum[J]. Mol Microbiol,2010,76(2):456−466. doi: 10.1111/j.1365-2958.2010.07109.x
[16] NIEHAUS E M, JANEVSKA S, VON BARGEN K W, et al. Apicidin F:Characterization and genetic manipulation of a new secondary metabolite gene cluster in the rice pathogen Fusarium fujikuroi[J]. PLoS One,2014,9(7):e103336. doi: 10.1371/journal.pone.0103336
[17] JIA L J, TANG H Y, WANG W Q, et al. A linear nonribosomal octapeptide from Fusarium graminearum facilitates cell-to-cell invasion of wheat[J]. Nat Commun,2019,10(1):922. doi: 10.1038/s41467-019-08726-9
[18] BAHADOOR A, BRAUER E K, BOSNICH W, et al. Gramillin A and B:Cyclic lipopeptides identified as the nonribosomal biosynthetic products of Fusarium graminearum[J]. J Am Chem Soc,2018,140(48):16783−16791. doi: 10.1021/jacs.8b10017
[19] 杨晓钰, 何佳宁, 牛雪梅. 真菌中PKS-NRPS杂合天然产物研究进展[J]. 中国科学:生命科学,2019,49(7):848−864. [YANG X Y, HE J N, NIU X M. Research progress on fungal PKS-NRPS hybrid metabolites[J]. Scientia Sinica (Vitae),2019,49(7):848−864.] doi: 10.1360/SSV-2019-0105 YANG X Y, HE J N, NIU X M. Research progress on fungal PKS-NRPS hybrid metabolites[J]. Scientia Sinica (Vitae), 2019, 49(7): 848−864. doi: 10.1360/SSV-2019-0105
[20] PANG G, SUN T T, DING M Y, et al. Characterization of an exceptional fungal mutant enables the discovery of the specific regulator of a silent PKS-NRPS hybrid biosynthetic pathway[J]. J Agric Food Chem,2022,70(37):11769−11781. doi: 10.1021/acs.jafc.2c03550
[21] JANEVSKA S, ARNDT B, BAUMANN L, et al. Establishment of the inducible tet-on system for the activation of the silent trichosetin gene cluster in Fusarium fujikuroi[J]. Toxins (Basel),2017,9(4):126. doi: 10.3390/toxins9040126
[22] CHENG J T, WANG H M, YU J H, et al. Discovery of a potential liver fibrosis inhibitor from a mushroom endophytic fungus by genome mining of a silent biosynthetic gene cluster[J]. J Agric Food Chem,2021,69(38):11303−11310. doi: 10.1021/acs.jafc.1c03639
[23] LI H, WEI H C, HU J Y, et al. Genomics-driven discovery of phytotoxic cytochalasans involved in the virulence of the wheat pathogen Parastagonospora nodorum[J]. ACS Chem Biol,2020,15(1):226−233. doi: 10.1021/acschembio.9b00791
[24] CHENG M, ZHAO S S, LIU H, et al. Functional analysis of a chaetoglobosin A biosynthetic regulator in Chaetomium globosum[J]. Fungal Biol,2021,125(3):201−210. doi: 10.1016/j.funbio.2020.10.010
[25] CARY J W, UKA V, HAN Z, et al. An Aspergillus flavus secondary metabolic gene cluster containing a hybrid PKS-NRPS is necessary for synthesis of the 2-pyridones, leporins[J]. Fungal Genet Biol,2015,81:88−97. doi: 10.1016/j.fgb.2015.05.010
[26] CHEN B, SUN Y L, LI S Q, et al. Inductive production of the iron-chelating 2-pyridones benefits the producing fungus to compete for diverse niches[J]. mBio,2021,12(6):e327921.
[27] SUN Y L, CHEN B, LI X L, et al. Orchestrated biosynthesis of the secondary metabolite cocktails enables the producing fungus to combat diverse bacteria[J]. mBio,2022,13(5):e180022.
[28] TANG S, ZHANG W, LI Z M, et al. Discovery and characterization of a PKS-NRPS hybrid in Aspergillus terreus by genome mining[J]. J Nat Prod,2020,83(2):473−480. doi: 10.1021/acs.jnatprod.9b01140
[29] HUANG X N, ZHANG W, TANG S, et al. Collaborative biosynthesis of a class of bioactive azaphilones by two separate gene clusters containing four PKS/NRPSs with transcriptional crosstalk in fungi[J]. Angew Chem Int Ed Engl,2020,59(11):4349−4353. doi: 10.1002/anie.201915514
[30] YAO G S, BAI X F, ZHANG B X, et al. Enhanced production of terrein in marine-derived Aspergillus terreus by refactoring both global and pathway-specific transcription factors[J]. Microb Cell Fact,2022,21(1):136. doi: 10.1186/s12934-022-01859-5
[31] HAUTBERGUE T, JAMIN E L, DEBRAUWER L, et al. From genomics to metabolomics, moving toward an integrated strategy for the discovery of fungal secondary metabolites[J]. Nat Prod Rep,2018,35(2):147−173. doi: 10.1039/C7NP00032D
[32] SHOSTAK K, BONNER C, SPROULE A, et al. Activation of biosynthetic gene clusters by the global transcriptional regulator TRI6 in Fusarium graminearum[J]. Mol Microbiol,2020,114(4):664−680. doi: 10.1111/mmi.14575
[33] RÖSLER S M, SIEBER C M, HUMPF H U, et al. Interplay between pathway-specific and global regulation of the fumonisin gene cluster in the rice pathogen Fusarium fujikuroi[J]. Appl Microbiol Biotechnol,2016,100(13):5869−5882. doi: 10.1007/s00253-016-7426-7
[34] BARDA O, MAOR U, SADHASIVAM S, et al. The pH-responsive transcription factor PacC governs pathogenicity and ochratoxin A biosynthesis in Aspergillus carbonarius[J]. Front Microbiol,2020,11:210. doi: 10.3389/fmicb.2020.00210
[35] MERHEJ J, RICHARD-FORGET F, BARREAU C. The pH regulatory factor Pac1 regulates Tri gene expression and trichothecene production in Fusarium graminearum[J]. Fungal Genet Biol,2011,48(3):275−284. doi: 10.1016/j.fgb.2010.11.008
[36] WANG B, HAN Z H, GONG D, et al. The pH signalling transcription factor PacC modulate growth, development, stress response and pathogenicity of Trichothecium roseum[J]. Environ Microbiol,2022,24(3):1608−1621. doi: 10.1111/1462-2920.15943
[37] ZEHETBAUER F, SEIDL A, BERGER H, et al. RimO (SrrB) is required for carbon starvation signaling and production of secondary metabolites in Aspergillus nidulans[J]. Fungal Genet Biol,2022,162:103726. doi: 10.1016/j.fgb.2022.103726
[38] RIES L, ALVES D C P, PEREIRA S L, et al. Aspergillus fumigatus acetate utilization impacts virulence traits and pathogenicity[J]. mBio,2021,12(4):e168221.
[39] HOU R, JIANG C, ZHENG Q, et al. The AreA transcription factor mediates the regulation of deoxynivalenol (DON) synthesis by ammonium and cyclic adenosine monophosphate (cAMP) signalling in Fusarium graminearum[J]. Mol Plant Pathol,2015,16(9):987−999. doi: 10.1111/mpp.12254
[40] YU Z Z, GAO J, IGBALAJOBI O, et al. The sulfur metabolism regulator MetR is a global regulator controlling phytochrome-dependent light responses in Aspergillus nidulans[J]. Sci Bull (Beijing),2021,66(6):592−602. doi: 10.1016/j.scib.2020.11.001
[41] ZHANG G, YAN P, LENG D D, et al. Functional roles of LaeA-like genes in fungal growth, cellulase activity, and secondary metabolism in Pleurotus ostreatus[J]. J Fungi (Basel),2022,8(9):902. doi: 10.3390/jof8090902
[42] WEI Z, SHU D, SUN Q, et al. The BcLAE1 is involved in the regulation of ABA biosynthesis in Botrytis cinerea TB-31[J]. Front Microbiol,2022,13:969499. doi: 10.3389/fmicb.2022.969499
[43] REYES-DOMINGUEZ Y, BOK J W, BERGER H, et al. Heterochromatic marks are associated with the repression of secondary metabolism clusters in Aspergillus nidulans[J]. Mol Microbiol,2010,76(6):1376−1386. doi: 10.1111/j.1365-2958.2010.07051.x
[44] HUANG R L, DING R R, LIU Y, et al. GATA transcription factor WC2 regulates the biosynthesis of astaxanthin in yeast Xanthophyllomyces dendrorhous[J]. Microb Biotechnol,2022,15(10):2578−2593. doi: 10.1111/1751-7915.14115
[45] DUSSART F, DOUGLAS R, SJÖKVIST E, et al. Genome-based discovery of polyketide-derived secondary metabolism pathways in the barley pathogen Ramularia collo-cygni[J]. Mol Plant Microbe Interact,2018,31(9):962−975. doi: 10.1094/MPMI-12-17-0299-R
[46] PERRIN R M, FEDOROVA N D, BOK J W, et al. Transcriptional regulation of chemical diversity in Aspergillus fumigatus by LaeA[J]. PLoS Pathog,2007,3(4):e50. doi: 10.1371/journal.ppat.0030050
[47] FENG Y Q, YIN Z Y, WU Y X, et al. LaeA controls virulence and secondary metabolism in apple canker pathogen Valsa mali[J]. Front Microbiol,2020,11:581203. doi: 10.3389/fmicb.2020.581203
[48] WANG K, CHI Z, LIU G L, et al. A novel PMA synthetase is the key enzyme for polymalate biosynthesis and its gene is regulated by a calcium signaling pathway in Aureobasidium melanogenum ATCC62921[J]. Int J Biol Macromol,2020,156:1053−1063. doi: 10.1016/j.ijbiomac.2019.11.188
[49] QI C Y, JIA S L, LIU G L, et al. Polymalate (PMA) biosynthesis and its molecular regulation in Aureobasidium spp.[J]. Int J Biol Macromol,2021,174:512−518. doi: 10.1016/j.ijbiomac.2021.02.008
[50] OAKLEY C E, AHUJA M, SUN W W, et al. Discovery of McrA, a master regulator of Aspergillus secondary metabolism[J]. Mol Microbiol,2017,103(2):347−365. doi: 10.1111/mmi.13562
[51] GRAU M F, ENTWISTLE R, OAKLEY C E, et al. Overexpression of an LaeA-like methyltransferase upregulates secondary metabolite production in Aspergillus nidulans[J]. ACS Chem Biol,2019,14(7):1643−1651. doi: 10.1021/acschembio.9b00380
[52] PERLATTI B, LAN N, JIANG Y Y, et al. Identification of secondary metabolites from Aspergillus pachycristatus by untargeted UPLC-ESI-HRMS/MS and genome mining[J]. Molecules,2020,25(4):913. doi: 10.3390/molecules25040913
[53] GRESSLER M, ZAEHLE C, SCHERLACH K, et al. Multifactorial induction of an orphan PKS-NRPS gene cluster in Aspergillus terreus[J]. Chem Biol,2011,18(2):198−209. doi: 10.1016/j.chembiol.2010.12.011
[54] GIL-SERNA J, GARCÍA-DÍAZ M, GONZÁLEZ-JAÉN M T, et al. Description of an orthologous cluster of ochratoxin A biosynthetic genes in Aspergillus and Penicillium species. A comparative analysis[J]. Int J Food Microbiol,2018,268:35−43. doi: 10.1016/j.ijfoodmicro.2017.12.028
[55] WANG G, ZHANG H Y, WANG Y L, et al. Requirement of LaeA, VeA, and VelB on asexual development, ochratoxin A biosynthesis, and fungal virulence in Aspergillus ochraceus[J]. Front Microbiol,2019,10:2759. doi: 10.3389/fmicb.2019.02759
[56] GERIN D, GARRAPA F, BALLESTER A R, et al. Functional role of Aspergillus carbonarius AcOTAbZIP Gene, a bZIP transcription factor within the OTA gene cluster[J]. Toxins,2021,13(2):111. doi: 10.3390/toxins13020111
[57] WANG L M, JIN J, LIU X, et al. Effect of cinnamaldehyde on morphological alterations of Aspergillus ochraceus and expression of key genes involved in ochratoxin A biosynthesis[J]. Toxins (Basel),2018,10(9):340. doi: 10.3390/toxins10090340
[58] CAI X Y, QI J R, XU Z, et al. Three stilbenes make difference to the antifungal effects on ochratoxin A and its precursor production of Aspergillus carbonarius[J]. Food Microbiol,2022,103:103967. doi: 10.1016/j.fm.2021.103967
[59] WEI S, HU C J, ZHANG Y G, et al. AnAzf1 acts as a positive regulator of ochratoxin A biosynthesis in Aspergillus niger[J]. Appl Microbiol Biotechnol,2023,107(7-8):2501−2514. doi: 10.1007/s00253-023-12404-8
-
期刊类型引用(0)
其他类型引用(1)





 下载:
下载:

 下载:
下载:



