Research Progress of Sensors Technology Based on DNA Walker in Food Safety Detection
-
摘要: 随着DNA纳米技术的快速发展,各种结构和功能的DNA分子被用于构建具有动态行为的可编程纳米机器。其中,DNA步行器因其能够借助驱动力在预定轨道上自主、逐步移动以产生级联信号放大的优势,成为了生物传感、生物成像以及药物递送等领域日益增长的研究热点,在食品污染物快速超灵敏检测方面展现出巨大的应用潜力。本综述首先简要介绍了DNA步行器的基本原理,并着重阐述了提升DNA步行器行走效率的策略,然后总结了其在食品安全检测中的应用,最后对DNA步行器未来研究方向进行了展望,为推动其实际应用提供了理论指导。Abstract: With the rapid development of DNA nanotechnology, DNA molecules with various structures and functions are used to construct programmable nanomachines with dynamic behaviors. Thereinto, DNA walker possesses the advantages of the cascade signal amplification generated by the autonomous movement along the predetermined track by driving force, which is widely applied in the biosensing, biological imaging, and drug delivery fields, thus showing the great application potential for rapid and ultrasensitive detection of contaminants in food. This review first briefly introduces the basic principles of DNA walker, and describes the strategies for improving the efficiency of DNA walker in detail, then summarizes its applications in the food safety detection. Finally, the future research directions of DNA walkers are prospected to provide theoretical guidance for its real applications.
-
Keywords:
- food safety /
- DNA walker /
- signal amplification /
- nucleic acid aptamers /
- biosensors
-
食品安全是关系国计民生的重大战略问题。食品安全检测具有时效性强、样品基质复杂、有害物质残留痕量等特点,高效可靠的污染物检测和实时连续监测过程对于食品生产可控、食品安全风险评估至关重要。目前,采用液相色谱-质谱联用、高效液相色谱法、气相色谱等方法虽能获得较高的灵敏度和准确性,但需要昂贵的专用仪器、专业的操作以及复杂的样品预处理过程,很难满足多样化食品安全检测的实际需求[1]。近年来,以酶[2]、抗体[3]或核酸适配体[4]为识别探针的生物传感器因其高灵敏度、操作简便、价格低廉以及能够现场实时监测而受到研究者们的广泛关注。核酸适配体是一类对靶标具有高亲和力和特异性的单链寡核苷酸。相比于抗体,它具有可体外人工合成、通量高、成本低、纯度高、结构稳定性高等优势,并且可以通过体外识别技术来筛选从小分子到蛋白质再到病原体的靶标[5]。然而,适配体有限的亲和力以及复杂的检测环境使食品中痕量甚至超痕量污染物的快速检测仍然面临挑战,需进一步引入信号放大技术以提高灵敏度。
DNA以其精确的碱基配对、序列可编程性、结构可控性、易合成和修饰等特性,被认为是构建人工分子机器的理想元件[6]。受天然马达蛋白可将货物沿着微管引导到细胞边缘这一现象的启发,一种能够在预设的纳米级轨道上自主行进进而产生放大信号的动态纳米机器——DNA步行器应运而生[7]。因其结构和功能的可编程性,可以设计分子步行行为以对不同的生物目标做出特定反应。DNA步行器主要包括行走链、轨道和驱动力三个组件,驱动力的加入会打破行走链的初始平衡,使其沿着轨道持续性机械性位移,通过消耗燃料分子,使反应平衡恢复。在行走过程中,平衡不断被破坏和恢复,直至反应结束[8−9]。与其他核酸等温扩增技术相比,利用适配体识别靶标这一过程来激活DNA步行器,可以在轨道界面上实现靶标识别和信号放大检测的集成以强化传质,并避免了均相反应带来的高背景信号干扰[10]。因此,将适配体与DNA步行器相结合构建生物传感器在食品污染物检测领域具有更大的应用潜力。
本综述首先基于不同的驱动力介绍了DNA步行器的基本原理,然后针对目前DNA步行器面临的挑战,分别从传感界面微环境调控、DNA行走链优化设计以及轨道纳米结构可控组装三个方面着重论述了提升DNA步行器行走效率的策略,并以食源性微生物、生物毒素以及抗生素为代表总结了其在食品安全检测中应用,最后对其未来发展提出了展望。
1. DNA步行器基本原理
1.1 链置换反应
立足点介导的链置换反应(toehold-mediated strand displacement,TSDR)基于Watson-Crick碱基配对原理,通过在寡核苷酸链间形成和断裂氢键来促进动力学和热力学平衡。催化发夹组装(catalytic hairpin assembly,CHA)可通过DNA碱基对形成时释放的自由能驱动DNA步行器[11−13]。Li等[13]将链霉亲和素(streptavidin,SA)与生物素修饰的DNA链结合以构建多腿DNA步行器,SA的末端保护作用可避免行走链被核酸外切酶I消化。随后,在CHA的驱动下开启行走过程将辣根过氧化物酶引入反应体系,从而实现SA的化学发光传感。然而,CHA扩增策略需要消耗大量发夹DNA,组装组分具有过多的互补碱基序列,这增加了生物分析设计的复杂性,并且容易发生非特异性结合[14]。燃料链驱动的靶标循环策略是通过额外释放分子的熵增强而不是靠碱基对形成的焓增加来驱动的,其采用多链复合结构而不是DNA发夹结构,优点在于序列设计简单灵活,可以适应不同的分析要求,有利于降低背景响应[15−17]。Yang等[16]开发了由适配体/靶标结合诱导的邻近位移DNA步行器来检测凝血酶。同时识别靶标的两个适配体相互靠近,使行走链局部浓度增加,通过TSDR将阻断链从DNA三链体轨道中置换出来,并暴露出预先锁定的toehold结构域,进而再次引发TSDR将亚甲基蓝(methylene blue,MB)标记的DNA燃料链引入电极表面以产生信号响应,同时释放的行走链将继续重复行走动作以实现信号累积。
1.2 酶催化水解
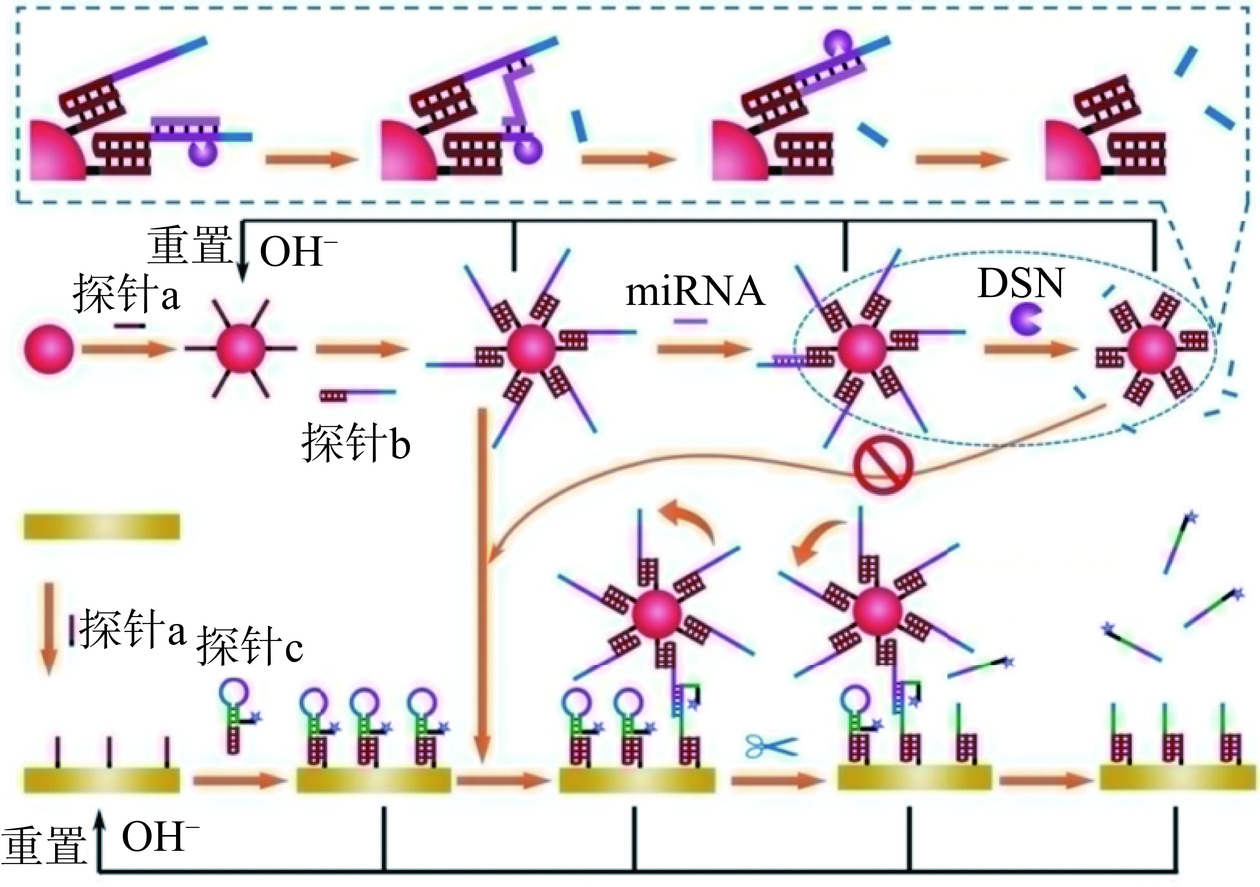
核酸酶(内切酶[18−19]、外切酶[20−22]、DNA酶[23])作用于DNA磷酸主链,利用其通用或特异性的识别和切割位点将底物链或燃料链中共价键裂解产生的自由能来驱动DNA步行器,其操作效率比基于核酸链置换反应驱动的DNA步行器高3个数量级。如图1所示,Miao等[18]制备并整合了由双链特异性核酸酶(duplex-specific nuclease,DSN)和切刻内切酶(nicking endonuclease,NEase)驱动的两个独立的行走和滚动纳米机器。靶标miRNA会触发DSN介导的行走反应以裂解DNA三链体探针,导致后续由NEase介导的滚动杂交过程被抑制,使MB的电化学信号恢复。Cheng等[19]设计了一种瓣状核酸内切酶1(flap endonuclease 1,FEN1)辅助的DNA步行器,靶标DNA在捕获探针的牵引下与轨道杂交,以形成可被FEN1识别的三碱基重叠结构,进而切割底物链释放带有荧光标记基团的寡核苷酸片段,产生荧光信号。相比于内切酶,核酸外切酶从DNA链末端切割磷酸二酯键,具有更快的水解速率。同时,它不需要特异性识别位点,这使得DNA探针的设计更加灵活。Qu等[20]基于“烧桥机制”,利用核酸外切酶III(exonuclease III,Exo III)识别DNA杂交轨道,逐步催化去除其钝3′末端的单核苷酸以释放行走链,而行走链尾部的寡T序列可防止其自身被消化。但外切酶对单/双链DNA均存在切割能力导致背景信号较高。此外,相比于生物酶,DNA酶具有易于合成且稳定性好的特性,且其驱动的行走过程无需外部添加燃料链。Yin等[23]以适配体作为阻断链对行走链中的DNA酶进行封锁,利用适配体识别靶标可以激活DNA酶,在Zn2+的辅助下切割底物链以释放荧光标记底物链片段,荧光信号因远离淬灭剂而得以恢复。
1.3 环境刺激响应
由pH变化[24−25]、光[26−28]、小分子[29−30]等环境刺激引起的构象变化也可以产生能量来驱动DNA步行器,并能够实现行走链与轨道之间关联和解离行为的调控。Cui等[25]开发了一种由DNA酶前体和pH敏感的DNA双链组成的活性可控的智能DNA步行器,DNA酶前体保持无活性的三链杂交状态。溶酶体内的酸性微环境可诱导DNA链的构象从线性转变成三重态并释放其互补链,随后将DNA酶前体上的锁定链置换下来以将其激活,从而实现活细胞中多种金属离子的可视化和定量分析。Zhang等[26]将“光拉链”封闭的DNA酶和末端修饰荧光淬灭剂的底物链共固定在多波长发射的上转换纳米颗粒(upconversion nanoparticles,UCNPs)上,构建了一种内参比型DNA步行器。DNA酶活性区域被含有光裂解基团的DNA链阻断,紫外光照射后可触发与靶miRNA的置换反应,释放出激活的DNA酶行走链以启动行走过程,使近红外光激发下UCNPs上的荧光团在580 nm处的发射光恢复,实现了miRNA活细胞内高灵敏成像。同时,UCNPs本身在658 nm处的发射光可以作为内标提高检测精度。此外,基于光敏剂芘分子能促进二硫键裂解这一特性,可将芘分子作为类DNA酶行走链来设计光控DNA步行器[27]。然而,借助光、金属离子等外源驱动力增加了检测的复杂性和不准确性。对此,Ma等[29]以内源性三磷酸腺苷为燃料分子,通过在DNA轨道上偶联其适配体来实现DNA步行器在活细胞内部自主运行,而无需任何添加剂辅助。
2. 提升DNA步行器行走效率的策略
目前,尽管基于DNA步行器信号放大策略的传感器的研究已经进入了较为成熟的发展阶段,但仍然面临一系列挑战,主要体现在以下几个方面:首先,对于单腿DNA步行器,单一识别位点降低了目标物与DNA行走链之间的碰撞效率,同时它依靠在均相溶液中与轨道链的随机碰撞行走,使得传感界面附近有效浓度低,在行走过程中容易脱离轨道而展现出有限的扩增效率[20];其次,DNA步行器的运动轨迹通常是将DNA分子随机组装在电极表面来制备的,这使得底物链处于分布不均的状态。无序的界面环境会干扰行走链,使行走过程缺乏方向性和可控性,极大地限制了DNA步行器的行走范围和信号放大能力[31−33];此外,伴随着底物链局部浓度的增加,容易导致其发生坍塌和纠缠。受空间位阻影响,DNA探针对目标物的捕获能力和杂交效率会大大降低[34−35]。基于上述讨论,DNA步行器的信号累积和运行效率很大程度上取决于行走链和轨道的负载量以及两者在传感界面附近的杂交效率。因此,为最大限度地发挥其信号放大作用,接下来将从传感界面微环境调控、DNA行走链优化设计以及轨道纳米结构可控组装三个方面来阐述提升DNA步行器行走效率的策略。
2.1 传感界面微环境调控
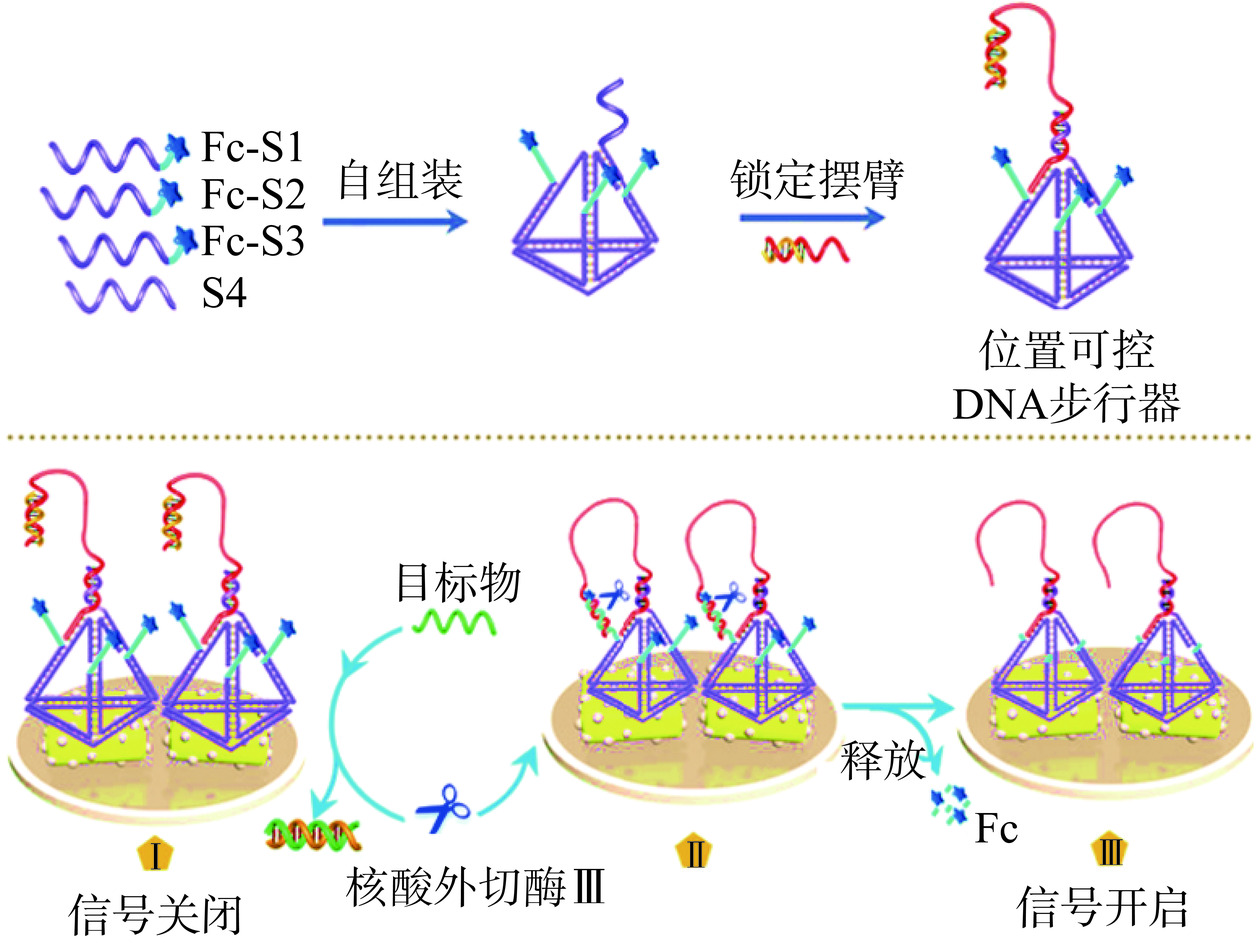
研究表明,相比于行走过程,行走链和轨道在传感界面上的动态识别过程是主要限速步骤,其效率与DNA底物链的构象和分布情况密切相关[36]。聚腺嘌呤(polyA)被用来修饰DNA探针以精确控制其在金纳米颗粒(gold nanoparticles,AuNPs)表面的密度并使其构象呈直立状态,从而提高杂交效率[37−38]。Yang等[37]通过优化polyA长度提高了DNA酶行走链活性,相比于巯基修饰方法,其检测限从98.6 pM降至8.9 pM。另外,行走链相对于轨道的位置也是决定识别效率的关键因素[31,39−40]。Oishi等[39]在轨道上修饰带正电氨基的聚乙二醇,借助其对DNA分子的静电相互作用阻止游离行走链向溶液中扩散,显著增加其局部浓度以强化邻近效应,从而加速TSDR反应,提高行走效率。除了调控传感界面的电荷性质外,如图2所示,Liao等[34]在四面体核酸框架中共定位行走链和底物链以形成反应单元。与随机固定行走链的DNA步行器相比,凭借对其空间分布的高可控性,反应效率提高了1.5倍,检测限低至4.92 fM。
此外,基于邻近效应,Lu等[28]将熵驱动DNA探针与DNA燃料链共同限域在空心硫化铜纳米颗粒(hollow copper sulfide nanoparticles,HCuSNPs)上,燃料链可以通过近红外光照射诱导HCuSNPs产生的光热效应有效释放,进而触发TSDR反应诱导靶标释放继续行走过程。与额外添加带有分子信标的燃料链相比,其灵敏度提高了两个数量级。Zhao等[17]设计了具有突出尾部序列的DNA燃料链,并将其作为行走链类比物触发邻近链间位移效应以激活相邻的DNA探针,诱导燃料链在电极上自推进。结果显示检测限最低为0.1 fM,这比传统由燃料链驱动的目标回收策略低两个数量级。此外,通过改变燃料链尾部序列长度,可以调整其电化学性能以满足不同分析需求。
值得注意的是,上述通过共价或配位作用将DNA链固定在刚性表面上来构筑轨道的方式易造成行走范围受限。受细胞膜上信号传导蛋白的启发,脂质双分子层提供的柔性流动界面能够赋予胆固醇功能化的DNA底物链以高迁移率,降低其扩散的空间位阻,从而表现出高的行走效率,同时也避免了DNA链和硬表面之间繁琐而耗时的连接过程,提高了组装效率[40−41]。然而,DNA轨道只能在单个脂质双层涂覆的纳米颗粒内扩散。同时,脂质双层在纳米颗粒上的涂覆增加了DNA步行器构建的难度。对此,Zhai等[42]提出了一种基于DNA胶束的步行器。胶束的动态结构赋予了行走链不受约束接近轨道的能力,使DNA组分扩散屏障完全去除,实现了粒子内和粒子间的行走运动以提高反应效率,从而显著缩短了行走时间,对miRNA-10b的检测限低至87 pM。
2.2 DNA行走链优化设计
与仅有单一识别位点的单腿DNA步行器相比,多识别位点强化了行走链与轨道链之间的有效碰撞与结合力,使多腿DNA步行器具有更好的行走稳定性与反应动力学。Zhou等[43]利用DNA自组装设计了四面体DNA步行器,其刚性和非平面多腿结构可以加速在多个方向上的迁移,因而具有较高的碰撞效率,其初始速率比游离双腿DNA步行器快了4.54倍。另外,有研究表明,通过引入AuNPs、磁珠以及金属有机骨架等三维(three-dimensional,3D)纳米材料在富集核酸链的同时,可以将步行器的运动模式从逐步行走转变为高速滚动,从而产生更快和更强的信号响应[44−46]。
尽管如此,3D纳米材料带来的高空间位阻仍会使行走速度受限。对此,Li等[47]研究发现通过调节纳米材料尺寸和DNA酶悬臂长度,赋予了DNA滚动纳米机器高驱动力和足够的反应空间,使行走过程更加高效可控。在此基础上,Zhang等[48]利用DNA四面体将DNA滚动纳米机器固定在电极表面,通过调节行走链密度和DNA四面体柔性结构域长度以降低行走反应的位阻,同时通过限制反应空间增加DNA组分局部浓度,从而进一步提高灵敏度。然而,固定型DNA步行器需要设计具有两种不同DNA链的复杂轨道,且行走链自由度较低,行走范围易受其长度的限制。Chang等[49]设计了由三个部分互补的寡核苷酸组成的三向连接DNA,并将识别区域预锁定在发夹结构中。目标物会触发发夹结构连续打开以形成具有多重识别区域的环状DNA行走链,它能在电极表面的整个范围内行走而不受限于腿长,同时也避免了行走链在纳米材料上复杂且耗时的固定化过程,实现了对miRNA-182-5p的超灵敏和快速检测,检测限为31.13 aM。
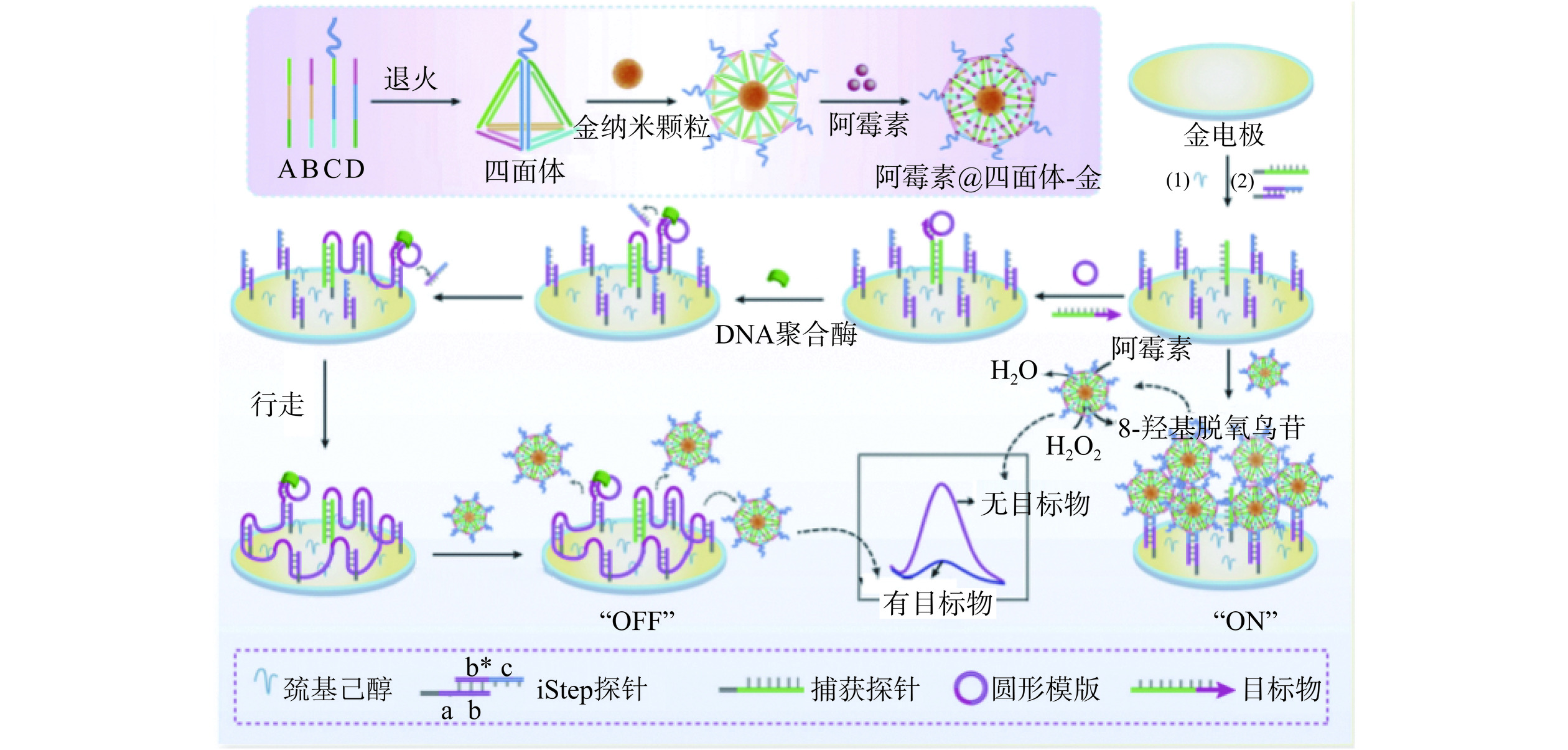
此外,利用靶标识别触发CHA[50−51]、滚环扩增(rolling circle amplification,RCA)[52−53]、指数扩增[54−55]等核酸等温扩增反应也能够合成多腿DNA步行器以实现级联信号放大。如图3所示,Li等[53]基于靶标识别触发RCA构建了多功能区域线性DNA步行器,通过phi29聚合酶介导的链置换聚合反应来驱动行走链在不同轨道链间运动。这能够克服传统单腿DNA步行器功能区域少和多腿DNA步行器高空间位阻的不足,同时随着扩增反应的进行,行走链与预定路径的结合力也逐渐增加,有效避免脱轨,从而拥有更大的行走范围和更高的放大效率。
2.3 轨道纳米结构可控组装
DNA轨道是引导DNA步行器运动的关键组成部分,DNA步行器的运动范围和效率取决于行走链和预设轨迹之间的动态相互作用。为了使两者更好地结合,需要构建一种有序且等距的DNA轨道以保证底物链在传感界面上的精确定位和均匀分布,进而赋予DNA步行器更多的行走步数[56−57]。Lü等[56]利用杂交链式反应(hybridization chain reaction,HCR)生成周期性双链DNA串联体以构筑有序轨道界面,轨迹步长与双腿DNA步行器中的酶切识别位点间距相匹配,从而提高了DNA行走效率,降低脱轨风险。
此外,如2.1节所述,通过引入刚性DNA纳米框架可以克服传统轨道的坍塌和纠缠,并辅助行走链和底物链的锚定并保证两者定量有序排列,从而有效提高其行走反应动力学[31,34]。然而,在整个传感界面上,每个刚性DNA单元之间的相互独立性使其空间距离难以调控,无法保证所有底物链有序排列,还是会造成长度有限的行走链与预设轨道分离,从而中断行走过程,降低结合效率。对此,可以设计带有突出末端的DNA单元,使其能够通过可控的定向自组装过程得到具有有序晶格结构的DNA轨道阵列[32−33]。Pan等[32]制备了由DNA立方体连接形成的线框轨道,保证了所有底物链均匀排列,从而加强了行走链与轨道的动态相互作用,使其能够以高可控性、方向性和快速反应速度移动。与基于单一DNA立方体框架的步行器相比,该传感器对DNA腺嘌呤甲基化酶具有较低的检测限(2.3×10−8 U/mL)和较宽的线性检测范围(10−7~10−1 U/mL)。
基于纳米颗粒的3D轨道使DNA步行器拥有广阔的移动空间和更高的行走连续性,但传统的制备方法容易造成底物链在传感界面上的无序分布,使其失去了1D轨道的高方向性和可控性。尽管在底物链中引入polyA能有效改善上述问题,但大尺寸的颗粒难以固定在DNA修饰的电极表面上,从而限制了其在电化学传感领域的应用。基于此,通过运用3D DNA纳米框架,使其通过可控的自组装过程形成类似于1D线性构型的纵向DNA立体管状结构[58−60]。Jiang等[58]以RCA生成的长链作为DNA纳米管的骨架,在其上组装了许多拥有3个特定杂交结构域的重复框架单元,并在一端设计含有行走链的始发单元。凭借对层内和层间底物链距离的精确控制,该3D轨道为DNA步行器提供了明确的行走方向和均匀的步伐以保证其有序运行。
3. DNA步行器在食品安全检测中的应用
表1总结了DNA步行器在食品安全检测中的应用,下文将以食源性致病菌、生物毒素以及抗生素三种典型的污染物为例来进行介绍。
表 1 DNA步行器在食品安全检测中的应用Table 1. Application of DNA walker in food safety detection危害因子 检测方法 线性范围 检出限 时间(min) 驱动力 参考文献 食源性致病菌 金黄色葡萄球菌 金纳米颗粒比色 1~105 CFU/mL 1 CFU/mL 15 Exo III [61] 鼠伤寒沙门氏菌 荧光法 100~108 CFU/mL 22.8 CFU/mL 50 CHA [62] 金黄色葡萄球菌 电化学 60~6×107 CFU/mL 9 CFU/mL 100 Exo III [63] 鼠伤寒沙门氏菌 荧光法 − 5 CFU/mL 50 NEase [64] 生物毒素 赭曲霉毒素 电化学发光法 10~108 fg/mL. 3.19 fg/mL 40 EDC [35] 玉米赤霉烯酮 催化显色法 0.02~80 µg/L 0.1 µg/L 120 Exo III [65] 玉米赤霉烯酮 电化学 0.0001~103 ng/mL 4.57×10−6 ng/mL 180 NEase [66] 黄曲霉毒素M1 电化学发光法 0.03~3×105 pg/mL 0.009 pg/mL 240 NEase [67] 棒曲霉素 电化学 0.05~500 pg/mL 4.14×10−5 ng/mL 180 NEase [68] 赭曲霉毒素 电化学发光法 0.01~100 pg/mL 4.7 fg/mL 30 Exo III & EDC [46] 抗生素 恩诺沙星 电化学 5×10−3~10 pg/mL 1.02×10−7 ng/mL 190 DNA酶 [69] 氨苄青霉素 电化学 1~104 pM 0.76 pM 210 Exo III [70] 卡那霉素 电化学 0.01~1000 ng/mL 8.4 fg/mL 190 NEase [71] 磺胺 电化学 10~106 pg/mL 3 pg/mL 90 DNA酶 [72] 卡那霉素 光电化学法 10~106 fg/mL 0.53 fg/mL 200 DNA酶 [73] 卡那霉素 电化学 10~106 fM 0.02 fM 180 CHA [74] 3.1 食源性致病菌
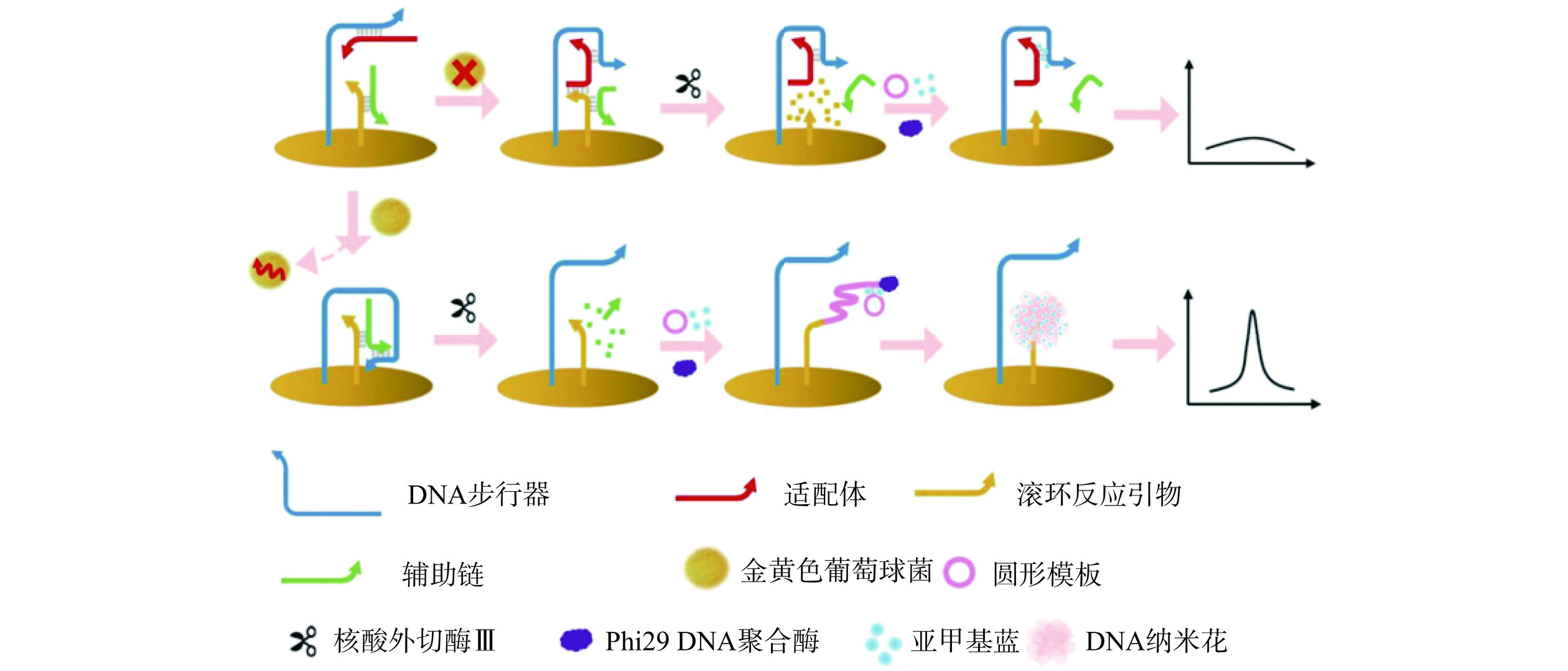
食源性致病菌主要包括大肠杆菌、金黄色葡萄球菌、沙门氏菌等,通常在食品生产、加工、贮存或运输和销售过程中,原辅料、中间产品、待包装产品、成品受到环境污染、杀菌不彻底、贮运方法不当以及不注意卫生操作等多方面因素影响而导致群体性食源性疾病的高发,并严重危害人类健康。Yang等[61]设计了一种基于随机双DNA步行器的比色生物传感器用于金黄色葡萄球菌的检测。为提高反应动力学,将多条适配体和行走链组装成DNA聚合物。当适配体识别靶标细菌时可触发多条行走链释放,从而加速Exo Ⅲ对AuNPs上底物链的切割效率,失去表面修饰的AuNPs会发生团聚并产生颜色变化,15 min内即可完成快速定量分析,检测限低至1 CFU/mL。如2.1节所述,向底物链中引入poly A可以实现对行走链在轨道上构象和分布的调控,从而提高行走效率。Li等[62]基于上述策略制备的DNA步行器对鼠伤寒沙门氏菌的检测限低至22.8 CFU/mL,且灵敏度相比巯基修饰方法提高了11.9倍。此外,可以将DNA步行器与其他信号放大策略联用以构筑级联反应。Cai等[63]在电极表面修饰适配体/行走链和引物链/底物链两组双链探针,金黄色葡萄球菌与适配体结合可激活DNA步行器,借助Exo III水解底物链以诱导引物链的释放,进而启动下游的RCA反应,形成的长单链延伸产物可自组装成DNA纳米花结构,其更大的比表面积增加了MB的结合位点以放大检测信号,检测限低至9 CFU/mL(图4)。Zhang等[64]将规律成簇间隔短回文重复序列及其关联蛋白(clustered regularly interspaced short palindromic repeats/CRISPR-associated protein,CRISPR-Cas12a)系统介导的扩增引入反应体系,NEase驱动DNA步行器产生的切割片段可以激活Cas12a的反式切割活性以产生荧光信号,对鼠伤寒沙门氏菌的检测限低至5 CFU/mL,降低了3个数量级。相比于RCA或HCR等耗时的扩增过程,CRISPR-Cas12a扩增只需要5 min,且整个检测过程可在50 min内完成。
3.2 生物毒素
生物毒素主要包括黄曲霉毒素(aflatoxins,AFs)、赭曲霉毒素(ochratoxins,OTs)、玉米赤霉烯酮(zearalenones,ZENs)等,它们具有化学稳定性高、难降解等特点,对细胞、肝脏及免疫系统有严重的毒副作用,甚至产生致畸致癌致突变效应。高脂肪、高蛋白或高淀粉类的农产品及制品更易受生物毒素污染,随后再以饲料喂养方式进入动物体内污染动物源食品。Shi等[35]设计了邻近连接的2D DNA轨道,OTA与双链复合物中的适配体结合以释放被封锁的双腿DNA步行器,随后在TSDR反应的驱动下将大量信号探针捕获在电极表面。得益于轨道优异的空间分布和刚性结构,它可在40 min内完成行走过程,反应动力学显著提高,检测限低至3.19 fg/mL。然而,适配体作为保护链预先对行走链进行封闭可能会影响其对靶标的亲和力。对此,Taghdisi等[65]基于“行走”适配体构建了非靶标诱导的DNA步行器用于ZEN的比色检测。当存在靶标时,“行走”适配体和底物链均以单链形式存在,底物链无法被Exo Ⅲ切割,导致4-硝基苯酚难以扩散到AuNPs表面发生催化褪色反应,检测限为10 ng/L。纳米材料能够作为电催化剂或电极改性剂增加电极响应面积并强化界面电子传递,同时也为轨道DNA提供了丰富的结合位点,从而提高了传感器的灵敏度和准确性[66−68]。Zeng等[67]使用基于共价有机骨架(covalent organic framework,COF)和DNA步行器的双重扩增策略开发了用于AFM1检测的电化学发光(Electrochemiluminescence,ECL)传感器。三联吡啶钌/三正丙胺(tris(2,2’-bipyridyl)ruthenium(II)/tri-n-propylamine,Ru(bpy)32+/TPrA)是经典的ECL反应体系,利用COF大表面和疏水多孔结构能够负载大量Ru(bpy)32+并富集TPrA,同时其纳米限域效应可以提高ECL反应效率。AFM1触发双腿DNA步行器运行以释放大量的单链DNA,与电极表面的DNA探针结合后在T7外切酶作用下将其水解,使淬灭剂二茂铁远离Ru(bpy)32+/TPrA@COF改性的电极表面,恢复ECL信号。COF的引入使信号强度提高了近5倍,检测限低至0.009 pg/mL。然而,行走过程中释放的单链DNA随机分散在反应溶液中很难被传感界面捕获。对此,Zhong等[46]利用靶标-适配体复合物的循环识别在由发夹DNA功能化AuNPs制成的3D轨道上持续移动以执行靶标转化过程,从而将上述单腿DNA步行器运动的轨道颗粒转化为3D多腿DNA步行器,其多位点锚定作用可以加快电极表面的TSDR反应以产生滚动放大信号,最终可在30 min内实现对OTA的高灵敏检测,反应速率提高了3.4倍,检测限低至4.7 fg/mL。
3.3 抗生素
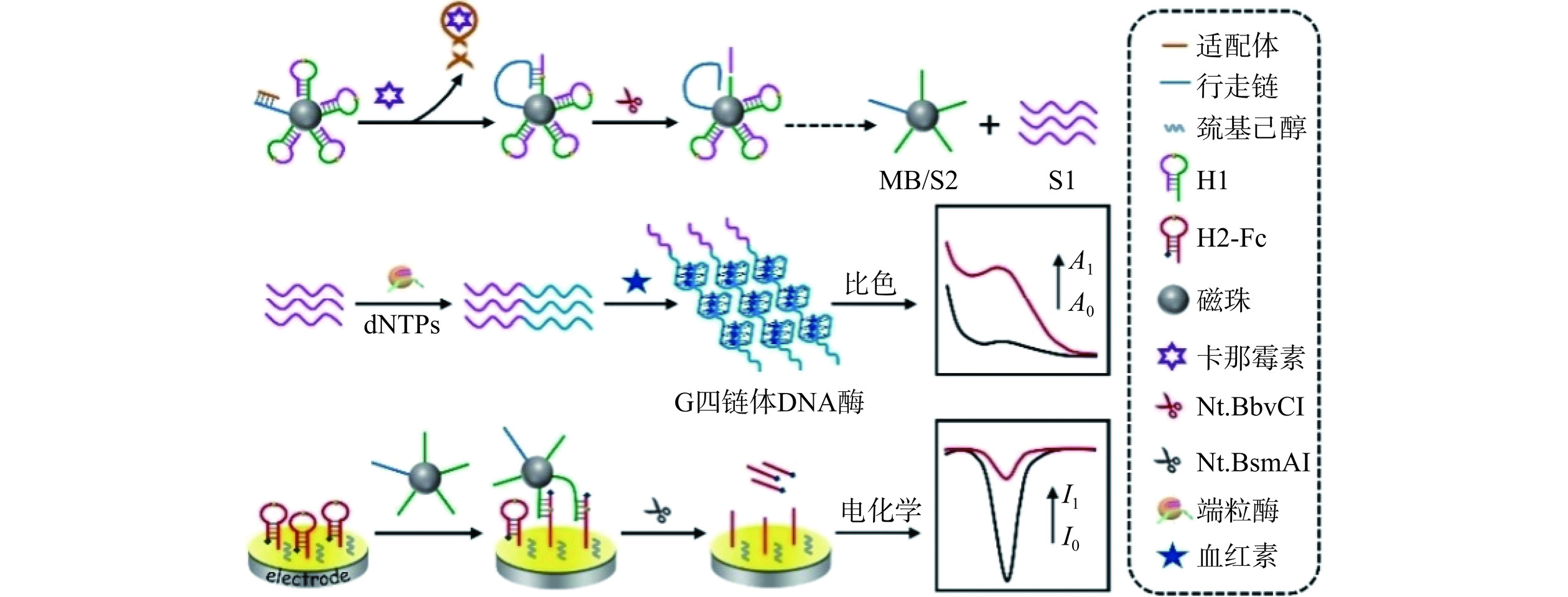
抗生素是由微生物在代谢过程中产生的能干扰致病菌正常生长的化学物质,被广泛用于预防和治疗人类或动物由细菌感染而引起的疾病,另外也被添加到动物饲料中以提高其出肉率。根据分子结构及其作用机制可分为β-内酰胺类、氨基糖苷类、喹诺酮类、磺胺类以及四环素类等。然而,抗生素滥用会导致含有耐药基因的超级细菌产生,对生态环境造成严重威胁。Zhang等[69]利用纳米材料修饰电极和信号探针,并结合DNA酶驱动的DNA步行器构建了具有三重信号放大作用的电化学传感器,实现了恩诺沙星的高灵敏检测,检测限低至1.02×10−7 ng/mL。利用血红素/G-四链体DNA酶作为催化标签,可以实现无标记生物传感,避免了信号标记核酸探针复杂的合成过程。Zhang等[70]利用靶标识别过程释放引发链,并通过Exo Ⅲ辅助的酶切循环过程将其转化为大量用于激活DNA步行器的引发链。随后在Exo Ⅲ驱动下将共固定在电极表面的发夹结构底物链降解,并诱导其形成G四链体/血红素复合物以催化过氧化氢还原产生响应电流,对氨苄青霉素的检测限低至0.76 pM。如图5所示,Wang等[71]建立了一种基于比色和电化学双模式的生物传感方法用于检测卡那霉素。以被靶标激活的DNA步行器在运动过程中产生的酶切片段为引物,在端粒酶的催化下在其末端添加TTAGGG重复序列以合成G-四链体DNA酶,实现比色传感。将上述运动轨迹颗粒转换为多腿DNA步行器,在NEase驱动下使二茂铁标记探针离开电极表面,导致电化学信号下降,检测限低至8.4 fg/mL。为了进一步提高灵敏度,有必要降低背景信号干扰[72−74]。Li等[72]制备了基于分裂DNA酶驱动的DNA步行器,只有当靶标与适配体结合后才能形成完整的DNA酶实现磺胺检测,从而有效降低假阳性信号,检测限低至3 pg/mL。此外,Liang等[73]设计了基于“开-关-超开”策略的光电化学生物传感器用于卡那霉素检测。将银纳米簇标记的发夹结构底物链固定在量子点修饰的电极表面以淬灭其初始光电流信号。适配体识别靶标后启动级联行走过程,在释放银纳米簇的同时在电极上引入量子点敏化剂,使信噪比大幅增强,检测限低至0.53 fg/mL。
4. 结论与展望
DNA步行器的可编程操作使信号级联放大成为可能,这对于超痕量分子的高灵敏检测至关重要。相比于其他核酸扩增过程,其独特优势在于能够克服传质阻碍,缩短样品响应时间。然而,这也在很大程度上受到了其行走效率低、行走范围有限、易脱轨等方面的限制。为应对上述挑战,大量研究通过调控其轨道界面微环境、设计多识别位点、低空间位阻的DNA行走链以及组装具有有序分布结构的刚性轨道纳米阵列来加速行走链和底物链之间的动态识别过程,并提高其反应动力学,以赋予行走过程高方向性和可控性。另外,通过结合CRISPR-Cas体系能在保证DNA步行器高灵敏度的同时大大缩短其响应时间。
为了推动DNA步行器的发展,今后的研究可以着重于以下几个方面:基于DNA步行器能够在纳米尺度上精确地执行分子任务这一特性,除了生物传感外,它在原位细胞成像以及靶向药物递送等方面也具有很大应用潜力。然而,这需要将所有组分集成在纳米器件中同步输送至靶标处以保证DNA步行器运行的稳定性和可控性;当底物链耗尽后DNA步行器作废,导致其重复利用率较低;与大多数基于信号开启或关闭的核酸信号放大过程不同,DNA步行器是由动态相互作用驱动的非平衡系统。因此,实际检测过程中复杂的基质环境可能会对其行走过程产生干扰,未来需要设计具有防污或自清洁功能的DNA步行器以保证其在长期连续检测中的稳定性和使用寿命;将DNA步行器与其他信号扩增策略联用时,通常需要多步移液和加样步骤,使检测过程复杂化并容易造成交叉污染。微流控芯片平台以封闭的微纳通道网络将特异性识别和富集、信号转导及放大和信号读取多个单元集成,自动完成分析全过程,最大限度地降低扩增转移过程中的污染风险并简化操作步骤,同时样品消耗量少,能实现多靶标高通量检测。在此基础上,通过植入现有的便携式商业传感器如侧向层析试纸条可以实现现场即时分析,以确保其准确性和时效性。
-
表 1 DNA步行器在食品安全检测中的应用
Table 1 Application of DNA walker in food safety detection
危害因子 检测方法 线性范围 检出限 时间(min) 驱动力 参考文献 食源性致病菌 金黄色葡萄球菌 金纳米颗粒比色 1~105 CFU/mL 1 CFU/mL 15 Exo III [61] 鼠伤寒沙门氏菌 荧光法 100~108 CFU/mL 22.8 CFU/mL 50 CHA [62] 金黄色葡萄球菌 电化学 60~6×107 CFU/mL 9 CFU/mL 100 Exo III [63] 鼠伤寒沙门氏菌 荧光法 − 5 CFU/mL 50 NEase [64] 生物毒素 赭曲霉毒素 电化学发光法 10~108 fg/mL. 3.19 fg/mL 40 EDC [35] 玉米赤霉烯酮 催化显色法 0.02~80 µg/L 0.1 µg/L 120 Exo III [65] 玉米赤霉烯酮 电化学 0.0001~103 ng/mL 4.57×10−6 ng/mL 180 NEase [66] 黄曲霉毒素M1 电化学发光法 0.03~3×105 pg/mL 0.009 pg/mL 240 NEase [67] 棒曲霉素 电化学 0.05~500 pg/mL 4.14×10−5 ng/mL 180 NEase [68] 赭曲霉毒素 电化学发光法 0.01~100 pg/mL 4.7 fg/mL 30 Exo III & EDC [46] 抗生素 恩诺沙星 电化学 5×10−3~10 pg/mL 1.02×10−7 ng/mL 190 DNA酶 [69] 氨苄青霉素 电化学 1~104 pM 0.76 pM 210 Exo III [70] 卡那霉素 电化学 0.01~1000 ng/mL 8.4 fg/mL 190 NEase [71] 磺胺 电化学 10~106 pg/mL 3 pg/mL 90 DNA酶 [72] 卡那霉素 光电化学法 10~106 fg/mL 0.53 fg/mL 200 DNA酶 [73] 卡那霉素 电化学 10~106 fM 0.02 fM 180 CHA [74] -
[1] SHAN Y K, LU Y N, YI W J, et al. On-site food safety detection:Opportunities, advancements, and prospects[J]. Biosensors and Bioelectronics:X,2023,14:100350.
[2] 张玉, 宋志敏, 杜衍, 等. 纳米酶传感器在现场即时检测领域的应用进展[J]. 分析化学,2023,51(5):800−810. [ZHANG Y, SONG Z M, DU Y, et al. Recent progress of nanozyme-based sensors in point-of-care testing[J]. Chinese Journal of Analytical Chemistry,2023,51(5):800−810.] ZHANG Y, SONG Z M, DU Y, et al. Recent progress of nanozyme-based sensors in point-of-care testing[J]. Chinese Journal of Analytical Chemistry, 2023, 51(5): 800−810.
[3] SHU R, LIU S J, HUANG L J, et al. Enzyme-Mimetic nano-immunosensors for amplified detection of food hazards:Recent advances and future trends[J]. Biosensors and Bioelectronics,2022,217:114577.
[4] 周迎卓, 王欣, 刘宝林. 基于适配体的功能化纳米探针在食品安全检测中的应用进展[J]. 食品工业科技,2018,39(10):335−341. [ZHOU Y Z, WANG X, LIU B L. Progress of functionalized nano probe based on aptamer in food safety and detection[J]. Science and Technology of Food Industry,2018,39(10):335−341.] ZHOU Y Z, WANG X, LIU B L. Progress of functionalized nano probe based on aptamer in food safety and detection[J]. Science and Technology of Food Industry, 2018, 39(10): 335−341.
[5] LYU C, KHAN I M, WANG Z P. Capture-SELEX for aptamer selection:A short review[J]. Talanta,2021,229:122274.
[6] 陈晶晶, 郑斌, 王瑜, 等. DNA 步行者在检测传感中的应用[J]. 分析试验室,2023,42(2):254−265. [CHEN J J, ZHEN B, WANG Y, et al. Application of DNA walker in detection and sensing[J]. Chinese Journal of Analysis Laboratory,2023,42(2):254−265.] CHEN J J, ZHEN B, WANG Y, et al. Application of DNA walker in detection and sensing[J]. Chinese Journal of Analysis Laboratory, 2023, 42(2): 254−265.
[7] MASON S D, TANG Y N, LI Y Y, et al. Emerging bioanalytical applications of DNA walkers[J]. TrAC Trends in Analytical Chemistry,2018,107:212−221.
[8] CHEN J, LUO Z W, SUN C J, et al. Research progress of DNA walker and its recent applications in biosensor[J]. TrAC Trends in Analytical Chemistry,2019,120:115626.
[9] SONG L, ZHUGE Y, ZUO X L, et al. DNA walkers for biosensing development[J]. Advanced Science,2022,9(18):2200327.
[10] 邓碧桃, 徐斐, 曹慧, 等. 核酸酶驱动DNA步行器的构建及在食品安全检测中的应用[J]. 分析化学,2023,51(6):1−14. [DENG B T, XU F, CAO H, et al. Construction of nuclease-powered DNA walker and its application in food safety[J]. Chinese Journal of Analytical Chemistry,2023,51(6):1−14.] DENG B T, XU F, CAO H, et al. Construction of nuclease-powered DNA walker and its application in food safety[J]. Chinese Journal of Analytical Chemistry, 2023, 51(6): 1−14.
[11] LI W, WANG L, JIANG W. A catalytic assembled enzyme-free three-dimensional DNA walker and its sensing application[J]. Chemical Communications,2017,53(40):5527−5530.
[12] SHEN Y, GONG J M, LI S Q, et al. Enzyme-free dual-DNA walker based on catalytic hairpin assembled DNAzyme for sensing telomerase activity[J]. Sensors and Actuators B:Chemical,2020,329:129078.
[13] LI N X, DU M Y, LIU Y C, et al. Multipedal DNA walker biosensors based on catalyzed hairpin assembly and isothermal strand-displacement polymerase reaction for the chemiluminescent detection of proteins[J]. ACS Sensors,2018,3(7):1283−1290.
[14] LI J, WENG X, MO F, et al. Superparamagnetic nanostructures coupled with an entropy-driven DNA circuit for elegant and robust photoelectrochemical biosensing[J]. Analytical Chemistry,2020,92(22):15145−15151.
[15] LIANG C P, MA P Q, LIU H, et al. Rational engineering of a dynamic, entropy-driven DNA nanomachine for intracellular microRNA imaging[J]. Angewandte Chemie,2017,56(31):9077−9081.
[16] YANG J M, DOU B T, YUAN R, et al. Aptamer/protein proximity binding-triggered molecular machine for amplified electrochemical sensing of thrombin[J]. Analytical Chemistry,2017,89(9):5138−5143.
[17] ZHAO Z, CHEN Z Q, LIU D R, et al. Fuel strand-powered self-propelled electrochemical DNA machine for enzyme-free and distinctly amplified detection of nucleic acid with tunable performance[J]. Biosensors and Bioelectronics,2021,171:112706.
[18] MIAO P, TANG Y G. DNA walking and rolling nanomachine for electrochemical detection of miRNA[J]. Small,2020,16(47):2004518.
[19] CHENG X Y, BAO Y F, LIANG S, et al. Flap endonuclease 1-assisted DNA walkers for sensitively and specifically sensing ctDNAs[J]. Analytical Chemistry,2021,93(27):9593−9601.
[20] QU X M, ZHU D, YAO G B, et al. An exonuclease III-powered, on-particle stochastic DNA walker[J]. Angewandte Chemie International Edition,2017,56(7):1855−1858.
[21] WANG Q, LIU Y Q, WANG X F, et al. Ternary electrochemiluminescence biosensor based on DNA walkers and AuPd nanomaterials as a coreaction accelerator for the detection of miRNA-141[J]. ACS Applied Materials & Interfaces,2021,13(22):25783−25791.
[22] FENG C, WANG Z H, CHEN T S, et al. A dual-enzyme-assisted three-dimensional DNA walking machine using T4 polynucleotide kinase as activators and application in polynucleotide kinase assays[J]. Analytical Chemistry,2018,90(4):2810−2815.
[23] YIN Y M, CHEN G F, GONG L, et al. DNAzyme-powered three-dimensional DNA walker nanoprobe for detection amyloid β-peptide oligomer in living cells and in vivo[J]. Analytical Chemistry,2020,92(13):9247−9256.
[24] YAO D B, BHADRA S, XIONG E, et al. Dynamic programming of a DNA walker controlled by protons[J]. ACS Nano,2020,14(4):4007−4013.
[25] CUI M R, LI X L, XU J J, et al. Acid-switchable DNAzyme nanodevice for imaging multiple metal ions in living cells[J]. ACS Applied Materials & Interfaces,2020,12(11):13005−13012.
[26] ZHANG Y, ZHANG Y, ZHANG X B, et al. A photo zipper locked DNA nanomachine with an internal standard for precise miRNA imaging in living cells[J]. Chemical Science,2020,11(24):6289−6296. doi: 10.1039/D0SC00394H
[27] ZHONG W K, WU J Y, HUANG Y Q, et al. Target-activated, light-actuated three-dimensional DNA walker nanomachine for amplified miRNA detection[J]. Langmuir,2022,38(3):1151−1157.
[28] LU H T, YANG F, LIU B H, et al. Intracellular low-abundance microRNA imaging by a NIR-assisted entropy-driven DNA system[J]. Nanoscale Horizons,2019,4(2):472−479. doi: 10.1039/C8NH00330K
[29] MA P Q, LIANG C P, ZHANG H H, et al. A highly integrated DNA nanomachine operating in living cells powered by an endogenous stimulus[J]. Chemical Science,2018,9(13):3299−3304. doi: 10.1039/C8SC00049B
[30] YE M Q, KONG Y J, ZHANG C L, et al. Near-infrared light controllable DNA walker driven by endogenous adenosine triphosphate for in situ spatiotemporal imaging of intracellular microRNA[J]. ACS Nano,2021,15(9):14253−14262. doi: 10.1021/acsnano.1c02229
[31] YANG F, YAO J S, BU S C, et al. Quadrilateral nucleic acid frame-accelerating DNAzyme walker kinetics for biosensing based on host–guest recognition-enhanced electrochemiluminescence[J]. Analytical Chemistry,2021,93(46):15493−15500. doi: 10.1021/acs.analchem.1c03525
[32] PAN M C, GAO M H, YANG X, et al. Wireframe orbit-accelerated bipedal DNA walker for electrochemiluminescence detection of methyltransferase activity[J]. ACS Sensors,2022,7(8):2475−2482. doi: 10.1021/acssensors.2c01262
[33] YAO T, CHEN J, KONG L Q, et al. Efficient three-dimensional DNA nanomachine guided by a robust tetrahedral DNA nanoarray structure for the rapid and ultrasensitive electrochemical detection of matrix metalloproteinase 2[J]. Analytical Chemistry,2023,95(35):13211−13219. doi: 10.1021/acs.analchem.3c02212
[34] LIAO N, PAN M C, WANG L, et al. Swing Arm location-controllable DNA walker for electrochemiluminescence biosensing[J]. Analytical Chemistry,2021,93(8):4051−4058. doi: 10.1021/acs.analchem.0c05051
[35] SHI X Y, ZHU X C, CHAI Y Q, et al. Non-enzymatic electrochemiluminescence biosensor for ultrasensitive detection of ochratoxin A based on efficient DNA walker[J]. Food Chemistry,2023,407:135113. doi: 10.1016/j.foodchem.2022.135113
[36] MASON S D, WANG G A, YANG P, et al. Probing and controlling dynamic interactions at biomolecule-nanoparticle interfaces using stochastic DNA walkers[J]. ACS Nano,2019,13(7):8106−8113. doi: 10.1021/acsnano.9b03053
[37] YANG K, WANG H Z, MA N, et al. Programmable target-initiated DNAzyme walker walking along a spatially isolated and highly hybridizable substrate track on a nanoparticle surface[J]. ACS Applied Materials & Interfaces,2018,10(51):44546−44553.
[38] LI D, LUO Z W, AN H F, et al. Poly-adenine regulated DNA density on AuNPs to construct efficient DNA walker for microRNA-21 detection[J]. Talanta,2020,217:121056. doi: 10.1016/j.talanta.2020.121056
[39] OISHI M, SAITO K. Simple single-legged DNA walkers at diffusion-limited nanointerfaces of gold nanoparticles driven by a DNA circuit mechanism[J]. ACS Nano,2020,14(3):3477−3489. doi: 10.1021/acsnano.9b09581
[40] PENG X, WEN Z B, YANG P, et al. Biomimetic 3D DNA nanomachine via free DNA walker movement on lipid bilayers supported by hard SiO2@CdTe nanoparticles for ultrasensitive microRNA detection[J]. Analytical Chemistry,2019,91(23):14920−14926. doi: 10.1021/acs.analchem.9b03263
[41] ZHANG Y Y, YANG G M, ZHAO J W, et al. Dynamic 3D DNA rolling walkers via directional movement on a lipid bilayer supported by Au@Fe3O4 nanoparticles for sensitive detection of miRNA-16[J]. Analytical Chemistry,2022,94(23):8346−8353. doi: 10.1021/acs.analchem.2c00831
[42] ZHAI F H, GUAN Y F, ZHU B B, et al. Intraparticle and interparticle transferable DNA walker supported by DNA micelles for rapid detection of microRNA[J]. Analytical Chemistry,2021,93(36):12346−12352. doi: 10.1021/acs.analchem.1c02104
[43] ZHOU X M, ZHUO Y, TU T T, et al. Construction of fast-walking tetrahedral DNA walker with four arms for sensitive detection and intracellular imaging of apurinic/apyrimidinic endonuclease 1[J]. Analytical Chemistry,2022,94(24):8732−8739. doi: 10.1021/acs.analchem.2c01171
[44] WU N, WANG K, WANG Y T, et al. Three-dimensional DNA nanomachine biosensor by integrating DNA walker and rolling machine cascade amplification for ultrasensitive detection of cancer-related gene[J]. Analytical Chemistry,2020,92(16):11111−11118. doi: 10.1021/acs.analchem.0c01074
[45] BAO T, FU R B, JIANG Y Y, et al. Metal-mediated polydopamine nanoparticles-DNA nanomachine coupling electrochemical conversion of metal–organic frameworks for ultrasensitive microRNA sensing[J]. Analytical Chemistry,2021,93(40):13475−13484. doi: 10.1021/acs.analchem.1c02125
[46] ZHONG X, YANG S S, LIAO N, et al. Development of hollow electrochemiluminescent nanocubes combined with a multisite-anchored DNA nanomachine for mycotoxin detection[J]. Analytical Chemistry,2021,93(12):5301−5308. doi: 10.1021/acs.analchem.1c00446
[47] LI D, ZHANG X L, CHAI Y Q, et al. Controllable three-dimensional DNA nanomachine-mediated electrochemical biosensing platform for rapid and ultrasensitive detection of microRNA[J]. Analytical Chemistry,2023,95(2):1490−1497.
[48] ZHANG X L, LI S S, LIU Y J, et al. High-efficiency 3D DNA walker immobilized by a DNA tetrahedral nanostructure for fast and ultrasensitive electrochemical detection of miRNA[J]. Analytical Chemistry,2023,95(8):4077−4085. doi: 10.1021/acs.analchem.2c04847
[49] CHANG Y Y, XU S, LI Y R, et al. DNA Three-way junction with multiple recognition regions mediated an unconfined DNA walker for electrochemical ultrasensitive detection of miRNA-182-5p[J]. Analytical Chemistry,2021,93(38):12981−12986. doi: 10.1021/acs.analchem.1c02668
[50] WANG L, LIU P F, LIU Z J, et al. Simple Tripedal DNA walker prepared by target-triggered catalytic hairpin assembly for ultrasensitive electrochemiluminescence detection of microRNA[J]. ACS Sensors,2020,5(11):3584−3590. doi: 10.1021/acssensors.0c01864
[51] ZHU L, ZHANG X L, YUAN R, et al. Y-shaped walker with abundant recognition domians mediated ultrasensitive electrochemical biosensor for miRNA-21 detection[J]. Sensors and Actuators B: Chemical,2023,375:132901. doi: 10.1016/j.snb.2022.132901
[52] HOU T L, ZHU L, ZHANG X L, et al. Multiregion linear DNA walker-mediated ultrasensitive electrochemical biosensor for miRNA detection[J]. Analytical Chemistry,2022,94(29):10524−10530. doi: 10.1021/acs.analchem.2c02004
[53] LI D D, XU Y X, FAN L, et al. Target-driven rolling walker based electrochemical biosensor for ultrasensitive detection of circulating tumor DNA using doxorubicin@tetrahedron-Au tags[J]. Biosensors and Bioelectronics,2020,148:111826. doi: 10.1016/j.bios.2019.111826
[54] CHEN A Y, ZHUO Y, CHAI Y Q, et al. Bipedal DNA walker mediated enzyme-free exponential isothermal signal amplification for rapid detection of microRNA[J]. Chemical Communications,2019,55(92):13932−13935. doi: 10.1039/C9CC06214A
[55] YANG L, FANG J, LI J J, et al. An integrated fluorescence biosensor for microRNA detection based on exponential amplification reaction-triggered three-dimensional bipedal DNA walkers[J]. Analytica Chimica Acta,2021,1143:157−165. doi: 10.1016/j.aca.2020.11.043
[56] LÜ H Y, CHEN A Y, CHENG W Q, et al. Efficient DNA walker guided with well-regulated interfacial tracks for ultrasensitive electrochemiluminescence biosensing[J]. Analytical Chemistry,2020,92(23):15624−15631. doi: 10.1021/acs.analchem.0c03893
[57] YAO T, KONG L Q, LIU Y, et al. Highly efficient quadruped DNA walker guided by ordered DNA tracks for rapid and ultrasensitive electrochemical detection of miRNA-21[J]. Analytical Chemistry,2022,94(35):12256−12262. doi: 10.1021/acs.analchem.2c03083
[58] JIANG J, ZHANG P, NIE Y M, et al. A well-directional three-dimensional DNA walking nanomachine that runs in an orderly manner[J]. Chemical Science,2020,11(8):2193−2199. doi: 10.1039/C9SC06328E
[59] JIANG J, ZHANG P, CHAI Y Q, et al. Electrochemiluminescence biosensor based on a 3D DNA walking nanomachine with a powerful payload capacity[J]. Sensors and Actuators B: Chemical,2021,330:129337. doi: 10.1016/j.snb.2020.129337
[60] KOU B B, CHAI Y Q, YUAN Y L, et al. A DNA nanopillar as a scaffold to regulate the ratio and distance of mimic enzymes for an efficient cascade catalytic platform[J]. Chemical Science,2021,12(1):407−411. doi: 10.1039/D0SC03584J
[61] YANG H H, XIAO M S, LAI W, et al. Stochastic DNA Dual-walkers for ultrafast colorimetric bacteria detection[J]. Analytical Chemistry,2020,92(7):4990−4995. doi: 10.1021/acs.analchem.9b05149
[62] LI D, YANG E L, LUO Z W, et al. An enzyme-mediated universal fluorescent biosensor template for pathogen detection based on a three-dimensional DNA walker and catalyzed hairpin assembly[J]. Nanoscale,2021,13(4):2492−2501. doi: 10.1039/D0NR07593K
[63] CAI R F, ZHANG S X, CHEN L, et al. Self-assembled DNA nanoflowers triggered by a DNA walker for highly sensitive electrochemical detection of Staphylococcus aureus[J]. ACS Applied Materials & Interfaces,2021,13(4):4905−4914.
[64] ZHANG H J, YAO S, SHENG R, et al. A cascade amplification strategy for ultrasensitive Salmonella typhimurium detection based on DNA walker coupling with CRISPR-Cas12a[J]. Journal of Colloid and Interface Science,2022,625:257−263. doi: 10.1016/j.jcis.2022.06.027
[65] TAGHDISI S M, DANESH N M, RAMEZANI M, et al. Novel colorimetric aptasensor for zearalenone detection based on nontarget-induced aptamer walker, gold nanoparticles, and exonuclease-assisted recycling amplification[J]. ACS Applied Materials & Interfaces,2018,10(15):12504−12509.
[66] YAN H, HE B S, ZHAO R Y, et al. Electrochemical aptasensor based on Ce3NbO7/CeO2@Au hollow nanospheres by using Nb. BbvCI-triggered and bipedal DNA walker amplification strategy for zearalenone detection[J]. Journal of Hazardous Materials,2022,438:129491. doi: 10.1016/j.jhazmat.2022.129491
[67] ZENG W J, WANG K, LIANG W B, et al. Covalent organic frameworks as micro-reactors:confinement-enhanced electrochemiluminescence[J]. Chemical Science,2020,11(21):5410−5414. doi: 10.1039/D0SC01817A
[68] HE B S, DONG X Z. Nb. BbvCI powered DNA walking machine-based Zr-MOFs-labeled electrochemical aptasensor using Pt@AuNRs/Fe-MOFs/PEI-rGO as electrode modification material for patulin detection[J]. Chemical Engineering Journal,2021,405:126642. doi: 10.1016/j.cej.2020.126642
[69] ZHANG B Z, LÜ L N, MA X Y, et al. Au@ZnNi-MOF labeled electrochemical aptasensor for detection of enrofloxacin based on AuPt@h-CeO2/MoS2 and DNAzyme-driven DNA walker triple amplification signal strategy[J]. Biosensors and Bioelectronics,2022,210:114296. doi: 10.1016/j.bios.2022.114296
[70] ZHANG R F, WANG Y, QU X N, et al. Exonuclease III-powered DNA walking machine for label-free and ultrasensitive electrochemical sensing of antibiotic[J]. Sensors and Actuators B:Chemical,2019,297:126771. doi: 10.1016/j.snb.2019.126771
[71] WANG X J, XUAN T T, HUANG W, et al. Endonuclease-driven DNA walking for constructing a novel colorimetric and electrochemical dual-mode biosensing method[J]. Analytica Chimica Acta,2022,1208:339835. doi: 10.1016/j.aca.2022.339835
[72] LI S Y, HE B S, LIANG Y, et al. Target-induced DNAzyme-driven DNA Walker triple signal amplification aptasensor for sulfanilamide detection based on Pt@Pd nanocubes and Au nanoflowers/ribbon-like PEI-CoSe2[J]. Sensors and Actuators B: Chemical,2023,376:133008. doi: 10.1016/j.snb.2022.133008
[73] LIANG P, HUANG W, LI C, et al. Dual cascade DNA walking-induced “super on” photocurrent response for constructing a novel antibiotic biosensing method[J]. Analytica Chimica Acta,2023,1264:341240. doi: 10.1016/j.aca.2023.341240
[74] CAI J Y, MA J P, DANG Y, et al. Magnetic DNA walker-engineered electrochemical sensor for highly sensitive detection of antibiotics[J]. Sensors and Actuators B: Chemical,2023,393:134215. doi: 10.1016/j.snb.2023.134215
-
期刊类型引用(1)
1. 康潇云. 食品安全检测中的生物传感器技术应用与创新. 食品安全导刊. 2024(29): 187-189 .  百度学术
百度学术
其他类型引用(0)





 下载:
下载:
 下载:
下载:



