Construction of a Food-grade Expression Vector Based on pyrF Gene in Lactococcus lactis
-
摘要: 基于pyrF筛选标记和来源于乳酸乳球菌(Lactococcus lactis,L. lactis)的基因组DNA为表达元件,构建L. lactis食品级表达载体,用于食品和药用多肽的表达和生产。首先,利用NZ3900 pyrF基因列构建同源重组突变盒,构建NZ3900 ΔpyrF突变株;然后,分别以来源于L. lactis的repA和repC基因为复制元件、pyrF基因为筛选标记、P32和P8为启动子、以及Tusp45和TpepN为终止子,构建食品级表达质粒pLD;最后以绿色荧光蛋白ZsGreen为报告基因,验证ZsGreen在NZ3900 ΔpyrF突变株的表达及pLD-ZsG的遗传稳定性。实验结果表明,原养型ZsGreen阳性转化子可在普通Elliker培养基中正常生长,在荧光显微镜下观察到明显的绿色荧光信号;此外,PCR和Western blotting也证实ZsGreen能在NZ3900中表达且能稳定传代至30代,说明pLD食品级表达载体构建成功且使得外源蛋白在L. lactis中稳定表达。综上所述,基于pyrF营养缺陷型标记构建的L. lactis食品级表达载体的方法切实可行,可为推动L. lactis在食品和药用多肽生产中的应用提供研究基础。Abstract: In this study, the pyrF screening marker and the genomic DNA fragments were used to construct the expression vectors in food-grade Lactococcus lactis (L. lactis). Such expression system could potentially be used to express and produce food-grade and medicinal polypeptides. Firstly, the NZ3900 ΔpyrF auxotrophic strain was created from the homologous recombination mutant cassette. Secondly, the repA and repC genes were used as the replication elements, the pyrF gene as the screening marker, the P32 and P8 elements from L. lactis as the promoters, and the Tusp45 and TpepN from L. lactis as the terminators, all of which were constructed in the expression plasmid pLD. Finally, the reporter gene ZsGreen (a fluorescent protein) was used to verify the expression of recombinant protein in the NZ3900 ΔpyrF mutant strain and the genetic stability of pLD-ZsG plasmid. The result showed that the prototrophic ZsGreen positive transformants could grow normally in common Elliker culture medium, and the green fluorescent signal was observed under a fluorescence microscope. In addition, ZsGreen protein could be highly expressed in NZ3900 ΔpyrF and the expression plasmid could be stably transmitted through at least 30 generations, according to the results of the PCR and Western blotting, indicating that the recombinant protein was expressed in L. lactis in a stable manner. Based on the above results, the approach for creating an L. lactis expression vector (without antibiotic resistance gene) based on the pyrF auxotrophic marker is feasible and offers a basis for further investigation into the use of L. lactis to manufacture food- and pharmaceutical-grade polypeptides.
-
乳酸乳球菌(Lactococcus lactis,L. lactis)是一种广泛存在于自然界的食品级微生物,因其具有营养和免疫等益生功能[1],被FDA(食品药品监督管理局,Food and Drug Administration)公认为GRAS(一般认为安全的,Generally Recognized as Safe)的微生物,在食品发酵、乳制品的生产和保存中的应用有着悠久的历史[2]。L. lactis是一种生长速度快、易于操作、遗传背景清楚的革兰氏阳性菌,某些菌株全基因组测序已经完成,因其良好的宿主基因工程能力,L. lactis常被用来作为表达异源蛋白的宿主菌,逐渐成为了食品和医药研究领域的热点[3−8]。
然而,传统的L. lactis表达系统存在一些问题,例如大多以抗生素作为筛选标记,抗性基因的横向转移导致抗生素污染已成为全球问题[9−12]。另外,传统的L. lactis表达系统的潜在危害主要是因其表达载体中含有来源于病原微生物或非安全型微生物DNA片段,这极大地限制了其实际应用。因此,寻找新的非抗性食品级筛选标记,构建食品级表达载体使得乳酸菌在食品和医疗行业具有更广泛的应用是乳酸菌基因工程需要探索的方向之一[13]。作为食品级表达系统,L. lactis必须具备:a.遗传特性清楚且稳定;b.表达载体中的表达元件等功能性DNA片段必须来自食品级微生物;c.选择标记必须是食品级的[14]。因此,为满足这三个基本条件,越来越多的研究者着手以来源于食品级微生物的DNA片段构建的食品级表达系统的研究[15]。
安全且稳定的筛选标记是食品级表达载体的关键因素之一。乳清酸核苷-5-磷酸脱羧酶基因(orotidine-5'-phosphate decarboxylase,pyrF)是目前应用最为广泛的一类营养缺陷型筛选标记之一,pyrF基因是L. lactis尿嘧啶核苷酸生物合成和代谢过程的关键酶,将pyrF基因突变或缺失,L. lactis突变菌株不能将体内底物乳清酸转化为乳清酸核苷酸,从而导致尿嘧啶营养缺陷型菌株的产生,其在培养时需要额外添加尿嘧啶才能正常生长,而出发菌株能将5-氟乳清酸(5-fluoroorotic acid,5-FOA)转化为对细胞有毒的5-氟尿嘧啶,从而导致野生型菌株不能在含5-FOA的培养基中生长[16]。基于以上筛选原理,可通过5-FOA筛选获得尿嘧啶营养缺陷型菌株,然后利用不加尿嘧啶的培养基筛选回补原养型菌株,在酵母、丝状真菌等和其他细菌物种中都有着重要的应用[16−17]。
因此,本研究以pyrF基因为筛选标记,通过同源重组法构建NZ3900 ΔpyrF营养缺陷型菌株,并利用食品级L. lactis的相关表达元件[18−19],构建L. lactis食品级表达载体,以ZsGreen为报告基因实现ZsGreen在NZ3900 ΔpyrF缺陷型菌株中的稳定表达。本研究为L. lactis食品级无抗性筛选标记的遗传改造提供新的思路。
1. 材料与方法
1.1 材料与仪器
乳酸乳球菌L. lactis NZ3900 购自德国MoBiTecGmbH公司,由本实验室−80 ℃保存;ZsGreen基因 来自本实验室保存的pLVX-IRES-ZsGreen质粒,经密码子优化并在C端加入6×His-tag蛋白标签,由华大基因公司合成;本研究所有DNA序列和所用引物(表1) 均由华大基因公司合成;Sac II和Hind III限制性内切酶(1 U/μL)、Taq酶(5 U/μL)、T4DNA连接酶(350 U/μL) TaKaRa公司;DNA Marker 南京诺唯赞生物科技股份有限公司;蛋白Marker 北京索莱宝科技有限公司;尿嘧啶、5-氟乳清酸、抗坏血酸、葡萄糖、蔗糖、甘油 上海生工生物工程技术服务有限公司;M17培养基 生物风;细菌基因组快速提取试剂盒、胶回收试剂盒、质粒提取试剂盒 OMEGA公司;His-Tag (D3I1O)偶联辣根过氧化物酶(HRP Conjugate)抗体 美国CST公司;本研究主要培养基及溶液:G17培养基、电转感受态SG17培养基、Elliker培养基、蔗糖-甘油溶液、电转复苏培养基、Elliker-FOA培养基具体配方参考文献[20]。
表 1 本研究所用到的引物Table 1. Primers used in this study名称 序列(5' to 3') 用途 pyrF sen1 TCTAGTTAAAGCTTGTAAAGCGG 扩增5'pyrF重组臂 pyrF anti1 GCGAAATTCTTTTCATAGGATCCCGATAACGATGTCGACTTTTTCTTCTTTCTTTTC 扩增5'pyrF重组臂 pyrF sen2 GAAAAGAAAGAAGAAAAAGTCGACATCGTTATCGGGATCCTATGAAAAGAATTTCGC 扩增3'pyrF重组臂 pyrF anti2 AAAATTTATCAAGGAAATCGC 扩增3'pyrF重组臂 ZsG-F ATGGCCCAGTCCAAGCACGGCC ZsGreen基因验证 ZsG-R TTAGTGATGGTGATGGTGATGGGGCAAGGCGGAGCCGGAGGC ZsGreen基因验证 T100+^TM+Thermal+Cycler PCR仪、MicroPulser电穿孔仪(1652100)、伯乐的电泳转化膜系统(165-8000)、ChemiDoc+XRS+凝胶成像及分析系统 美国Bio-Rad公司;Leica DM2500荧光显微镜 德国徕卡;SW-CJ-1BU超净工作台 苏州净化设备有限公司;LBI-80恒温培养箱 上海龙跃仪器设备有限公司;TS-200B空气浴摇床 上海天呈实验仪器制造有限公司;DELTA320pH计 Mettler Toledo。
1.2 实验方法
1.2.1 pLD载体的合成
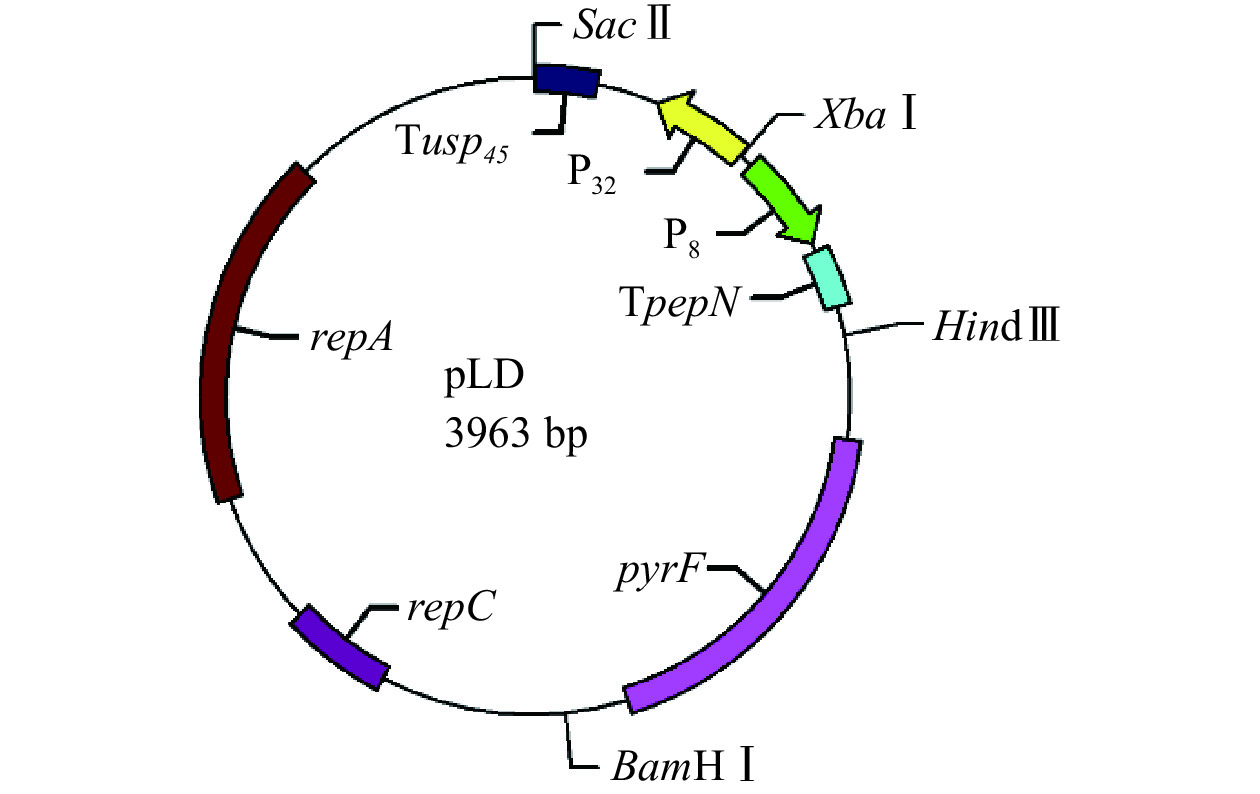
根据L. lactis的基因组DNA全序列(GenBank:CP002094.1)查询得到repA、repC、pyrF、P8、Tusp45和TpepN等相关基因的序列,P32的基因组DNA全序列从GenBank: AP018499.1查询得到、由深圳华大基因公司合成,pLD载体详细结构示意图如图1所示。
1.2.2 电转感受态L. lactis NZ3900的制备
将NZ3900甘油冻存菌划线于G17平板上,置于30 ℃静置培养活化2 d。挑取单克隆菌落,接种到50 mL的G17培养基中,30 ℃静置培养,当菌液OD600=0.3时,于室温下3000×g离心5 min收集NZ3900。用5 mL蔗糖-甘油溶液(冰上预冷)重悬和洗涤菌体,4 ℃,6000×g离心3 min,弃上清液,用1 mL蔗糖-甘油溶液(冰上预冷)重悬菌体,此时即为电转感受态,80 μL/EP管分装,储存于−80 ℃保存备用。
1.2.3 pyrF突变盒pyrF-PT的构建
用OMEGA的细菌基因组快速抽提试剂盒提取,按照说明书操作提取NZ3900 gDNA,以其为模板,利用pyrF sen1/pyrF anti1和pyrF sen2/pyrF anti2引物分别扩增出5'pyrF重组臂pyrF-P和3'pyrF重组臂pyrF-T,目标片段DNA序列大小分别为658 bp和604 bp,PCR扩增条件:预变性94 ℃,5 min;94 ℃ 30 s,55 ℃ 50 s,72 ℃ 40 s,产物进行25个循环,72 ℃延伸7 min。将pyrF-P和pyrF-T的DNA片段进行电泳并切胶纯化回收,作为overlap PCR模板,引物为pyrF sen1/pyrF anti2,PCR扩增条件:预变性94 ℃,5 min;94 ℃ 30 s,55 ℃ 50 s,72 ℃ 1 min,产物进行25个循环,72 ℃延伸7 min,扩增出pyrF突变盒pyrF-PT的DNA序列,为1262 bp。然后电泳并切胶纯化回收,将所得的DNA片段定量并于−20 ℃保存备用。
1.2.4 L. lactis NZ3900 ΔpyrF突变株的构建
将纯化的pyrF-PT DNA片段电转化入L. lactis NZ3900:吸取冰上融化的80 μL感受态NZ3900于事先预冷的电转杯中,再加入10 μL(含5 μg的pyrF-PT DNA)待转化DNA。设置电转程序如下:2000 V,25 μF,200 Ω,5 ms。电击后加入1 mL电转复苏培养基混匀,将电转杯置于冰上5 min,然后放入培养箱30 ℃孵育2 h,3000×g离心5 min,弃去上清液体然后用200 μL生理盐水重悬,涂布于Elliker-FOA平板上,30 ℃培养72 h。
1.2.5 L. lactis NZ3900 ΔpyrF突变株的鉴定
挑取Elliker-FOA平板上的单菌落,接种于100 mL含有50 mg/L的尿嘧啶的G17培养基中,30 ℃静置培养2 d,提取L. lactis NZ3900 ΔpyrF gDNA,用1%琼脂糖电泳鉴定,部分送往华大基因公司进行序列鉴定。然后将构建成功的L. lactis NZ3900 ΔpyrF突变株制备为电转化感受态细胞。
1.2.6 pLD-ZsG载体的构建
将pLD质粒与P32-ZsG-Tusp45 DNA片段分别进行Sac II和Hind III双酶切反应后对产物进行琼脂糖凝胶电泳并切胶回收备用。将纯化的pLD质粒酶切片段与P32-ZsG-Tusp45 DNA酶切片段进行连接反应,16 ℃水浴连接过夜,重组载体命名为pLD-ZsG。
1.2.7 LL-pLD-ZsG工程菌株的构建及鉴定
将pLD和pLD-ZsG质粒分别电转入NZ3900 ΔpyrF感受态细胞中,涂布于Elliker平板上。30 ℃培养72 h,将重组菌株分别命名为LL-pLD和LL-pLD-ZsG。然后提取质粒DNA,提取步骤参考任志娟等[21]的研究。将质粒DNA进行酶切,反应完成后取5 μL酶切产物进行1%琼脂糖凝胶电泳鉴定,部分质粒DNA送去华大基因公司进行测序。LL-pLD-ZsG重组工程菌株蛋白样的制备:在室温下5000×g离心3 min收集LL-pLD-ZsG菌体,液氮研磨成细粉状后取菌体50 mg至1.5 mL离心管中,加入1 mL RIPA细胞裂解液振荡均匀,按比例加入5×蛋白上样缓冲液(4:1 v/v)充分振荡混匀,100 ℃水浴变性10 min,室温下5000×g离心3 min,取上清液作为蛋白样保存。ZsGreen蛋白验证步骤参考文献[22]。
1.2.8 LL-pLD-ZsG重组工程菌的荧光观察
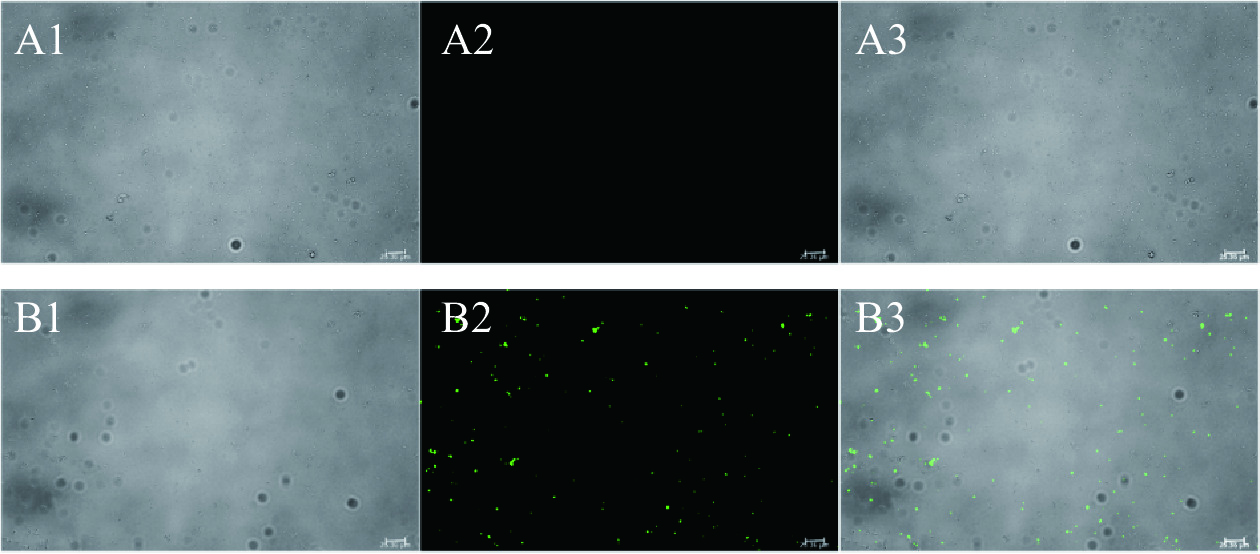
将转化pLD-ZSG片段的Elliker筛选平板上长出的阳性转化子转接到3 mL含有40 mg/L的尿嘧啶的Elliker液体培养基中, 30 ℃下静置培养48 h后,室温下3000×g离心1 min,弃上清液,用1 mL 的PBS溶液重悬菌体,将菌液制片并于Leica DM2500荧光显微镜下观察绿色荧光蛋白在菌体中的表达情况(对照组为相同培养条件的NZ3900 ΔpyrF),激发光波长为493 nm。
1.2.9 LL-pLD-ZsG工程菌株的连续传代培养稳定性测定
将LL-pLD-ZsG重组工程菌株从Elliker平板上挑取阳性菌落,接种于100 mL的Elliker培养基中,在30 ℃培养箱进行培养24 h,至OD600约0.4的时候即作为第1代LL-pLD-ZsG重组工程菌株,然后取1 mL的第1代工程菌株培养菌液于室温下5000×g离心1 min,弃上清液留菌体沉淀,然后用PBS洗涤,室温下5000×g离心1 min,然后用PBS重悬菌体。取1×104 CFU PBS重悬的第1代工程菌株接种到100 mL的Elliker培养基中,在30 ℃培养箱进行培养24 h,至OD600约0.4的时候即作为第2代工程菌株,按照上述步骤收集工程菌株并传代,重复培养到30代。然后分别提取1、5、10、15、20、25、30代的重组工程菌的质粒并进行Sac II单酶切,将酶切产物进行琼脂糖凝胶鉴定,将第30代重组工程菌的质粒送华大基因公司进行测序。以第1代培养菌液和第30代培养菌液制作为蛋白样,然后进行Western blotting鉴定。
1.3 数据处理
本文采用的数据库GenBank(www.ncbi.nlm.nih.gov);PCR引物设计软件:Primer premier 5;基因和蛋白序列处理软件:DNAssist(v3.0);密码子优化JCat online tool(http://www.jcat.de/)。
2. 结果与分析
2.1 突变盒pyrF-PT的构建
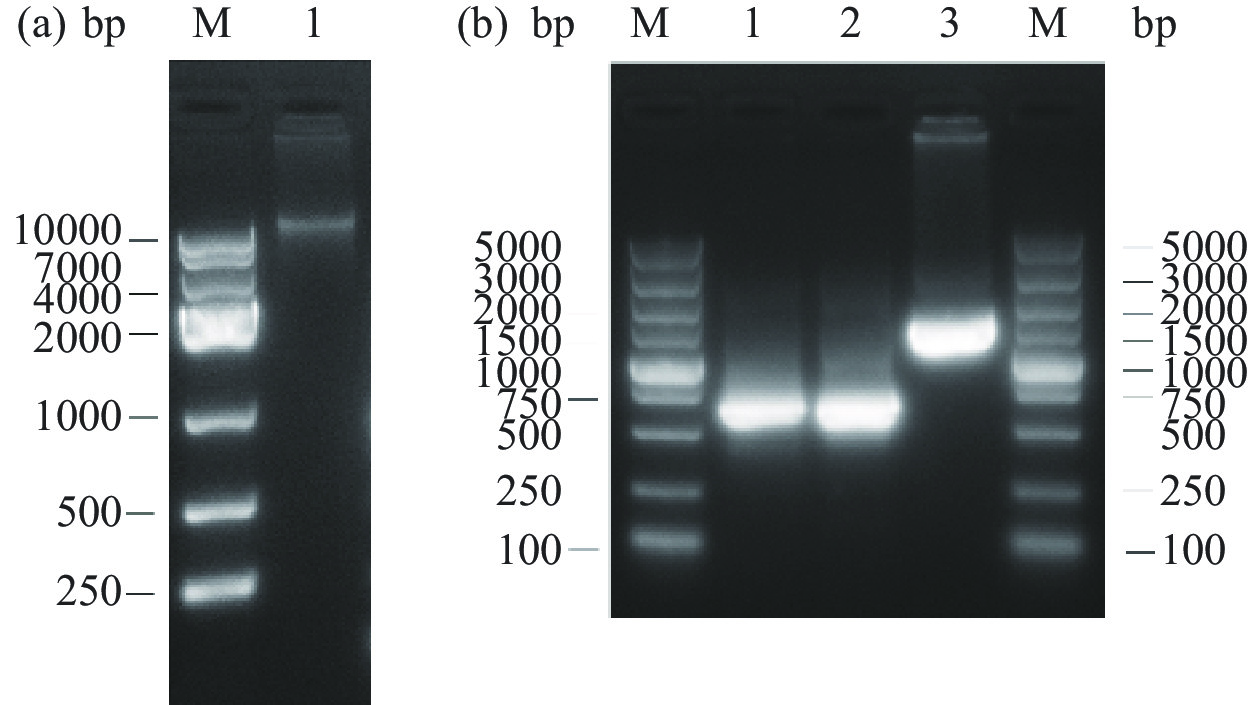
利用基因组DNA提取试剂盒提取NZ3900 gDNA,通过琼脂糖凝胶电泳鉴定结果如图2a,在10 kbp上方可以看到条带,说明NZ3900 gDNA提取成功。以NZ3900 gDNA为模板,扩增5'pyrF重组臂pyrF-P和3'pyrF重组臂pyrF-T的DNA片段,并以pyrF-P/pyrF-T为模板进行overlap PCR,扩增出pyrF突变盒pyrF-PT,琼脂糖凝胶电泳鉴定结果如图2b,目的序列的DNA片段分别约为658、604和1262 bp,与预期大小一致,且测序结果表明pyrF-P、pyrF-T和突变盒pyrF-PT扩增成功。
2.2 NZ3900 ΔpyrF突变株的鉴定
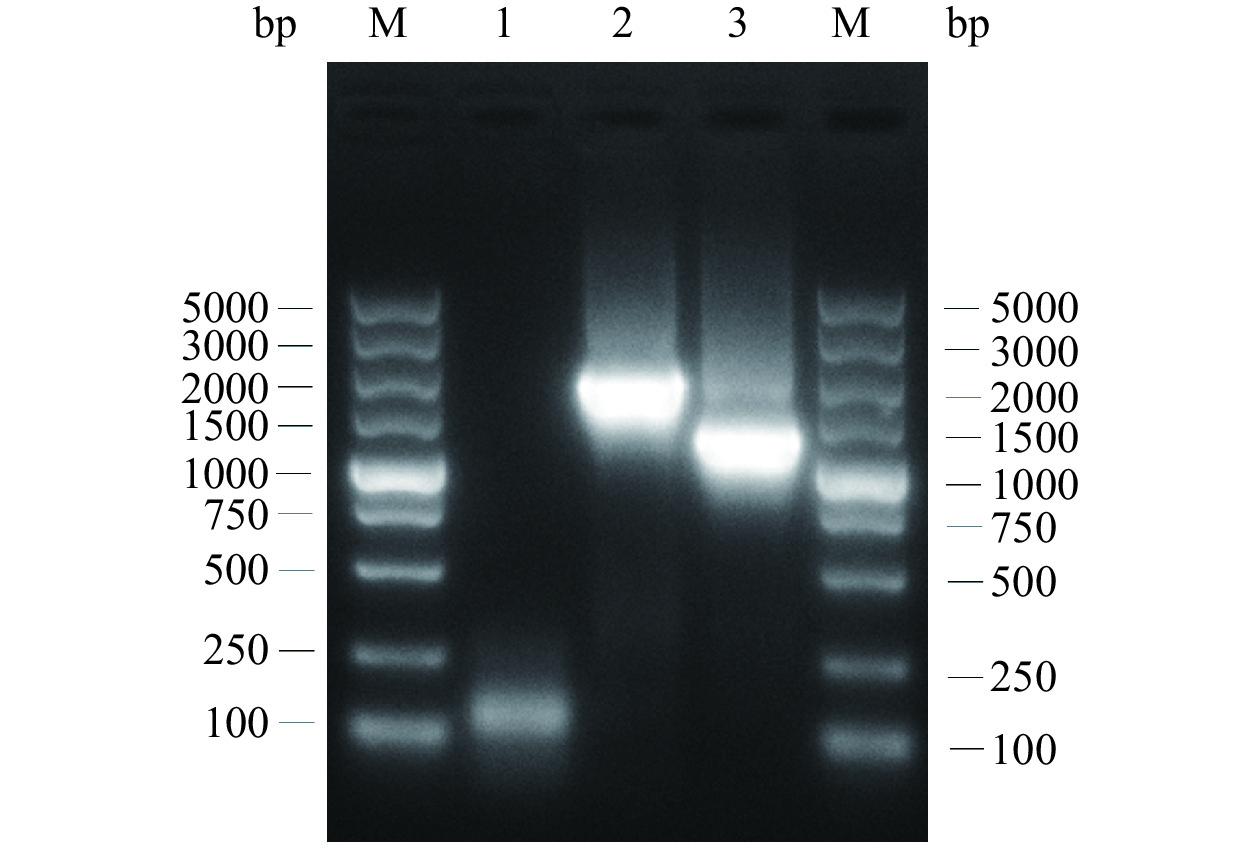
将突变盒pyrF-PT电转化至NZ3900出发菌中,构建NZ3900 ΔpyrF突变株,在含有尿嘧啶和5-FOA的固体培养基中进行NZ3900 ΔpyrF突变株筛选。基因组PCR鉴定如图3所示,结果表明NZ3900出发菌的pyrF基因大小约为1976 bp,而NZ3900 ΔpyrF突变株的pyrF基因区大小约为1262 bp左右,比出发菌减少了714 bp,经过测序表明,pyrF基因的编码序列被成功地敲除。
2.3 pLD-ZSG载体的构建
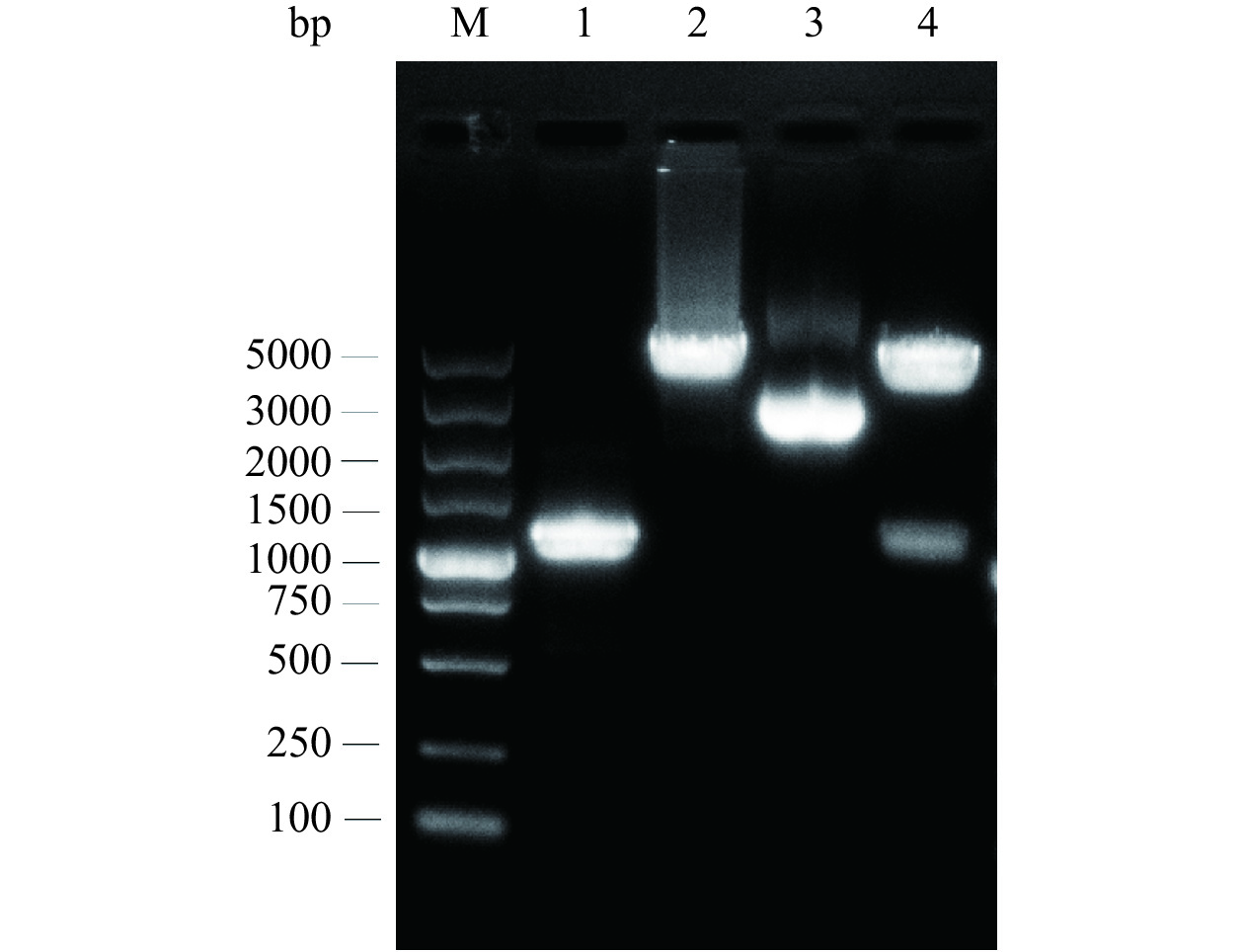
将合成的P32-ZsG-Tusp45 DNA片段和pLD载体分别进行Sac II和Hind III双酶切,纯化后由连接酶连接,构建pLD-ZsG。将其电转至NZ3900 ΔpyrF突变株,构建原养型LL-pLD-ZsG工程菌株并扩增pLD-ZsG质粒,提取pLD-ZsG进行双酶切鉴定,琼脂糖凝胶电泳结果如图4,在约1073 bp和3686 bp处可见目标条带,与双酶切的P32-ZsG-Tusp45 DNA和pLD质粒的酶切片段大小一致,且测序结果表明pLD-ZsG载体构建成功。
2.4 LL-pLD-ZsG重组工程菌中绿色荧光的观察
本研究以ZsGreen绿色荧光蛋白作为报告基因,构建的LL-pLD-ZsG原养型重组工程菌在荧光显微镜下可观察到明亮的绿色荧光菌体,而NZ3900 ΔpyrF突变株在相同条件下无绿色荧光(见图5)且NZ3900 ΔpyrF出发菌和LL-pLD-ZsG重组工程菌在形态上并无差别,表明pLD-ZsG表达载体成功转入NZ3900 ΔpyrF突变株,使得ZsGreen基因在NZ3900 ΔpyrF突变株进行有效的转录和表达。
2.5 LL-pLD-ZsG重组工程菌中ZsGreen蛋白的表达
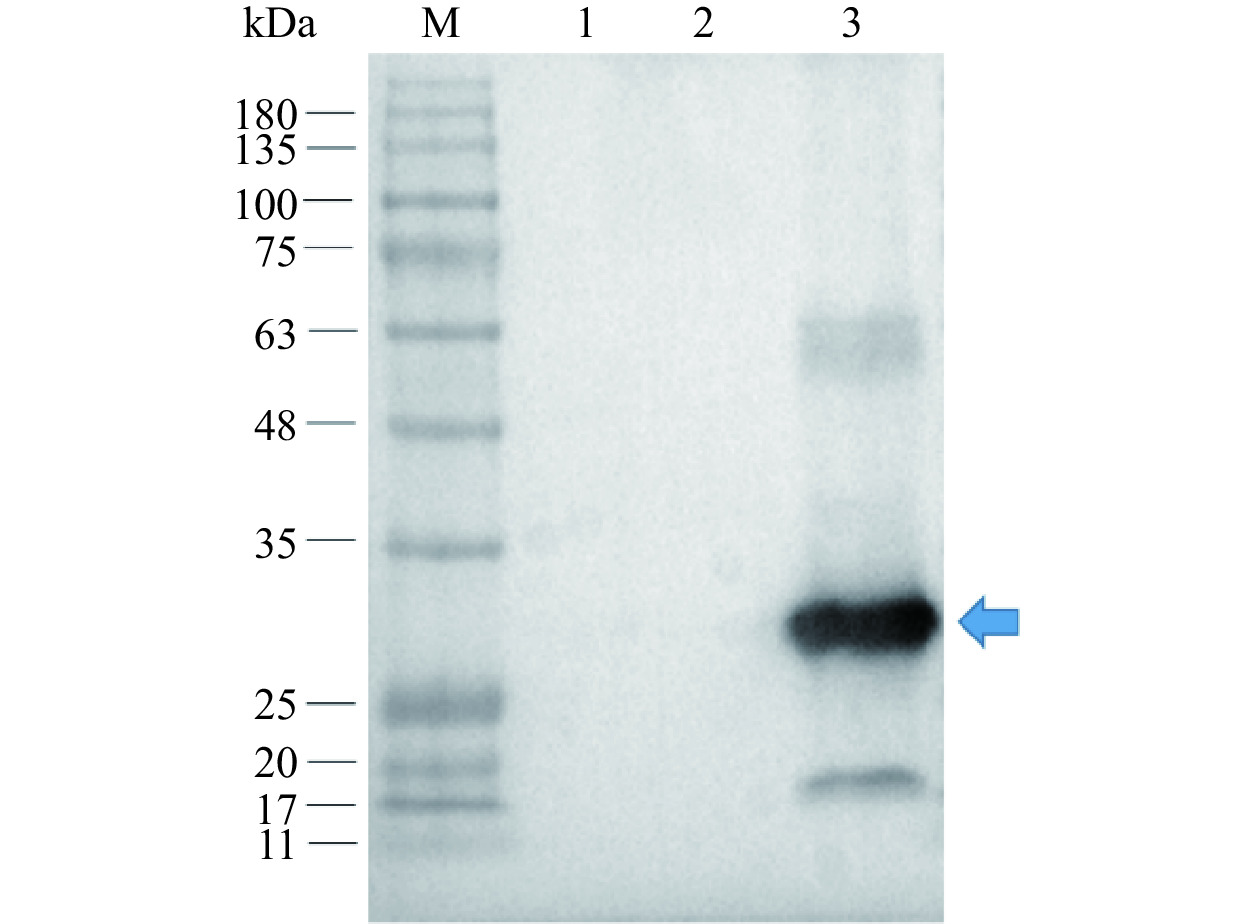
通过Western blotting验证ZsGreen蛋白在NZ3900 ΔpyrF突变株中的表达情况,结果如图6所示,在NZ3900出发菌、LL-pLD空载工程菌、LL-pLD-ZsG重组工程菌中只有LL-pLD-ZsG工程菌的菌体蛋白在约28 kDa处有特异性目的条带出现,该条带的大小与预期相符,表明绿色荧光蛋白ZsGreen能在NZ3900 ΔpyrF中表达,同时也说明pLD能实现外源基因在NZ3900 ΔpyrF突变株中的转录和表达。
2.6 LL-pLD-ZsG重组工程菌传代稳定性的鉴定
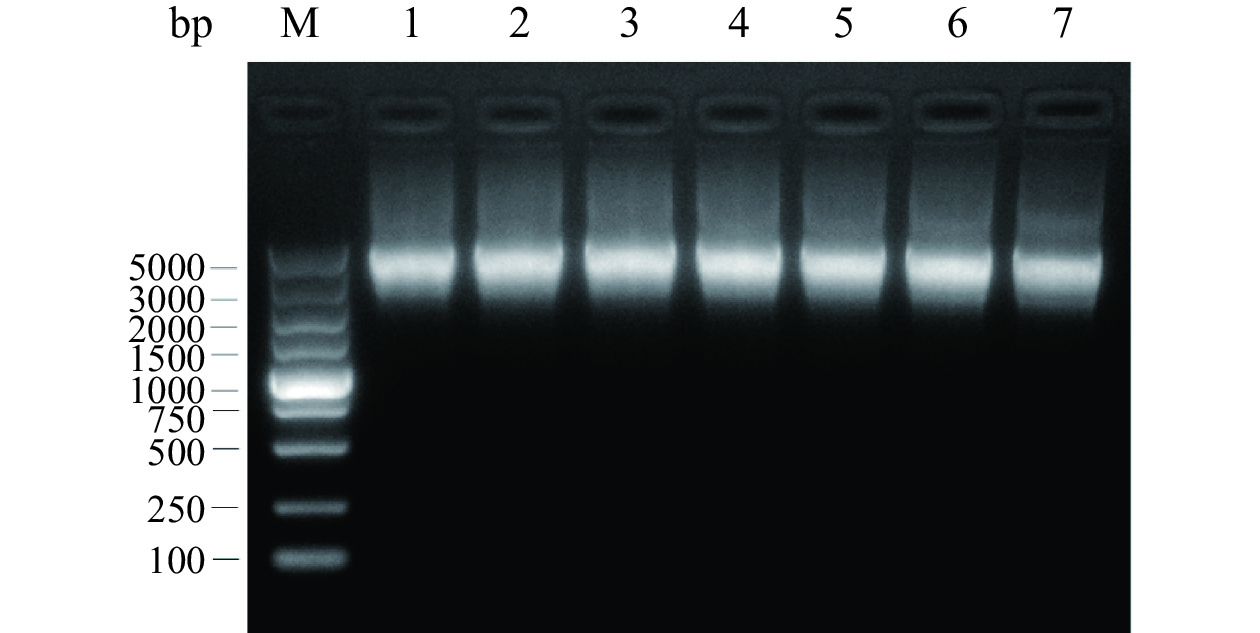
提取不同代的LL-pLD-ZsG重组工程菌的pLD-ZsG质粒进行Sac II单酶切,酶切产物进行琼脂糖胶电泳鉴定,结果如图7所示,1、5、10、15、20、25、30代的重组工程菌的质粒经过单酶切后大小均一致约为4759 bp,表明LL-pLD-ZsG工程菌在30代以内质粒没有丢失或发生改变,具有一定的遗传稳定性。
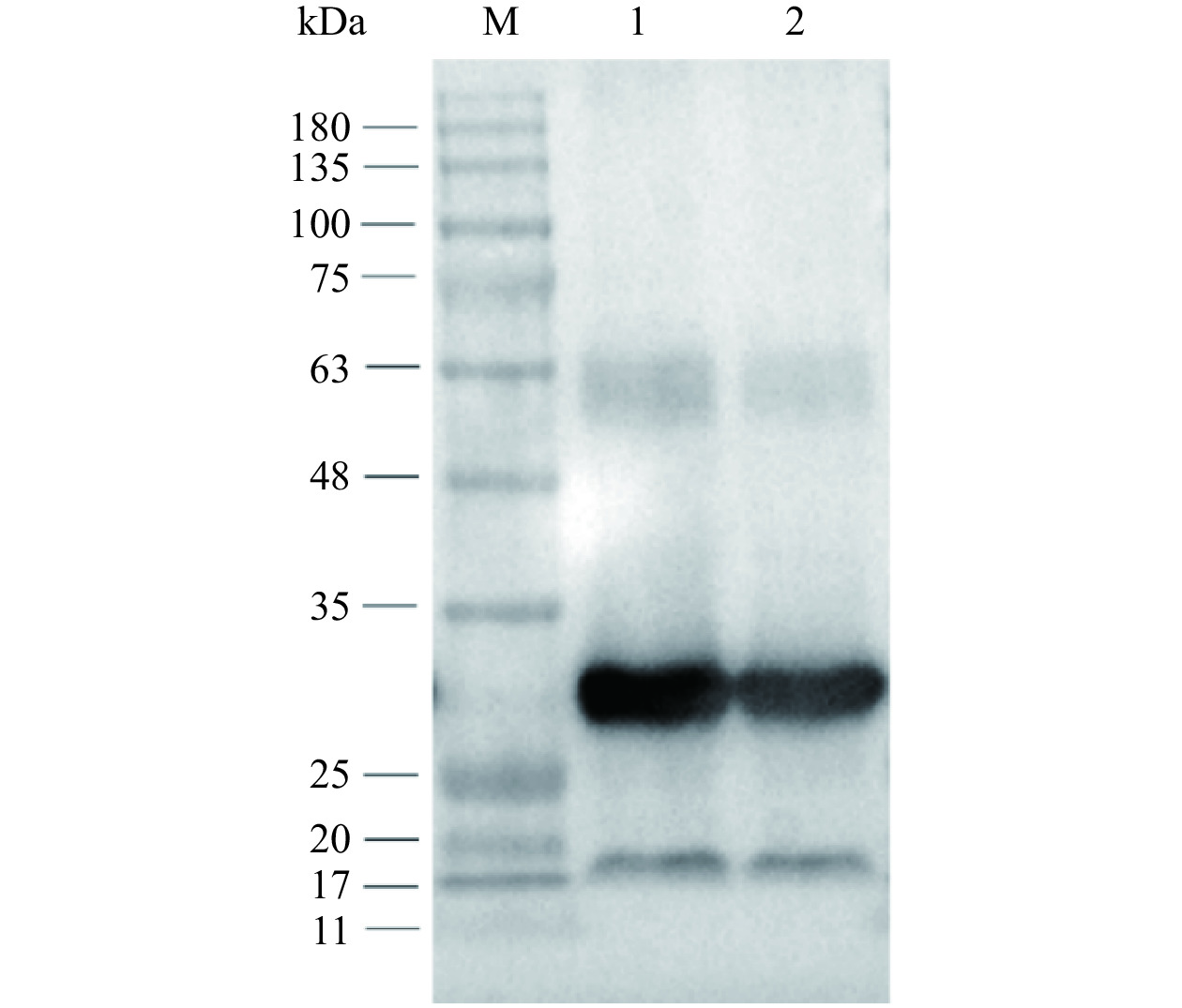
通过Western blotting验证LL-pLD-ZsG工程菌经过30次传代后,是否仍能稳定表达外源蛋白,结果如图8所示,以第1代LL-pLD-ZsG工程菌为阳性对照,结果表明LL-pLD-ZsG工程菌经过30次传代后仍能正确表达ZsGreen,分子量大小约为28 kDa,再次证实了LL-pLD-ZsG重组工程菌至少在传代培养的30代以内保持质粒不丢失,具有一定的遗传稳定性。
3. 讨论与结论
在L. lactis的前期研究中,以抗生素作为筛选标记的非食品级表达载体,构建了大量的乳酸菌抗性基因工程菌株,在多肽、酶、多糖、细菌素等代谢物质的合成表达方面的大量应用,促进了乳酸菌工业化的应用进展,为制备酶制剂、乳酸菌口服疫苗提供了理论基础和实验依据[23]。然而,抗生素在生物体或者环境中漂移扩散,破坏生态,影响生物多样性,给全球生态和世界经济带来了沉重的负担[9−10]。
目前,研究者为了构建L. lactis食品级表达载体开发了糖类筛选标记、细菌素筛选标记、抗金属离子筛选标记、噬菌体筛选标记以及营养缺陷型筛选标记等[24−27]。营养缺陷型筛选标记是理想的安全选择性标记,Dickely等[28]以supB基因作为选择标记,构建pFG1表达载体,进行克隆表达。Sorensen等[29]用supD基因构建了pFG200载体,实现了多种外源蛋白的过表达。以上两种选择标记均属无义突变抑制基因,可在多种培养基上进行选择,由于其使用灵活,相比较于其他“食品级”筛选标记有许多优点,但tRNA抑制基因可能会导致多重效应[30]。而孙强正等[14]以营养缺陷型thyA基因为筛选标记,构建pSQZ食品级表达载体,实现具有保护活性蛋白的表达。以上的研究可以看出,营养缺陷型筛选标记在L. lactis的食品级表达载体构建中的应用较少,因此,本实验利用pyrF/5-FOA营养缺陷型筛选标记,构建L. lactis食品级表达载体,为L. lactis食品级表达载体的构建提供新的选择。尿嘧啶营养缺陷型是一种在微生物中常用的营养缺陷型筛选标记,因其来自于宿主本身,属于同源转化,避免了甲基化[31−32],是理想的安全型筛选标记。本研究基于pyrF/5-FOA营养缺陷型筛选标记,构建L. lactis NZ3900 ΔpyrF突变株,并以其为出发菌,通过电转化法转染包含pyrF回补标记和报告基因ZsGreen的pLD-ZsG质粒构建LL-pLD-ZsG原养型工程菌株,成功实现外源基因的表达,为L. lactis的遗传改造提供了有效的食品级筛选标记。
L. lactis NZ3900/pNZ8149是目前食品工业和医学领域中最常用且唯一商业化的NICE表达系统[33],由于其需要添加诱导剂Nisin来诱导外源蛋白的表达,不适用于口服疫苗、生物活性多肽的应用;且在食品工业发酵过程中需要添加诱导剂,操作过程麻烦、增加生产成本[5]。而相较于NICE表达系统,pyrF/5-FOA表达体系的优势在于它不需要添加任何的诱导剂和抗生素的条件下,可实现外源基因的稳定表达,这为L. lactis食品级表达系统的实际应用提供了可能。L. lactis组成型表达系统表达外源蛋白产量较低是目前亟待解决的问题。本研究所构建的表达载体pLD也存在这样的问题。因此,在后续研究中继续改造该组成型表达载体,从强启动子筛选、增加多拷贝数等方向进行改造,以期提高外源蛋白在L. lactis组成型表达系统的表达量,进而提高该食品级表达载体的表达能力。
本研究基于pyrF/5-FOA筛选机制构建的食品级表达载体pLD不含有任何抗生素抗性基因、各组分均来自食品级微生物,且宿主菌也为食品级微生物,完全符合FDA所规定的严格的食品级规范,很好地解决了生物安全性问题,提供了一种高效简捷的构建可供口服的食品级表达载体的方法,为利用L. lactis生产食品级药物多肽提供了研究基础和理论依据。
-
表 1 本研究所用到的引物
Table 1 Primers used in this study
名称 序列(5' to 3') 用途 pyrF sen1 TCTAGTTAAAGCTTGTAAAGCGG 扩增5'pyrF重组臂 pyrF anti1 GCGAAATTCTTTTCATAGGATCCCGATAACGATGTCGACTTTTTCTTCTTTCTTTTC 扩增5'pyrF重组臂 pyrF sen2 GAAAAGAAAGAAGAAAAAGTCGACATCGTTATCGGGATCCTATGAAAAGAATTTCGC 扩增3'pyrF重组臂 pyrF anti2 AAAATTTATCAAGGAAATCGC 扩增3'pyrF重组臂 ZsG-F ATGGCCCAGTCCAAGCACGGCC ZsGreen基因验证 ZsG-R TTAGTGATGGTGATGGTGATGGGGCAAGGCGGAGCCGGAGGC ZsGreen基因验证 -
[1] 杨文晓. 乳酸菌在食品工业中的运用研究[J]. 工业微生物,2023,53(1):61−63. [YANG W X. On the application of lactic acid bacteria in food industry[J]. Industrial Microbiology,2023,53(1):61−63.] doi: 10.3969/j.issn.1001-6678.2023.01.019 YANG W X. On the application of lactic acid bacteria in food industry[J]. Industrial Microbiology, 2023, 53(1): 61−63. doi: 10.3969/j.issn.1001-6678.2023.01.019
[2] KOK J, VAN GIJTENBEEK L A, DE JONG A, et al. The evolution of gene regulation research in Lactococcus lactis[J]. FEMS Microbiology Reviews,2017,41(Supp_1):S220−S243. doi: 10.1093/femsre/fux028
[3] LINARES D M, KOK J, POOLMAN B. Genome sequences of Lactococcus lactis MG1363 (revised) and NZ9000 and comparative physiological studies[J]. Journal of Bacteriology,2010,192(21):5806−5812. doi: 10.1128/JB.00533-10
[4] BOLOTIN A, WINCKER P, MAUGER S, et al. The complete genome sequence of the lactic acid bacterium Lactococcus lactis ssp. lactis IL1403[J]. Genome Research,2001,11(5):731−753. doi: 10.1101/gr.169701
[5] 高莹, 李淼, 孙元, 等. 乳酸乳球菌表达系统的发展现状与前景展望[J]. 微生物学报,2022,62(3):895−905. [GAO Y, LI M, SUN Y, et al. Development and prospects of Lactococcus lactis expression system[J]. Acta Microbiologica Sinica,2022,62(3):895−905.] GAO Y, LI M, SUN Y, et al. Development and prospects of Lactococcus lactis expression system[J]. Acta Microbiologica Sinica, 2022, 62(3): 895−905.
[6] 梁琰, 崔欣, 王哲, 等. 乳酸菌食品级表达载体的研究与应用[J]. 微生物学通报,2021,48(3):906−915. [LIANG Y, CUI X, WANG Z, et al. Research and application of food-grade expression vectors of Lactic acic bacteria[J]. Microbiology China,2021,48(3):906−915.] LIANG Y, CUI X, WANG Z, et al. Research and application of food-grade expression vectors of Lactic acic bacteria[J]. Microbiology China, 2021, 48(3): 906−915.
[7] SUN N, ZHANG R, DUAN G, et al. An engineered food-grade Lactococcus lactis strain for production and delivery of heat-labile enterotoxin B subunit to mucosal sites[J]. BMC Biotechnology,2017,17(1):25. doi: 10.1186/s12896-017-0345-6
[8] CAO W Y, DONG M, HU Z Y, et al. Recombinant Lactococcus lactis NZ3900 expressing bioactive human FGF21 reduced body weight of Db/Db mice through the activity of brown adipose tissue[J]. Beneficial Microbes,2020,11(1):67−78. doi: 10.3920/BM2019.0093
[9] KLEIN E Y, VAN BOECKEL T P, MARTINEZ E M, et al. Global increase and geographic convergence in antibiotic consumption between 2000 and 2015[J]. Proceedings of the National Academy of Sciences of the United States of America,2018,115(15):E3463−E3470.
[10] KRAEMER S A, RAMACHANDRAN A, PERRON G G. Antibiotic pollution in the environment:From microbial ecology to public policy[J]. Microorganisms,2019,7(6):180. doi: 10.3390/microorganisms7060180
[11] STOWER H. Antibiotic tolerance leads to antibiotic resistance[J]. Nature Medicine,2020,26(2):163.
[12] KUMAR S B, ARNIPALLI S R, ZIOUZENKOVA O. Antibiotics in food chain:The consequences for antibiotic resistance[J]. Antibiotics (Basel, Switzerland),2020,9(10):688. doi: 10.3390/antibiotics9100688
[13] KAZI T A, ACHARYA A, MUKHOPADHYAY B C, et al. Plasmid-based gene expression systems for lactic acid bacteria:A review[J]. Microorganisms,2022,10(6):1132. doi: 10.3390/microorganisms10061132
[14] 孙强正, 熊衍文, 叶长芸, 等. 乳酸乳球菌食品级分泌性表达载体pSQZ的构建及报告蛋白的表达[J]. 中国人兽共患病学报,2008,6(3):224−228,275. [SUN Q Z, XIONG Y W, YE C Y, et al. Construction of a food-grade secretion expression vector and the expression of reporter protein in Lactococcus lactis[J]. Chinese Journal of Zoonoses,2008,6(3):224−228,275.] SUN Q Z, XIONG Y W, YE C Y, et al. Construction of a food-grade secretion expression vector and the expression of reporter protein in Lactococcus lactis[J]. Chinese Journal of Zoonoses, 2008, 6(3): 224−228,275.
[15] 苏松坤, 晏励民, 刘芳. 乳酸菌食品级表达系统的研究进展[J]. 食品与生物技术学报,2012,31(12):1233−1238. [SU S K, YAN L M, LIU F. Research development of LAB food-grade expression system[J]. Journal of Food Science and Biotechnology,2012,31(12):1233−1238.] doi: 10.3969/j.issn.1673-1689.2012.12.001 SU S K, YAN L M, LIU F. Research development of LAB food-grade expression system[J]. Journal of Food Science and Biotechnology, 2012, 31(12): 1233−1238. doi: 10.3969/j.issn.1673-1689.2012.12.001
[16] 毕付提, 史亚楠, 张久祎, 等. 米曲霉3.042尿苷/尿嘧啶营养缺陷型遗传转化体系的构建[J]. 食品研究与开发,2021,42(3):189−195. [BI F T, SHI Y N, ZHANG J Y, et al. Construction of Aspergillus oryzae 3.042 uridine/uracil auxotrophy genetic transformation system[J]. Food Research and Development,2021,42(3):189−195.] doi: 10.12161/j.issn.1005-6521.2021.03.031 BI F T, SHI Y N, ZHANG J Y, et al. Construction of Aspergillus oryzae 3.042 uridine/uracil auxotrophy genetic transformation system[J]. Food Research and Development, 2021, 42(3): 189−195. doi: 10.12161/j.issn.1005-6521.2021.03.031
[17] YANG Y, SUN Q, LIU Y, et al. Development of a pyrF-based counterselectable system for targeted gene deletion in Streptomyces rimosus[J]. Journal of Zhejiang University Science B,2021,22(5):383−396. doi: 10.1631/jzus.B2000606
[18] WANG C, ZHOU H, GUO F, et al. Oral immunization of chickens with Lactococcus lactis expressing cjaA temporarily reduces Campylobacter jejuni colonization[J]. Foodborne Pathogens and Disease,2020,17(6):366−372. doi: 10.1089/fpd.2019.2727
[19] SHA Z, SHANG H, MIAO Y, et al. Recombinant Lactococcus lactis expressing M1-HA2 fusion protein provides protective mucosal immunity against H9N2 avian influenza virus in chickens[J]. Frontiers in Veterinary Science,2020,7:153. doi: 10.3389/fvets.2020.00153
[20] 韦云莹, 王立峰, 熊智强, 等. 响应面法优化乳酸乳球菌电转化效率研究[J]. 上海理工大学学报,2018,40(6):566−571. [WEI W Y, WANG L F, XIONG Z Q, et al. Optimization of electroporation efficiency of Lactococcus lactis using response surface methodology[J]. Journal of University of Shanghai for Science and Technology,2018,40(6):566−571.] WEI W Y, WANG L F, XIONG Z Q, et al. Optimization of electroporation efficiency of Lactococcus lactis using response surface methodology[J]. Journal of University of Shanghai for Science and Technology, 2018, 40(6): 566−571.
[21] 任志娟, 李轶杰, 马正海. 乳酸乳球菌质粒提取方法的改进与优化[J]. 新疆农业科学,2011,48(4):734−738. [REN Z J, LI Y J, MA Z H. Improvement and optimization of a method for extraction of plasmid from Lactococcus lactis[J]. Xinjiang Agricultural Sciences,2011,48(4):734−738.] doi: 10.6048/j.issn.1001-4330.2011.04.026 REN Z J, LI Y J, MA Z H. Improvement and optimization of a method for extraction of plasmid from Lactococcus lactis[J]. Xinjiang Agricultural Sciences, 2011, 48(4): 734−738. doi: 10.6048/j.issn.1001-4330.2011.04.026
[22] 邓木兰, 梁志成, 梁秀怡, 等. 基于URA3基因快速构建表达载体及其在里氏木霉的应用[J]. 微生物学通报,2020,47(2):649−658. [DENG M L, LIANG Z C, LIANG X Y, et al. Rapid construction of an expression vector based on URA3 gene for application in Trichodermium reesei[J]. Microbiology China,2020,47(2):649−658.] DENG M L, LIANG Z C, LIANG X Y, et al. Rapid construction of an expression vector based on URA3 gene for application in Trichodermium reesei[J]. Microbiology China, 2020, 47(2): 649−658.
[23] 崔月倩, 王菁蕊, 王艳萍. 乳酸菌基因表达载体及其应用研究进展[J]. 食品科学,2015,36(9):224−229. [CUI Y Q, WANG J R, WANG Y P. A review of research on lactic acid bacteria vectors for gene expression and their applications[J]. Food Science,2015,36(9):224−229.] doi: 10.7506/spkx1002-6630-201509042 CUI Y Q, WANG J R, WANG Y P. A review of research on lactic acid bacteria vectors for gene expression and their applications[J]. Food Science, 2015, 36(9): 224−229. doi: 10.7506/spkx1002-6630-201509042
[24] 王莹, 胡桂学. 食品级乳酸菌表达系统的研究进展[J]. 中国畜牧兽医,2010,37(7):80−83. [WANG Y, HU G X. Advance in food-grade lactic acid bacteria expression system[J]. China Animal Husbandry & Veterinary Medicine,2010,37(7):80−83.] WANG Y, HU G X. Advance in food-grade lactic acid bacteria expression system[J]. China Animal Husbandry & Veterinary Medicine, 2010, 37(7): 80−83.
[25] BRAVO D, LANDETE J M. Genetic engineering as a powerful tool to improve probiotic strains[J]. Biotechnology & Genetic Engineering Reviews,2017,33(2):173−189.
[26] LANDETE J M. A review of food-grade vectors in lactic acid bacteria:from the laboratory to their application[J]. Critical Reviews in Biotechnology,2017,37(3):296−308. doi: 10.3109/07388551.2016.1144044
[27] TAGLIAVIA M, NICOSIA A. Advanced strategies for food-grade protein production:A new E. coli/lactic acid bacteria shuttle vector for improved cloning and food-grade expression[J]. Microorganisms,2019,7(5):116. doi: 10.3390/microorganisms7050116
[28] DICKELY F, NILSSON D, HANSEN E B, et al. Isolation of Lactococcus lactis nonsense suppressors and construction of a food-grade cloning vector[J]. Molecular Microbiology,1995,15(5):839−847. doi: 10.1111/j.1365-2958.1995.tb02354.x
[29] SORENSEN K I, LARSEN R, KIBENICH A, et al. A food-grade cloning system for industrial strains of Lactococcus lactis[J]. Applied and Environmental Microbiology,2000,66(4):1253−1258. doi: 10.1128/AEM.66.4.1253-1258.2000
[30] 郝凤奇, 杨桂连, 叶丽萍, 等. “食品级”乳酸菌表达载体系统的研究进展[J]. 中国预防兽医学报,2008,30(10):824−830. [HAO F Q, YANG G L, YE L P, et al. Advance in food-grade lactic acid bacteria expression system[J]. Chinese Journal of Preventive Veterinary Medicine,2008,30(10):824−830.] HAO F Q, YANG G L, YE L P, et al. Advance in food-grade lactic acid bacteria expression system[J]. Chinese Journal of Preventive Veterinary Medicine, 2008, 30(10): 824−830.
[31] 周陈力, 万佳宁, 卢绪志, 等. 灵芝尿嘧啶营养缺陷型菌株的筛选和分子鉴定[J]. 微生物学通报,2019,46(10):2657−2664. [ZHOU C L, WAN J N, LU X Z, et al. Screening and molecular verification of uracil auxotrophic mutants of Ganoderma lucidum[J]. Microbiology China,2019,46(10):2657−2664.] ZHOU C L, WAN J N, LU X Z, et al. Screening and molecular verification of uracil auxotrophic mutants of Ganoderma lucidum[J]. Microbiology China, 2019, 46(10): 2657−2664.
[32] 杨镒婴, 李晓乐, 李子龙, 等. 反选标记法在微生物基因敲除中的应用[J]. 中国生物工程杂志,2023,43(4):101−111. [YANG Y Y, LI X L, LI Z L, et al. Application of the efficient counter-selection method in gene deletion of microorganism[J]. China Biotechnology,2023,43(4):101−111.] YANG Y Y, LI X L, LI Z L, et al. Application of the efficient counter-selection method in gene deletion of microorganism[J]. China Biotechnology, 2023, 43(4): 101−111.
[33] KUIPERS O P, BEERTHUYZEN M M, DE RUYTER P G, et al. Autoregulation of nisin biosynthesis in Lactococcus lactis by signal transduction[J]. The Journal of Biological Chemistry,1995,270(45):27299−27304. doi: 10.1074/jbc.270.45.27299





 下载:
下载:
 下载:
下载:



