Application Progresses on Near-infrared Spectroscopy in Quality Detection of Edible Fungi
-
摘要: 食用菌是一类高蛋白、低脂肪且兼具药用价值的大型真菌,因其多营养、鲜风味、低热量、多功效等特点而成为餐桌上常备食材之一。随着消费理念不断转变,食用菌的高品质和高营养需求成为必然。近红外光谱(near-infrared spectroscopy,NIRS)技术因其快捷、无损、多组分同时检测、绿色无污染等优点,被广泛应用于食用菌品质检测研究。本文全面归纳综述了近5年来NIRS技术结合化学计量学方法在食用菌理化组分、活性成分、品种鉴别、产地溯源、病原菌污染、掺假识别等及其他方面的研究及应用进展,同时提出了NIRS技术在食用菌品质检测应用方面的发展策略,以期为完善NIRS技术检测食用菌理论、研发专用食用菌检测设备提供方法参考和思路借鉴。Abstract: Edible fungi is a kind of large fungi with high protein, low fat and medicinal value. It has become one of the common food ingredients on the table because of its characteristics of more nutrition, fresh flavor, low calories and multi-functions. With the continuous upgrading of the consumption concept, high quality and nutrition have become necessary requirements for edible fungi consumption. Near-infrared spectroscopy (NIRS) technology has been widely used in the quality detection of edible fungi because of its advantages of fast, non-destructive, multi-component simultaneous detection, green and pollution-free. This paper comprehensively summarizes the research and application progresses of NIRS technology combined with chemometrics in quality evaluation of edible fungi in terms of physical and chemical components, active ingredients, variety identification, origin tracing, pathogen contamination and adulteration recognition in recent five years. At the same time, the development strategy of NIRS technology in the quality detection of edible fungi is proposed, which will provide methodology and ideas reference for improving the detection theory of NIRS technology and developing special testing equipment.
-
Keywords:
- near-infrared (NIR) /
- chemometrics /
- edible fungi /
- quality /
- inspection
-
食用菌是一类风味独特且富含蛋白质、氨基酸、多糖、矿质元素及维生素等多种营养成分的大型可食用真菌,是目前国际上公认的理想蛋白质和组织营养来源[1]。食用菌还兼具药用价值,其含有的生理活性物质如多糖具有调节机体免疫、预防心血管疾病、抗炎防癌等保健功效[2−4],已成为日常生活中常备食材之一。我国是食用菌生产和消费大国,2020年全国食用菌总产量高达4061.43万吨[5],食用菌产业已成为大健康产业的重要组成部分。随着健康消费理念普及和消费需求增长,高品质的食用菌及其制品受到当今消费者的普遍青睐。品质检测是食用菌产业化生产和产品质量控制的必备环节。近红外光谱(near-infrared spectroscopy,NIRS)技术作为一种快捷、高效、绿色无污染的无损检测手段,被大量用于食品品质的快速检测研究[6−8]。
红外光谱是介于可见光区和微波区之间的电磁波谱,其光谱的产生是由于分子振动的非谐振性,使分子从基态向高能级跃迁。NIRS技术能够进行定性和定量分析的实质是利用原子间的相对振动和分子转动等信息以确定分子结构。用于食品质量检测主要是基于食品组分中化学基团(主要指C-H、N-H、O-H等含氢基团)在近红外波段(780~2526 nm)的倍频和合频吸收[9],通过化学计量学算法挖掘光谱吸收信息,构建高稳定性、高精度的数学模型(线性模型和非线性模型)实现对未知样品的快速预测。食品样品在近红外波段的吸收谱峰重叠严重,且易受背景、噪声和外界环境等因素干扰,需借助化学计量学方法挖掘光谱信息与研究目标之间的内在联系[10]。化学计量学算法旨在通过寻找特征光谱信息与品质指标之间的相关性,发现内在规律并实现较合理准确的未知预测(定性和定量),在NIRS技术用于食品研究中发挥着重要作用[11]。构建的预测或识别模型需通过相关系数(R)或决定系数(R2)、均方根误差(root mean square error,RMSE)以及剩余预测偏差(residual predictive deviation,RPD)等参数进行评价。一般而言,R或R2越接近1,RMSE越接近于0,模型预测性能越好[12]。
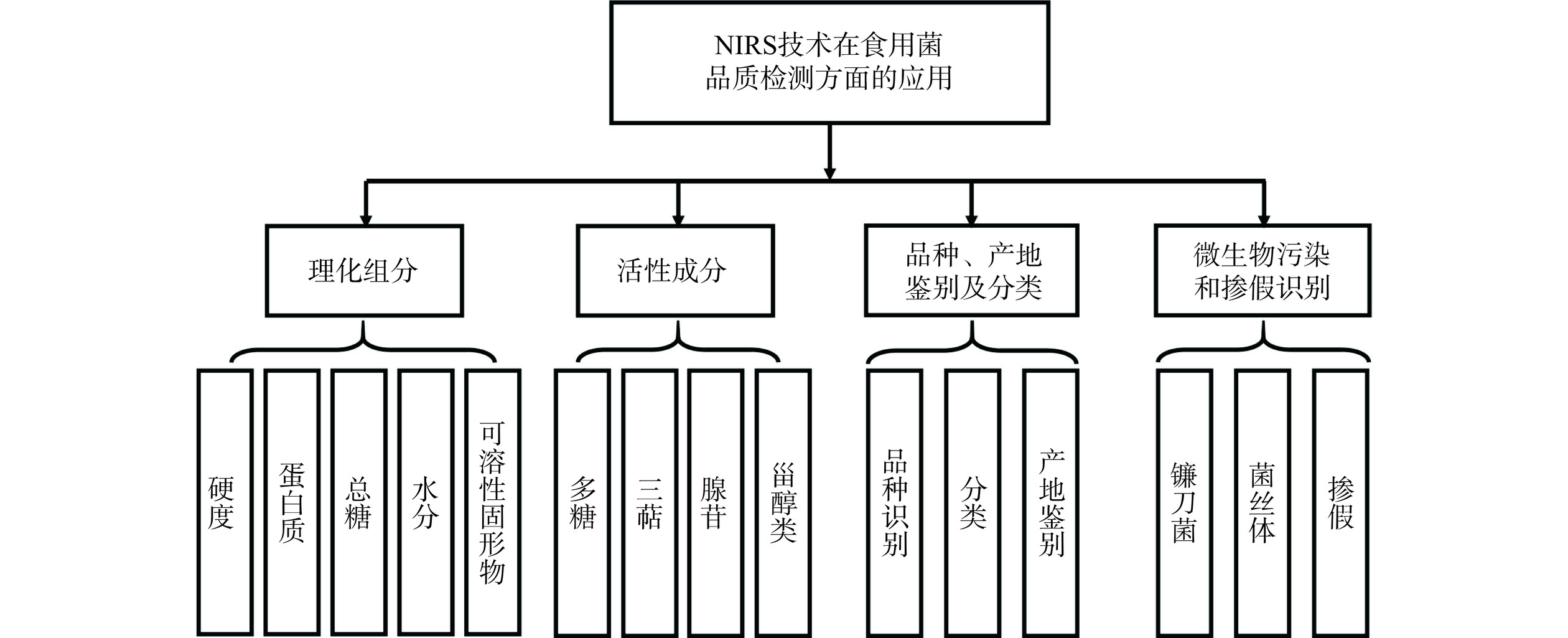
NIRS技术主要具有如下特点:a.无损快速:可直接利用近红外光的强穿透性对样品进行光谱采集,再将测量光谱代入建立好的校正模型,可完成对物质快速分析检测。b.绿色环保:光谱技术无需使用化学试剂对样品进行处理,降低各种生物、化学污染。c.重现性好:NIRS是一种二次分析检测技术,通过建立稳定的预测模型,由该模型对相关指标进行快速检测,所得结果具有较高的重现性。NIRS技术结合化学计量学算法已被用于研究食品种类及产地鉴别[13]、掺假[14]、质量评估[15]、等级分级[16]等,研究结果显示NIRS技术在部分指标检测应用方面具有较高可行性和适用性,应用潜力巨大。本文全面归纳综述了近5年来NIRS技术在食用菌品质检测方面的应用研究进展,主要包括理化组分(如水分、糖、硬度、固形物含量等)、活性成分(如多糖、三萜类、甾醇类等)、品种识别、产地鉴别和其他方面(图1),为进一步完善NIRS技术在食用菌质控中的应用提供方法参考。
1. NIRS技术用于检测食用菌理化组分
食用菌的化学组成主要包括水分、蛋白质、脂肪、碳水化合物、矿物质和维生素等。这些理化组分会影响食用菌的生产加工及食用品质,如水分含量直接影响食用菌的嫩度、鲜度和口感。食用菌理化组分的检测目前使用国标法较多[17−18],NIRS技术被用于食用菌检测研究已有报道,结果各不相同。如Giovenzana等[19]分别采集双孢菇菌帽和菌柄的400~1000 nm光谱信息,构建偏最小二乘(partial least squares,PLS)模型预测不同部位的硬度、可溶性固形物和水分等理化品质,结果显示在菌帽中,硬度的预测效果良好,而其他两个指标不是很理想(R2P:硬度>水分>可溶性固形物);在菌柄中,水分的预测效果明显好于其他两个指标(R2P:水分>可溶性固形物>硬度),这可能由于菌帽和菌柄的不同理化组分对模型精度产生了影响。相比之下,Shinoda等[20]通过获取1450 nm处的光谱信息构建PLS模型预测香菇中水分含量效果更好,具体如表1所示。
表 1 NIRS技术用于检测食用菌理化组分Table 1. Application of NIRS technology for detection of physicochemical index in edible fungi食用菌 指标 光谱范围 样本量 光谱预处理方法 最优波长/波段 建模方法 模型性能 参考文献 双孢菇 硬度/可溶性固形物/水分 400~1000 nm 248 Smooth+SNV — PLS 菌帽硬度
R2P =0.78
RMSEP=8.81%
菌帽可溶性固形物
R2P =0.33
RMSEP=1.39°Brix
菌帽水分
R2P =0.55
RMSEP=3.12%
菌柄硬度
R2P =0.41
RMSEP=13.10%
菌柄可溶性固形物
R2P =0.65
RMSEP=1.32°Brix
菌柄水分
R2P=0.78
RMSEP=2.14%[19] 香菇 水分 1450 nm 48 None — PLS R2>0.85
SEC=3.1%~8.2%[20] 蛹虫草 蛋白质 900~1700 nm 278 None — PLS RP=0.9774
RMSECV=0.0222 g·g−1
RMSEP=0.5670 g·g−1[21] 黑木耳 总糖 1000~2500 nm 166 SNV+(2nd Der) 1000~2500 nm PLS R2C=0.9092
RMSEC=1.405%
R2P=0.9048
RMSEP=1.507%
RPD=3.32[22] 香菇 总糖 1000~2500 nm 106 MSC+(2nd Der) 1000~2500 nm PLS R2C=0.94004
RMSEC=1.393%
RMSEP=1.557%
RPD=4.08[23] 注:RC:校正集相关系数;RCV:交叉验证集相关系数;R2CV:交叉验证集决定系数;RP:验证集相关系数;R2C:校正集决定系数;R2P:预测集决定系数;SEP:标准偏差;RMSEC:校正集均方根误差;RMSECV:交叉验证集均方根误差;RMSEP:预测集均方根误差;PLS:偏最小二乘;2nd Der:二阶导数;SNV:标准正态变量变换;MSC:多元散射校正,表2~表5同。 食用菌富含蛋白质,营养价值高,是高品质蛋白质的理想来源。马建等[21]利用NIRS技术对278株蛹虫草样品的成分进行快速定量分析,经对比发现,采用原始光谱预测蛹虫草蛋白质含量效果最好。碳水化合物也是食用菌重要成分之一,是组成食用菌所占比重最高的大分子物质。为推动黑木耳精深加工和技术提升,杨根等[22]尝试基于1000~2500 nm波长信息研发一种快速检测黑木耳总糖的NIRS方法,结果显示构建的PLS预测模型R2超过0.90,效果良好。卢洁等[23]使用相同的光谱波段构建PLS模型预测香菇总糖含量,模型R2C最高达0.94004,模型验证结果显示,样品预测值和实测值具有良好的线性关系,该试验建立的NIRS分析模型可用于预测香菇样品的总糖含量。对比近5年研究结果发现,NIRS技术在预测食用菌总糖和蛋白质含量方面效果更好,但是总体上对于食用菌理化组分的NIRS检测研究较少。
2. NIRS技术用于检测食用菌活性成分
食用菌含有多糖、萜类、黄酮类等多种生理活性成分,具有提升机体免疫力、阻止和防御疾病等功效。活性成分的快速检测对食用菌的药理开发及应用具有重要意义。食用菌多糖是食用菌细胞体内的一类大分子物质,具有良好的降血糖功效[24]。张龙等[25]研究了NIRS快速检测杏鲍菇多糖含量的可行性,结合三种算法构建多糖的定量校正模型,结果显示径向基神经网络模型预测效果最好(R2P=0.89,RMSEP=7.73 mg/g),但仍有提升空间,这为快速检测杏鲍菇多糖含量提供了初步依据。黑木耳是一种胶质食用菌,其多糖占比最大、含量最高[26]。孙丽萍等[27]以NIRS对样本进行光谱采集(900~1700 nm),运用支持向量机(support vector machines,SVM)算法进行建模仿真,对黑木耳多糖品质进行分类研究,测得模型识别精确率为85.7%,具有较好的预测性,但该研究采集样本总量略有不足。灵芝是一种药用真菌,其多糖是主要活性成分之一,可通过调节免疫系统增强宿主抗病降糖降脂能力[28]。赖长江生等[29]采用NIRS(1000~2500 nm)结合化学计量学算法构建灵芝多糖含量(质量分数为1.93%~3.99%)快速预测模型,通过标准正太变量校正(standard normal variate,SNV)光谱预处理所建立的PLS模型预测结果最佳,具备应用推广潜力。马玉涵等[30]研究发现灵芝多糖NIRS的特征吸收位点在2321.80、2270.15 nm,结合多糖化学测量结果,构建基于NIRS的灵芝菌丝体多糖定量分析模型,校正模型和预测模型表现非常好(R2均>0.95)。利用校正模型继续预测诱变灵芝菌丝体多糖含量,效果依然良好。因灵芝多糖大多存在于水提物中,为了更加准确全面评估灵芝多糖含量,张倩倩等[31]采用NIRS对灵芝子实体水提物进行定性和定量分析,结果显示基于水提物优选波段1830.93~2361.22 nm 构建的 NIRS 模型定量检测灵芝多糖效果良好,可以帮助解决灵芝子实体多糖定量不准的问题。
灵芝三萜是另一类重要的药用活性成分,具有广泛的抗癌特性[32]。研究发现灵芝三萜在近红外谱区内的1689.19、1730.10、2265、2304.15及2350.73 nm处有特征吸收峰,马玉涵等[30]通过选取两个波段1676.98~1766.07、2266.26~2298.40 nm的光谱信息构建PLS模型预测灵芝菌丝体中的灵芝三萜含量,经过一阶导数预处理光谱,模型预测效果更好。张倩倩等[33]通过采集灵芝子实体及其提取纯化物的NIRS(833.33~2500 nm),分析得出了灵芝三萜NIRS和中红外光谱(mid-infrared spectroscopy,MIRS)之间存在较强的相关性,并提出基于灵芝子实体提取物,可建立灵芝子实体三萜的NIRS定量分析模型。
蛹虫草含有腺苷、多糖、虫草酸等多种活性成分,在降血脂、抗菌、抗癌、抗衰老等方面功效显著[34−35]。快速定量分析蛹虫草有效成分可避免大量损耗,对开发蛹虫草的药用价值具有重要意义。马健等[21]通过获得化学诱变蛹虫草的NIRS,并经过不同的光谱预处理,分别构建了蛹虫草腺苷、虫草酸、多糖等的PLS定量分析模型,结果显示使用原始光谱(900~1700 nm)和预处理光谱在预测这三种活性成分方面效果差异不显著,建议直接使用原始光谱分析效率更高。除了定量预测,NIRS技术还被引入蛹虫草加工过程分析,用以提高生产效率,如史畑女等[36]对发酵虫草菌粉生产全过程中的总核苷、总多糖及甾醇类含量进行了NIRS定量检测研究,针对不同成分选择不同的波段信息构建PLS定量分析模型,通过模型参数(RC和RMSEC、RP和RMSEP)及模型验证(准确度、重复性、精密度、稳定性)得出结论,NIRS技术结合PLS算法用于对发酵虫草菌粉生产过程中核苷类成分、总多糖含量、甾醇类成分的定量检测效果良好(详见表2),模型应用具有可行性,可提高生产过程可控性和高效性。
表 2 NIRS技术用于检测食用菌活性成分Table 2. Application of NIRS technology for detection of active ingredients in edible fungi食用菌 指标 光谱范围 样本量 光谱预
处理方法最优波长/波段 建模方法 模型性能 参考文献 杏鲍菇 多糖 833.33~2500 nm 85 小波转换 1428.57~2500 nm RBF-NN R2C=1
RMSEC=9.02E-10
R2P=0.890075
RMSEP=7.728933 mg/g[25] 木耳 多糖 900~1700 nm — None — SVM 识别精确率85.7%
召回率87.3%
F1-score=0.864[27] 灵芝 多糖 1000~2500 nm 47 SNV — PLS R2C=0.9592
RMSEC=0.0185%
R2P=0.8516
RMSECP=0.0236%[29] 灵芝 多糖 1111.11~2500 nm 76 1st Der 1897.97~2500 nm PLS 灵芝菌丝体
R2C=0.9779
RMSECV=0.467%
R2P=0.9554
RMSEP=0.603%
RPD=3.13
诱变灵芝菌丝体
R2P=0.9684
RMSEP=0.445%
RPD=2.75[30] 灵芝 多糖 833.33~2500 nm 70 矢量归一法+(1st Der) 1830.93~2361.22 nm PLS R2C=0.9007
RMSECV=2.54%
R2P=0.9181
RMSEP=3.45%
RPD=3.17[31] 灵芝 三萜 1111.11~2500 nm — 1st Der (1676.98~1766.07)+
(2266.26~2298.40) nmPLS R2=0.8857
RMSECV=0.267 mg/g
RPD=4.74[30] 灵芝 三萜 833.33~2500 nm — 2nd Der — — 灵芝三萜NIRS和MIRS之间存在较强相关性 [33] 蛹虫草 虫草酸/
腺苷/
多糖900~1700 nm 278 SGS — PLS 虫草酸
RP=0.8807
RMSECV=0.0088 g.g−1
RMSEP=0.0082 g.g−1
腺苷
RP=0.9743
RMSECV=0.0232 mg/g
RMSEP=0.0189 mg/g
多糖
RP =0.8819
RMSECV=0.0086 g.g−1
RMSEP=0.0085 g.g−1[21] 蛹虫草 总核苷/
多糖/
甾醇类800~2500 nm 210 None 总核苷
(1366.68~1443.52)+
(1484~1544.12)+(1706.78~1810.48)+(2039.78~
2075.70)+(2109.49~
2434.33) nm
总多糖
(950~1200)+(1009.70~
1533.30)+(1500~2200) nm
甾醇类
1123.30~2500 nmPLS 总核苷
RC=0.9967
RMSEC=0.113 mg/g
RP =0.9923
RMSEP=0.157 mg/g
总多糖
RC =0.9418
RMSEC=0.0186%
RP =0.9800
RMSEP=0.0133%
甾醇类
RC =0.9626
RMSEC=0.0449 mg/g
RP =0.9872
RMSEP=0.0434 mg/g[36] 注:RBF-NN:径向基神经网络;SVM:支持向量机;1st Der:一阶导数;SGS:卷积平滑,表3~表5同。 3. NIRS技术用于检测食用菌品种鉴别及分类
食用菌品种繁多,其野生和人工栽培的功效差异较大,目前常用于食用菌品种鉴别及分类的方法主要有感官识别、色谱和质谱联用等[37−38]。感官识别人为主观因素影响较大,色谱和质谱联用前处理复杂、耗时耗力,无法实现快速检测。NIRS技术作为一种快捷有效的检测手段,在食用菌品种鉴别分类方面的应用研究具有重要现实意义。杨吉等[39]利用NIRS法结合主成分分析(PCA)及聚类分析对9种540个灵芝样品快速识别研究,分类归属正确识别率达100%,为灵芝药用功效评价提供了一种有效方法。王乾龙等[40]研究基于800~2500 nm波段信息构建快速鉴别冬虫夏草品级的可行性,通过采集西藏和青海两地的冬虫夏草的NIRS数据,经二阶导数预处理后进行主成分分析,构建的模型鉴别冬虫夏草品级准确率可达100%。李鱼强等[41]在900~1700 nm波长范围下,选取4种不同的食用菌构建反向传播神经网络模型和K最邻近算法模型,结果显示构建的两种模型对松茸真假辨别准确率可达100%和78.26%,对食用菌分类正确率可达89.1%和80.43%,模型精度和稳定性均高于PCA法(详见表3)。
表 3 NIRS技术用于检测食用菌品种鉴别及分类分级Table 3. Application of NIRS technology for identification and classification of edible fungi食用菌 指标 光谱范围 样本量 光谱预
处理方法最优波长/波
段筛选方法最优波
长/波段建模方法 模型性能 参考
文献灵芝 品种识别 1000~
2500 nm540 MSC — — MSC-PCA 分类正确识别率达到100% [39] 冬虫夏草虫草 品种识别 800~2500 nm 30 2nd Der — — PCA 鉴别品级准确率达到100% [40] 松茸、杏鲍菇、老人头、姬松茸 品种识别
及分类900~
1700 nm166 SNV LASSO, PCA — BPNN,
KNN基于原始光谱鉴别松茸真伪正确率为69.57%
(BPNN)和60.87%(KNN)
基于原始光谱分类食用菌正确率为67.39%
(BPNN)和65.22%(KNN)
基于特征波长鉴别松茸正确率为100%
(BPNN)和78.26%(KNN)
基于特征波长分类食用菌正确率为89.1%
(BPNN)和80.43%(KNN)[41] 牛肝菌 品种识别 1000~
2500 nm418 SGS, 去趋势,
2nd Der— 1428.57~
2500 nmPLS-DA, ResNet 两种模型的识别准确率均达到100% [42] 牛肝菌 品种鉴别 1000~
2500 nm801 SNV, MSC, 1st Der, 2nd Der, SGS — — PLS-DA, SVM, ResNet PLS-DA、SVM和ResNet对训练集样本的正确识别率分别为99.63%、98.5%和100%;对测试集样本的正确识别率分别为97.38%、93.63%和100% [43] 牛肝菌 品种鉴别 1000~
2500 nm187 2D-COS — 1428.57~
2500 nmResNet 准确率为100% [44] 注:PCA:主成分分析;BPNN:返向传播神经网络;KNNK:最邻近算法;2D-COS:二维相关光谱;ResNet:残差卷积神经网络,表4~表5同。 除了上述几种食用菌,近几年牛肝菌品种鉴别也被研究报道。Chen等[42]试图开发一种基于傅里叶变换NIRS的牛肝菌品种快速鉴别方法,通过预处理5种不同品种牛肝菌的1000~2500 nm光谱信息,构建偏最小二乘法判别分析(partial least squares discriminant analysis,PLS-DA)和构建残差卷积神经网络(ResNet)两种识别模型。结果显示这两种模型均能100%识别牛肝菌试样品种,但是ResNet模型更容易操作,效率更高,NIRS结合ResNet更具有应用潜质。与Chen等[42]同一课题组Yan等[43]基于相同的光谱信息,继续对比了PLS-DA、SVM和ResNet三种模型在5种常食用牛肝菌品种方面的识别能力,结果依然是ResNet表现最佳,识别准确率依然为100%。傅里叶NIRS结合二维相关光谱(two-dimensional correlation spectroscopy,2D-COS)算法,以总多酚含量为鉴别参数构建ResNet模型识别云南野生牛肝菌试样品种,正确识别率也可达100%[44]。
4. NIRS技术用于检测食用菌产地鉴别
我国食用菌种植范围广,不同产地间的食用菌品质差异很大,市场上售价也因此存在差别,有些不法商家为了追逐更高经济利益,混淆食用菌产地。为促进食用菌市场稳健发展,迫切需要建立一种准确、快速、高效的食用菌产地鉴别技术。NIRS技术以便捷无损等优点在替代常规技术手段方面具有很大潜力。夏珍珍等[45]采集3个不同产地干香菇样品的NIRS(1000~2500 nm)结合不同的光谱预处理方法和波长选择方法,建立了香菇产地鉴别模型,结果表明基于小波变换(continuous wavelet transform,CWT)预处理和随机测试(randomization test,RT)筛选特征波长构建的PLS-DA模型鉴别正确率更高(96.97%),但该模型仅覆盖3个香菇主产地,模型适用性有限,后续研究应扩大样品覆盖范围。在相同的波长范围内,Fu等[46]对来自15个产地的灵芝样品进行了NIRS分类鉴别研究,采用一对一最小二乘支持向量机模型与SNV预处理相结合使得产地鉴别正确率达93.17%。依然使用1000~2500 nm波段信息,赖长江生等[29]则选择利用随机森林(random forest,RF)算法对来自4个产地的灵芝进行判别分析研究,训练集预测准确率达到96.87%,独立测试集判别准确率达到93.33%,与Fu等[46]的研究结果相似。这说明基于1000~2500 nm谱区的NIRS技术结合化学计量学能够较准确鉴别灵芝产地。
对于牛肝菌产地鉴别,云南农业大学一课题组使用同一波段光谱信息(1000~2500 nm)进行了多项研究,胡翼然等[47−48]先后对绒柄牛肝菌产地(云南4个气候带)和美味牛肝菌产地(云南8个产地)进行了NIRS鉴别分析。对于绒柄牛肝菌而言,基于特征变量的高级数据融合策略结合RF产地鉴别正确率更高,为99.6%;对于美味牛肝菌而言,基于特征变量的中级数据融合策略结合PLS-DA产地鉴别正确率更高,为100%。陈凤霞等[49]对美味牛肝菌也做了产地鉴别研究(云南6个产地),采用NIRS和紫外光谱中级数据融合后鉴别正确率有所提升,训练集正确率为100%,预测集正确率为92.31%,分类效果明显。Chen等[50]筛选使用最优波段光谱信息(1428.57~2500 nm)构建ResNet模型鉴别亚洲兰茂牛肝菌产地,训练集和预测集正确率均为100%,外部验证集正确率为97.92%。总之,近年来的食用菌产地鉴别研究均基于1000~2500 nm波段信息,尽管构建识别模型的方法不同,但总体的鉴别正确率很高,这些研究结果可为进一步开发食用菌产地溯源技术提供方法借鉴(表4)。
表 4 NIRS技术用于检测食用菌产地鉴别Table 4. Application of NIRS technology for origin identification of edible fungi食用菌 指标 光谱范围 样本量 光谱预
处理方法最优波长/波段筛选方法 最优波长/
波段建模方法 模型性能 参考
文献香菇 产地鉴别 1000~2500 nm 113 1st Der, CWT, MSC, SNV RT — PLS-DA 基于原光谱+RT的PLS-DA模型正确鉴别率为95.96%
基于CWT+RT的PLS-DA模型正确鉴别率为96.97%[45] 灵芝 产地鉴别 1000~2500 nm 25 SNV — — IC-OVO-LS-SVM 鉴别正确率为93.17% [46] 灵芝 产地鉴别 1000~2500 nm 47 — — — RF 训练集正确率为96.87%, 测试集正确率为93.33% [29] 牛肝菌 产地鉴别 1000~2500 nm 87 — — — RF 基于单一光谱结合RF鉴别正确率为81.9%
基于特征变量的高级融合策略结合RF鉴别正确率为99.6%,灵敏性为0.969,特异性为0.986[47] 牛肝菌 产地鉴别 1000~2500 nm 141 — — — PLS-DA 基于特征变量的中级融合策略结合PLS-DA鉴别正确率为100% [48] 牛肝菌 产地鉴别 1000~2500 nm 79 SGS+2nd Der, SGS+SNV — — PLS-DA 基于为NIRS和紫外光谱的低级数据融合鉴别正确率分别为,训练集100%,预测集88.46%
基于为NIRS和紫外光谱的中级数据融合鉴别正确率分别为,训练集100%,预测集92.31%[49] 牛肝菌 产地鉴别 1000~2500 nm 497 — — 1428.57~
2500 nmResNet 训练集和预测集正确率均为100.00%,外部验证集正确率为97.92% [50] 注:CWT:小波变换;RT:随机测试;IC-OVO-LS-SVM:一对一最小二乘支持向量机;RF:随机森林。 5. NIRS技术用于检测食用菌病原菌污染和掺假识别
除了以上四个方面的研究,NIRS技术还用于检测食用菌的病原菌污染、掺假等方面。刘晨等[51]基于傅里叶NIRS技术建立了木耳受镰刀菌(5种常见菌)侵染程度的定性与定量分析方法,线性判别分析(LDA)模型定性分析总体正确率在80%以上,单一镰刀菌侵染木耳样品中,串珠镰刀菌的PLS定量预测效果最好(R2P=0.902,RMSEP=0.187 lg CFU/g),5种镰刀菌混合侵染木耳样品的PLS定量预测性能有所降低,详见表5。Shi等[52]探讨了NIRS技术结合化学计量学方法快速检测灵芝孢子粉掺假(掺入干淀粉)的可行性,建立回归模型计算灵芝孢子粉中淀粉的掺假量,结果显示基于SNV预处理构建的PLS模型预测性能略好于ANN模型。Yang等[53]利用NIRS技术识别杏鲍菇菌丝体的6个生长阶段,竞争性自适应加权算法(CARS)筛选最优波长构建的BPNN模型识别准确率可达99.67%。这些研究结果均说明NIRS技术在食用菌检测方面具有很大的潜力,可为进一步拓展NIRS技术应用提供数据支撑。
表 5 NIRS技术用于检测食用菌病原菌污染和掺假识别Table 5. Application of NIRS technology for detection of contamination and adulterants in edible fungi食用菌 指标 光谱范围 样本量 光谱预
处理方法最优波长/
波段筛选方法最优波
长/波段建模方法 模型性能 参考
文献木耳 镰刀菌 1000~2500 nm 120 MSC, 2nd Der — — LDA定性, PLS定量 LDA模型:
建模集和验证集的正确率分别为 85.8%和 80%
单一镰刀菌PLS预测模型:
R2P=0.825~0.923
RMSEP=0.187~0.406 lg CFU/g
RPD=2.85~3.57
5种镰刀菌PLS预测模型:
R2P =0.849
RMSEP=0.364 lg CFU/g
RPD=3.14[51] 灵芝 掺假 1000~2500 nm 59 平滑, 1st Der, 2nd Der, MSC, SNV, 2nd Der+SNV SI-PLS — PLS, ANN PLS模型
R2C=0.9994
RMSECV=0.9218%
R2P =0.9938
RMSEP=3.1127%
ANN模型
R2C =0.9999
RMSECV=0.0116%
R2P =0.9975
RMSEP=4.3591%[52] 杏鲍菇 菌丝体 865.80~
2531.65 nm86 MSC CARS 86个 BPNN 生长阶段鉴别正确率99.67% [53] 注:LDA:线性判别分析;CARS:竞争性自适应加权算法。 6. 结论与展望
纵观NIRS技术在食用菌检测方面的成果可以看出,NIRS技术在食用菌生物活性成分检测和产地鉴别方面研究较多,这也间接反映了消费者更加注重健康饮食的理念。从各项研究结果看出,NIRS技术具备在食用菌品质检测方面的应用可行性,但仍存在以下问题有待进一步研究:a.由于食用菌成分复杂,其近红外光谱的数据有效信息较为分散,干扰信号较多,模型的准确度和稳定性有待提高。b.在定量分析中,多数研究的样品为干样或粉质产品,适用范围具有局限性,可进一步研究生鲜食用菌产品。c.目前食用菌的近红外快速检测研究,基本处于可行性探索,系统性研究较少。未来将试验模型应用在实际检测工作中,仍需多方面的探索。未来的研究可以选用大量且有代表性的样品,并扩充样品集的种类和数量,对建模的分析软件加以优化等方法来提高模型的准确度和稳定性。
目前市面上已有成熟稳定的NIRS检测设备,可实现对部分食品的快速检测,但大多为进口设备,自主研发设备甚少,尤其针对食用菌品质检测的设备几乎没有。当前市场对快检设备需求依旧旺盛,针对某种或某类食品的专业化NIRS检测设备依然短缺,如目前还未发现可以用于食用菌品质快检的专用NIRS设备。未来深化NIRS技术快速检测食用菌研究应从以下几个方面重点考虑:a.我国食用菌品种繁多,因产地不同导致理化组分差异大,可根据不同检测指标采集代表性样品,构建可实现地方标准或企业标准的快速检测模型。b.智能化时代背景下,应融合最新的硬件和软件技术,开发新的最佳波长筛选算法,构建更加稳健可靠的食用菌检测模型。c.近红外数据融合化学计量学算法后的模型稳定转化、模型集成、小型便捷专用化食用菌检测设备研发及生产应用依然是未来发展趋势。
-
表 1 NIRS技术用于检测食用菌理化组分
Table 1 Application of NIRS technology for detection of physicochemical index in edible fungi
食用菌 指标 光谱范围 样本量 光谱预处理方法 最优波长/波段 建模方法 模型性能 参考文献 双孢菇 硬度/可溶性固形物/水分 400~1000 nm 248 Smooth+SNV — PLS 菌帽硬度
R2P =0.78
RMSEP=8.81%
菌帽可溶性固形物
R2P =0.33
RMSEP=1.39°Brix
菌帽水分
R2P =0.55
RMSEP=3.12%
菌柄硬度
R2P =0.41
RMSEP=13.10%
菌柄可溶性固形物
R2P =0.65
RMSEP=1.32°Brix
菌柄水分
R2P=0.78
RMSEP=2.14%[19] 香菇 水分 1450 nm 48 None — PLS R2>0.85
SEC=3.1%~8.2%[20] 蛹虫草 蛋白质 900~1700 nm 278 None — PLS RP=0.9774
RMSECV=0.0222 g·g−1
RMSEP=0.5670 g·g−1[21] 黑木耳 总糖 1000~2500 nm 166 SNV+(2nd Der) 1000~2500 nm PLS R2C=0.9092
RMSEC=1.405%
R2P=0.9048
RMSEP=1.507%
RPD=3.32[22] 香菇 总糖 1000~2500 nm 106 MSC+(2nd Der) 1000~2500 nm PLS R2C=0.94004
RMSEC=1.393%
RMSEP=1.557%
RPD=4.08[23] 注:RC:校正集相关系数;RCV:交叉验证集相关系数;R2CV:交叉验证集决定系数;RP:验证集相关系数;R2C:校正集决定系数;R2P:预测集决定系数;SEP:标准偏差;RMSEC:校正集均方根误差;RMSECV:交叉验证集均方根误差;RMSEP:预测集均方根误差;PLS:偏最小二乘;2nd Der:二阶导数;SNV:标准正态变量变换;MSC:多元散射校正,表2~表5同。 表 2 NIRS技术用于检测食用菌活性成分
Table 2 Application of NIRS technology for detection of active ingredients in edible fungi
食用菌 指标 光谱范围 样本量 光谱预
处理方法最优波长/波段 建模方法 模型性能 参考文献 杏鲍菇 多糖 833.33~2500 nm 85 小波转换 1428.57~2500 nm RBF-NN R2C=1
RMSEC=9.02E-10
R2P=0.890075
RMSEP=7.728933 mg/g[25] 木耳 多糖 900~1700 nm — None — SVM 识别精确率85.7%
召回率87.3%
F1-score=0.864[27] 灵芝 多糖 1000~2500 nm 47 SNV — PLS R2C=0.9592
RMSEC=0.0185%
R2P=0.8516
RMSECP=0.0236%[29] 灵芝 多糖 1111.11~2500 nm 76 1st Der 1897.97~2500 nm PLS 灵芝菌丝体
R2C=0.9779
RMSECV=0.467%
R2P=0.9554
RMSEP=0.603%
RPD=3.13
诱变灵芝菌丝体
R2P=0.9684
RMSEP=0.445%
RPD=2.75[30] 灵芝 多糖 833.33~2500 nm 70 矢量归一法+(1st Der) 1830.93~2361.22 nm PLS R2C=0.9007
RMSECV=2.54%
R2P=0.9181
RMSEP=3.45%
RPD=3.17[31] 灵芝 三萜 1111.11~2500 nm — 1st Der (1676.98~1766.07)+
(2266.26~2298.40) nmPLS R2=0.8857
RMSECV=0.267 mg/g
RPD=4.74[30] 灵芝 三萜 833.33~2500 nm — 2nd Der — — 灵芝三萜NIRS和MIRS之间存在较强相关性 [33] 蛹虫草 虫草酸/
腺苷/
多糖900~1700 nm 278 SGS — PLS 虫草酸
RP=0.8807
RMSECV=0.0088 g.g−1
RMSEP=0.0082 g.g−1
腺苷
RP=0.9743
RMSECV=0.0232 mg/g
RMSEP=0.0189 mg/g
多糖
RP =0.8819
RMSECV=0.0086 g.g−1
RMSEP=0.0085 g.g−1[21] 蛹虫草 总核苷/
多糖/
甾醇类800~2500 nm 210 None 总核苷
(1366.68~1443.52)+
(1484~1544.12)+(1706.78~1810.48)+(2039.78~
2075.70)+(2109.49~
2434.33) nm
总多糖
(950~1200)+(1009.70~
1533.30)+(1500~2200) nm
甾醇类
1123.30~2500 nmPLS 总核苷
RC=0.9967
RMSEC=0.113 mg/g
RP =0.9923
RMSEP=0.157 mg/g
总多糖
RC =0.9418
RMSEC=0.0186%
RP =0.9800
RMSEP=0.0133%
甾醇类
RC =0.9626
RMSEC=0.0449 mg/g
RP =0.9872
RMSEP=0.0434 mg/g[36] 注:RBF-NN:径向基神经网络;SVM:支持向量机;1st Der:一阶导数;SGS:卷积平滑,表3~表5同。 表 3 NIRS技术用于检测食用菌品种鉴别及分类分级
Table 3 Application of NIRS technology for identification and classification of edible fungi
食用菌 指标 光谱范围 样本量 光谱预
处理方法最优波长/波
段筛选方法最优波
长/波段建模方法 模型性能 参考
文献灵芝 品种识别 1000~
2500 nm540 MSC — — MSC-PCA 分类正确识别率达到100% [39] 冬虫夏草虫草 品种识别 800~2500 nm 30 2nd Der — — PCA 鉴别品级准确率达到100% [40] 松茸、杏鲍菇、老人头、姬松茸 品种识别
及分类900~
1700 nm166 SNV LASSO, PCA — BPNN,
KNN基于原始光谱鉴别松茸真伪正确率为69.57%
(BPNN)和60.87%(KNN)
基于原始光谱分类食用菌正确率为67.39%
(BPNN)和65.22%(KNN)
基于特征波长鉴别松茸正确率为100%
(BPNN)和78.26%(KNN)
基于特征波长分类食用菌正确率为89.1%
(BPNN)和80.43%(KNN)[41] 牛肝菌 品种识别 1000~
2500 nm418 SGS, 去趋势,
2nd Der— 1428.57~
2500 nmPLS-DA, ResNet 两种模型的识别准确率均达到100% [42] 牛肝菌 品种鉴别 1000~
2500 nm801 SNV, MSC, 1st Der, 2nd Der, SGS — — PLS-DA, SVM, ResNet PLS-DA、SVM和ResNet对训练集样本的正确识别率分别为99.63%、98.5%和100%;对测试集样本的正确识别率分别为97.38%、93.63%和100% [43] 牛肝菌 品种鉴别 1000~
2500 nm187 2D-COS — 1428.57~
2500 nmResNet 准确率为100% [44] 注:PCA:主成分分析;BPNN:返向传播神经网络;KNNK:最邻近算法;2D-COS:二维相关光谱;ResNet:残差卷积神经网络,表4~表5同。 表 4 NIRS技术用于检测食用菌产地鉴别
Table 4 Application of NIRS technology for origin identification of edible fungi
食用菌 指标 光谱范围 样本量 光谱预
处理方法最优波长/波段筛选方法 最优波长/
波段建模方法 模型性能 参考
文献香菇 产地鉴别 1000~2500 nm 113 1st Der, CWT, MSC, SNV RT — PLS-DA 基于原光谱+RT的PLS-DA模型正确鉴别率为95.96%
基于CWT+RT的PLS-DA模型正确鉴别率为96.97%[45] 灵芝 产地鉴别 1000~2500 nm 25 SNV — — IC-OVO-LS-SVM 鉴别正确率为93.17% [46] 灵芝 产地鉴别 1000~2500 nm 47 — — — RF 训练集正确率为96.87%, 测试集正确率为93.33% [29] 牛肝菌 产地鉴别 1000~2500 nm 87 — — — RF 基于单一光谱结合RF鉴别正确率为81.9%
基于特征变量的高级融合策略结合RF鉴别正确率为99.6%,灵敏性为0.969,特异性为0.986[47] 牛肝菌 产地鉴别 1000~2500 nm 141 — — — PLS-DA 基于特征变量的中级融合策略结合PLS-DA鉴别正确率为100% [48] 牛肝菌 产地鉴别 1000~2500 nm 79 SGS+2nd Der, SGS+SNV — — PLS-DA 基于为NIRS和紫外光谱的低级数据融合鉴别正确率分别为,训练集100%,预测集88.46%
基于为NIRS和紫外光谱的中级数据融合鉴别正确率分别为,训练集100%,预测集92.31%[49] 牛肝菌 产地鉴别 1000~2500 nm 497 — — 1428.57~
2500 nmResNet 训练集和预测集正确率均为100.00%,外部验证集正确率为97.92% [50] 注:CWT:小波变换;RT:随机测试;IC-OVO-LS-SVM:一对一最小二乘支持向量机;RF:随机森林。 表 5 NIRS技术用于检测食用菌病原菌污染和掺假识别
Table 5 Application of NIRS technology for detection of contamination and adulterants in edible fungi
食用菌 指标 光谱范围 样本量 光谱预
处理方法最优波长/
波段筛选方法最优波
长/波段建模方法 模型性能 参考
文献木耳 镰刀菌 1000~2500 nm 120 MSC, 2nd Der — — LDA定性, PLS定量 LDA模型:
建模集和验证集的正确率分别为 85.8%和 80%
单一镰刀菌PLS预测模型:
R2P=0.825~0.923
RMSEP=0.187~0.406 lg CFU/g
RPD=2.85~3.57
5种镰刀菌PLS预测模型:
R2P =0.849
RMSEP=0.364 lg CFU/g
RPD=3.14[51] 灵芝 掺假 1000~2500 nm 59 平滑, 1st Der, 2nd Der, MSC, SNV, 2nd Der+SNV SI-PLS — PLS, ANN PLS模型
R2C=0.9994
RMSECV=0.9218%
R2P =0.9938
RMSEP=3.1127%
ANN模型
R2C =0.9999
RMSECV=0.0116%
R2P =0.9975
RMSEP=4.3591%[52] 杏鲍菇 菌丝体 865.80~
2531.65 nm86 MSC CARS 86个 BPNN 生长阶段鉴别正确率99.67% [53] 注:LDA:线性判别分析;CARS:竞争性自适应加权算法。 -
[1] ZHANG Y, WANG D, CHEN Y, et al. Healthy function and high valued utilization of edible fungi[J]. Food Science and Human Wellness,2021,10(4):408−420. doi: 10.1016/j.fshw.2021.04.003
[2] YIN Z, SUN-WATERHOUSE D, WANG J, et al. Polysaccharides from edible fungi Pleurotus spp. :Advances and perspectives[J]. Journal of Future Foods,2021,1(2):128−140. doi: 10.1016/j.jfutfo.2022.01.002
[3] LIU J, WANG Y, WU J, et al. Isolation, structural properties, and bioactivities of polysaccharides from mushrooms Termitomyces:A review[J]. Journal of Agriculture and Food Chemistry,2022,70(1):21−33. doi: 10.1021/acs.jafc.1c06443
[4] YIN Z, LIANG Z, LI C, et al. Immunomodulatory effects of polysaccharides from edible fungus:A review[J]. Food Science and Human Wellness,2021,10(4):393−400. doi: 10.1016/j.fshw.2021.04.001
[5] 中国食用菌协会. 2020年度全国食用菌统计调查结果分析[J]. 中国食用菌,2022,41(1):85−91. [China Edible Fungi Association. Analysis of the results of the 2020 national edible fungi statistical survey[J]. Edible Fungi of China,2022,41(1):85−91.] doi: 10.13629/j.cnki.53-1054.2022.01.017 China Edible Fungi Association. Analysis of the results of the 2020 national edible fungi statistical survey[J]. Edible Fungi of China, 2022, 41(1): 85−91. doi: 10.13629/j.cnki.53-1054.2022.01.017
[6] HE H J, WANG Y, ZHANG M, et al. Rapid determination of reducing sugar content in sweet potatoes using NIR spectra[J]. Journal of Food Composition and Analysis,2022,111:104641. doi: 10.1016/j.jfca.2022.104641
[7] CARVALHO L C, LEITE M L, MORAIS C, et al. Non-destructive assessment of the oxidative stability of intact macadamia nuts during the drying process by near-infrared spectroscopy[J]. LWT,2019,103:101−107. doi: 10.1016/j.lwt.2018.12.056
[8] SUNETTE M, MARK D, LEMBE S, et al. Development of NIRS models for rapid quantification of protein content in sweet potato [Ipomoea batatas (L.) LAM. ][J]. LWT,2016,72:63−70. doi: 10.1016/j.lwt.2016.04.032
[9] 曾斯杰, 马金爽, 王玥, 等. 基于近红外光谱技术快速检测青金桔果粉中微生物菌数[J]. 海南师范大学学报(自然科学版),2020,33(1):30−35. [ZENG S J, MA J S, WANG Y, et al. Detection of total microorganism counts in kumquat powder by near-infrared spectroscopy[J]. Journal of Hainan Normal University (Natural Science),2020,33(1):30−35.] doi: 10.12051/j.issn.1674-4942.2020.01.007 ZENG S J, MA J S, WANG Y, et al. Detection of total microorganism counts in kumquat powder by near-infrared spectroscopy[J]. Journal of Hainan Normal University (Natural Science), 2020, 33(1): 30−35. doi: 10.12051/j.issn.1674-4942.2020.01.007
[10] HE H J, WANG Y, OU X, et al. Rapid determination of chemical compositions in chicken flesh by mining hyperspectral data[J]. Journal of Food Composition and Analysis,2023,116:105069. doi: 10.1016/j.jfca.2022.105069
[11] ALEIXANDRE-TUDO J L, CASTELLO-COGOLLOS L, ALEIXANDRE J L, et al. Chemometrics in food science and technology:A bibliometric study[J]. Chemometrics and Intelligent Laboratory Systems,2022,222:104514. doi: 10.1016/j.chemolab.2022.104514
[12] TSEGAY G, AMMARE Y, MESFIN S. Development of non-destructive NIRS models to predict oil and major fatty acid contents of Ethiopian sesame[J]. Journal of Food Composition and Analysis,2023,115:104908. doi: 10.1016/j.jfca.2022.104908
[13] PAN S, ZHANG X, XU W, et al. Rapid on-site identification of geographical origin and storage age of tangerine peel by near-infrared spectroscopy[J]. Spectrochimica Acta Part A:Molecular and Biomolecular Spectroscopy,2022,271:120936. doi: 10.1016/j.saa.2022.120936
[14] HE H J, CHEN Y, WANG Y, et al. Hyperspectral imaging combined with chemometrics for rapid detection of talcum powder adulterated in wheat flour[J]. Food Control,2023,144:109378. doi: 10.1016/j.foodcont.2022.109378
[15] 曾斯杰, 刘红, 王友法, 等. 近红外光谱在食品工业应用的研究进展[J]. 中国调味品, 2019, 44(7):179−183. [ZENG S J, LIU H, WANG Y F, et al. Research progress on the application of near-infrared spectroscopy in food industry[J].China Condiment,2019, 44(7):179−183.] ZENG S J, LIU H, WANG Y F, et al. Research progress on the application of near-infrared spectroscopy in food industry[J].China Condiment,2019, 44(7): 179−183.
[16] ZHANG J, LIU G, LI Y, et al. Rapid identification of lamb freshness grades using visible and near-infrared spectroscopy (Vis-NIR)[J]. Journal of Food Composition and Analysis,2022,111:104590. doi: 10.1016/j.jfca.2022.104590
[17] BARROS L, BAPTISTA P, CORREIA D M, et al. Fatty acid and sugar compositions, and nutritional value of five wild edible mushrooms from Northeast Portugal[J]. Food Chemistry,2007,105:140−145. doi: 10.1016/j.foodchem.2007.03.052
[18] JACINTO-AZEVEDO B, VALDERRAMA N, HENRÍQUEZ K, et al. Nutritional value and biological properties of Chilean wild and commercial edible mushrooms[J]. Food Chemistry,2021,356:129651. doi: 10.1016/j.foodchem.2021.129651
[19] GIOVENZANA V, TUGNOLO A, CASSON A, et al. Application of visible-near infrared spectroscopy to evaluate the quality of button mushrooms[J]. Journal of Near Infrared Spectroscopy,2019,27(1):38−45. doi: 10.1177/0967033518811921
[20] SHINODA K, KONNO N, SUZUKI T. Non-destructive analysis of the moisture content in shiitake mushrooms (Lentinula edodes) using near-infrared imaging at 1450 nm[J]. Mycoscience,2020,61(5):235−239. doi: 10.1016/j.myc.2020.04.005
[21] 马健, 李尽哲. 基于近红外技术的蛹虫草成分快速定量分析研究[J]. 中国食用菌,2019,38(10):22−25. [MA J, LI J Z. Rapid quantitative analysis of Cordyceps militaris based on near-infrared technology[J]. Edible Fungl of China,2019,38(10):22−25.] doi: 10.13629/j.cnki.53-1054.2019.10.007 MA J, LI J Z. Rapid quantitative analysis of Cordyceps militaris based on near-infrared technology[J]. Edible Fungl of China, 2019, 38(10): 22−25. doi: 10.13629/j.cnki.53-1054.2019.10.007
[22] 杨根. 近红外和电子鼻技术在黑木耳功效成分的快速测定及产地鉴别中的应用[D]. 开封:河南大学, 2020. [YANG G. Application of near infrared and electronic nose technology in rapid determination of the effective components and origin identification of Auricularia auricular[D]. Kaifeng:Henan University, 2020.] YANG G. Application of near infrared and electronic nose technology in rapid determination of the effective components and origin identification of Auricularia auricular[D]. Kaifeng: Henan University, 2020.
[23] 卢洁, 田婧, 梁振华, 等. 近红外光谱法快速测定香菇总糖含量[J]. 食品科学,2021,42(12):189−194. [LU J, TIAN J, LIANG Z H, et al. Application of near infrared spectroscopy in the rapid detection of total sugar content in Lentinula edodes[J]. Food Science,2021,42(12):189−194.] doi: 10.7506/spkx1002-6630-20210115-167 LU J, TIAN J, LIANG Z H, et al. Application of near infrared spectroscopy in the rapid detection of total sugar content in Lentinula edodes[J]. Food Science, 2021, 42(12): 189−194. doi: 10.7506/spkx1002-6630-20210115-167
[24] GARCIA J, RODRIGUES F, SAAVEDRA M J, et al. Bioactive polysaccharides from medicinal mushrooms:A review on their isolation, structural characteristics and antitumor activity[J]. Food Bioscience,2022,49:101955. doi: 10.1016/j.fbio.2022.101955
[25] 张龙, 吴琦翡, 姜华年. 基于近红外光谱快速检测杏鲍菇多糖含量的研究[J]. 丽水学院学报,2018,40(5):30−35. [ZHANG L, WU Q F, JIANG H N. Rapid detection of polysaccharide content of Pleurotus eryngii based on near infrared spectroscopy[J]. Journal of Lishui University,2018,40(5):30−35.] doi: 10.3969/j.issn.2095-3801.2018.05.007 ZHANG L, WU Q F, JIANG H N. Rapid detection of polysaccharide content of Pleurotus eryngii based on near infrared spectroscopy[J]. Journal of Lishui University, 2018, 40(5): 30−35. doi: 10.3969/j.issn.2095-3801.2018.05.007
[26] MA J, QIAO Z, XIANG X. Optimisation of extraction procedure for black fungus polysaccharides and effect of the polysaccharides on blood lipid and myocardium antioxidant enzymes activities[J]. Carbohydrate Polymers,2011,84(3):1061−1068. doi: 10.1016/j.carbpol.2010.12.068
[27] 孙丽萍, 张希萌, 何睿, 等. 基于SVM的近红外黑木耳多糖含量分类[J]. 电子科技,2019,32(8):16−21. [SUN L P, ZHANG X M, HE R. Near-infrared scanning polysaccharide content classification of Auricularia auricular based on SVM[J]. Electronic Science and Technology,2019,32(8):16−21.] doi: 10.16180/j.cnki.issn1007-7820.2019.08.004 SUN L P, ZHANG X M, HE R. Near-infrared scanning polysaccharide content classification of Auricularia auricular based on SVM[J]. Electronic Science and Technology, 2019, 32(8): 16−21. doi: 10.16180/j.cnki.issn1007-7820.2019.08.004
[28] CHEN S, GUAN X, YONG T, et al. Structural characterization and hepatoprotective activity of an acidic polysaccharide from Ganoderma lucidum[J]. Food Chemistry:X,2022,13:100204. doi: 10.1016/j.fochx.2022.100204
[29] 赖长江生, 周融融, 余意, 等. 基于近红外分析和化学计量学方法对不同产地灵芝快速鉴别及多糖含量测定的研究[J]. 中国中药杂志,2018,43(16):3243−3248. [LAI C J S, ZHOU R R, YU Y, et al. Study on rapid identification and polysaccharide content determination of Ganoderma lucidum from different producing areas based on near infrared analysis and chemometrics[J]. China Journal of Chinese Materia Medica,2018,43(16):3243−3248.] doi: 10.19540/j.cnki.cjcmm.20180514.002 LAI C J S, ZHOU R R, YU Y, et al. Study on rapid identification and polysaccharide content determination of Ganoderma lucidum from different producing areas based on near infrared analysis and chemometrics[J]. China Journal of Chinese Materia Medica, 2018, 43(16): 3243−3248. doi: 10.19540/j.cnki.cjcmm.20180514.002
[30] 马玉涵. 等离子体诱变灵芝及其药用成分的红外光谱分析[D]. 合肥:中国科学技术大学, 2018. [MA Y H. Plasma mutagenesis of Ganoderma lingzhi and screening through infrared spectroscopy for medicinal components[D]. Hefei:University of Science and Technology of China, 2018.] MA Y H. Plasma mutagenesis of Ganoderma lingzhi and screening through infrared spectroscopy for medicinal components[D]. Hefei: University of Science and Technology of China, 2018.
[31] 张倩倩, 黄青. 近红外光谱测定灵芝子实体多糖含量[J]. 菌物学报,2021,40(1):252−260. [ZHANG Q Q, HUANG Q. Quantitative analysis of Ganoderma polysaccharides content in fruiting bodies by near-infrared spectroscopy[J]. Mycosystema,2021,40(1):252−260.] doi: 10.13346/j.mycosystema.200218 ZHANG Q Q, HUANG Q. Quantitative analysis of Ganoderma polysaccharides content in fruiting bodies by near-infrared spectroscopy[J]. Mycosystema, 2021, 40(1): 252−260. doi: 10.13346/j.mycosystema.200218
[32] CAO L, JIN H, LIANG Q, et al. A new anti-tumor cytotoxic triterpene from Ganoderma lucidum[J]. Natural Product Research,2022,36(16):4125−4131. doi: 10.1080/14786419.2021.1976175
[33] 张倩倩. 灵芝子实体三萜的红外光谱分析与研究[D]. 合肥:中国科学技术大学, 2020. [ZHANG Q Q. Analytical study of triterpenoids from Ganoderma lingzhi fruiting bodies through infrared spectroscopy[D]. Hefei:University of Science and Technology of China, 2020.] ZHANG Q Q. Analytical study of triterpenoids from Ganoderma lingzhi fruiting bodies through infrared spectroscopy[D]. Hefei: University of Science and Technology of China, 2020.
[34] YU Y, WEN Q, SONG A, et al. Isolation and immune activity of a new acidic Cordyceps militaris exopolysaccharide[J]. International Journal of Biological Macromolecules,2022,194:706−714. doi: 10.1016/j.ijbiomac.2021.11.115
[35] ZHANG X, ZHANG X, GU S, et al. Structure analysis and antioxidant activity of polysaccharide-iron (III) from Cordyceps militaris mycelia[J]. International Journal of Biological Macromolecules: Structure, Function and Interactions, 2021, 178:170−179.
[36] 史畑女. 发酵虫草菌粉生产过程近红外光谱分析技术质量控制应用及滤过工艺优化研究[D]. 南昌:江西中医药大学, 2020. [SHI T N. Study on quality control application of NIRS analysis technology and filtration process optimization in the production process of fermented Cordyceps sinensis[D]. Nanchang:Jiangxi University of Traditional Chinese Medicine, 2020.] SHI T N. Study on quality control application of NIRS analysis technology and filtration process optimization in the production process of fermented Cordyceps sinensis[D]. Nanchang: Jiangxi University of Traditional Chinese Medicine, 2020.
[37] WEI Y, LI L, LIU Y, et al. Identification techniques and detection methods of edible fungi species[J]. Food Chemistry,2022,374:131803. doi: 10.1016/j.foodchem.2021.131803
[38] ZHANG H, HUANG D, PU D, et al. Multivariate relationships among sensory attributes and volatile components in commercial dry porcini mushrooms (Boletus edulis)[J]. Food Research International,2020,133:109112. doi: 10.1016/j.foodres.2020.109112
[39] 杨吉, 冼玲, 钟天圣, 等. 近红外光谱指纹图谱的灵芝品种快速鉴别方法[J]. 时珍国医国药,2017,28(6):1359−1361. [YANG J, XIAN L, ZHONG T S, et al. Rapid identification of different kinds of Ganoderma lucidum based on near infrared spectroscopy fingerprint information[J]. LISHIZHEN Medicine and Materia Medica Research,2017,28(6):1359−1361.] doi: 10.3969/j.issn.1008-0805.2017.06.027 YANG J, XIAN L, ZHONG T S, et al. Rapid identification of different kinds of Ganoderma lucidum based on near infrared spectroscopy fingerprint information[J]. LISHIZHEN Medicine and Materia Medica Research, 2017, 28(6): 1359−1361. doi: 10.3969/j.issn.1008-0805.2017.06.027
[40] 王乾龙, 汪亚芳, 安丽灿, 等. 基于近红外高光谱技术的冬虫夏草品级快速鉴定[J]. 现代养生(下半月版),2018(10):62−64. [WANG Q L, WANG Y F, AN L C, et al. Rapid identification of Cordyceps sinensis grade based on near-infrared hyperspectral technology[J]. Health Protection and Promotion,2018(10):62−64.] doi: 10.3969/j.issn.1671-0223(x).2018.10.050 WANG Q L, WANG Y F, AN L C, et al. Rapid identification of Cordyceps sinensis grade based on near-infrared hyperspectral technology[J]. Health Protection and Promotion, 2018(10): 62−64. doi: 10.3969/j.issn.1671-0223(x).2018.10.050
[41] 李鱼强, 潘天红, 李浩然, 等. 近红外光谱LASSO特征选择方法及其聚类分析应用研究[J]. 光谱学与光谱分析,2019,39(12):3809−3815. [LI Y Q, PAN T H, LI H R, et al. NIR spectral feature selection using lasso method and its application in the classification analysis[J]. Spectroscopy and Spectral Analysis,2019,39(12):3809−3815.] doi: 10.3964/j.issn.1000-0593(2019)12-3809-07 LI Y Q, PAN T H, LI H R, et al. NIR spectral feature selection using lasso method and its application in the classification analysis[J]. Spectroscopy and Spectral Analysis, 2019, 39(12): 3809−3815. doi: 10.3964/j.issn.1000-0593(2019)12-3809-07
[42] CHEN J, LIU H, LI J, et al. A rapid and effective method for species identification of edible boletes:FT-NIR spectroscopy combined with ResNet[J]. Journal of Food Composition and Analysis,2022,112:104698. doi: 10.1016/j.jfca.2022.104698
[43] YAN Z, LIU H, LI T, et al. Two dimensional correlation spectroscopy combined with ResNet:Efficient method to identify bolete species compared to traditional machine learning[J]. LWT,2022,162:113490. doi: 10.1016/j.lwt.2022.113490
[44] CHEN X, LIU H G, LI J Q, et al. Rapid identification of total phenolic content levels in boletes by two-dimensional correlation spectroscopy combined with deep learning[J]. Vibrational Spectroscopy,2022,121:103404. doi: 10.1016/j.vibspec.2022.103404
[45] 夏珍珍, 郑丹, 夏虹, 等. 基于近红外光谱分析技术的栽培香菇产地快速鉴别[J]. 分析测试学报,2020,39(11):1371−1377. [XIA Z Z, ZHENG D, XIA H, et al. Rapid identification of shiitake geographical origins based on near infrared spectroscopy[J]. Journal of Instrumental Analysis,2020,39(11):1371−1377.] doi: 10.3969/j.issn.1004-4957.2020.11.009 XIA Z Z, ZHENG D, XIA H, et al. Rapid identification of shiitake geographical origins based on near infrared spectroscopy[J]. Journal of Instrumental Analysis, 2020, 39(11): 1371−1377. doi: 10.3969/j.issn.1004-4957.2020.11.009
[46] FU H, YIN Q, XU L, et al. A comprehensive quality evaluation method by FT-NIR spectroscopy and chemometric:Fine classification and untargeted authentication against multiple frauds for Chinese Ganoderma lucidum[J]. Spectrochimica Acta Part A: Molecular and Biomolecular Spectroscopy,2017,182:17−25. doi: 10.1016/j.saa.2017.03.074
[47] 胡翼然, 李杰庆, 刘鸿高, 等. 红外光谱的随机森林算法与数据融合策略对绒柄牛肝菌产地鉴别[J]. 光谱学与光谱分析,2020,40(5):1495−1502. [HU Y R, LI J Q, LIU H G, et al. Infrared spectral study on the origin identification of Boletus tomentipes based on the random forest algorithm and data fusion strategy[J]. Spectroscopy and Spectral Analysis,2020,40(5):1495−1502.] doi: 10.3964/j.issn.1000-0593(2020)05-1495-08 HU Y R, LI J Q, LIU H G, et al. Infrared spectral study on the origin identification of Boletus tomentipes based on the random forest algorithm and data fusion strategy[J]. Spectroscopy and Spectral Analysis, 2020, 40(5): 1495−1502. doi: 10.3964/j.issn.1000-0593(2020)05-1495-08
[48] 胡翼然, 李杰庆, 刘鸿高, 等. 红外光谱数据融合对美味牛肝菌产地鉴别[J]. 光谱学与光谱分析,2020,40(4):1276−1282. [HU Y R, LI J Q, LIU H G, et al. The origin identification study of Boletus edulis based on the infrared spctrum data fusion strategy[J]. Spectroscopy and Spectral Analysis,2020,40(4):1276−1282.] doi: 10.3964/j.issn.1000-0593(2020)04-1276-07 HU Y R, LI J Q, LIU H G, et al. The origin identification study of Boletus edulis based on the infrared spctrum data fusion strategy[J]. Spectroscopy and Spectral Analysis, 2020, 40(4): 1276−1282. doi: 10.3964/j.issn.1000-0593(2020)04-1276-07
[49] 陈凤霞, 杨天伟, 李杰庆, 等. 美味牛肝菌矿质元素富集能力及产地鉴别[J]. 食品科学,2021,42(10):290−296. [CHEN F X, YANG T W, LI J Q, et al. Mineral enrichment capacity and geographical origin identification of Boletus edulis[J]. Food Science,2021,42(10):290−296.] doi: 10.7506/spkx1002-6630-20191104-039 CHEN F X, YANG T W, LI J Q, et al. Mineral enrichment capacity and geographical origin identification of Boletus edulis[J]. Food Science, 2021, 42(10): 290−296. doi: 10.7506/spkx1002-6630-20191104-039
[50] CHEN X, LIU H, LI J, et al. A geographical traceability method for Lanmaoa asiatica mushrooms from 20 township-level geographical origins by near infrared spectroscopy and ResNet image analysis techniques[J]. Ecological Informatics,2022,71:101808. doi: 10.1016/j.ecoinf.2022.101808
[51] 刘晨. 木耳中镰刀菌快速识别检测与控制方法研究[D]. 咸阳:西北农林科技大学, 2022. [LIU C. Research on rapid identification, detection and control of Fusarium in black fungus[D]. Xianyang:Northwest A&F University, 2022.] LIU C. Research on rapid identification, detection and control of Fusarium in black fungus[D]. Xianyang: Northwest A&F University, 2022.
[52] SHI X, GAN X, WANG X, et al. Rapid detection of Ganoderma lucidum spore powder adulterated with dyed starch by NIR spectroscopy and chemometrics[J]. LWT,2022,167:113829. doi: 10.1016/j.lwt.2022.113829
[53] YANG C, MA X, GUAN H, et al. Rapid detection method of Pleurotus eryngii mycelium based on near infrared spectral characteristics[J]. Spectrochimica Acta Part A:Molecular and Biomolecular Spectroscopy,2022,271:120919. doi: 10.1016/j.saa.2022.120919
-
期刊类型引用(0)
其他类型引用(1)





 下载:
下载:
 下载:
下载:



