Identification of A Novel Capsular Polysaccharide Cluster in Lactiplantibacillus plantarum YZH81
-
摘要: 为了研究植物乳植杆菌胞外多糖(EPS)的合成机制,本文选取一株具有较高EPS产量的植物乳植杆菌YZH81开展其EPS合成基因簇(cps)的研究。经全基因组测序、比对和分析,结果确定了YZH81菌株基因组含有两个cps基因簇,其中一个cps基因簇(cps1)的结构与功能尚未鉴定,而另一个cps基因簇(cps2)与其他已报道的植物乳植杆菌同源性很高。本研究实验敲除了cps1基因簇,与野生型YZH81菌株相比,YZH81∆cps1菌株的EPS产量降低了52.28%,自聚集能力加速,粘附性降低,且清除1,1-二苯基-2-三硝基苯肼(1,1-diphenyl-2-picryl-hydrazyl radical,DPPH·)能力下降了32.42%。该结果说明YZH81菌株的cps1基因簇与EPS合成相关,也为进一步研究该菌株EPS生物合成机制建立有利条件。
-
关键词:
- 植物乳植杆菌 /
- 胞外多糖(EPS) /
- 荚膜多糖簇(cps cluster) /
- 粘附性 /
- 益生菌
Abstract: In order to investigate the mechanism of extracellular polysaccharide (EPS) synthesis in Lactobacillus plantarum, a strain of Lactobacillus plantarum YZH81 with high EPS production was selected for the study of extracellular polysaccharide synthesis gene cluster (cps). After whole-genome sequencing, alignment and analysis, it was determined that the genome of YZH81 contained two cps gene clusters, one of which (cps1) had yet to be identified in terms of structure and function, while the other (cps2) was highly homologous to other reported Lactobacillus plantarum strains. The conclusions in this study, the cps1 gene cluster resulted 52.28% reduction in EPS production, accelerated self-aggregation, reduced adhesion and 32.42% reduction in the ability to generated 1,1-diphenyl-2-trinitrophenylhydrazine (DPPH·) in the YZH81∆cps1 strain compared to the wild-type YZH81 strain. The results suggested that the cps1 gene cluster of strain YZH81 was associated with EPS synthesis and established favorable conditions for further studies on the mechanism of EPS biosynthesis in this strain. -
胞外多糖(exopolysaccharides,EPS)是乳酸菌的重要代谢产物,一般包括释放到培养基中的粘液多糖(slime polysaccharide,SPS)和附着在细胞壁的荚膜多糖(capsular polysaccharide,CPS)两类[1]。在食品工业中,乳酸菌EPS是一类安全的功能性添加剂,应用范围广泛,通常用作稳定剂[2−3]、增稠剂[4−5]和乳化剂[6−7],能赋予产品良好的质地、风味和口感。植物乳植杆菌作为一类食源性微生物,因其安全性高,在工业、食品和医药等重要领域占有一席之地,在众多乳酸菌EPS研究中,植物乳植杆菌来源的EPS产量高,研究报道较多,其在发酵过程中产生的EPS被广泛应用于食品生产[8]、制药[9]及化工生产等领域[10]。
细菌EPS合成相关基因及其功能最先在肺炎链球菌等致病菌中得到了详细的阐述[11]。近年来,随着许多乳酸菌菌株的全基因组序列已经公布[12−15],EPS合成相关基因的研究也在不断推进。乳酸菌EPS合成基因簇的大小通常为15~20 kb,所含基因数不超过30个,这些基因往往方向相同并以单个mRNA的形式转录。植物乳植杆菌来源的EPS合成基因簇研究报道也较多,目前结果显示植物乳植杆菌的EPS合成基因簇类型为荚膜多糖基因簇(cps cluster),不同菌株基因组通常含有1~4个cps基因簇[16]。一个典型的cps基因簇结构上可以分为四个功能区域,即链长调节、组装、合成和输出区域,多个基因共同完成EPS重复单元的合成、聚合和输出。植物乳植杆菌WCFS1(以下简称WCFS1)是第一个进行全基因组测序的乳酸菌,也是目前报道含cps基因簇最多的植物乳植杆菌,其基因组包含四个不同的cps基因簇(cps1A-I,cps2A-J,cps3A-J和cps4A-J),每个基因簇片段大小约10 kb,包含10个左右负责编码调控因子或酶的基因。此外,还有一些植物乳植杆菌的cps被较多报道[17],如ST-III,JDM1,ZJ316等,各菌株含1~3个不等的cps基因簇。目前研究显示植物乳植杆菌的cps基因簇的多样性与其菌株特异性存在一定相关性[18],因此针对不同菌株的cps需要开展更多的功能询证实验,为更多潜在益生菌的开发利用奠定基础。
本实验室前期开展高产EPS乳酸菌筛选,筛选获得一株植物乳植杆菌YZH81(简称YZH81),其EPS产量为380.00±47.16 mg/L,与现有文献报道的EPS产量较高的乳酸菌水平相当[19−20]。对YZH81进行全基因组测序,分析其cps基因簇结构,发现其基因组上有一个cps基因簇(cps2)与报道菌株同源性较高[21]。另外,还存在一个区域含多个与EPS合成相关基因串联一起,推测是YZH81菌株的另一个cps基因簇(cps1)。进一步比对结果显示,cps1基因簇中的很多基因也在一些植物乳植杆菌菌株中存在,推测cps1基因簇是植物乳植杆菌中普遍存在的一个cps基因簇,其结构和功能亟待鉴定。因此,本文首先利用同源重组的手段对YZH81菌株cps1基因簇进行敲除,从菌株生长特性、自凝集能力、粘附特性和DPPH自由基清除能力等不同方面开展研究,旨在揭示cps1基因簇的功能,为进一步研究YZH81菌株高产EPS的特性奠定基础。
1. 材料和方法
1.1 材料与仪器
本实验所用菌株和质粒见表1。本研究中使用的所有植物乳植杆菌均在MRS培养基中培养,添加的红霉素和氯霉素终浓度均为10 µg/mL,培养温度为37 ℃。大肠杆菌的培养用LB 培养基,添加的红霉素或氯霉素终浓度分别为250和10 µg/mL;MRS培养基 中国青岛海博有限公司;LB培养基 英国Oxoid有限公司;红霉素、氯霉素、透析袋(14 kD)、溶菌酶、PBS(磷酸缓冲盐溶液)、细菌DNA提取试剂盒、琼脂糖(Agarose H)、4S Red核酸染料(4S Red Plus Nucleic Acid Stain) 生工生物工程(上海)股份有限公司;ExTaq、DNA Marker、Bacteria Genomic DNA Extraction Kit 宝生物工程(大连)有限公司;三氯乙酸、1,1-二苯基-2-三硝基苯肼(1,1-diphenyl-2-picryl-hydrazyl radical,DPPH·) 上海麦克林生化试剂有限公司;无水乙醇 天津市天力化学试剂有限公司;浓硫酸 哈尔滨理工化学试剂有限公司;人结肠腺癌细胞系Caco-2细胞 生兴生物技术有限公司;
表 1 菌株和质粒Table 1. Bacterial strains and plasmids used in this study菌株和质粒 描述* 参考文献 植物乳植杆菌 WCFS1 人体唾液中筛选分离 [14] YZH81 鸡的直肠组织中筛选分离 实验室保藏 YZH81∆cps1 lox66-P 32-cat-lox71代替 N7E54_08415至N7E54_08485(N7E54_08415-08485: lox66-P 32-cat-lox71),Eryr 本研究 大肠杆菌 DH10B 克隆宿主 [22] 质粒 pNZ5319 包含lox66-P32-cat-lox71 片段 [21] pNZ5319∆cps1 包含N7E54_08415至N7E54_08485上下游同源区域片段 本研究 *注:Eryr为红霉素抗性。 本实验所用的引物由生工生物工程(上海)股份有限公司合成(表2)。
表 2 引物序列Table 2. Sequence of primers引物 5'~3' 参考文献 YZH81-Up-F AGGACCGATAACGCGCTCGAGGTGATTCATCAGCTTTTGGCAG 本研究 YZH81-Up-R GGCTTCTGTTTCTATCAGGCAATACAACATTCAAAGCCACGTT 本研究 CAT-F GCCTGATAGAAACAGAAGCCACTGG 本研究 CAT-R ATCTTCACGTTACTAAAGGGAAT
GTAGA本研究 YZH81-Down-F CCCTTTAGTAACGTGAAGATAGATATATTTATACTCAGTTACCATTGTTTTTG 本研究 YZH81-Down-R CCCCGTCAGCTTTAGAGATCTCACCTAAAATAAGGCAGACTGAATCA 本研究 83 GCAGCAGTTGATAAAGCAATTAC [23] 85 GTTTTTTTCTAGTCCAAGCTCACA [23] 87 GCCGACTGTACTTTCGGATCCT [23] 90 GCTAAAGAAGAATTTGCTTCTGC [23] N7E54_07200-F CGTCTGGCAGCTACTATAGTGA 本研究 N7E54_07200-R GTGAAATGTCCGTGATCC 本研究 N7E54_11140-F GTGGGAAATGCTGGTGGTAA 本研究 N7E54_11140-R CTGAGTCACCATTCCAACCA 本研究 N7E54_01205-F ACAGTTACTAAGACCGCGAC 本研究 N7E54_01205-R TTGAAGCAGTACCGACTTCG 本研究 N7E54_06005-F TCTGATGGTAATGTGACGGC 本研究 N7E54_06005-R AGTACCATCAGCACCTTCTG 本研究 N7E54_11140-F GGAGACCATCATAAACGGTG 本研究 N7E54_11140-R AGAATCAACCGTAACGCTCG 本研究 N7E54_11400-F CACAACGACGACTGATACTG 本研究 N7E54_11400-R GCTATCAGCAGTCCAATC 本研究 N7E54_11445-F ATCAAGACACGACCAGTGCAAC 本研究 N7E54_11445-R CACGTTACTCAGGTCACCATTC 本研究 N7E54_03885-F CAACACCACAAGTAACCGTG 本研究 N7E54_03885-R AGTCGCATCTCCAATGACAG 本研究 N7E54_00975-F GGAGTACTTGGTATCCAGGT 本研究 N7E54_00975-R AGTTAGCAATACCGGCACTG 本研究 N7E54_00570-F TGGCACGACATAGTAACG 本研究 N7E54_00570-R CGGTGTAAATGGGTCTTG 本研究 N7E54_05645-F GTCAGGATGATGTCAAGCAACC 本研究 N7E54_05645-R GGTATCCGTCGTTAATCCCA 本研究 N7E54_10785-F CATCGGTCATCAATGGTCCT 本研究 N7E54_10795-R GTCTCAGCATCAGTCACATC 本研究 rpoB-F CACCGTACCCGTAGAAGTTATGC [24] rpoB-R GGAGACCTTCATCCAAGAACCA [24] HE-90型电泳仪、水平电泳槽 上海天能科技有限公司;SHP-150型生化培养箱 上海精宏实验设备有限公司;ZQTY-70T振荡培养箱 上海知楚仪器有限公司;MCO-15AC型CO2培养箱 日本三洋公司;XD-202型倒置生物显微镜 江南永新光学有限公司;HH-2型电热数显恒温水浴锅 常州亚特实验仪器有限公司;SW-CJ-ZF型双人双面净化工作台 苏州净化设备有限公司;AG 4309型高压脉冲电击转化仪、Personal PCR仪、CL-22M型高速冷冻离心机 德国Eppendorf公司;Nano-300微量分光光度计 杭州奥盛仪器有限公司。
1.2 实验方法
1.2.1 基因组DNA提取
使用细菌全基因组提取试剂盒提取YZH81全基因组DNA,具体步骤参照使用说明书。取提取后的DNA样本进行1%琼脂糖凝胶电泳,检测基因组提取质量及完整性。
1.2.2 基因组的测序与分析
YZH81菌株GenBank编号为JAODTM000000000。基因组的测序在生工生物工程(上海)股份有限公司进行,采用Illumina测序技术;基因组拼接使用SPAdes拼接二代测序数据,采用GapFiller对拼接得到的contig补GAP;利用PrInSeS-G进行序列矫正,修正拼接过程中的剪辑错误及小片段的插入缺失。使用Blastn(http://blast.ncbi.nlm.nih.gov/)将YZH81基因组的cps基因簇与WCFS1(AL935263.2)、LP90(CP_015857)、ST-Ⅲ(NC_014554)和JDM1(CP001617.1)进行对比,由TB Tools(v1.108)进行基因组共线性分析。
1.2.3 cps基因簇缺失株的构建
本研究中使用的引物列于表2。YZH81∆cps1的构建基于同源重组原理进行[21],以YZH81的全基因组DNA为模板,利用YZH81-Up-F/R、YZH81-Down-F/R和CAT-F/R引物通过PCR扩增cps1基因簇的上游和下游侧翼区域,先后克隆至质粒pNZ5319 XhoI和BgIII酶切位点,构建敲除质粒pNZ5319∆cps1。按照电击转化法,将质粒电转化YZH81感受态细胞中,电转产物37 ℃孵育2 h后涂布于含10 µg/mL氯霉素的MRS培养基上,37 ℃静置培养24~36 h,阳性克隆采用菌落PCR验证长度约1.7 kb同源臂序列,最终获得缺失cps1基因簇的YZH81突变株 ,命名为YZH81∆cps1。
为了进一步验证cps1敲除的结果,将YZH81和YZH81∆cps1序列上传到Galaxy平台进行基因组测序。根据条形码分离来自Illumina测序的原始Illumina序列片段,并使用Bowtie 2将YZH81∆cps1序列的每个读数对应的核苷酸片段映射到YZH81基因组。参考Zhang等[25]的方法,整合基因组学查看器(IGV)使用核苷酸窗口大小对比对结果进行排序和计数,然后对基因求和。读取映射到基因的最后10%被丢弃,因为这些插入可能不会使基因功能失活。然后,通过以下公式计算归一化读取计数RPKM(每百万输入读取每千碱基读取数),将每个基因的读取计数标准化为映射到每个重复中基因组的读取总数:RPKM=(映射到基因×读取数106)/(样本中映射的总输入读取数×基因长度,单位为kb)。
1.2.4 EPS产量测定
参考李卫娜等[26]的实验方法并做适量改动。将−80 ℃冰箱保藏的YZH81和YZH81∆cps1菌种划线相应抗性的MRS平板,单菌落挑取至液体MRS培养基活化,活化后的种子液以1%接种量发酵培养,37 ℃静置培养32 h,离心(10000×g,4 ℃,15 min)取上清液。向上清液中加入80%三氯乙酸至终浓度为4%(w/v),4 ℃下静置8 h。通过离心(10000×g,4 ℃,15 min)除去沉淀的蛋白质。随后上清加入3倍体积的冷乙醇在4 ℃下醇沉过夜,离心(10000×g,4 ℃,20 min)取沉淀。将沉淀用适当体积去离子水溶解,在4 ℃下透析48 h,然后冻干,最终获得粗EPS样品。采用苯酚-硫酸法测定粗EPS的碳水化合物含量。绘制标准曲线,以葡萄糖含量(mg/mL)为横坐标,吸光度(A490 nm)作为纵坐标,建立回归方程,y=0.014x−0.021,R2=0.99。
1.2.5 生长曲线及pH测定
参考Yang等[27]的实验方法并适当改动。将−80 ℃冰箱保藏的YZH81和YZH81∆cps1菌种划线相应抗性的MRS平板,单菌落挑取至液体MRS培养基活化,活化后的种子液以1%接种量发酵培养,接种后即刻混匀,取1 mL于比色皿中测定OD600 nm值,作为初始零时间点值,随后每2 h测定一次OD600 nm值和MRS液体培养基的pH,直至24 h实验结束。以时间为横坐标,OD600 nm数值和pH为纵坐标绘制生长曲线。
1.2.6 自凝集能力检测
参考Gao等[28]的实验方法并做适量改动。将−80 ℃冰箱保藏的YZH81和YZH81∆cps1菌种划线相应抗性的MRS平板,单菌落挑取至液体MRS培养基活化,活化后的种子液以1%接种量发酵培养,37 ℃静置培养12 h ,离心收集菌体(4500×g,15 min),使用PBS(pH7.2)洗涤2次后于PBS中重悬,调整菌液浓度至108 CFU/mL,将4 mL等量的细胞菌悬液装入5 mL的离心管中,混匀后静置于室温。自凝集测定的时间为4和16 h,每次取0.5 mL离心管上部菌悬液,加入到1.5 mL的PBS中,混匀,测定600 nm下的吸光值。以 PBS作为空白对照,凝集率(%)的计算公式为:
凝集率(%)=(1−ATA0)×100 注: A0是凝集前的上清溶液在600 nm下测定的OD600 nm值;AT是凝集后的上清溶液在600 nm下测定的OD600 nm值。
1.2.7 Caco-2细胞粘附性测定
Caco-2细胞水浴解冻,8000×g离心5 min收集细胞,加入5 mL完全培养液,吸出上清,保留细胞沉淀。加入1 mL完全培养液轻轻吹吸,混匀并移至培养皿中。放置于37 ℃培养,每隔2 d更换培养液,显微镜镜检,待细胞培养皿上Caco-2的覆盖率达到80%后,可进行粘附实验。
细胞粘附实验参照Yang等[27]的方法。将YZH81和YZH81∆cps1的菌悬液(已稀释调整至菌浓度为1×108 CFU/mL),于12000×g离心1 min,弃上清,菌体经无菌PBS缓冲液(pH7.2)清洗3次,然后重悬于不完全DMEM培养液。待细胞培养皿中的细胞长至单层时,用无菌PBS缓冲液(pH7.2)清洗后,添加1 mL含有1×108 CFU/mL重组乳酸菌的DMEM培养物,于二氧化碳培养箱中孵育2 h;PBS洗涤去除未粘附细菌,随后加入0.5% Triton X-100处理5 min,使粘附细菌从细胞脱落,收集孔内液体。用细胞粘附数表示粘附性,公式如下:
粘附数(CFU/cell)=粘附菌株细胞数Caco-2细胞数 1.2.8 粘附蛋白的表达量
将活化后的YZH81和YZH81∆cps1菌株分别转接于相应的MRS培养基中,37 ℃培养至OD600 nm吸光度为0.8~1。根据试剂盒说明书提取YZH81和YZH81∆cps1的总RNA,使用带有gDNA的PrimeScript™ RT试剂盒合成cDNA。以cDNA为模板,检测粘附蛋白的表达。然后,以rpoB mRNA为参考基因[24],采用2−ΔΔCT相对定量方法计算各组基因表达,用于RT-qPCR的引物如表2所示。
1.2.9 DPPH自由基清除实验
取2 mL的DPPH无水乙醇溶液(400 µmol/L),加入2 mL待测菌液混匀,室温避光静置30 min,8000×g离心10 min,测定上清液在517 nm的吸光度值。DPPH 自由基清除率计算公式为:
DPPH自由基清除率(%)=[1−(A1−A2)A0]×100 注:A0为等体积无水乙醇代替样品的吸光度值; A1为实验组吸光度值;A2为等体积无水乙醇代替DPPH 乙醇溶液的吸光度值。
1.3 数据处理
每组实验重复3次,实验结果为3次重复的平均值,采用SPSS 22.0软件进行实验数据的统计与分析。检测水准为0.05,运用最小显著差异(Least significant difference,LSD)法比较,以P<0.05为差异有统计学意义。实验作图均采用Prism 8.0软件绘制。
2. 结果与分析
2.1 YZH81菌株cps基因簇分析
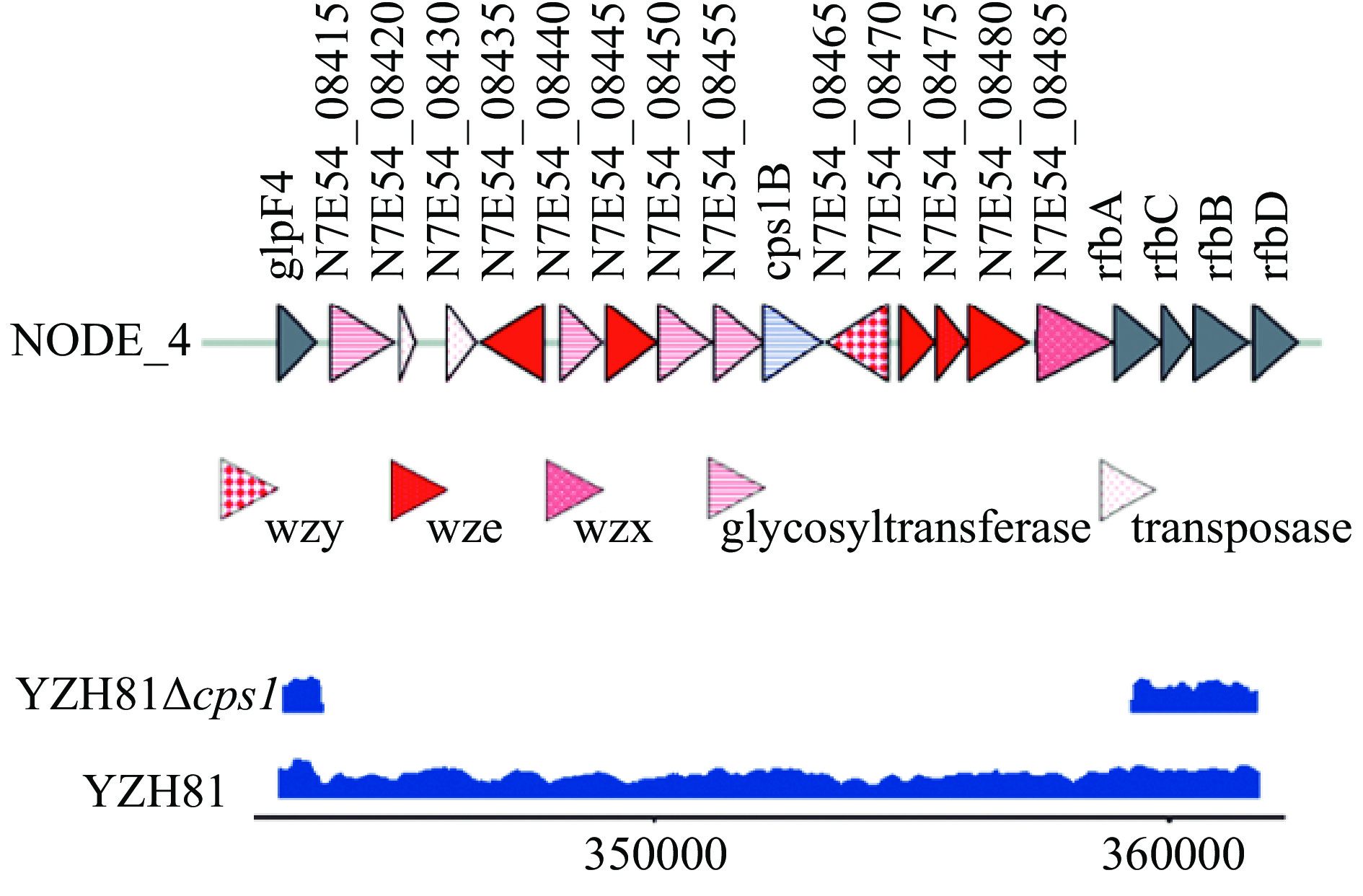
YZH81菌株经全基因组测序,与cps基因簇研究最为深入的WCFS1菌株进行比对,结果显示如图1。YZH81基因组上除基因N7E54_08460与WCFS1的cps1B基因有相似性(36.94%)以外,比对获得一个基因簇(自N7E54_12115至N7E54_12070),含10个基因,其基因组成、功能、大小及排列与WCFS1菌株cps4基因簇高度相似,单个基因序列同源性很高,为94.75%~99.62%,故推测该基因簇是YZH81的一个完整的cps基因簇。
![]() 图 1 YZH81菌株cps基因簇的功能对比图示注:比对的植物乳植杆菌参考基因组accession number如下:YZH81 (AL935263.2;N7E54_08410~08485),LP90 (CP_015857;Lp90_RS04080~RS09095),JDM1 (NC_012984;JDM1_JDM1_RS05090~RS08755) and ST-III (NC_014554;LPST_LPST_RS04720~LPST_RS08645),实线框表示YZH81的cps1基因簇,推测为植物乳植杆菌新的cps基因簇,虚线框表示YZH81的高同源性的cps2基因簇。Figure 1. Comparative illustration of cps gene clusters of L. plantarum YZH81
图 1 YZH81菌株cps基因簇的功能对比图示注:比对的植物乳植杆菌参考基因组accession number如下:YZH81 (AL935263.2;N7E54_08410~08485),LP90 (CP_015857;Lp90_RS04080~RS09095),JDM1 (NC_012984;JDM1_JDM1_RS05090~RS08755) and ST-III (NC_014554;LPST_LPST_RS04720~LPST_RS08645),实线框表示YZH81的cps1基因簇,推测为植物乳植杆菌新的cps基因簇,虚线框表示YZH81的高同源性的cps2基因簇。Figure 1. Comparative illustration of cps gene clusters of L. plantarum YZH81然而,与其他植物乳植杆菌比对的结果显示YZH81基因组上还存在一些连续排列的基因(共计14个,含上述基因N7E54_08460),其功能注释与植物乳植杆菌cps基因簇的功能基因相符,如编码糖基转移酶、多糖生物合成蛋白、翻转酶等,检索比对的基因功能注释和与其他代表性植物乳植杆菌基因相似性分析结果列于表3。因此,推测其为YZH81另一个cps基因簇。
表 3 YZH81菌株cps1基因簇功能比对Table 3. Functional alignment of cps1 gene clusters of YZH81 strainYZH81 功能注释 JDM1 LP90 N7E54_08415 glycosyltransferase RS05095 [100] RS04085 [100] N7E54_08420 transposase RS05100 [100] RS04090 [100] N7E54_08430 transposase RS05105 [100] RS05230 [99.45] N7E54_08435 hypothetical protein RS05110 [100] RS05235 [99.75] N7E54_08440 glycosyltransferase RS05115 [100] RS05240 [100] N7E54_08445 capsular biosynthesis protein RS05120 [100] RS05245 [100] N7E54_08450 glycosyltransferase RS05125 [100] RS05250 [99.71] N7E54_08455 glycosyltransferase RS05130 [100] RS05255 [99.35] N7E54_08460 glycosyltransferase family 4 protein RS05135 [100] RS05260 [98.99] N7E54_08465 Wzy RS05140 [100] RS05265 [100] N7E54_08470 polysaccharide biosynthesis protein RS05145 [100] RS05270 [100] N7E54_08475 CpsD/CapB family tyrosine-protein kinase(Wze) RS05150 [100] RS05275 [100] N7E54_08480 serine hydrolase RS05155 [100] RS05280 [100] N7E54_08485 oligosaccharide flippase family protein (Wzx) RS05160 [100] RS05285 [100] 注:比对的植物乳植杆菌参考基因组accession number如下:YZH81,AL935263.2;LP90,CP_015857;JDM1,NC_012984;“[ ]”中数字为与YZH81同源蛋白基因序列相似性。 综上所述,YZH81菌株推测含有两个cps基因簇,分别命名为cps1和cps2,其中cps1是尚未报道的植物乳植杆菌cps基因簇,遂开展对YZH81菌株cps1的功能鉴定研究。
2.2 YZH81∆cps1的构建
根据方法1.2.3,构建带有YZH81∆cps1基因簇上下游侧翼序列的敲除质粒pNZ5319∆cps1,电击转化YZH81感受态细胞,利用红霉素和氯霉素双抗性MRS平板筛选,经PCR验证,获得阳性菌株YZH81∆cps1。
由于敲除的cps1基因簇片段较长,为了验证敲除cps1的完整性,对YZH81∆cps1的基因组进行了测序,与野生型YZH81进行比对,使用IGV进行定位,如图2所示,显示整个cps1簇在基因水平上被完全去除。
2.3 cps1对YZH81产EPS的影响
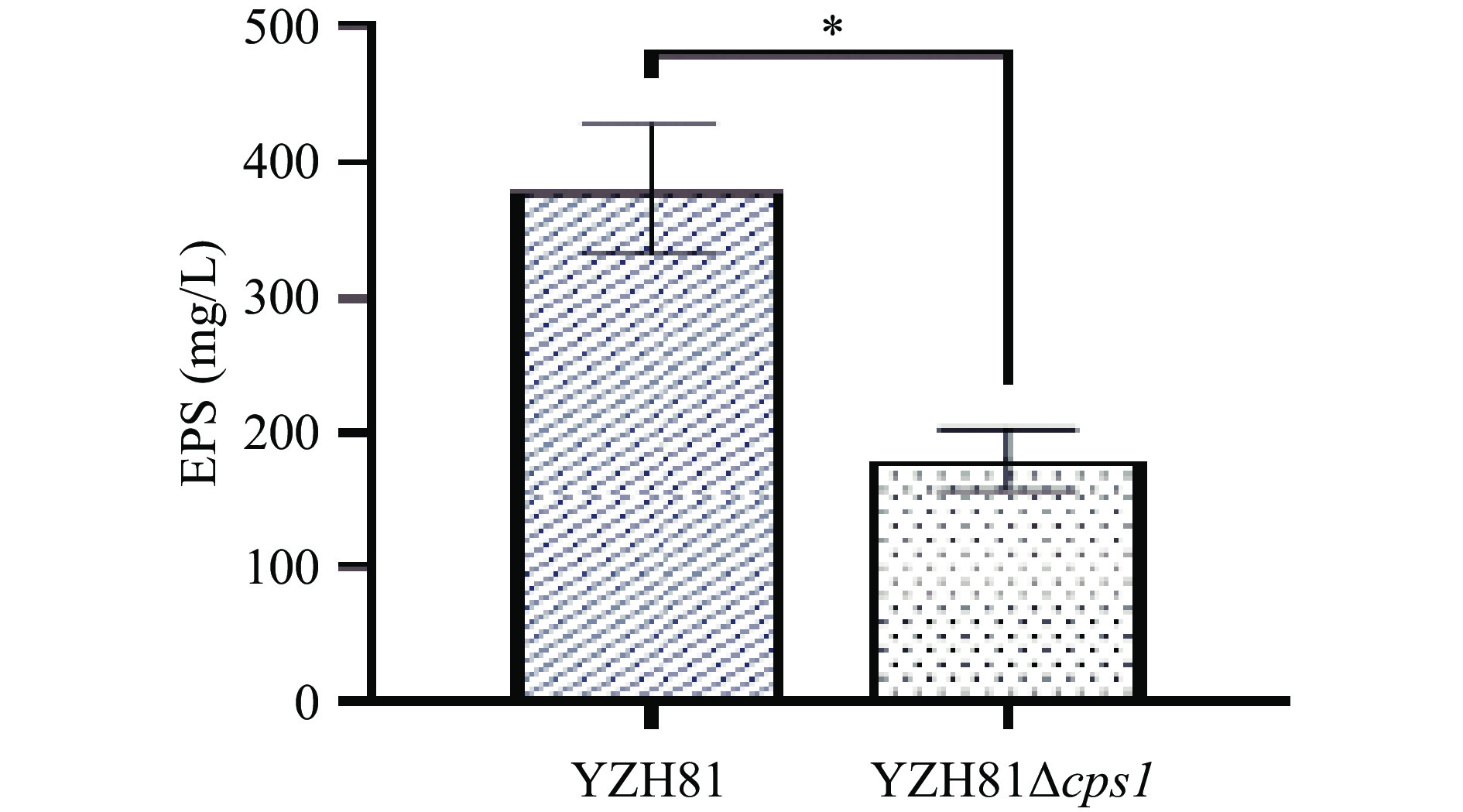
根据方法1.2.4,对YZH81和YZH81∆cps1的EPS产量进行测定,结果如图3。结果显示YZH81∆cps1的EPS产量为181.33±22.03 mg/L,相比野生型YZH81的380.00±47.16 mg/L,EPS产量显著下降了52.28%(P<0.05)。该结果表明cps1基因簇与菌株EPS的产量非常相关。
2.4 cps1对YZH81生长的影响
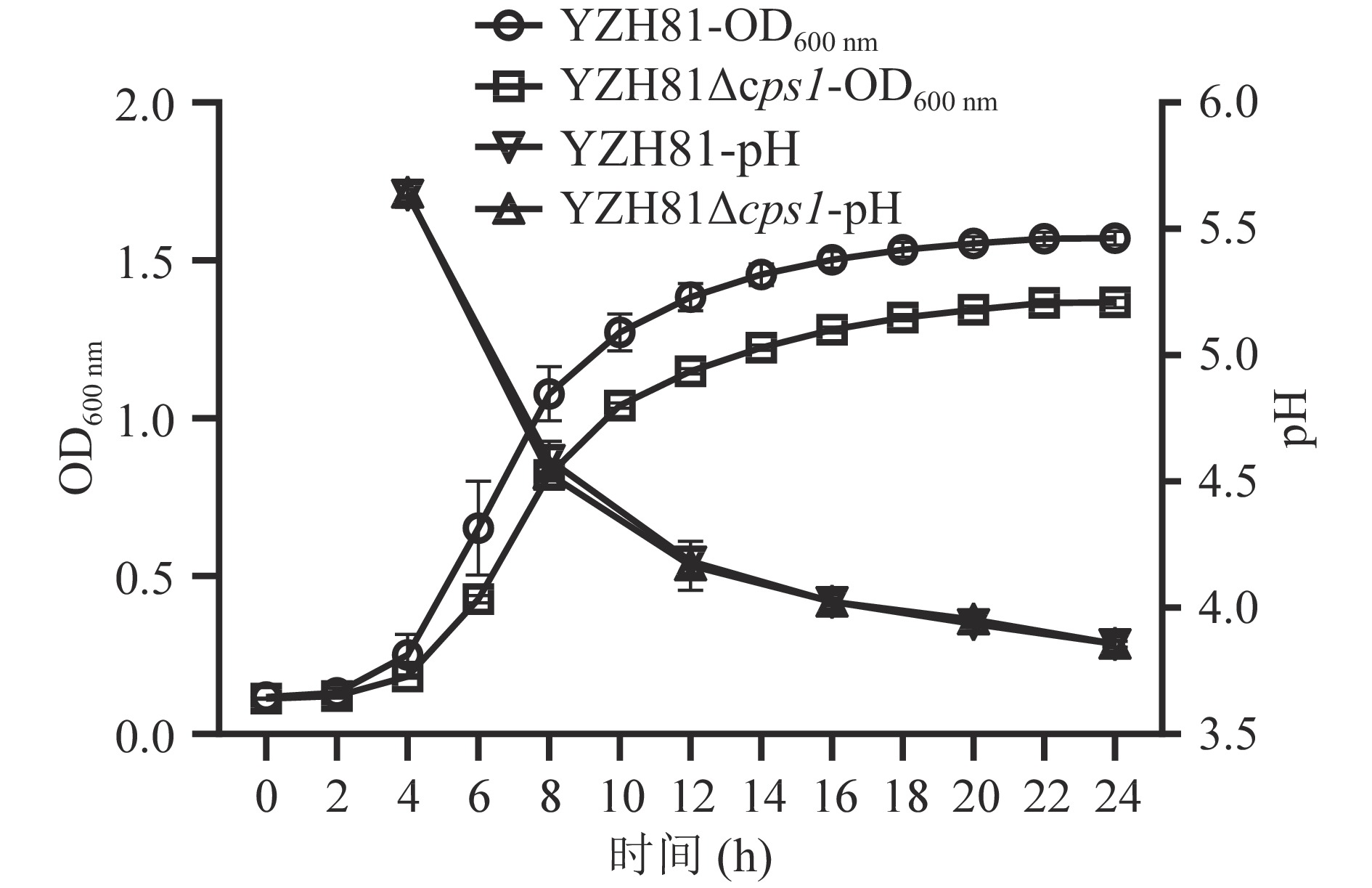
YZH81和YZH81∆cps1分别在相应的MRS培养基中培养24 h,每2 h测定OD600 nm绘制生长曲线,结果如图4所示。从OD600 nm数值来看, YZH81∆cps1生长不如YZH81,培养24 h的OD600 nm吸光值差异显著(P<0.05)。然而,从培养基pH数据来看,这两种菌株没有显著差异(P>0.05),随后对两个菌24 h发酵液进行菌落平板计数,结果为YZH81和YZH81∆cps1的菌落数分别为(9.57±0.40)×109和(9.53±0.25)×109 CFU/mL,无明显差异(P>0.05),该结果与pH结果一致。因此,本研究最终结果表明虽然吸光值OD600 nm有显著(P<0.05)下降,但cps1缺失没有影响菌株YZH81的生长量。
2.5 cps1对菌株自凝集的影响
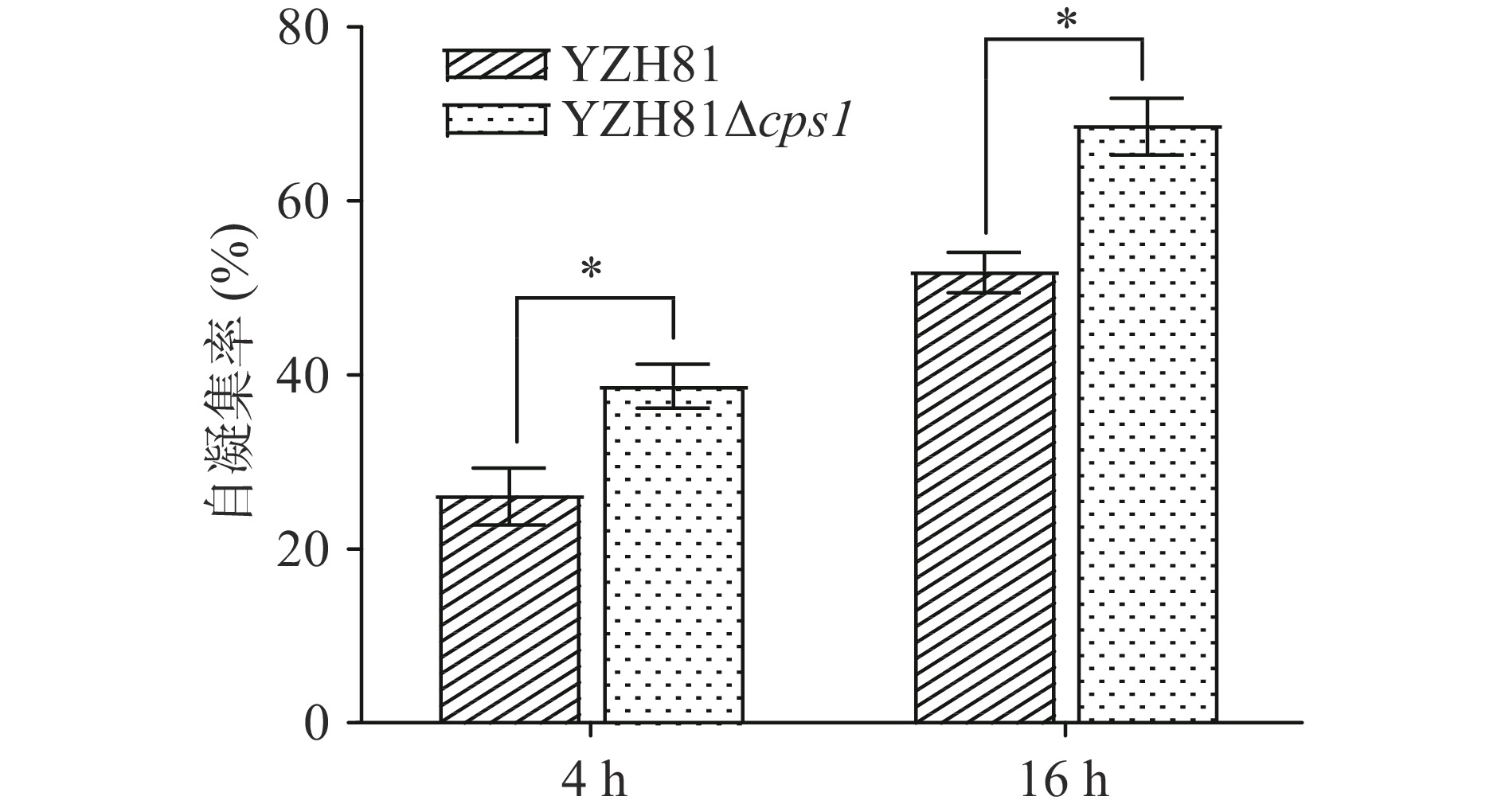
菌株的自凝集是指同一种菌间相互凝集形成多细胞簇的现象,与菌株表面结构相关[28]。YZH81和YZH81∆cps1的自凝集结果见图5。在4 h时,YZH81和YZH81∆cps1的自凝集率分别是26.30%±3.22%、38.90%±2.31%;在16 h时,YZH81和YZH81∆cps1的凝集速率分别是52.07%±2.03%、68.90%±3.14%。YZH81∆cps1在4 h和16 h的自凝集率均显著高于YZH81(P<0.05),分别是YZH81的1.48和1.32倍。该结果说明cps1基因簇的缺失加速了菌株自凝集,分析其原因可能由于EPS的减少改变了菌株表面分子的相互作用力。自凝集速率的增加可能是导致YZH81∆cps1菌株生长时OD600 nm值降低的原因。
2.6 cps1对菌株粘附性的影响
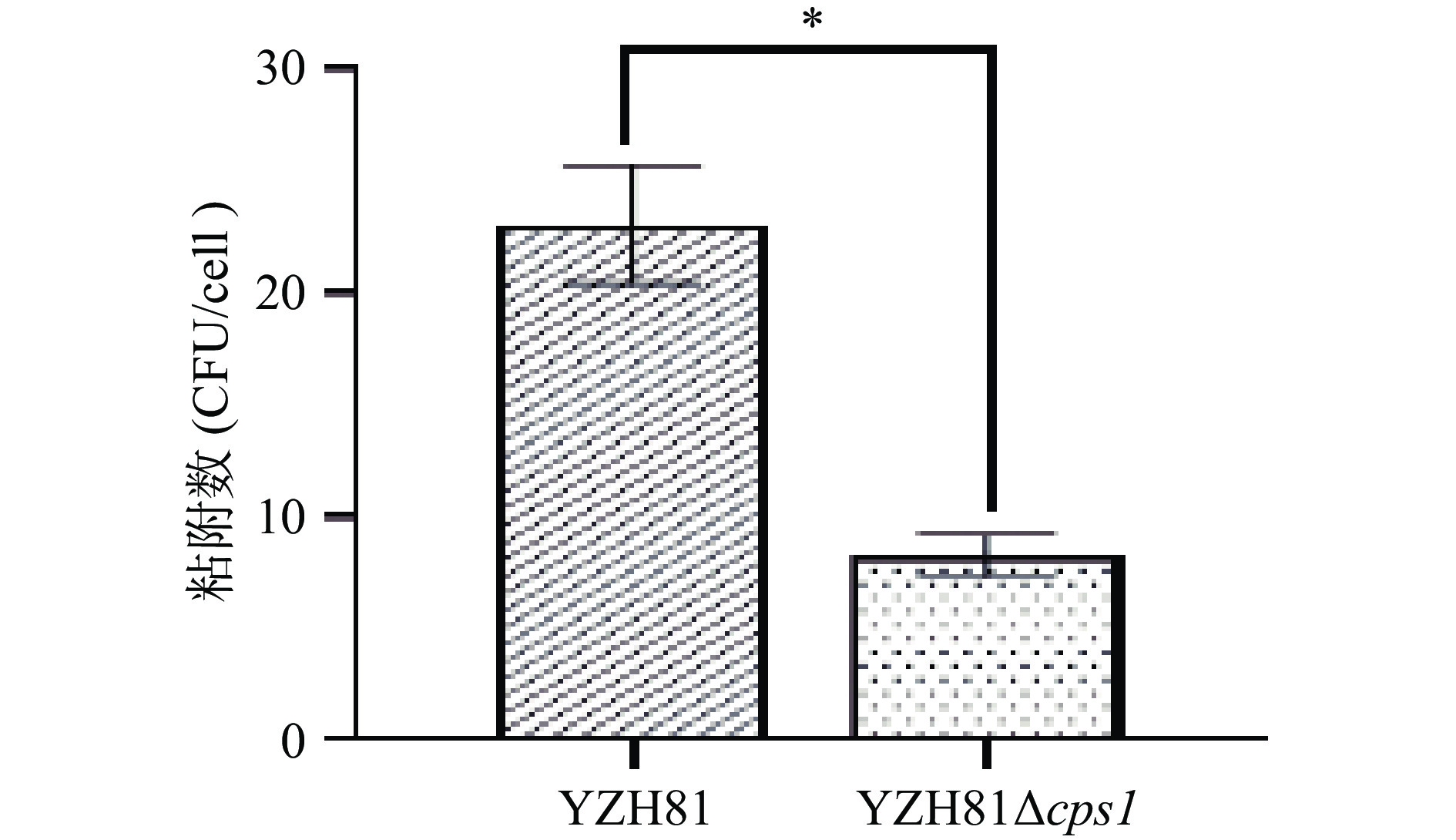
粘附性是乳酸菌发挥益生作用的前提,与菌体细胞表面结构相关[29]。为了研究cps1基因簇在YZH81粘附肠上皮细胞的作用,选择肠上皮细胞系(Caco-2细胞)为研究对象,对YZH81∆cps1的粘附性进行了测定。如图6所示,YZH81∆cps1的粘附性为8.52±0.20 CFU/cell,显著低于YZH81的粘附性21.50±1.80 CFU/cell,该结果与Gao 等[28]的实验结果一致,即cpsWC可以促进植物乳植杆菌在Caco-2细胞上粘附。
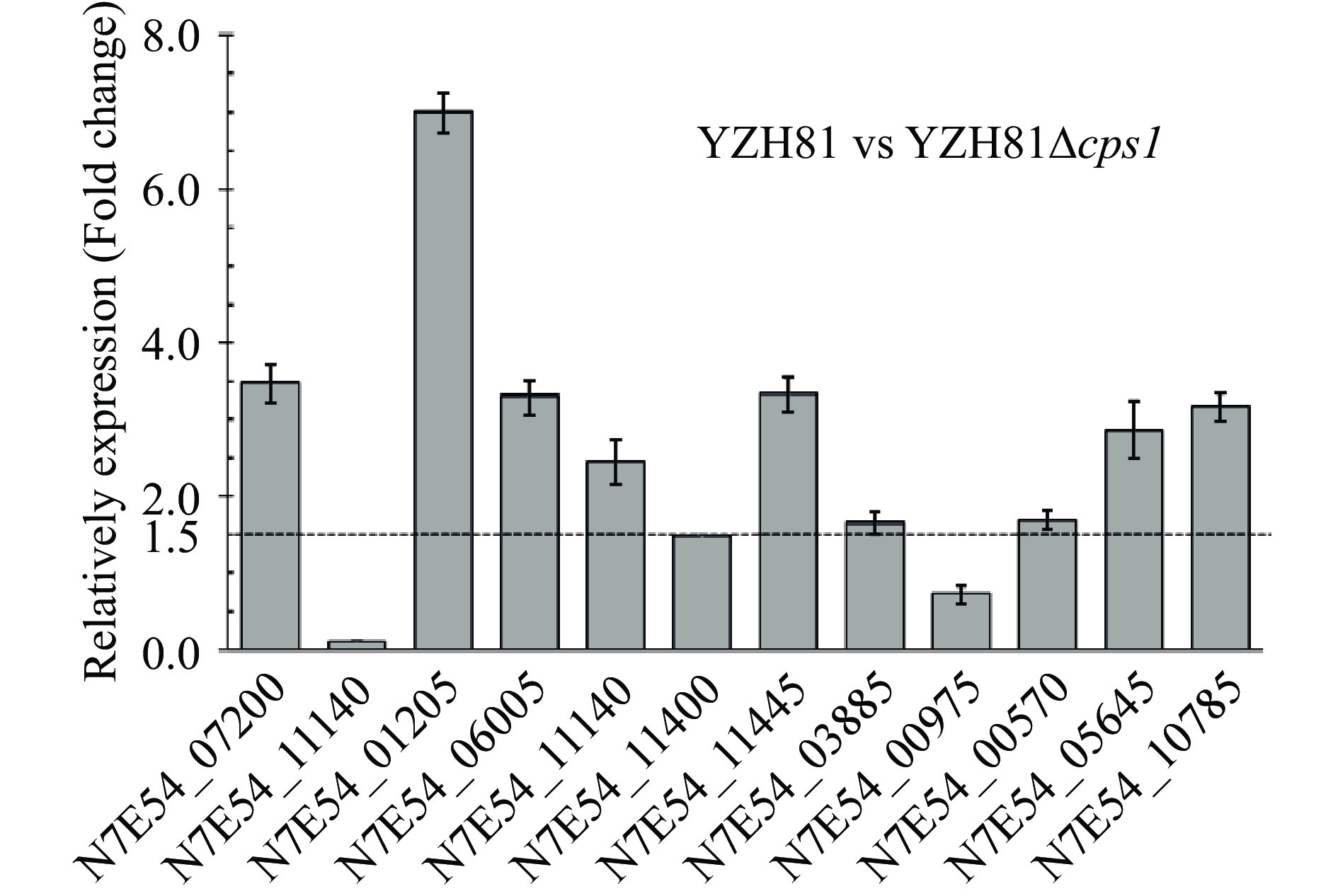
此外,实验研究了YZH81菌株可能的粘附蛋白基因表达情况。首先,根据YZH81基因组序列与WCFS1的比对结果,检索获得YZH81菌株中有12个与WCFS1粘附蛋白[14]同源的粘附蛋白基因(表4)。结果显示,除基因N7E54_11140外,其余11个粘附蛋白基因序列与WCFS1菌株同源性较高,相似性高于75%。通过RT-qPCR研究cps1的缺失对YZH81粘附蛋白表达的影响,如图7所示,YZH81∆cps1菌株的12个粘附蛋白基因中有10个基因的转录水平显著低于野生型YZH81表达水平(Fold change比≥1.5),该结果表明cps1基因簇可能通过影响YZH81中粘附蛋白的表达,最终导致该菌株与Caco-2细胞的粘附性显著下降。
表 4 粘附蛋白基因Table 4. Adhesion protein genes编号 WCFS1 功能注释 YZH81 相似性(%) 1 LP_RS04070 Internalin-J precursor N7E54_07200 85.26 2 LP_RS05225 mannose-specific adhesin, LPXTG-motif cell wall anchor N7E54_11140 30.87 3 LP_RS07025 MucBP domain protein N7E54_01205 98.51 4 LP_RS10640 Internalin-J precursor N7E54_06005 98.79 5 LP_RS12860 IgA FC receptor precursor N7E54_11140 98.53 6 LP_RS13100 MucBP domain protein N7E54_11400 79.03 7 LP_RS13150 Internalin-J precursor N7E54_11445 95.90 8 LP_RS00795 Collagen adhesin precursor N7E54_03885 97.74 9 LP_RS07255 GlcNAc-binding protein A precursor N7E54_00975 99.01 10 LP_RS07650 hypothetical protein N7E54_00570 99.30 11 LP_RS10955 Cna protein B-type domain protein N7E54_05645 96.88 12 LP_RS12445 Collagen binding domain protein N7E54_10785 91.59 2.7 YZH81∆cps1对DPPH自由基清除活性的影响
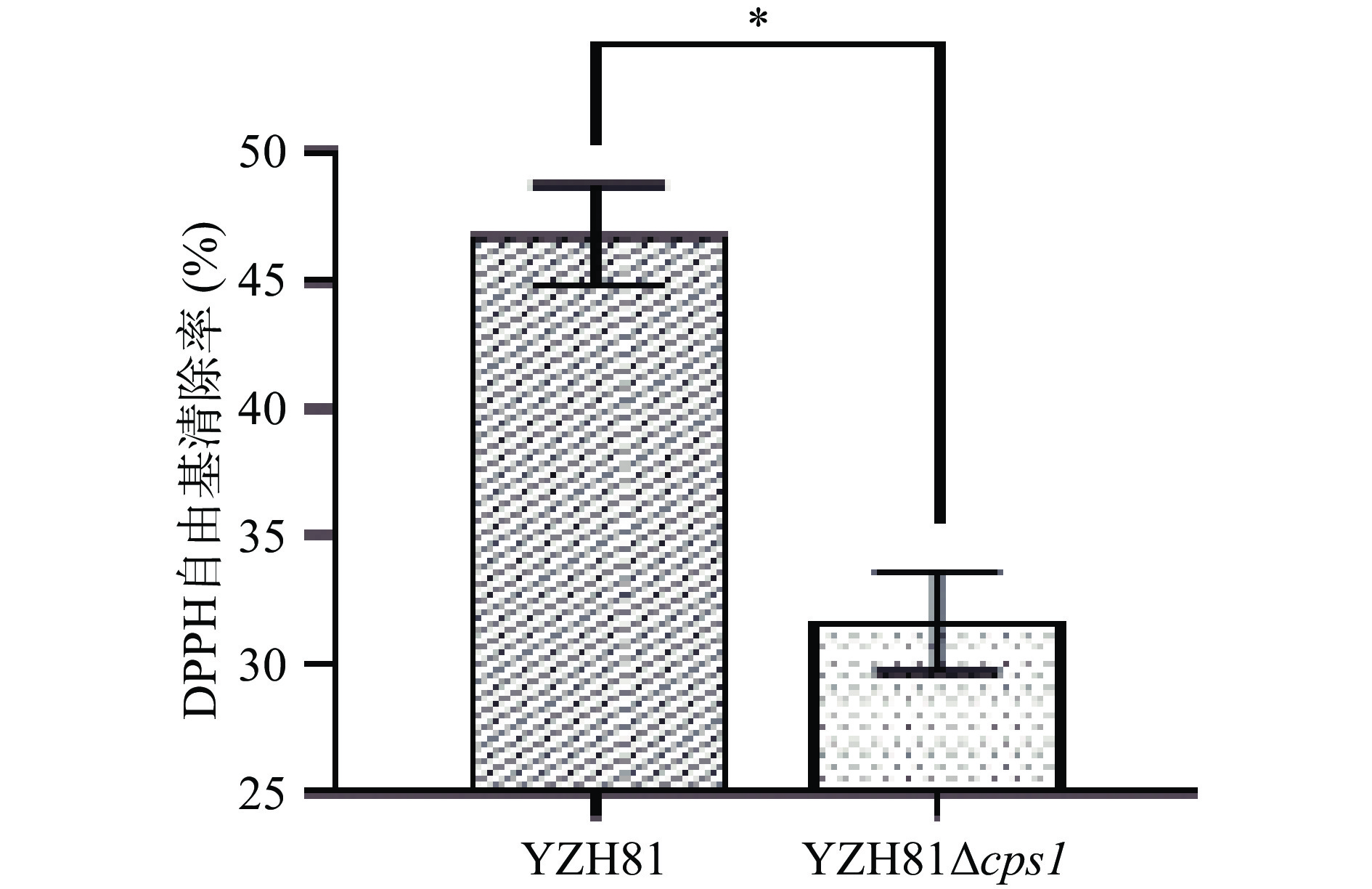
YZH81和YZH81∆cps1清除DPPH自由基能力如图8所示,两株菌株均有一定的清除DPPH自由基的能力。但是,YZH81的DPPH自由基清除率是46.82%±1.95%,显著高于YZH81∆cps1,清除率为31.64%±1.97(P<0.05)。乳酸菌的DPPH自由基清除能力与其分泌到菌体表面和代谢产物中的EPS有关,这些多糖类物质通过氢原子和电子转移中和DPPH自由基[30]。因此,推测YZH81菌株cps1的缺失引起的EPS产量的降低,影响了YZH81对DPPH自由基清除的能力。本实验的结果与Zhang等[31]对植物乳植杆菌C88 EPS抗氧化活性分析的结果一致。
3. 讨论
乳酸菌广泛应用于生产发酵乳制品,很多菌株被报道可以产生EPS。目前,植物乳植杆菌来源的EPS研究较为广泛,而且,由于相对简单的基因操作,如成熟的异源表达载体、成功的基因敲除方法[21]和简单的电转化方法,现阶段对植物乳植杆菌的EPS合成基因簇(cps簇)研究报道较多[32−33]。本实验室前期从鸡的直肠中分离获得一株高产EPS的乳酸菌,经鉴定为植物乳植杆菌,命名为YZH81,其EPS产量为380 mg/L,本研究开展了该菌株cps基因簇的研究,为进一步了解该菌株的EPS生产特性奠定基础。
植物乳植杆菌基因组通常含有1~4个EPS合成基因簇即cps 基因簇。典型的cps基因簇通常包含EPS合成基因(wzd,wze,wzh)、翻转酶(wzx)和聚合酶(wzy)等相关功能基因,单向转录。WCFS1菌株是植物乳植杆菌中cps簇研究最为透彻的菌株,也是被报道含有最多cps簇的菌株[18]。因此,本文首先将YZH81菌株与WCFS1进行比对,结果显示YZH81的基因组仅含1个cps基因簇,与WCFS1 cps4基因簇高度相似(单个基因相似度高达94.75%~99.62%)。但是,与其他植物乳植杆菌(LP90和JDM1)比对结果显示,YZH81基因组上还有一个区域聚集一连串与EPS合成相关基因,功能分别为wze、wzx、wzy和一些编码糖基转移酶家族蛋白的基因,与典型的产EPS的基因簇结构一致。因此,推测YZH81具有一个尚未鉴定的cps基因簇,根据基因组上的物理顺序命名为cps1基因簇。
为了鉴定cps1基因簇的功能,本研究构建了cps1敲除菌YZH81∆cps1,并随即开展了该菌株EPS产量、菌株生长量、菌株自凝集特性和菌株粘附性的体外实验。结果表明,与野生型YZH81相比,YZH81∆cps1的EPS产量下降,细胞自聚集加速,且粘附性降低。以上结果不仅说明了cps1基因簇参与了YZH81菌株的EPS合成,更为重要的是由于cps1的缺失造成了EPS的减少,也显著影响了菌株的表面结构,导致了自凝集的加剧和粘附性的降低。考虑到EPS产量并不是影响菌株粘附性的唯一因素,本文进一步研究了YZH81∆cps1粘附蛋白的表达情况。经检索比对获得的12个YZH81菌株粘附蛋白中,与野生型相比,cps1的缺失导致了10个粘附蛋白基因表达量显著下降,其余2个无明显变化。该结果说明了YZH81菌株的cps1基因簇可能通过生产的EPS产量和调节粘附蛋白表达两个方面的作用来共同影响菌株的粘附性。在此基础上,本研究继续测定了cps1缺失后DPPH自由基清除效果,结果显示野生型菌株具有一定的抗氧化活性,但cps1基因簇敲除后DPPH自由基清除率下降了32.42%。该结果显示cps1基因簇的缺失造成EPS的产量下降影响了菌株的抗氧化功能。
综上所述,YZH81菌株基因组上发现一个尚未鉴定的EPS合成基因簇cps1,本研究结果显示该基因簇可影响菌株EPS的产量,造成菌株粘附性和抗氧化性的改变,为深入研究该菌株EPS的生物学功能奠定了一定的理论基础。乳酸菌EPS的合成产生是一个复杂的过程,相关基因簇的调控可能涉及各种调控机制。本研究发现了一个新的cps1基因簇,其功能与EPS的产生和菌株的功能相关。在此基础上,需要进一步地研究单一cps簇的基因结构与功能以及不同cps簇在EPS合成中的互作关系,从而深入揭示其合成EPS的分子机制。
4. 结论
本研究针对一株高产EPS的植物乳植杆菌YZH81的EPS合成基因簇(cps cluster)开展研究,对其中一个cps基因簇(cps1)的结构与功能进行初步鉴定。利用同源重组方法,构建pNZ5319∆cps1敲除质粒并成功构建了cps1敲除菌YZH81∆cps1。结果显示,与野生型YZH81菌株相比,YZH81∆cps1的EPS产量显著降低,自聚集能力加速,粘附性降低,且清除DPPH自由基能力明显下降。本研究证明了YZH81菌株的cps1基因簇在EPS合成中的重要作用,也为进一步研究该菌株EPS合成基因簇及其作用机制建立有利条件。
-
图 1 YZH81菌株cps基因簇的功能对比图示
注:比对的植物乳植杆菌参考基因组accession number如下:YZH81 (AL935263.2;N7E54_08410~08485),LP90 (CP_015857;Lp90_RS04080~RS09095),JDM1 (NC_012984;JDM1_JDM1_RS05090~RS08755) and ST-III (NC_014554;LPST_LPST_RS04720~LPST_RS08645),实线框表示YZH81的cps1基因簇,推测为植物乳植杆菌新的cps基因簇,虚线框表示YZH81的高同源性的cps2基因簇。
Figure 1. Comparative illustration of cps gene clusters of L. plantarum YZH81
表 1 菌株和质粒
Table 1 Bacterial strains and plasmids used in this study
菌株和质粒 描述* 参考文献 植物乳植杆菌 WCFS1 人体唾液中筛选分离 [14] YZH81 鸡的直肠组织中筛选分离 实验室保藏 YZH81∆cps1 lox66-P 32-cat-lox71代替 N7E54_08415至N7E54_08485(N7E54_08415-08485: lox66-P 32-cat-lox71),Eryr 本研究 大肠杆菌 DH10B 克隆宿主 [22] 质粒 pNZ5319 包含lox66-P32-cat-lox71 片段 [21] pNZ5319∆cps1 包含N7E54_08415至N7E54_08485上下游同源区域片段 本研究 *注:Eryr为红霉素抗性。 表 2 引物序列
Table 2 Sequence of primers
引物 5'~3' 参考文献 YZH81-Up-F AGGACCGATAACGCGCTCGAGGTGATTCATCAGCTTTTGGCAG 本研究 YZH81-Up-R GGCTTCTGTTTCTATCAGGCAATACAACATTCAAAGCCACGTT 本研究 CAT-F GCCTGATAGAAACAGAAGCCACTGG 本研究 CAT-R ATCTTCACGTTACTAAAGGGAAT
GTAGA本研究 YZH81-Down-F CCCTTTAGTAACGTGAAGATAGATATATTTATACTCAGTTACCATTGTTTTTG 本研究 YZH81-Down-R CCCCGTCAGCTTTAGAGATCTCACCTAAAATAAGGCAGACTGAATCA 本研究 83 GCAGCAGTTGATAAAGCAATTAC [23] 85 GTTTTTTTCTAGTCCAAGCTCACA [23] 87 GCCGACTGTACTTTCGGATCCT [23] 90 GCTAAAGAAGAATTTGCTTCTGC [23] N7E54_07200-F CGTCTGGCAGCTACTATAGTGA 本研究 N7E54_07200-R GTGAAATGTCCGTGATCC 本研究 N7E54_11140-F GTGGGAAATGCTGGTGGTAA 本研究 N7E54_11140-R CTGAGTCACCATTCCAACCA 本研究 N7E54_01205-F ACAGTTACTAAGACCGCGAC 本研究 N7E54_01205-R TTGAAGCAGTACCGACTTCG 本研究 N7E54_06005-F TCTGATGGTAATGTGACGGC 本研究 N7E54_06005-R AGTACCATCAGCACCTTCTG 本研究 N7E54_11140-F GGAGACCATCATAAACGGTG 本研究 N7E54_11140-R AGAATCAACCGTAACGCTCG 本研究 N7E54_11400-F CACAACGACGACTGATACTG 本研究 N7E54_11400-R GCTATCAGCAGTCCAATC 本研究 N7E54_11445-F ATCAAGACACGACCAGTGCAAC 本研究 N7E54_11445-R CACGTTACTCAGGTCACCATTC 本研究 N7E54_03885-F CAACACCACAAGTAACCGTG 本研究 N7E54_03885-R AGTCGCATCTCCAATGACAG 本研究 N7E54_00975-F GGAGTACTTGGTATCCAGGT 本研究 N7E54_00975-R AGTTAGCAATACCGGCACTG 本研究 N7E54_00570-F TGGCACGACATAGTAACG 本研究 N7E54_00570-R CGGTGTAAATGGGTCTTG 本研究 N7E54_05645-F GTCAGGATGATGTCAAGCAACC 本研究 N7E54_05645-R GGTATCCGTCGTTAATCCCA 本研究 N7E54_10785-F CATCGGTCATCAATGGTCCT 本研究 N7E54_10795-R GTCTCAGCATCAGTCACATC 本研究 rpoB-F CACCGTACCCGTAGAAGTTATGC [24] rpoB-R GGAGACCTTCATCCAAGAACCA [24] 表 3 YZH81菌株cps1基因簇功能比对
Table 3 Functional alignment of cps1 gene clusters of YZH81 strain
YZH81 功能注释 JDM1 LP90 N7E54_08415 glycosyltransferase RS05095 [100] RS04085 [100] N7E54_08420 transposase RS05100 [100] RS04090 [100] N7E54_08430 transposase RS05105 [100] RS05230 [99.45] N7E54_08435 hypothetical protein RS05110 [100] RS05235 [99.75] N7E54_08440 glycosyltransferase RS05115 [100] RS05240 [100] N7E54_08445 capsular biosynthesis protein RS05120 [100] RS05245 [100] N7E54_08450 glycosyltransferase RS05125 [100] RS05250 [99.71] N7E54_08455 glycosyltransferase RS05130 [100] RS05255 [99.35] N7E54_08460 glycosyltransferase family 4 protein RS05135 [100] RS05260 [98.99] N7E54_08465 Wzy RS05140 [100] RS05265 [100] N7E54_08470 polysaccharide biosynthesis protein RS05145 [100] RS05270 [100] N7E54_08475 CpsD/CapB family tyrosine-protein kinase(Wze) RS05150 [100] RS05275 [100] N7E54_08480 serine hydrolase RS05155 [100] RS05280 [100] N7E54_08485 oligosaccharide flippase family protein (Wzx) RS05160 [100] RS05285 [100] 注:比对的植物乳植杆菌参考基因组accession number如下:YZH81,AL935263.2;LP90,CP_015857;JDM1,NC_012984;“[ ]”中数字为与YZH81同源蛋白基因序列相似性。 表 4 粘附蛋白基因
Table 4 Adhesion protein genes
编号 WCFS1 功能注释 YZH81 相似性(%) 1 LP_RS04070 Internalin-J precursor N7E54_07200 85.26 2 LP_RS05225 mannose-specific adhesin, LPXTG-motif cell wall anchor N7E54_11140 30.87 3 LP_RS07025 MucBP domain protein N7E54_01205 98.51 4 LP_RS10640 Internalin-J precursor N7E54_06005 98.79 5 LP_RS12860 IgA FC receptor precursor N7E54_11140 98.53 6 LP_RS13100 MucBP domain protein N7E54_11400 79.03 7 LP_RS13150 Internalin-J precursor N7E54_11445 95.90 8 LP_RS00795 Collagen adhesin precursor N7E54_03885 97.74 9 LP_RS07255 GlcNAc-binding protein A precursor N7E54_00975 99.01 10 LP_RS07650 hypothetical protein N7E54_00570 99.30 11 LP_RS10955 Cna protein B-type domain protein N7E54_05645 96.88 12 LP_RS12445 Collagen binding domain protein N7E54_10785 91.59 -
[1] 王烁, 姜静, 杜仁鹏, 等. 乳酸菌胞外多糖的生物合成、特性和应用[J]. 生物技术,2022,32(3):394−401. [WANG S, JIANG J, DU R P, et al. Biosynthesis, characterization and application of exopolysaccharides from lactic acid bacteria[J]. Biotechnology,2022,32(3):394−401. WANG S, JIANG J, DU R P, et al . Biosynthesis, characterization and application of exopolysaccharides from lactic acid bacteria[J]. Biotechnology,2022 ,32 (3 ):394 −401 .[2] PRETE R, ALAM M K, PERPETUINI G, et al. Lactic acid bacteria exopolysaccharides producers:A sustainable tool for functional foods[J]. Foods,2021,10(7):1653. doi: 10.3390/foods10071653
[3] 张丽, 张兰威, 韩雪. 乳酸菌胞外多糖的研究进展[J]. 食品工业科技,2012,33(17):378−381, 387. [ZHANG L, ZHANG L W, HAN X. Research and development of lactic acid bacteria exopolysaccharides[J]. Science and Technology of Food Industry,2012,33(17):378−381,387. ZHANG L, ZHANG L W, HAN X . Research and development of lactic acid bacteria exopolysaccharides[J]. Science and Technology of Food Industry,2012 ,33 (17 ):378 −381,387 .[4] 钱超. 乳酸菌产胞外多糖及其在食品中的应用[J]. 现代食品,2022,28(17):18−22. [QIAN C. Production of exopolysaccharide by lactic acid bacteria and its application in food[J]. Modern Food,2022,28(17):18−22. QIAN C . Production of exopolysaccharide by lactic acid bacteria and its application in food[J]. Modern Food,2022 ,28 (17 ):18 −22 .[5] 邓凯波. 乳酸菌胞外多糖的功能及在食品工业中的应用[J]. 食品安全导刊,2009(7):49−50. [DENG K B. The function of Lactobacillus extracellular polysaccharide and its application in food industry[J]. China Food Safety Magazine,2009(7):49−50. DENG K B . The function of Lactobacillus extracellular polysaccharide and its application in food industry[J]. China Food Safety Magazine,2009 (7 ):49 −50 .[6] POURJAFAR H, ANSARI F, SADEGHI A, et al. Functional and health-promoting properties of probiotics' exopolysaccharides; isolation, characterization, and applications in the food industry[J]. Critical Reviews in Food Science and Nutrition, 2022.
[7] XU Y M, CUI Y L, YUE F F, et al. Exopolysaccharides produced by lactic acid bacteria and Bifidobacteria:Structures, physiochemical functions and applications in the food industry[J]. Food Hydrocolloids,2019,94:475−499. doi: 10.1016/j.foodhyd.2019.03.032
[8] 曹永强, 张健, 赵雯, 等. 植物乳植杆菌胞外多糖的分离纯化及其乳化特性[J]. 食品科学,2016,37(17):7−13. [CAO Y Q, ZHANG J, ZHAO W, et al. Isolation, purification and emulsifying properties of exopolysaccharides produced by Lactobacillus plantarum[J]. Food Science,2016,37(17):7−13. CAO Y Q, ZHANG J, ZHAO W, et al . Isolation, purification and emulsifying properties of exopolysaccharides produced by Lactobacillus plantarum[J]. Food Science,2016 ,37 (17 ):7 −13 .[9] 兰冬雪, 瞿茜楠, 黄天, 等. 益生菌活性代谢产物的研究及应用进展[J]. 食品工业科技,2022,43(24):11−20. [LAN D X, QU X N, HUANG T, et al. Research and application progress of the active metabolites of probiotics[J]. Science and Technology of Food Industry,2022,43(24):11−20. LAN D X, QU X N, HUANG T, et al . Research and application progress of the active metabolites of probiotics[J]. Science and Technology of Food Industry,2022 ,43 (24 ):11 −20 .[10] 朱玉婷, 文欣, 向俊, 等. 植物乳植杆菌胞外多糖的单糖组成及抗氧化活性[J]. 乳业科学与技术,2022,45(6):7−11. [ZHU Y T, WEN X, XIANG J, et al. Monosaccharide composition and antioxidant activity of exopolysaccharides of Lactobacillus plantarum[J]. Journal of Dairy Science and Technology,2022,45(6):7−11. ZHU Y T, WEN X, XIANG J, et al . Monosaccharide composition and antioxidant activity of exopolysaccharides of Lactobacillus plantarum[J]. Journal of Dairy Science and Technology,2022 ,45 (6 ):7 −11 .[11] KAWAI Y, MARLES W J, CLEVERLEY R M, et al. A widespread family of bacterial cell wall assembly proteins[J]. The EMBO Journal,2011,30(24):4931−4941. doi: 10.1038/emboj.2011.358
[12] ZHANG Z Y, LIU C, ZHU Y Z, et al. Complete genome sequence of Lactobacillus plantarum JDM1[J]. Journal of Bacteriology,2009,191(15):5020−5021. doi: 10.1128/JB.00587-09
[13] WANG Y Y, CHEN C, AI L Z, et al. Complete genome sequence of the probiotic Lactobacillus plantarum ST-III[J]. Journal of Bacteriology,2011,193(1):313−314. doi: 10.1128/JB.01159-10
[14] KLEEREBEZEM M, BOEKHORST J, VAN K R, et al. Complete genome sequence of Lactobacillus plantarum WCFS1[J]. Proceedings of the National Academy of Sciences of the United States of America,2003,100(4):1990−1995.
[15] LAMONTANARA A, CAGGIANIELLO G, ORRÙ L, et al. Draft genome sequence of Lactobacillus plantarum Lp90 isolated from wine[J]. Genome Announcements,2015,3(2):e00097−15.
[16] 田佳乐, 刘洋, 李嘉雯, 等. 产胞外多糖嗜热链球菌 eps基因簇的表达及生物信息学分析[J]. 中国食品学报,2021,21(11):244−251. [TIAN J L, LIU Y, LI J W, et al. Expression of eps gene cluster in Streptococcus thermophilus exopolysaccharide and bioinformatics analysis[J]. Journal of Chinese Institute of Food Science and Technology,2021,21(11):244−251. TIAN J L, LIU Y, LI J W, et al . Expression of eps gene cluster in Streptococcus thermophilus exopolysaccharide and bioinformatics analysis[J]. Journal of Chinese Institute of Food Science and Technology,2021 ,21 (11 ):244 −251 .[17] ZEIDAN A A, POULSEN V K, JANZEN T, et al. Polysaccharide production by lactic acid bacteria:From genes to industrial applications[J]. FEMS Microbiology Reviews,2017,41(Supp_1):S168−S200. doi: 10.1093/femsre/fux017
[18] JIANG Y Y, YANG Z N. A functional and genetic overview of exopolysaccharides produced by Lactobacillus plantarum[J]. Journal of Functional Foods,2018,47:229−240. doi: 10.1016/j.jff.2018.05.060
[19] OLEKSY M, KLEWICKA E. Exopolysaccharides produced by Lactobacillus sp.:Biosynthesis and applications[J]. Critical Reviews in Food Science and Nutrition,2018,58(3):450−462.
[20] UMMUGULSUM T, BUSRA A, BELMA A, et al. The relationship between the structural characteristics of Lactobacilli-EPS and its ability to induce apoptosis in colon cancer cells in vitro[J]. Scientific Reports,2019,9(1):1−14. doi: 10.1038/s41598-018-37186-2
[21] REMUS D M, VAN KRANENBURG R, VAN SWAM I I, et al. Impact of 4 Lactobacillus plantarum capsular polysaccharide clusters on surface glycan composition and host cell signaling[J]. Microbial Cell Factories,2012,11(1):149. doi: 10.1186/1475-2859-11-149
[22] DURFEE T, NELSON R, BALDWIN S, et al. The complete genome sequence of Escherichia coli DH10B:Insights into the biology of a laboratory workhorse[J]. Journal of Bacteriology,2008,190(7):2597−2606. doi: 10.1128/JB.01695-07
[23] LAMBERT J M, BONGERS R S, KLEEREBEZEM M. Cre-lox-based system for multiple gene deletions and selectable-marker removal in Lactobacillus plantarum[J]. Applied and Environmental Microbiology,2007,73(4):1126−1135. doi: 10.1128/AEM.01473-06
[24] MARCO M L, BONGERS R S, DE VOS W M, et al. Spatial and temporal expression of Lactobacillus plantarum genes in the gastrointestinal tracts of mice[J]. Applied and Environmental Microbiology,2007,73(1):124−132. doi: 10.1128/AEM.01475-06
[25] ZHANG X, PRIETO A, MAAT V D, et al. Fitness determinants of vancomycin-resistant Enterococcus faecium during growth in human serum[J]. BioMed Central,2017,18(1):893.
[26] 李卫娜, 赵波, 柳陈坚, 等. 副干酪乳杆菌胞外多糖抗氧化活性分析[J]. 食品工业科技,2019,40(24):34−39. [LI W N, ZHAO B, LIU C J, et al. Antioxidant activity analysis of exopolysaccharide from Lactobacillus paracasei strains[J]. Science and Technology of Food Industry,2019,40(24):34−39. LI W N, ZHAO B, LIU C J, et al . Antioxidant activity analysis of exopolysaccharide from Lactobacillus paracasei strains[J]. Science and Technology of Food Industry,2019 ,40 (24 ):34 −39 .[27] YANG Y, ZHANG W J, HUAN H L, et al. Construction of an integrated mCherry red fluorescent protein expression system for labeling and tracing in Lactiplantibacillus plantarum WCFS1[J]. Frontiers in Microbiology,2021,12:690270−690270. doi: 10.3389/fmicb.2021.690270
[28] GAO Y Q, NIU M Z, YU X H, et al. Horizontally acquired polysaccharide-synthetic gene cluster from Weissella cibaria boosts the probiotic property of Lactiplantibacillus plantarum[J]. Frontiers in Microbiology,2021,12:692957−692957. doi: 10.3389/fmicb.2021.692957
[29] LEE I-C, CAGGIANIELLO G, VAN SWAM I I, et al. Strain-specific features of extracellular polysaccharides and their impact on Lactobacillus plantarum-host interactions[J]. Applied and Environmental Microbiology,2016,82(13):3959−3970. doi: 10.1128/AEM.00306-16
[30] 高云云, 李宝坤, 卢士玲, 等. 新疆传统乳品中产胞外多糖乳酸菌的筛选及益生特性的研究[J]. 中国酿造,2020,39(5):28−34. [GAO Y Y, LI B K, LU S L, et al. Screening and probiotic characteristics of extracellular polysaccharide-producing lactic acid bacteria from traditional dairy products in Xinjiang[J]. China Brewing,2020,39(5):28−34. GAO Y Y, LI B K, LU S L, et al . Screening and probiotic characteristics of extracellular polysaccharide-producing lactic acid bacteria from traditional dairy products in Xinjiang[J]. China Brewing,2020 ,39 (5 ):28 −34 .[31] ZHANG L, LIU C H, LI D, et al. Antioxidant activity of an exopolysaccharide isolated from Lactobacillus plantarum C88[J]. International Journal of Biological Macromolecules,2013,54:270−275. doi: 10.1016/j.ijbiomac.2012.12.037
[32] 代安娜尔, 乔少婷, 孙思霖, 等. 嗜热链球菌IMAU20246胞外多糖基因簇及其表达分析[J]. 微生物学报,2023,63(1):403−418. [DAI A, QIAO S T, SUN S L, et al. ldentification and expression profiling of exopolysaccharide gene cluster in Streptococcus thermophilus IMAU20246[J]. Acta Microbiologica Sinica,2023,63(1):403−418. DAI A, QIAO S T, SUN S L, et al . ldentification and expression profiling of exopolysaccharide gene cluster in Streptococcus thermophilus IMAU20246[J]. Acta Microbiologica Sinica,2023 ,63 (1 ):403 −418 .[33] 李敏, 李伟程, 刘亚华, 等. 乳酸乳球菌乳酸亚种IMAU11823全基因组测序及胞外多糖基因簇分析[J]. 中国食品学报,2021,21(4):256−266. [LI M, LI W C, LIU Y H, et al. Complete genome sequence of Lactococcus lactis subsp. lactis lMAU11823 and analysis of the exopolysaccharide biosynthesis gene cluster[J]. Journal of Chinese Institute of Food Science and Technology,2021,21(4):256−266. LI M, LI W C, LIU Y H, et al . Complete genome sequence of Lactococcus lactis subsp. lactis lMAU11823 and analysis of the exopolysaccharide biosynthesis gene cluster[J]. Journal of Chinese Institute of Food Science and Technology,2021 ,21 (4 ):256 −266 . -
期刊类型引用(0)
其他类型引用(1)





 下载:
下载:

 下载:
下载:
