Recombinant Expression and Characterization of A Novel Cold-Adapted β-Galactosidase from Paenibacillus polymyxa
-
摘要: 目的:本实验旨在挖掘一个适冷性新型β-半乳糖苷酶,为低温生产低乳糖乳制品或合成低聚半乳糖(Galactooligosaccharides,GOS)提供基础。方法:从多粘类芽孢杆菌(Paenibacillus polymyxa)基因组中克隆一个β-半乳糖苷酶(PpBgal42A)基因,构建重组表达质粒pET-28a-PpBgal42A在大肠杆菌BL21(DE3)中表达,经亲和层析纯化后研究重组β-半乳糖苷酶的酶学性质,利用高效液相色谱检测各糖分浓度,以GOS转化率为指标,评价PpBgal42A的转糖苷能力。结果:PpBgal42A与酸热脂环酸芽孢杆菌(Alicyclobacillus acidocaldarius)来源的糖苷水解酶42家族β-半乳糖苷酶同源性最高,为59.9%。纯化后,PpBgal42A的比活力为163.7 U/mg,分子量约为79 kDa。其最适pH和温度分别为pH7.5和35 ℃,在pH7.0~9.5范围内和4~35 ℃以下保持稳定,50 ℃时快速失活,35 ℃时的半衰期为1777 min。PpBgal42A利用350 g/L乳糖于30 ℃反应6 h后合成GOS的转化率为31.6%。结论:PpBgal42A是一个适冷性新型β-半乳糖苷酶,可水解乳糖并合成低聚半乳糖,具有潜在应用价值。Abstract: Objective: To analyze a novel cold-adapted β-galactosidase, which provided the basis for producing low lactose dairy products or synthesizing galactooligosaccharides (GOS) at low temperatures. Methods: The β-galactosidase (PpBgal42A) gene was cloned from the genome of Paenibacillus polymyxa. The recombinant plasmid (pET-28a-PpBgal42A) was constructed and successfully expressed in E.coli BL21(DE3). After purification by affinity chromatography, the enzymatic properties of recombinant β-galactosidase were studied. To evaluate the transglycosylation ability of PpBgal42A, the concentration of each component was determined by high performance liquid chromatography with the conversion rate of GOS as evaluation index. Results: PpBgal42A shared the highest identity of 59.9% with the GH family 42 β-galactosidase from Alicyclobacillus acidocaldarius. After purification, the specific activity of PpBgal42A was 163.7 U/mg and the molecular weight was about 79 kDa. Its optimal pH and temperature were pH7.5 and 35 ℃, respectively. It was stable in the range of pH7.0~9.5 and 4~35 ℃. It was rapidly inactivated at 50 ℃. The half-life of PpBgal42A at 35 ℃ was 1777 min. PpBgal42A synthesized 31.6% of GOS using 350 g/L lactose as the substrate at 30 ℃ for 6 h. Conclusion: PpBgal42A is a novel cold-adapted enzyme. It can hydrolyze lactose and synthesize galactooligosaccharides, which has potential application value.
-
β-半乳糖苷酶(EC 3.2.1.23,β-Galactosidase)具有水解和转糖苷的活性[1],可用于生产低乳糖乳制品,减轻乳糖不耐者食用乳制品后出现的腹胀、腹泻等不良反应,还可用于制备低聚半乳糖(Galactooligosaccharides,GOS)[2]。GOS是由一个或多个半乳糖基与葡萄糖或半乳糖连接形成的功能性低聚糖[3],可被肠道中双歧杆菌(Bifidobacteria)和乳杆菌(Lactobacilli)等利用,具有调节肠道菌群、促进矿物质吸收、增强免疫力等功效[4]。
《食品营养强化剂使用标准GB14880-2012》已批准GOS可添加至婴幼儿配方食品和谷类辅助食品中。米曲霉(Aspergillus oryzae)、环状芽孢杆菌(Bacillus circulans)和乳酸克鲁维酵母(Kluyveromyces lactis)来源的商业化β-半乳糖苷酶分别利用300、500和300 g/L乳糖于45、60和40 ℃合成GOS,转化率分别为19.5%、48.3%和34.9%[5]。婴儿双歧杆菌(Bifidobacterium infantis)来源的β-半乳糖苷酶利用300 g/L乳糖于60 ℃合成GOS的转化率为63.3%[6]。巴伦葛兹类芽孢杆菌(Paenibacillus barengoltzii)来源的β-半乳糖苷酶在40 ℃下催化350 g/L乳糖反应8 h合成GOS的转化率为47.9%[7]。硫磺矿硫化叶菌(Sulfolobus solfataricus)来源的β-半乳糖苷酶为耐热酶,于75 ℃高效合成GOS,转化率为60.5%[8]。目前多数β-半乳糖苷酶合成GOS的最适温度高于40 ℃,挖掘在较低温度下合成GOS的β-半乳糖苷酶可有效避免反应过程中非酶褐变并减少能耗[9]。
适冷性β-半乳糖苷酶在0~30 ℃之间保持高催化活性,适于在低温下制备低乳糖乳制品或合成GOS,同时适冷酶的构象稳定性差,中等温度即可破坏其二级和三级结构而导致失活[10−11]。如嗜冷乳糖节杆菌(Arthrobacter psychrolactophilus)F2产生的一种适冷性β-半乳糖苷酶在10 ℃下24 h水解牛奶中80%乳糖,在45 ℃下5 min失活[12]。南极节杆菌(Arthrobacter sp.)32cB来源的GH2家族β-半乳糖苷酶在高于45 ℃下极不稳定,但可在10 ℃下高效制备无乳糖乳制品和合成GOS[13]。适冷酶在食品工业生产中具有较大的应用潜力,可有效降低生产成本,减少能源浪费,保留产品风味和营养价值。因此挖掘适冷性β-半乳糖苷酶具有重要意义[10]。
多粘类芽孢杆菌(Paenibacillus polymyxa)是一种无致病性的植物根际促生菌,可分泌胞外多糖、抗菌化合物和植物激素等,这些代谢产物具有抑制肿瘤细胞和病原菌生长、提高农作物产量等功能[14−15]。迄今已有一些多粘类芽孢杆菌产糖苷水解酶的报道,如β-葡聚糖酶(β-Glucanase)[16]、β-甘露聚糖酶(β-Mannase)[17]和普鲁兰酶(Pullulanase)[18]等,但尚无多粘类芽孢杆菌来源β-半乳糖苷酶的报道。实验室筛选获得一株多粘类芽孢杆菌,可胞外分泌β-半乳糖苷酶,通过PDB(Protein Data Bank Proteins)数据库分析其与拉恩氏菌(Rahnella sp.)R3来源的适冷性β-半乳糖苷酶同源性较高(57.2%),推测该蛋白可能具有适冷酶的特性,本文将该基因在大肠杆菌中表达,研究酶学性质,分析该酶的温度稳定性,并用于合成低聚半乳糖。
1. 材料与方法
1.1 材料与仪器
多粘类芽孢杆菌L709 由本实验室自行筛选并于-80 ℃保藏;大肠杆菌DH5α、BL21(DE3)感受态细胞 北京全式金生物技术公司;载体pET-28a(+) Invitrogen公司;细菌基因组DNA提取试剂盒 天根生化科技有限公司;TransStart FastPfu DNA Polymerase 北京全式金生物技术公司;限制性内切酶和T4 DNA连接酶 TaKaRa公司;琼脂糖凝胶DNA/PCR产物中量回收试剂盒 杭州倍沃医学科技有限公司;Ni-NTA亲和层析柱 GE公司;葡萄糖测定试剂盒 上海荣盛生物药业股份有限公司;异丙基-β-D-硫代半乳糖苷(IPTG) 美国Sigma-Aldrich公司;邻硝基苯-β-D-吡喃半乳糖苷(oNP-β-Galactopyranoside,CAS号:369-07-3)、对硝基苯-β-D-吡喃半乳糖苷(pNP-β-Galactopyranoside,CAS号:3150-24-1)、对硝基苯-β-吡喃葡萄糖苷(pNP-β-Glucopyranoside,CAS号:2492-87-7)、对硝基苯-β-吡喃甘露糖苷(pNP-β-Mannopyranosidae,CAS号:252-633-9)、对硝基苯-β-吡喃木糖苷(pNP-β-Xylopyranosidae,CAS号:2001-96-9)、对硝基苯-α-吡喃半乳糖苷(pNP-α-Galactopyranoside,CAS号:7493-95-0)、对硝基苯-α-吡喃葡萄糖苷(pNP-α-Glucopyranoside,CAS号:3767-28-0) 分析纯,上海麦克林生化科技有限公司;其他试剂若无特殊说明均为分析纯。
T100TM Thermal Cycler 梯度PCR仪 Bio-Rad公司;JY92-IIDN型超声波细胞粉碎机 宁波新芝生物科技股份有限公司;TU-1901紫外-可见分光光度计 北京普析通用仪器设备有限公司;ÄKTA蛋白纯化系统 美国GE Healthcare公司;高效液相色谱(High Performance Liquid Chromatography,HPLC)系统 Agilent Technologies公司。
1.2 实验方法
1.2.1 β-半乳糖苷酶基因的克隆和表达载体的构建
多粘类芽孢杆菌L709接种于LB液体培养基中,30 ℃培养16 h,离心收集菌体,使用细菌基因组DNA提取试剂盒提取多粘类芽孢杆菌基因组DNA。依据基因组序列信息,设计上游引物PpBgal42A-F(5’ CGCGATCTC CATATGACGTATAGTAATATCACCAAATATC 3’)和下游引物PpBgal42A-R(5’ CAGATACCGCTCGAGTTAATTATTGCGTTCGGCTTTC 3’),以基因组DNA为模版扩增β-半乳糖苷酶基因。PCR反应体系:5×TransStart FastPfu Buffer 10 μL,2.5 mmol/L dNTPs 4 μL,引物PpBgal42A-F/R各1 μL,基因组DNA 1 μL,TransStart FastPfu DNA Polymerase 1 μL,补ddH2O至50 μL。PCR条件为:95 ℃预变性3 min;95 ℃变性20 s,55 ℃退火20 s,72 ℃延伸60 s,循环33次;72 ℃延伸5 min。PCR产物经琼脂糖凝胶电泳分析后切胶回收目的条带,用Nde I和Xho I酶切目的片段和载体pET-28a(+),由T4 DNA连接酶连接构建重组表达质粒pET-28a-PpBgal42A,将重组表达质粒转入大肠杆菌DH5α,在LB平板培养基中筛选出含有目的基因的阳性转化子,并提取质粒和测序。
1.2.2 β-半乳糖苷酶的序列分析
β-半乳糖苷酶的基因测序结果提交至NCBI数据库,利用NCBI ORF finder(http://www.ncbi.nlm.nih.gov/gorf/gorf.html)查找开放阅读框(Open Reading Frame,ORF);利用BLAST工具(https://blast. ncbi.nlm.nih.gov/Blast.cgi)进行序列同源性比对;通过NCBI CD-Search(https://www.ncbi.nlm. nih.gov/Structure/cdd/wrpsb.cgi)分析序列的保守结构域;将蛋白序列提交至SignalP 5.0(http://www.cbs.dtu.dk/services/SignallP/)预测信号肽序列;利用在线软件ExPASy ProtParam(http://web.expasy.org/protparam/)预测基因所编码蛋白质的分子量和等电点等;使用DNAMAN进行多序列比对分析并绘图。
1.2.3 β-半乳糖苷酶(PpBgal42A)的重组表达和纯化
重组表达质粒pET-28a-PpBgal42A转入大肠杆菌表达宿主BL21(DE3)中,挑取单菌落至20 mL含50 μg/mL卡那霉素的LB液体培养基中37 ℃培养12 h作为种子液,按1%(v/v)接种量转接至300 mL上述培养基中。当菌体密度OD600处于0.6~0.8时加入终浓度为1 mmol/L的IPTG,在16 ℃、200 r/min的条件下诱导培养16 h。4 ℃下10000×g离心5 min收集菌体,将菌体重悬在缓冲液A(20 mmol/L Na2HPO4-NaH2PO4,500 mmol/L NaCl,20 mmol/L咪唑,pH8.0)中,超声破壁后4 ℃、10000×g离心10 min得到粗酶液。预先用10个柱体积的缓冲液A平衡Ni-NTA亲和柱,粗酶液以0.5 mL/min流速上样,用缓冲液A洗脱未结合蛋白,再用缓冲液B(20 mmol/L Na2HPO4-NaH2PO4,500 mmol/L NaCl,100 mmol/L咪唑,pH8.0)以1.0 mL/min流速洗脱目的蛋白。在Na2HPO4-NaH2PO4缓冲液(20 mmol/L,pH8.0)中透析(透析袋MD77,8000~14000 Da),用SDS-PAGE法分析蛋白纯度[19]。
1.2.4 PpBgal42A酶活力和蛋白浓度的测定
以邻硝基苯-β-D-吡喃半乳糖苷(oNP-β-Galactopyranoside,oNPG)为底物测定β-半乳糖苷酶的酶活力[20]:75 μL 15 mmol/L oNPG和150 μL 50 mmol/L Na2HPO4-NaH2PO4缓冲液(pH7.5)混合。35 ℃保温3 min后加入25 μL适当稀释的酶液,35 ℃反应10 min后加入750 μL 2 mol/L Na2CO3溶液终止反应,用紫外-可见分光光度计在410 nm下测定邻硝基苯酚的量。一个酶活力单位定义为:在上述反应条件下,每分钟催化oNPG水解释放1 μmol邻硝基苯酚所需的酶量。参照Lowry法[21]测定蛋白浓度,以牛血清蛋白(Bovine Serum Albumin,BSA)作为标准蛋白。
1.2.5 PpBgal42A酶学性质的测定
最适pH和pH稳定性测定:按照1.2.4中酶活力测定方法,以酶活力最大值为100%,分别计算不同pH值下的相对酶活力。所用缓冲液体系和pH缓冲范围如下:柠檬酸-柠檬酸三钠缓冲液(Citrate,pH3.0~6.0),乙酸-乙酸钠缓冲液(Acetate,pH4.0~5.5),2-吗啉乙磺酸缓冲液(MES,pH5.0~6.5),Na2HPO4-NaH2PO4缓冲液(PBS,pH6.0~8.0),三羟甲基氨基甲烷-盐酸缓冲液(Tris-HCl,pH7.0~9.0),甘氨酸-氢氧化钠缓冲液(Glycine-NaOH,pH8.5~10.5),3-环己胺-1-丙磺酸缓冲液(CAPS,pH10.0~11.0)。在上述不同pH缓冲液中测定pH稳定性,35 ℃保温30 min后迅速置于冰水浴中冷却30 min,按照上述酶活力测定方法测定残余酶活力,以未经处理的酶液为对照,分别计算不同pH条件下的相对酶活力。
最适温度和温度稳定性的测定:用最适pH缓冲液配制15 mmol/L oNPG,按照1.2.4中酶活力测定方法在不同温度(20~70 ℃)下测定酶活力,以酶活力最大值为100%,分别计算不同温度下的相对酶活力。温度稳定性在最适pH和不同温度(20~70 ℃)下测定,保温30 min后迅速置于冰水浴中冷却30 min,在最适条件下测定残余酶活力。在4、10、20 ℃下处理30、60、120、180 min后测定残余酶活力,进一步探究低温下PpBgal42A的稳定性,以未经处理的酶液为对照,计算不同温度处理后残余酶活力的相对值。
半衰期的测定:用最适pH缓冲液稀释酶液,在30、35、40、45、50 ℃下进行保温反应,分别在5、15、30、60、90、120、150和180 min取样并迅速置于冰水浴中冷却30 min,在最适条件下测定残余酶活力。酶液在不同温度下酶活力降低至50%时所需的时间即为该温度条件下的半衰期[22]。
1.2.6 PpBgal42A的底物特异性
用50 mmol/L Na2HPO4-NaH2PO4缓冲液(pH7.5)配制15 mmol/L邻硝基苯-β-D-吡喃半乳糖苷(β-oNPGal)、对硝基苯-β-D-吡喃半乳糖苷(β-pNPGal)、对硝基苯-β-吡喃葡萄糖苷(β-pNPGlu)、对硝基苯-β-吡喃甘露糖苷(β-pNPMan)、对硝基苯-β-吡喃木糖苷(β-pNPXyl)、对硝基苯-α-吡喃半乳糖苷(α-pNPGal)、对硝基苯-α-吡喃葡萄糖苷(α-pNPGlu)和10 mg/mL乳糖,pNP合成底物酶活力测定按照1.2.4中酶活力测定方法进行,乳糖底物酶活力测定参照葡萄糖检测试剂盒(GOD-POD比色法)说明书:将适当稀释的酶液与乳糖混合,35 ℃保温10 min后用葡萄糖测定试剂盒测定葡萄糖浓度,通过检测溶液中释放出的葡萄糖浓度来计算酶活力。oNPG为底物时酶活力为100%,分别计算β-半乳糖苷酶对不同底物的比活力和相对酶活力。一个酶活力单位定义为:在上述反应条件下,每分钟生成1 μmol硝基苯(oNP或pNP)或葡萄糖所需的酶量。
1.2.7 PpBgal42A水解乳糖
用50 mmol/L Na2HPO4-NaH2PO4缓冲液(pH8.0)配制50 g/L乳糖溶液,添加10 U/mL的PpBgal42A,混合均匀后于30 ℃反应0.25、0.5、1、2、4、6、8、12和24 h后取样煮沸5 min灭酶,适当稀释样品后进行定性定量分析。
薄层层析法(Thin Layer Chromatography,TLC)定性分析水解产物[7]:样品于TLC分析板上展层两次,用显色剂完全浸湿吹干后在180 ℃显色。展层剂为正丁醇:乙醇:水=5:3:2,显色剂为硫酸、甲醇溶液(5%,v/v),标准品为半乳糖、葡萄糖和乳糖的混合溶液(各组分终浓度为1 mg/mL)。
高效液相色谱(HPLC)定量分析水解产物[7]:色谱柱为BP-800Pb++(Benson Polymeric,Reno,NE,USA),流动相为超纯水,进样量10 μL,流速0.6 mL/min,柱温80 ℃,检测器为示差折光检测器,标准品为半乳糖、葡萄糖和乳糖的混合溶液(各组分终浓度为1 mg/mL)。计算公式如下:
水解率(%)=初始乳糖浓度(g/L)−取样时乳糖浓度(g/L)初始乳糖浓度(g/L)×100 1.2.8 PpBgal42A合成GOS
利用PpBgal42A的转糖苷能力合成GOS,合成条件优化主要包括初始乳糖浓度(250、300、350、400、450 g/L)、反应温度(25、30、35、40、45 ℃)、反应pH(7.0、7.5、8.0、8.5、9.0)、加酶量(2、3、4、5、6 U/mL)和反应时间(0.25、0.5、1、2、4、6、8、12、24 h)五个因素,单因素优化实验固定其他参数为:350 g/L、35 ℃、pH7.5、5 U/mL、4 h。用TLC和HPLC定性、定量分析待测样品中GOS的浓度和转化率,确定最优合成条件。TLC和HPLC的检测条件同1.2.7所示。计算公式如下[23]:
GOS的转化率(%)=A1−A2−A3−A4A1×100 式中:A1表示初始乳糖浓度,g/L;A2表示取样时乳糖浓度,g/L;A3表示取样时葡萄糖浓度,g/L;A4表示取样时半乳糖浓度,g/L。
1.3 数据处理
采用GraphPad Prism进行数据整理和图片处理,每组数据均有设3次平行。
2. 结果与分析
2.1 β-半乳糖苷酶基因的克隆和序列分析
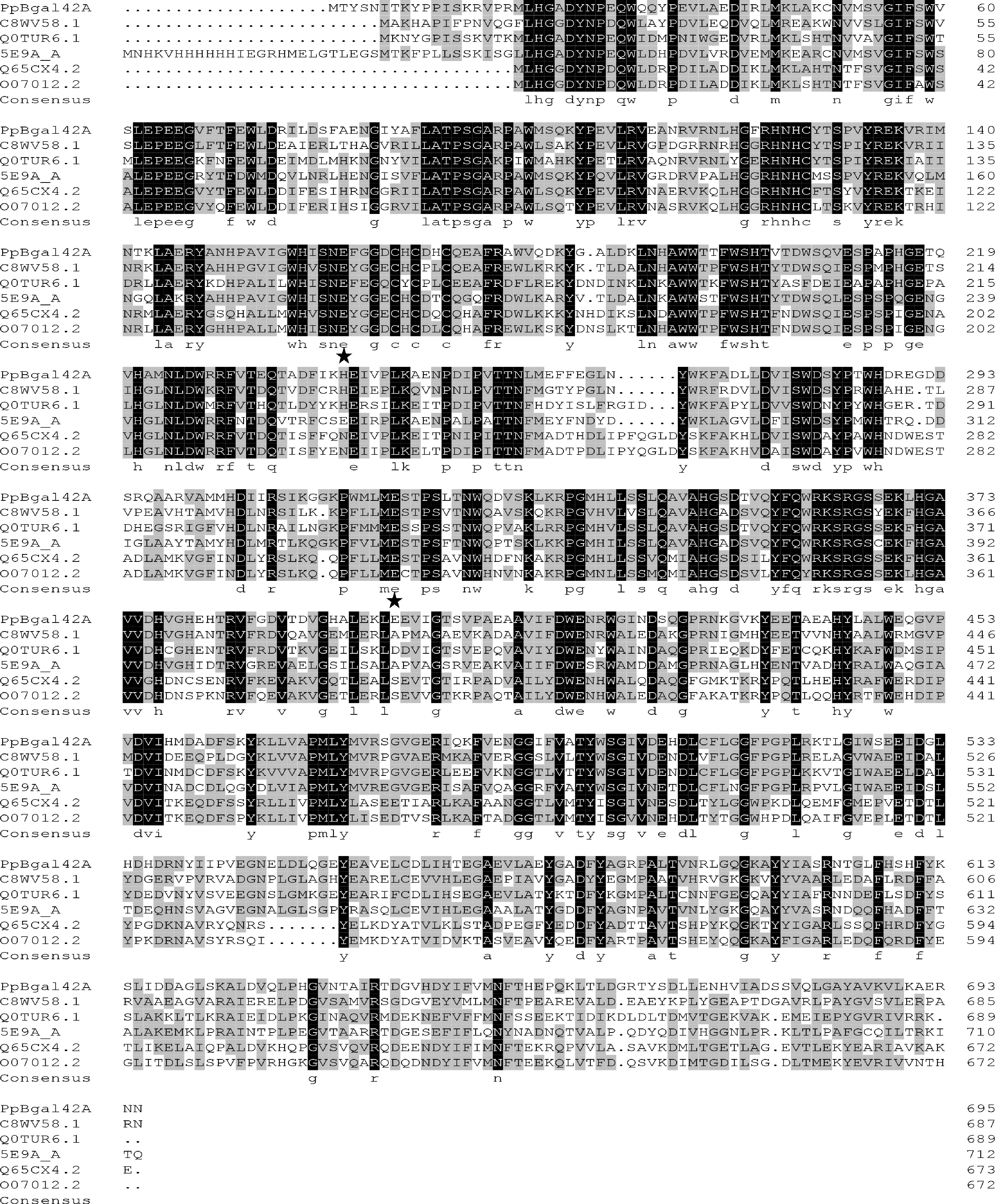
以多粘类芽孢杆菌L709基因组为模版,利用PCR扩增出一条长度为2088 bp的基因片段PpBgal42A,该基因片段编码695个氨基酸。SignalP 5.0预测PpBgal42A不含信号肽,ExPASy分析表明该蛋白的理论分子量和等电点分别为79 kDa和5.58。NCBI CD-Search分析表明PpBgal42A含有GH42家族保守结构域。BLAST蛋白序列比对表明(图1),该基因编码的蛋白与酸热脂环酸芽孢杆菌(Alicyclobacillus acidocaldarius)来源的β-半乳糖苷酶(GenBank: C8WV58.1)同源性最高,为59.9%;其次,与产气荚膜梭菌(Clostridium perfringens)(GenBank: Q0TUR6.1)、拉恩氏菌(Rahnella sp.)R3(PDB:5E9A_A)、地衣芽孢杆菌(Bacillus licheniformis)DSM 13(GenBank: Q65CX4.2)和枯草芽孢杆菌(Bacillus subtilis)(GenBank: O07012.2)来源的β-半乳糖苷酶的同源性分别为58.2%、57.2%、49.3%和49.2%。此外,两个保守的谷氨酸残基(E162和E320)可能作为广义酸/碱和亲核试剂,在酶分子与底物的结合和催化中起关键作用。
![]() 图 1 多粘类芽孢杆菌β-半乳糖苷酶(PpBgal42A)与其他β-半乳糖苷酶的多重序列比对注:黑色阴影表示相同残基,灰色阴影表示保守残基。PpBgal42A多重序列比对中所用的序列分别为:酸热脂环酸芽孢杆菌来源的β-半乳糖苷酶(C8WV58.1)、产气荚膜梭菌来源的β-半乳糖苷酶(Q0TUR6.1)、拉恩氏菌R3来源的β-半乳糖苷酶(5E9A_A)、地衣芽孢杆菌DSM 13来源的β-半乳糖苷酶(Q65CX4.2)和枯草芽孢杆菌来源的β-半乳糖苷酶(O07012.2)。两个保守的催化残基用黑色五角星标记。Figure 1. Multiple alignment of amino acid sequences of PpBgal42A and other β-galactosidases
图 1 多粘类芽孢杆菌β-半乳糖苷酶(PpBgal42A)与其他β-半乳糖苷酶的多重序列比对注:黑色阴影表示相同残基,灰色阴影表示保守残基。PpBgal42A多重序列比对中所用的序列分别为:酸热脂环酸芽孢杆菌来源的β-半乳糖苷酶(C8WV58.1)、产气荚膜梭菌来源的β-半乳糖苷酶(Q0TUR6.1)、拉恩氏菌R3来源的β-半乳糖苷酶(5E9A_A)、地衣芽孢杆菌DSM 13来源的β-半乳糖苷酶(Q65CX4.2)和枯草芽孢杆菌来源的β-半乳糖苷酶(O07012.2)。两个保守的催化残基用黑色五角星标记。Figure 1. Multiple alignment of amino acid sequences of PpBgal42A and other β-galactosidases2.2 β-半乳糖苷酶(PpBgal42A)的重组表达和纯化
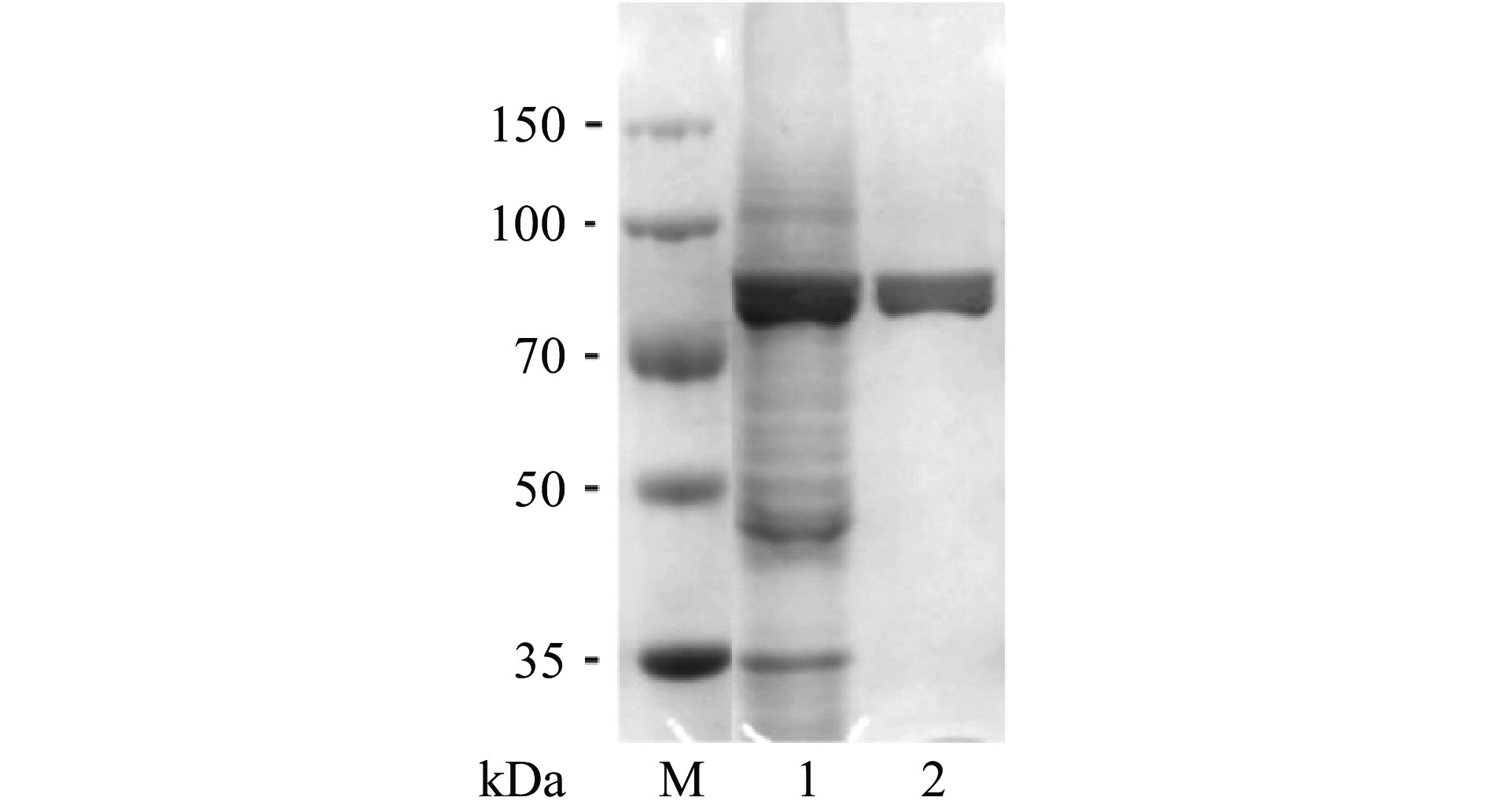
PpBgal42A成功在大肠杆菌BL21(DE3)中可溶表达,用ÄKTA蛋白纯化系统将目标蛋白和杂蛋白分离,得到电泳级纯酶,纯化结果见表1。SDS-PAGE表明在80 kDa处显示单一条带,与预测分子量79 kDa相近(图2)。酶活力回收率为60.1%,比活力由31.3 U/mg提高到163.7 U/mg,纯化倍数为5.2。
表 1 重组β-半乳糖苷酶(PpBgal42A)纯化过程Table 1. Purification of the recombinant β-galactosidase (PpBgal42A)纯化步骤 总酶活力
(U)总蛋白
(mg)比活力
(U/mg)纯化倍数 回收率
(%)a粗酶液 3376.8 107.88 31.3 1.0 100.0 亲和层析 2029.9 12.4 163.7 5.2 60.1 注: a:回收率为纯化后总酶活力占纯化前总酶活力的百分比。 2.3 PpBgal42A的酶学性质
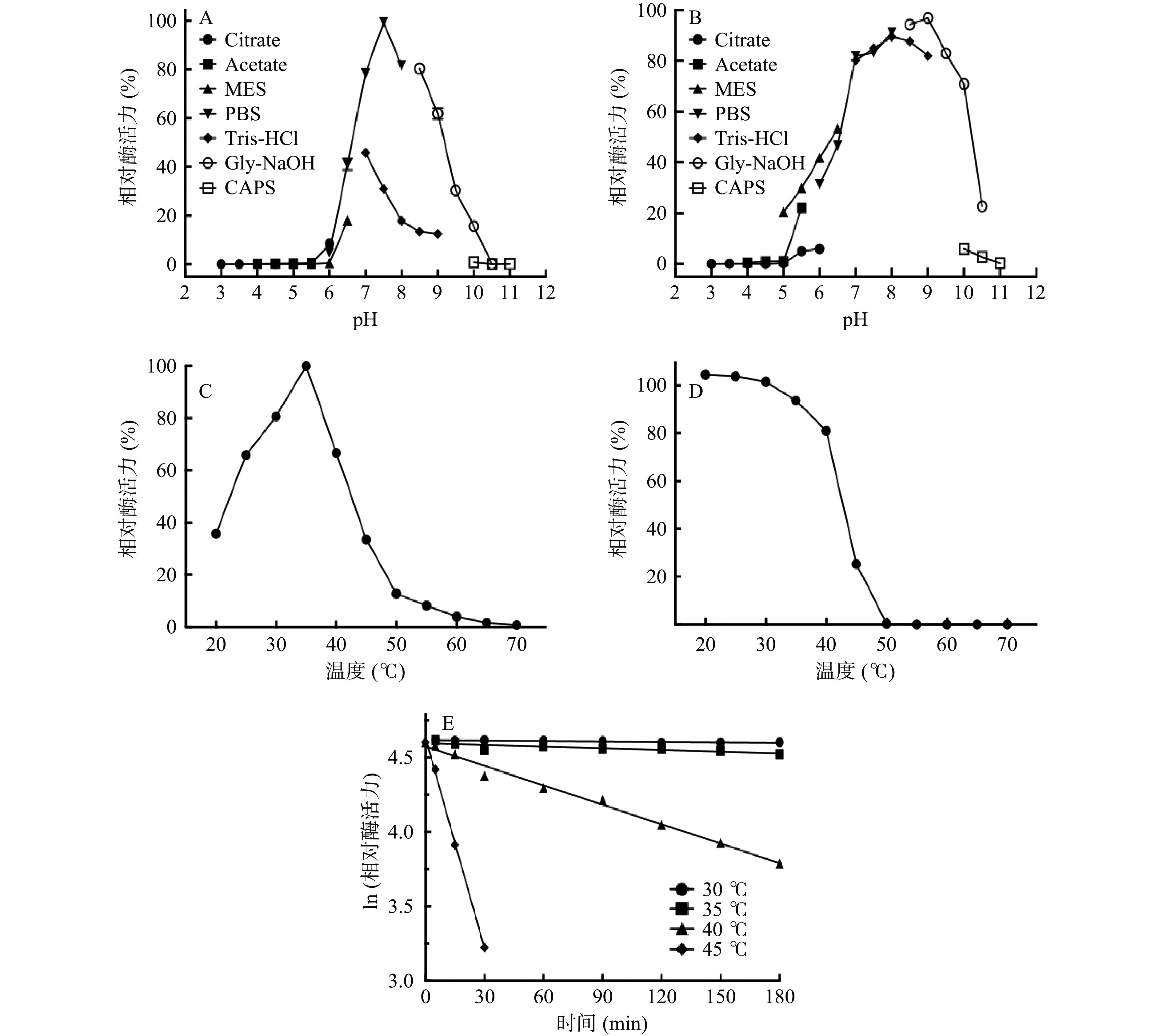
PpBgal42A的基本酶学性质如图3。PpBgal42A最适pH为7.5,在pH7.0~9.5范围内保持80%以上相对酶活力。PpBgal42A在35 ℃时酶活力最高,20 ℃时相对酶活力为36%。该酶在35 ℃以下能保留90%以上相对酶活力,高于40 ℃时酶活力急剧下降。PpBgal42A在30、35、40和45 ℃下的半衰期分别为7145、1777、157和15 min(50 ℃下5 min失活)。进一步分析PpBgal42A的温度稳定性(图3E,表2),其在20 ℃以下保温180 min后无酶活力损失,45 ℃处理15 min后相对酶活力仅为50%,50 ℃下快速失活。
表 2 低温下PpBgal42A的稳定性Table 2. Stability of PpBgal42A at low temperatures时间(min) 相对酶活力(%) 4 ℃ 10 ℃ 20 ℃ 30 104.5±0.5 102.3±0.5 104.5±0.6 60 104.6±0.4 104.7±0.5 101.3±0.7 120 105.2±0.5 102.8±0.5 100.5±0.4 180 104.3±0.6 102.6±0.3 100.3±0.6 2.4 PpBgal42A的底物特异性
PpBgal42A的底物特异性见表3。对β键连接的合成底物β-oNPGal的催化活性最高(100%),其次为β-pNPGal,比活力为102.5 U/mg。以乳糖为底物时,PpBgal42A仅表现出7.0%的相对酶活力。该酶对β-pNPGlu、β-pNPMan、β-pNPXyl、α-pNPGal和α-pNPGlu均没有表现出水解活性,表明其对β键连接的半乳糖苷化合物的高度专一性。
表 3 PpBgal42A底物特异性Table 3. Substrate specificity of PpBgal42A底物 比活力(U/mg) 相对酶活力(%) 邻硝基苯-β-D-吡喃半乳糖苷 (β-oNPGal) 163.7±1.1 100 对硝基苯-β-D-吡喃半乳糖苷(β-pNPGal) 102.5±0.3 62.6 对硝基苯-β-吡喃葡萄糖苷 (β-pNPGlu) − 0 对硝基苯-β-吡喃甘露糖苷 (β-pNPMan) − 0 对硝基苯-β-吡喃木糖苷 (β-pNPXyl) − 0 对硝基苯-α-吡喃半乳糖苷 (α-pNPGal) − 0 对硝基苯-α-吡喃葡萄糖苷 (α-pNPGlu) − 0 乳糖 11.5±0.3 7.0 2.5 PpBgal42A的水解特性
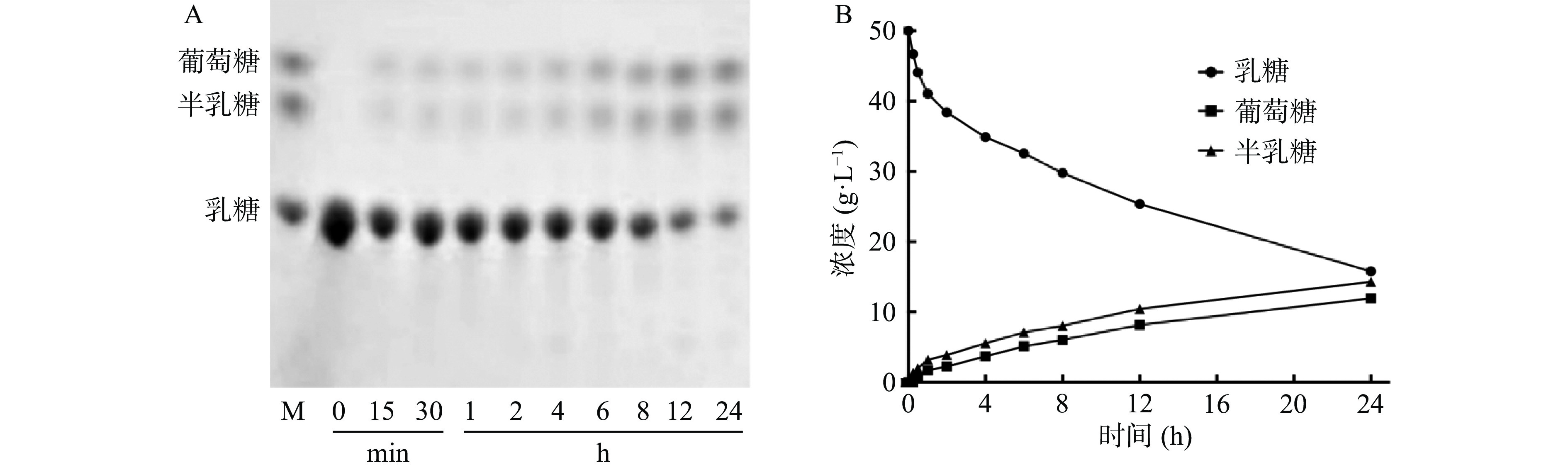
以50 g/L乳糖溶液考察PpBgal42A的水解特性(图4)。该酶能水解乳糖生成葡萄糖和半乳糖,HPLC定量分析表明,在pH8.0和30 ℃的条件下反应24 h,PpBgal42A可水解68.3%的乳糖。
2.6 PpBgal42A的转糖苷特性
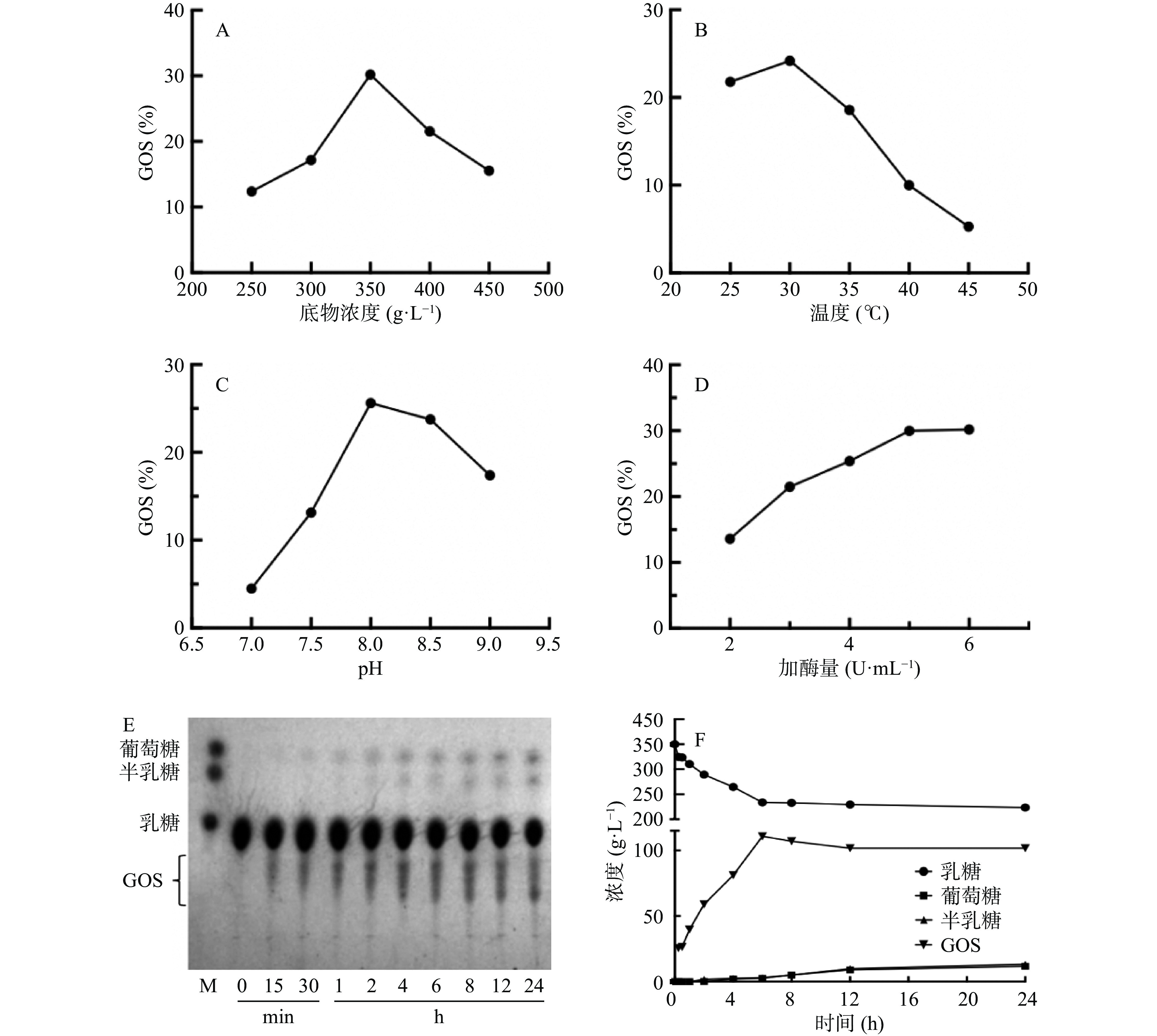
底物浓度、温度、pH和加酶量对PpBgal42A发挥转糖苷能力合成GOS的影响如图5。GOS的转化率随着底物浓度的增加而增加,当底物浓度高于350 g/L时,转化率逐渐降低;30 ℃时PpBgal42A合成GOS的转化率最高,当反应温度高于30 ℃时,GOS的转化率降低;当pH由中性变为碱性时,GOS转化率先增加后降低,在pH8.0时最高;随着加酶量的增加,GOS转化率先增加后趋于平稳,加酶量为5 U/mL时转化率较高。经反应条件优化后,HPLC检测结果表明加入5 U/mL的PpBgal42A,以350 g/L乳糖为底物,在pH8.0和30 ℃的条件下反应6 h,合成GOS的转化率为31.6%。
3. 讨论
β-半乳糖苷酶在乳品工业中应用广泛[24−25]。本文从多粘类芽孢杆菌中克隆了一个β-半乳糖苷酶(PpBgal42A)基因,并在大肠杆菌中表达,纯化后研究其酶学性质。PpBgal42A含有GH42家族保守结构域,与酸热脂环酸芽孢杆菌来源的β-半乳糖苷酶具有最高的同源性(59.9%,图1),表明PpBgal42A是一个新型的GH42家族β-半乳糖苷酶。重组β-半乳糖苷酶PpBgal42A经Ni-NTA亲和层析纯化得到纯酶,比酶活为163.7 U/mg(表1),高于乳酸杆菌(Lactobacillus sp. B164)(122.90 U/mg)[26]、芽孢杆菌(Bacillus sp.)KW1(86.99 U/mg)[27]和乙酰微小杆菌(Exiguobacterium acetylicum)(82.6 U/mg)[28]等来源的GH42家族β-半乳糖苷酶。
PpBgal42A最适pH为7.5(图3A),与海栖热袍菌(Thermotoga maritima)[29]、乙酰微小杆菌[28]等来源的GH42家族β-半乳糖苷酶的最适pH相近。该酶在pH7.0~9.5具有较好的稳定性(图3B),与交替单胞菌(Alteromonas sp. ML52)(pH7.0~8.5)[30]和南极节杆菌32cB(pH7.0~8.5)[13]来源的β-半乳糖苷酶的pH稳定范围相近。PpBgal42A在35 ℃时表现出最高酶活力(图3C),与拉恩氏菌R3来源的β-半乳糖苷酶的最适温度相同[10]。该酶在30 ℃以下高度稳定,温度上升至40 ℃时酶活力急剧下降(图3D),30 ℃以下保温3 h后仍十分稳定,35 ℃处理29 h后可保留50%酶活力,45 ℃处理15 min后仅剩50%酶活力,50 ℃以上迅速失活(图3E,表2),与南极节杆菌32cB[13]和拉恩氏菌R3[10]来源的适冷性β-半乳糖苷酶的热稳定性相似,易热失活。PpBgal42A对温度变化敏感,具有在低温下保持稳定,在中高温时易变性失活的适冷性特征[10−11],是一个新型适冷酶。目前用于生产低乳糖乳制品的商业β-半乳糖苷酶,如Lactozym Pure®(Novozymes)和Maxilact®(DSM Food Specialties)在37 ℃具有较高活性,但当温度降低时活性迅速下降[30]。
PpBgal42A对人工合成底物β-oNPGal和β-pNPGal表现出较高的水解活性,但对天然底物乳糖的相对酶活力仅为β-oNPGal的7.0%(表3)。大多数GH42家族β-半乳糖苷酶对乳糖的水解能力较差,如土壤来源的β-半乳糖苷酶zd410(对乳糖的相对酶活力为β-oNPGal的10.5%)[31]和乙酰微小杆菌来源的β-半乳糖苷酶β-galEa(无乳糖水解活性)[28]。GH42家族β-半乳糖苷酶的活性易受乳糖水解产物的强烈抑制,因此水解乳糖的能力较弱[32],已报道的对乳糖有较强水解能力的GH42家族β-半乳糖苷酶主要来源于拉恩氏菌R3[10]和冷杆菌(Cryobacterium sp. LW097)[32]。
β-半乳糖苷酶在合成GOS的同时也会将GOS水解成单糖。单因素实验表明在pH8.0和30 ℃条件下,以350 g/L乳糖为底物,加入5 U/mL的PpBgal42A反应6 h合成GOS的转化率最高,为31.6%(图5F)。许多GH42家族β-半乳糖苷酶合成GOS的转化率低,甚至无法作用于乳糖发挥转糖苷能力,如乙酰微小杆菌[28]和枯草芽孢杆菌来源[33]的β-半乳糖苷酶,而地衣芽孢杆菌DSM 13来源的β-半乳糖苷酶在60 ℃下合成GOS的转化率仅为12%[34]。本研究中重组酶PpBgal42A在较低温度下合成GOS的转化率高,在乳制品加工中具有很大的应用潜力。
4. 结论
从多粘类芽孢杆菌中克隆一个新型GH42家族β-半乳糖苷酶基因并在大肠杆菌中表达。重组β-半乳糖苷酶PpBgal42A是一个新型适冷酶,具有较高的比活力(163.7 U/mg),其最适pH和最适温度分别为pH7.5和35 ℃。该酶在低温下稳定,高于45 ℃时易失活。 PpBgal42A在最适条件下利用350 g/L乳糖合成GOS的转化率为31.6%。本研究为β-半乳糖苷酶在低温乳品中的应用提供基础。
-
图 1 多粘类芽孢杆菌β-半乳糖苷酶(PpBgal42A)与其他β-半乳糖苷酶的多重序列比对
注:黑色阴影表示相同残基,灰色阴影表示保守残基。PpBgal42A多重序列比对中所用的序列分别为:酸热脂环酸芽孢杆菌来源的β-半乳糖苷酶(C8WV58.1)、产气荚膜梭菌来源的β-半乳糖苷酶(Q0TUR6.1)、拉恩氏菌R3来源的β-半乳糖苷酶(5E9A_A)、地衣芽孢杆菌DSM 13来源的β-半乳糖苷酶(Q65CX4.2)和枯草芽孢杆菌来源的β-半乳糖苷酶(O07012.2)。两个保守的催化残基用黑色五角星标记。
Figure 1. Multiple alignment of amino acid sequences of PpBgal42A and other β-galactosidases
表 1 重组β-半乳糖苷酶(PpBgal42A)纯化过程
Table 1 Purification of the recombinant β-galactosidase (PpBgal42A)
纯化步骤 总酶活力
(U)总蛋白
(mg)比活力
(U/mg)纯化倍数 回收率
(%)a粗酶液 3376.8 107.88 31.3 1.0 100.0 亲和层析 2029.9 12.4 163.7 5.2 60.1 注: a:回收率为纯化后总酶活力占纯化前总酶活力的百分比。 表 2 低温下PpBgal42A的稳定性
Table 2 Stability of PpBgal42A at low temperatures
时间(min) 相对酶活力(%) 4 ℃ 10 ℃ 20 ℃ 30 104.5±0.5 102.3±0.5 104.5±0.6 60 104.6±0.4 104.7±0.5 101.3±0.7 120 105.2±0.5 102.8±0.5 100.5±0.4 180 104.3±0.6 102.6±0.3 100.3±0.6 表 3 PpBgal42A底物特异性
Table 3 Substrate specificity of PpBgal42A
底物 比活力(U/mg) 相对酶活力(%) 邻硝基苯-β-D-吡喃半乳糖苷 (β-oNPGal) 163.7±1.1 100 对硝基苯-β-D-吡喃半乳糖苷(β-pNPGal) 102.5±0.3 62.6 对硝基苯-β-吡喃葡萄糖苷 (β-pNPGlu) − 0 对硝基苯-β-吡喃甘露糖苷 (β-pNPMan) − 0 对硝基苯-β-吡喃木糖苷 (β-pNPXyl) − 0 对硝基苯-α-吡喃半乳糖苷 (α-pNPGal) − 0 对硝基苯-α-吡喃葡萄糖苷 (α-pNPGlu) − 0 乳糖 11.5±0.3 7.0 -
[1] KOLEV P, ROCHA-MENDOZA D, RUIZ-RAMÍREZ S, et al. Screening and characterization of β-galactosidase activity in lactic acid bacteria for the valorization of acid whey[J]. JDS Communications,2022,3(1):1−6. doi: 10.3168/jdsc.2021-0145
[2] LU L L, GUO L C, WANG K, et al. β-Galactosidases:A great tool for synthesizing galactose-containing carbohydrates[J]. Biotechnology Advances,2020,39:107465. doi: 10.1016/j.biotechadv.2019.107465
[3] GAO X, WU J, WU D. Rational design of the β-galactosidase from Aspergillus oryzae to improve galactooligosaccharide production[J]. Food Chemistry,2019,286:362−367. doi: 10.1016/j.foodchem.2019.01.212
[4] ARSOV A, IVANOV I, TSIGORIYNA L, et al. In vitro production of galactooligosaccharides by a novel β-galactosidase of Lactobacillus bulgaricus[J]. International Journal of Molecular Sciences,2022,23(22):14308. doi: 10.3390/ijms232214308
[5] YIN H F, BULTERMA J B, DIJKHUIZEN L, et al. Reaction kinetics and galactooligosaccharide product profiles of the β-galactosidases from Bacillus circulans, Kluyveromyces lactis and Aspergillus oryzae[J]. Food Chemistry,2017,225:230−238. doi: 10.1016/j.foodchem.2017.01.030
[6] HUNG M N, LEE B H. Purification and characterization of a recombinant β-galactosidase with transgalactosylation activity from Bifidobacterium infantis HL96[J]. Applied Microbiology and Biotechnology,2002,58(4):439−445. doi: 10.1007/s00253-001-0911-6
[7] LIU Y, CHEN Z, JIANG Z Q, et al. Biochemical characterization of a novel β-galactosidase from Paenibacillus barengoltzii suitable for lactose hydrolysis and galactooligosaccharides synthesis[J]. International Journal of Biological Macromolecules,2017,104:1055−1063. doi: 10.1016/j.ijbiomac.2017.06.073
[8] SUN X J, DUAN X G, WU D, et al. Characterization of Sulfolobus solfataricus β-galactosidase mutant F441Y expressed in Pichia pastoris[J]. Journal of the Science of Food and Agriculture,2014,94(7):1359−1365. doi: 10.1002/jsfa.6419
[9] RUTKIEWICZ M, BUJACZ A, BUJACZ G. Structural features of cold-adapted dimeric GH2 β-D-galactosidase from Arthro bacter sp. 32cB[J]. BBA - Proteins and Proteomics,2019,1867(9):776−786. doi: 10.1016/j.bbapap.2019.06.001
[10] FAN Y T, HUA X, ZHANG Y Z, et al. Cloning, expression and structural stability of a cold-adapted β-galactosidase from Rahnella sp. R3[J]. Protein Expression and Purification,2015,115:158−164. doi: 10.1016/j.pep.2015.07.001
[11] MANGIAGALLI M, LOTTI M. Cold-active β-galactosidases:insight into cold adaption mechanisms and biotechnological exploitation[J]. Marine Drugs,2021,19(1):43. doi: 10.3390/md19010043
[12] NAKAGAWA T, FUJIMOTO Y, IKEHATA R, et al. Purification and molecular characterization of cold-active β-galactosidase from Arthrobacter psychrolactophilus strain F2[J]. Applied Microbiology and Biotechnology,2006,72(4):720−725. doi: 10.1007/s00253-006-0339-0
[13] PAWLAK-SZUKALSKA A, WANARSKA M, TOMASZ A, et al. A novel cold-active β-d-galactosidase with transglycosylation activity from the Antarctic Arthrobacter sp. 32cB-gene cloning, purification and characterization[J]. Process Biochemistry,2014,49(12):2122−2133. doi: 10.1016/j.procbio.2014.09.018
[14] TIMMUSK S, COPOLOVICI D, COPOLOVICI L, et al. Paenibacillus polymyxa biofilm polysaccharides antagonise Fusarium graminearum[J]. Scientific Reports,2019,9(1):1−11. doi: 10.1038/s41598-018-37186-2
[15] SAAD M A, ABDRABOU H S, ELKHTAB E, et al. Occurrence of toxic biogenic amines in various types of soft and hard cheeses and their control by Bacillus polymyxa D05-1[J]. Fermentation,2022,8(7):327. doi: 10.3390/fermentation8070327
[16] YUAN Y, ZHANG X Y, ZHANG H, et al. Degradative GH5 β-1, 3-1, 4-glucanase PpBglu5A for glucan in Paenibacillus polymyxa KF-1[J]. Process Biochemistry,2020,98:183−192. doi: 10.1016/j.procbio.2020.08.008
[17] HORI K, KAWABATA Y, NAKAZAWA Y, et al. A novel β-1, 4-mannanase isolated from Paenibacillus polymyxa KT551[J]. Food Science and Technology Research,2014,20(6):1261−1265. doi: 10.3136/fstr.20.1261
[18] 张春园, 张鸣明, 胡先望, 等. 多粘类芽孢杆菌新普鲁兰酶基因克隆及其在大肠杆菌中的表达[J]. 食品工业科技,2020,41(15):119−123,128 doi: 10.13386/j.issn1002-0306.2020.15.019 ZHANG C Y, ZHANG M M, HU X W, et al. Cloning of neopullulanase gene from Paenibacillus polymyxa and its expression in Escherichia coli[J]. Science and Technology of Food Industry,2020,41(15):119−123,128. doi: 10.13386/j.issn1002-0306.2020.15.019
[19] LAEMMLI U K. Cleavage of structural proteins during the assembly of the head of bacteriophage T4[J]. Nature,1970,227(5259):680−685. doi: 10.1038/227680a0
[20] KATROLIA P, YAN Q J, JIA H Y, et al. Molecular cloning and high-level expression of a β-galactosidase gene from Paecilomyces aerugineus in Pichia pastoris[J]. Journal of Molecular Catalysis. B:Enzymatic, 2011, 69(3):112-119.
[21] LOWRY O H, ROSEBROUGH N J, FARR A L, et al. Protein measurement with the folin phenol reagent[J]. The Journal of Biological Chemistry,1951,193(1):265−275. doi: 10.1016/S0021-9258(19)52451-6
[22] LIU Y, YAN Q J, GUAN L Y, et al. Biochemical characterization of a novel protease-resistant α-galactosidase from Paecilomyces thermophila suitable for raffinose family oligosaccharides degradation[J]. Process Biochemistry,2020,94:370−379. doi: 10.1016/j.procbio.2020.04.013
[23] QIN Z M, LI S F, HUANG X, et al. Improving galactooligosaccharide synthesis efficiency of β-galactosidase Bgal1-3 by reshaping the active site with an intelligent hydrophobic amino acid scanning[J]. Journal of Agriculture and Food Chemistry,2019,67(40):11158−11166. doi: 10.1021/acs.jafc.9b04774
[24] KOLEV P, ROCHA-MENDOZA D, RUIZ-RAMÍREZ S, et al. Screening and characterization of β-galactosidase activity in lactic acid bacteria for the valorization of acid whey[J]. JDS Communications,2021,3(1):1−6.
[25] ZERVA A, LIMNAIOS A, KRITIKOU A S, et al. A novel thermophile β-galactosidase from Thermothielavioides terrestris producing galactooligosaccharides from acid whey[J]. New Biotechnology,2021,63:45−53. doi: 10.1016/j.nbt.2021.03.002
[26] PEPRAH A F, WANG T T, KOSIBA A A, et al. Integration of elastin-like polypeptide fusion system into the expression and purification of Lactobacillus sp. B164 β-galactosidase for lactose hydrolysis[J]. Bioresource Technology,2020,311:123513. doi: 10.1016/j.biortech.2020.123513
[27] LIN Q B, WANG S D, WANG M L, et al. A novel glycoside hydrolase family 42 enzyme with bifunctional β-galactosidase and α-L-arabinopyranosidase activities and its synergistic effects with cognate glycoside hydrolases in plant polysaccharides degradation[J]. International Journal of Biological Macromolecules,2019,140:129−139. doi: 10.1016/j.ijbiomac.2019.08.037
[28] ABURTO C, CASTILLO C, CORNEJO F, et al. β-Galactosidase from Exiguobacterium acetylicum:cloning, expression, purification and characterization[J]. Bioresource Technology,2019,277:211−215. doi: 10.1016/j.biortech.2019.01.005
[29] KATROLIA P, ZHANG M, YAN Q J, et al. Characterisation of a thermostable family 42 β-galactosidase (BgalC) family from Thermotoga maritima showing efficient lactose hydrolysis[J]. Food Chemistry,2011,125(2):614−621. doi: 10.1016/j.foodchem.2010.08.075
[30] SUN J J, YAO C Y, WANG W, et al. Cloning, expression and characterization of a novel cold-adapted β-galactosidase from the deep-sea bacterium Alteromonas sp. ML52[J]. Marine Drugs,2018,16(12):469. doi: 10.3390/md16120469
[31] WANG K, LI G, YU S Q, et al. A novel metagenome-derived β-galactosidase:gene cloning, overexpression, purification and characterization[J]. Applied Microbiology and Biotechnology,2010,88(1):155−165. doi: 10.1007/s00253-010-2744-7
[32] WANG L J, MOU Y Z, GUAN B, et al. Genome sequence of the psychrophilic Cryobacterium sp. LW097 and characterization of its four novel cold-adapted β-galactosidases[J]. International Journal of Biological Macromolecules,2020,163:2068−2083. doi: 10.1016/j.ijbiomac.2020.09.100
[33] CARNEIRO L A B C, LI Y, DUPREE P, et al. Characterization of a β-galactosidase from Bacillus subtilis with transgalactosylation activity[J]. International Journal of Biological Macromolecules,2018,120:279−287. doi: 10.1016/j.ijbiomac.2018.07.116
[34] JUAJUN O, NGUYEN T H, MAISCHBERGER T, et al. Cloning, purification, and characterization of β-galactosidase from Bacillus licheniformis DSM 13[J]. Applied Microbiology and Biotechnology,2011,89(3):645−654. doi: 10.1007/s00253-010-2862-2
-
期刊类型引用(0)
其他类型引用(1)





 下载:
下载:
 下载:
下载:
