Bioinformatics Analysis of Extracellular Keratinase KerQH2 from Rheinheimera sp.QH
-
摘要: 目的:预测海洋菌Rheinheimera sp.QH分泌的角蛋白酶KerQH2的结构特征及与底物的互作位点,为深入研究KerQH2对角蛋白底物的降解机理提供理论依据。方法:应用生物信息学技术对KerQH2的结构特征、作用于角蛋白底物的可能位点及关键氨基酸等进行分析。结果:KerQH2为S8家族丝氨酸蛋白酶,其分子表面无规则卷曲占比较高,α-螺旋与β-折叠占比较低且多位于酶分子内部。KerQH2具有保守的催化三联体(Asp8、His41和Ser197),催化三联体区域表面静电势为中性。KerQH2催化结构域中含有大量保守的极性氨基酸及芳香族氨基酸,与底物结合有关的序列富含Gly和Ala。KerQH2主要切割疏水性氨基酸Val(V)/ILe(I)和极性氨基酸Cys(C)/Gln(Q)之间形成的肽键。结论:角蛋白酶KerQH2的特殊结构有助于其降解结构复杂角蛋白分子,这可能是海洋菌对海洋寡营养环境的一种适应性。
-
关键词:
- 角蛋白酶 /
- KerQH2 /
- 海洋菌 /
- Rheinheimera sp. /
- 生物信息学
Abstract: Objective: To analyze and predict the structural characteristics and the site of interaction with the substrate of keratinase KerQH2 from marine bacteria Rheinheimera sp.QH, and to provide theoretical basis for further study on the degradation mechanism of keratin substrate by KerQH2. Methods: The bioinformatics technology was used to analyze the structural characteristics of keratinase KerQH2 and its possible sites and key amino acids on the keratin substrate. Results: KerQH2 was an S8 family serine protease, and the proportion of random coil on the molecular surface of KerQH2 was high; The proportion of α-helix and β-sheet was low, which were mainly located inside the enzyme molecule. KerQH2 contained a conserved catalytic triad in the order of Asp8, His41, Ser197. The surface electrostatic potential in the catalytic triad area was neutral. The KerQH2 catalytic domain contained a large number of conserved polar amino acids and aromatic amino acids, in addition to the sequences related to substrate binding were rich in Gly and Ala. KerQH2 mainly cut the peptide bond formed between hydrophobic amino acid Val(V)/ILe(I) and polar amino acid Cys(C)/Gln(Q). Conclusion: The special structure of KerQH2 helps to degrade complex keratins, which may be an adaptation of marine bacteria to marine oligotrophic environment.-
Keywords:
- keratinase /
- KerQH2 /
- marine bacteria /
- Rheinheimera sp. /
- bioinformatics
-
角蛋白(keratin)广泛存在于生物体的组织结构中,包括软角蛋白和硬角蛋白两大类[1-3]。其中,纤维化的硬角蛋白在自然界中有多种存在形式,包括动物的毛发、羽毛、指甲、蹄、壳、爪、角和鳞片等。由于角蛋白富含二硫键并且高度交联,使其结构非常稳定,不易被普通蛋白酶水解[4-7]。近年来,随着养殖业的
迅速发展,在畜禽产品加工过程中产生大量的毛发和羽毛等角蛋白废弃物,如不加以有效利用,既浪费资源又污染环境[8]。用传统的物理化学法降解角蛋白不仅存在工艺繁琐、条件剧烈、污染严重等问题,且氨基酸破坏严重,产物不均一,利用率低[9-11]。因此,探究高效、环保的角蛋白降解方法具有重要的应用价值。 角蛋白酶是一种特殊的蛋白酶类,能特异性降解角蛋白,在饲料和食品工业、医疗业、美容业、环境治理等方面具有广阔的应用前景[12-13]。如角蛋白酶可以降解羽毛等角蛋白,将其转化为动物蛋白质饲料;羽毛角蛋白中还含有丰富的谷氨酸、天冬氨酸等鲜味氨基酸,可以用来制作新型食品添加剂应用到食品加工业中;角蛋白酶可用于肉制品嫩化、燕窝脱毛以及蚕丝脱胶;角蛋白酶可改善药物在皮肤角质层中的通透性,提高皮肤外用药物的疗效;另外,角蛋白酶还可以用于头发护理品,脱毛膏的制作等[14-15]。目前市售角蛋白酶主要来源于陆地微生物,如Bacillus sp.,Xanthomonas maltophilia和Stenotrophomonas sp.[16-18]等。有关海洋菌来源的角蛋白酶报道较少,且缺乏系统的研究。海洋菌分泌的蛋白酶具有陆地菌无法比拟的优越性,如对盐、去污剂及有机溶剂等的耐受性比较好,在皮革生产、污水处理等盐离子浓度和有机溶剂浓度较高的工业环境中具有更为实际的应用价值和广阔的应用前景。此外,海洋菌分泌的蛋白酶在较低的温度下仍具有较高的酶活性[19-20],在实际应用中可有效节约成本。因此,探索海洋菌来源的角蛋白酶及其催化机制具有重要的意义。
角蛋白酶KerQH2来源于海洋菌Rheinheimera sp.QH,前期研究发现KerQH2对羽毛角蛋白具有高效降解能力,且对有机试剂和去污剂具有很强的耐受性,具有潜在的工业应用价值。本文利用生物信息学软件对角蛋白酶KerQH2的氨基酸组成特点、结构特征,对不同不可溶角蛋白底物的酶切位点、与底物分子的相互作用及发挥作用的可能关键氨基酸位点进行了预测,以期为深入探究角蛋白酶KerQH2高效降解角蛋白的催化机制提供理论依据。
1. 材料与方法
1.1 材料与仪器
Rheinheimera sp.QH分离自渤海潮间带,将菌株Rheinheimera sp.QH活化后接种于发酵培养基,20 ℃,200 r/min, 培养96 h,8000×g离心10 min,上清即为粗酶液。粗酶液经离子交换层析、SDS-PAGE纯化后得到角蛋白酶KerQH2,对KerQH2进行质谱鉴定,结果显示KerQH2为S8家族丝氨酸蛋白酶。
1.2 实验方法
利用相关生物信息学分析软件及在线工具对KerQH2氨基酸序列进行分析,表1列出分析项目所应用的在线工具及网址。
表 1 生物信息学分析工具Table 1. Bioinformatics analysis tools分析项目 分析工具 序列查询 https://www.ncbi.nlm.nih.gov/Protein 结构查询 http://www.rcsb.org/ 理化性质 ExPASy:ProtParam(https://web.expasy.org/Protparam/) 亲疏水性 ExPASy:ProtSale(https://web.expasy.org/Protscale/) 多序列比对 MEGA6.0(http://www.megasoftware.net/mega) 切割位点预测 Prosper(https://Prosper.erc.monash.edu.au/home.html) 二级结构预测 PredictProtein(https://www.predictProtein.org/) 三级结构预测 SWISS-MODEL(https://swissmodel.expasy.org/) 可视化处理 Discovery Studio 2016软件 分子对接 Discovery Studio 2016软件 表面静电势分析 PyMol 2.5.0 2. 结果与分析
2.1 角蛋白酶KerQH2的理化性质分析
通过ExPASy: ProtParam(https://web.expasy.org/Protparam/)对角蛋白酶KerQH2的氨基酸组成及理化性质进行分析(表2),发现Gly(11.41%)、Ala(11.98%)、Val(7.79%)与Ser(10.27%)含量相对较高,Cys(1.33%)与Trp(1.52%)含量较低,碱性氨基酸(Arg、Lys、His)含量略高于酸性氨基酸(Asp、Glu),等电点7.18,脂肪指数70.2,蛋白酶不稳定系数小于40,稳定性较好。由ProtScale的K-D预测算法对KerQH2的疏水性进行分析,结果显示为亲水性蛋白质。
表 2 角蛋白酶KerQH2的氨基酸组成Table 2. Amino acid composition of keratinase KerQH2氨基酸 占比(%) 氨基酸 占比(%) 氨基酸 占比(%) 氨基酸 占比(%) Ala 11.98 Thr 7.22 Pro 3.8 Arg 2.47 Gly 11.41 Leu 5.32 Tyr 3.8 Phe 2.28 Ser 10.27 Gln 4.37 Asp 3.23 His 2.28 Val 7.79 Lys 3.8 Glu 3.04 Trp 1.52 Asn 7.79 Ile 3.8 Met 2.47 Cys 1.33 2.2 角蛋白酶KerQH2的二级结构分析
通过PredictProtein(https://www.predictProtein.org/)网站对角蛋白酶KerQH2的二级结构进行预测,结果显示KerQH2中α-螺旋和β-折叠总的占比较低(分别为14.1%和23.2%),无规则卷曲的含量为62.7%。其中溶剂可及性分析显示α-螺旋与β-折叠多分布于酶分子的内部,而无规则卷曲多处于酶分子表面。有研究表明,β-折叠和α-螺旋等二级结构形式更有利于维持蛋白质结构的稳定性[21-22],而无规则卷曲则更易于去折叠而使蛋白质构象发生改变[23],推测该结构特征有助于KerQH2对结构复杂角蛋白的降解。此外,前期酶学性质研究发现角蛋白酶KerQH2相较于已报道的角蛋白酶在催化性能方面具有的一些优越性,如对有机溶剂DMSO、氯仿、乙醇的耐受性极好,SDS对酶具有一定的促进作用等,这些特性可能与其结构特征有一定的相关性。
2.3 角蛋白酶KerQH2的底物裂解位点预测
从UniProt数据库中选取角蛋白序列P02451(Larus novaehollandiae)、P02450(Gallus gallus)、Q9BYR7(Homo sapiens)、Q24JX8(Bos taurus)、P02445(Ovis aries)和P02447(Capra hircus),利用蛋白酶特异性预测服务器Prosper (https://Prosper.erc.monash.edu.au/home.html),对角蛋白酶KerQH2的底物裂解位点进行预测[24]。不同类型蛋白酶具有不同的酶切位点,蛋白酶主要包括天冬氨酸蛋白酶、半胱氨酸蛋白酶、金属蛋白酶和丝氨酸蛋白酶等类型,角蛋白酶KerQH2为S8家族丝氨酸蛋白酶,图1和表3显示了角蛋白酶KerQH2对不同来源角蛋白的裂解位点。角蛋白酶KerQH2主要切割疏水性氨基酸Val(V)/ILe(I)和极性氨基酸Cys(C)/Gln(Q)之间形成的肽键。KerQH2在底物角蛋白Q9BYR7、P02445和Q24JX8上的酶切位点更丰富,因此推测KerQH2对人、牛和绵羊来源角蛋白的降解效率更高。
表 3 角蛋白酶KerQH2对角蛋白的裂解位点Table 3. Keratinase KerQH2 cleavage sites on the keratin来源 氨基酸
登录号名称 位点 切割位点 得分 Larus novaehollandiae
(Silver gull)P02451 羽毛角
蛋白33 RVVI 
1.10 56 STAV 
1.04 40 TVVV 
0.97 64 SSSV 
0.94 Gallus gallus
(Chicken)P02450 羽毛角
蛋白41 RVVI 
1.15 76 GVPI 
1.12 34 RVVI 
1.10 Homo sapiens
(Human)Q9BYR7 头发角
蛋白19 ATTI 
1.10 40 PHTV 
1.06 30 RCGV 
1.03 46 LEPI 
1.01 64 CVPT 
0.99 Bos taurus
(Cattle)Q24JX8 头发角
蛋白80 LSGI 
1.23 19 ATTI 
1.10 64 YVPT 
1.10 30 RCGV 
1.03 86 TTFV 
1.00 Ovis aries
(Sheep)P02445 羊毛角
蛋白80 LESI 
1.12 18 ATTI 
1.10 83 INLT 
1.07 29 RCGV 
1.03 4 ACCA 
1.02 39 PHTV 
1.01 Capra hircus
(Goat)P02447 羊毛角
蛋白80 LSGI 
1.23 19 ATTI 
1.10 30 RCGV 
1.03 53 NSPV 
1.02 2.4 多序列比对分析
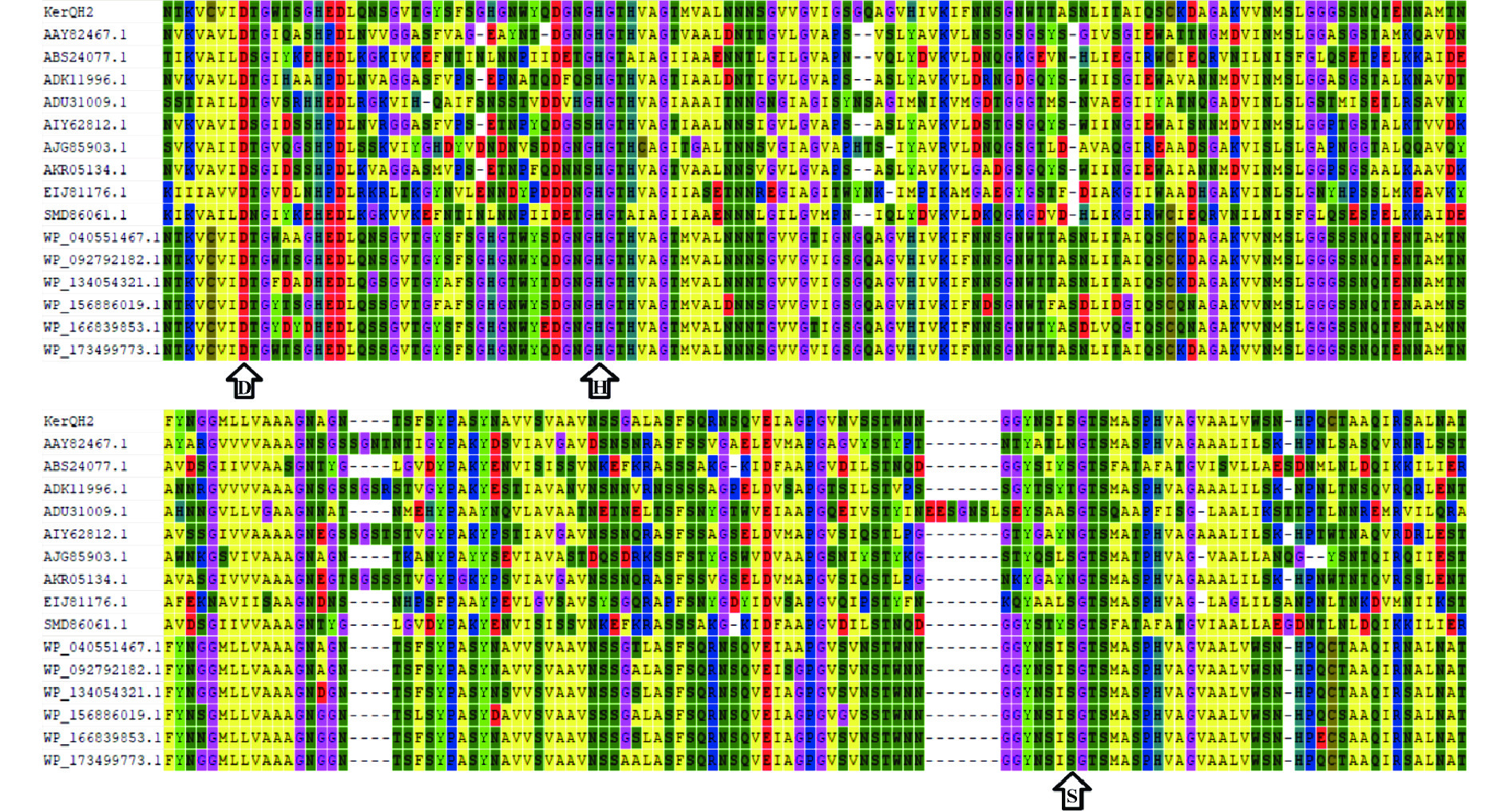
对KerQH2以及从NCBI下载的15个Rheinheimera属和Bacillus属来源的角蛋白酶催化结构域进行多序列比对,结果如图2所示,不同来源角蛋白酶由Asp(D8)、His(H41)和Ser(S197)构成的催化三联体高度保守。其中,Asp(D8)位于基序Xaa-Xaa-Asp-Xbb-Gly中(Xaa为非极性氨基酸,Xbb为极性氨基酸),His(H41)位于基序His-Gly-Thr-Xcc-Xaa-Ala-Gly中(Xcc为His/Ala),Ser(S197)位于基序Gly-Thr-Ser-Met-Ala-Xbb-Xaa中。一些极性氨基酸和芳香族氨基酸也高度保守,如:Asp(17、37)、Thr(43、184、196)、Asn(54、135)、Ser(105、166、183)、Pro(144、177)和Tyr(147、190)等。这些氨基酸可能在底物结合中发挥着重要作用[25-26]。而大量保守的Gly和Ala,则由于其分子侧链较小,因此赋予了角蛋白酶较大的自由度。此外,还存在一些共有序列如:Ser105-Gly109、Ala132-Ala136和Ser142-Ala145等。上述结构特征,可能在KerQH2降解不可溶底物中发挥着重要作用。
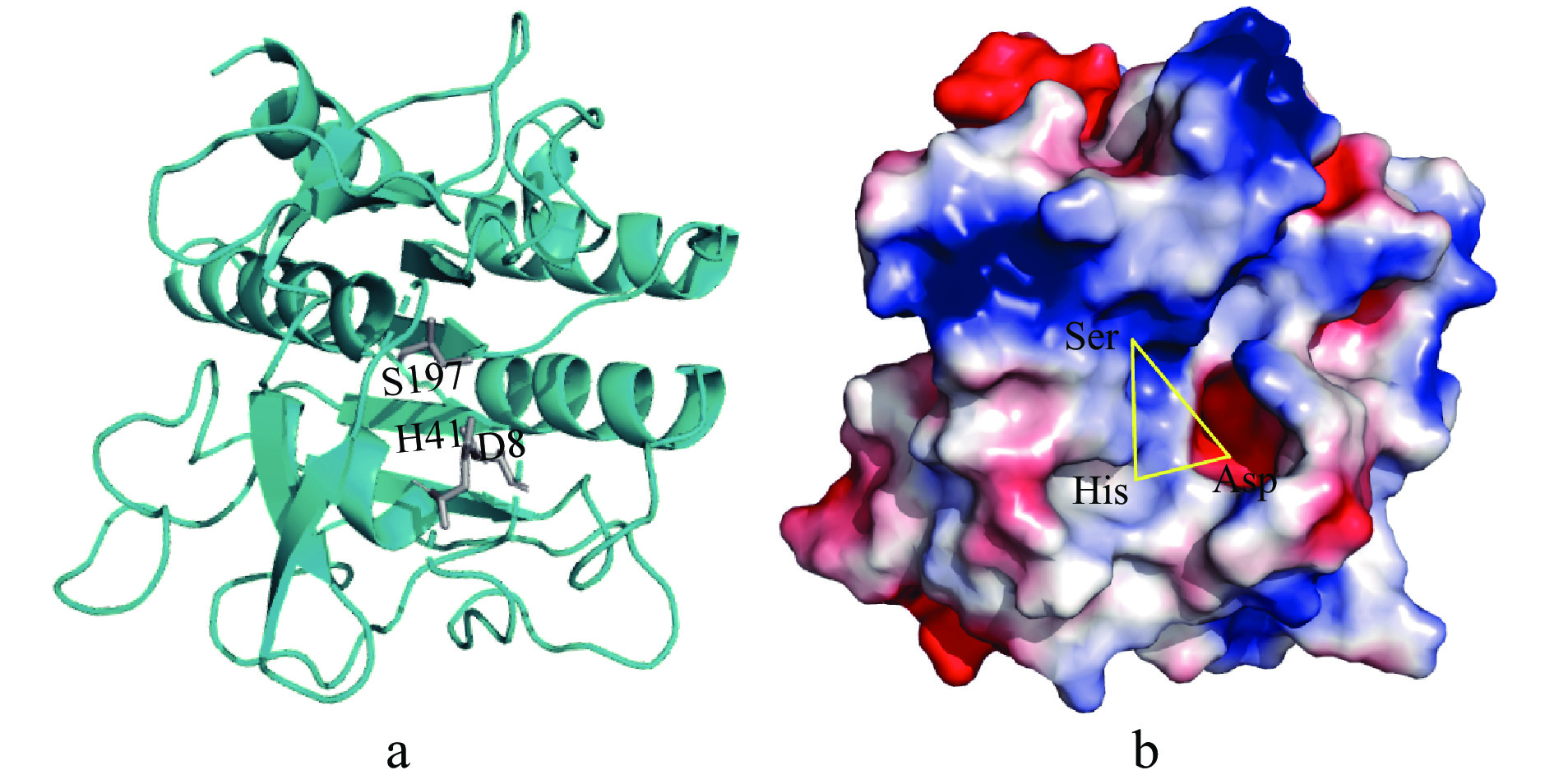
2.5 角蛋白酶KerQH2的同源建模及表面静电势分析
利用在线软件SWISS-MODEL(https://swissmodel.expasy.org/)蛋白质建模数据库,以枯草杆菌蛋白酶(PDB:1SCN.1.N,相似度45.98%)为模板进行同源建模,获得角蛋白酶KerQH2催化结构域的模型。利用蛋白质分析软件Discovery Studio 2016分析其结构,发现KerQH2催化结构域含有5个α-螺旋、8个β-折叠,其中Asp8、His41和Ser197构成催化三联体。通过PyMol 2.5.0 软件对KerQH2进行表面静电势分析,结果如图3所示,角蛋白酶KerQH2中催化三联体所处空穴多为不带电荷区域,这与文献报道的来自Pyrococcus furiosus(PMDB ID: PM0075943)的丝氨酸蛋白酶有所不同,同时研究中还提到蛋白酶的功能多样性及环境适应性与其表面静电势分布有关[27],因此推测角蛋白酶KerQH2催化三联体区域的表面静电势分布可能与其对不可溶角蛋白底物的特异性吸附、降解有关。
2.6 角蛋白酶KerQH2与底物Q9BYR7的分子对接
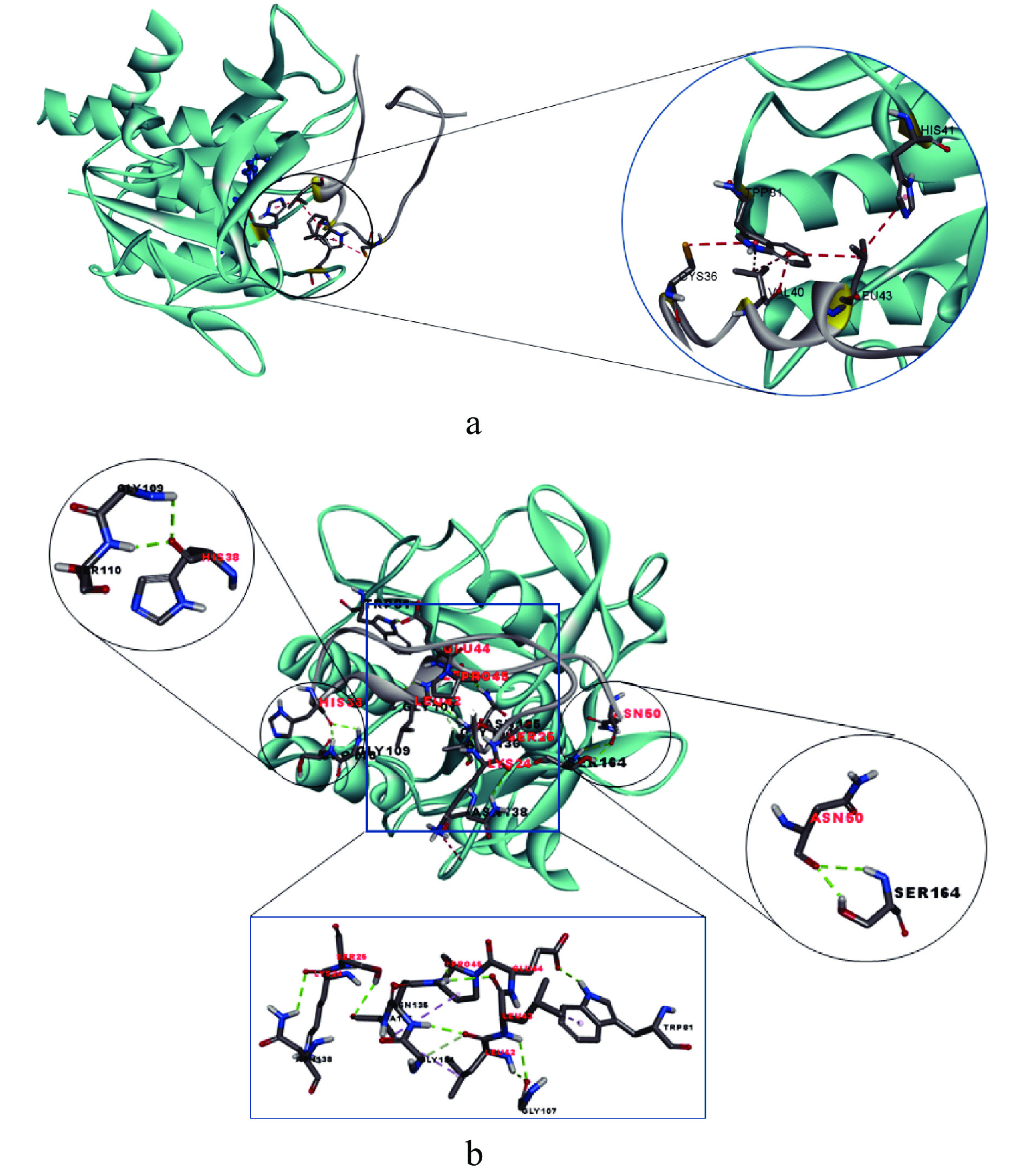
以枯草杆菌蛋白酶为模板进行同源建模,获得角蛋白酶KerQH2催化结构域的模型,以PDB:6u9w.1.A为模板进行同源建模,获得底物Q9BYR7的结构模型。通过Discovery studio分别生成KerQH2及底物Q9BYR7的拉氏图,以评价模型的合理性。如图4c中允许区(蓝色区域)和最大允许区(紫色区域)内的氨基酸为合理氨基酸,不允许区域(空白区域)的氨基酸为不合理氨基酸。从拉氏图可以看出,超过90%的氨基酸残基都落在允许区域和最大允许区域内,KerQH2与Q9BYR7的三维结构模型是可信的,可以用于进一步研究。
将角蛋白酶KerQH2的催化结构域和底物角蛋白Q9BYR7同时置于一个CELL晶格中,在 DOCK PROTEINS 模块中进行对接,欧拉角度设置为 6°。蛋白对接结果显示(图5),KerQH2与Q9BYR7的Zdock Score为12.14,E-Rdock为-24.7547,结合较好。分析对接结果,KerQH2有32个氨基酸残基在理论上能够与Q9BYR7的17个氨基酸残基发生相互作用,维持结合稳定性。KerQH2中互作氨基酸分别为:His41、Phe75、Gly79、Trp81、Thr82、Ser105-Ser111、Gly134-Thr139、Ser142、Tyr143、Val156、Asn157、Leu162-Phe165、Gln167、Val179、Asn180、Ile193、Ser194、Gly195和Ser197。Q9BYR7中互作氨基酸分别为:Lys24、Ser25、Arg27、Cys36、His38-Cys48、Asn50和Cys51。KerQH2与Q9BYR7的氨基酸残基形成三组分子间氢键,其中,Ser164:Asn50和Gly109-Ser110:His38两组氢键单独存在,位于相互作用面的两侧,使酶催化结构域的构象发生了轻微改变,“凹”字型的凹陷处向外撑开,暴露出酶的催化三联体(Asp8、His41和Ser197)。而Gly134:Leu42、Asn135:(Leu42、Leu43、Pro45)、Ala136:Ser25、Asn138:Lys24、Trp81:Glu44和Gly107:(Leu42、Leu43)这六个氢键构成一组氢键,位于相互作用面中心,使酶-底物复合物结构更加稳定。KerQH2与Q9BYR7形成His41:Leu43和Trp81:(Cys36、Val40、Leu43)两处Pi键,维持着酶-底物复合物的稳定性。
此外,结合KerQH2与底物Q9BYR7的分子对接结果进行分析,发现共有序列Ser105-Gly109、Ala132-Ala136和Ser142-Ala145参与了底物的结合,同时,共有序列中富含的Gly和Ala不仅增加了结合位点的疏水性,而且减少了侧链空间位阻,更有利于角蛋白酶与底物的结合,与文献中的报道一致[28]。
2.7 系统发育分析
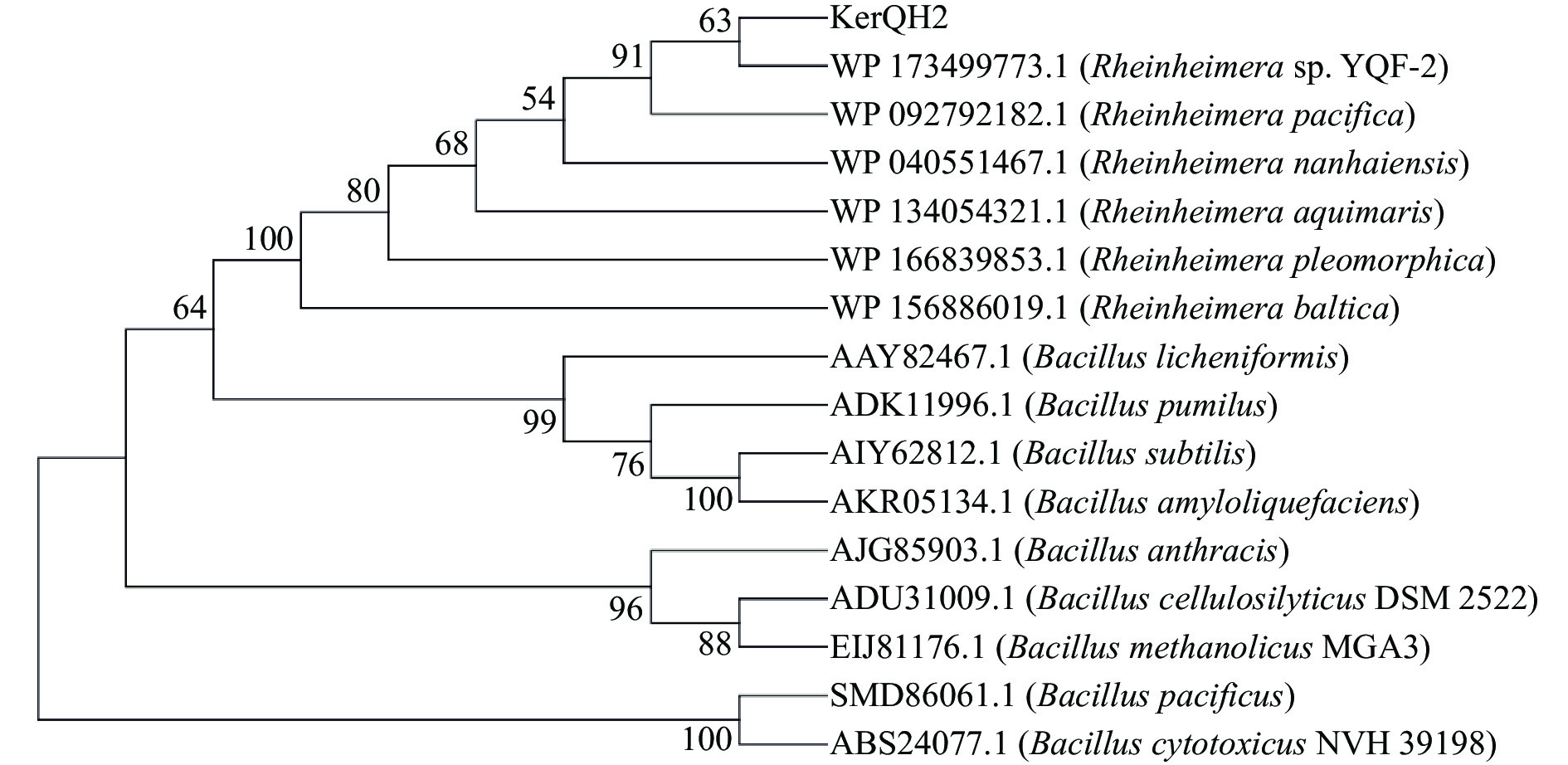
将KerQH2的氨基酸序列以及从NCBI下载的15个Rheinheimera属和Bacillus属来源的角蛋白酶氨基酸序列,运用MEGA 6.0软件中的邻接法(neighbor-joining,NJ)进行分子系统发育分析,采用默认参数,得到分子进化树(图6),可以较为精确地确定其进化地位[29-30]。角蛋白酶KerQH2和Rheinheimera属来源的蛋白WP173499773.1分支聚在一起,具有较近的亲缘关系, 且不同种属来源的角蛋白酶具有一定的保守性。
3. 结论
对海洋菌Rheinheimera sp.QH分泌的角蛋白酶KerQH2的结构特征、角蛋白底物的作用位点及关键氨基酸位点的预测结果表明,角蛋白酶KerQH2的特殊结构有助于其降解结构复杂的角蛋白分子,这可能是海洋菌对海洋寡营养环境的一种适应性[31-32]。本研究结果为进一步深入研究KerQH2的降解机制提供了一定的理论依据,为角蛋白酶KerQH2的推广和应用奠定了一定的理论基础。后期本团队将针对这些位点,构建一系列突变体,进一步验证这些氨基酸对角蛋白酶KerQH2活性的影响。
-
表 1 生物信息学分析工具
Table 1 Bioinformatics analysis tools
分析项目 分析工具 序列查询 https://www.ncbi.nlm.nih.gov/Protein 结构查询 http://www.rcsb.org/ 理化性质 ExPASy:ProtParam(https://web.expasy.org/Protparam/) 亲疏水性 ExPASy:ProtSale(https://web.expasy.org/Protscale/) 多序列比对 MEGA6.0(http://www.megasoftware.net/mega) 切割位点预测 Prosper(https://Prosper.erc.monash.edu.au/home.html) 二级结构预测 PredictProtein(https://www.predictProtein.org/) 三级结构预测 SWISS-MODEL(https://swissmodel.expasy.org/) 可视化处理 Discovery Studio 2016软件 分子对接 Discovery Studio 2016软件 表面静电势分析 PyMol 2.5.0 表 2 角蛋白酶KerQH2的氨基酸组成
Table 2 Amino acid composition of keratinase KerQH2
氨基酸 占比(%) 氨基酸 占比(%) 氨基酸 占比(%) 氨基酸 占比(%) Ala 11.98 Thr 7.22 Pro 3.8 Arg 2.47 Gly 11.41 Leu 5.32 Tyr 3.8 Phe 2.28 Ser 10.27 Gln 4.37 Asp 3.23 His 2.28 Val 7.79 Lys 3.8 Glu 3.04 Trp 1.52 Asn 7.79 Ile 3.8 Met 2.47 Cys 1.33 表 3 角蛋白酶KerQH2对角蛋白的裂解位点
Table 3 Keratinase KerQH2 cleavage sites on the keratin
来源 氨基酸
登录号名称 位点 切割位点 得分 Larus novaehollandiae
(Silver gull)P02451 羽毛角
蛋白33 RVVI 
1.10 56 STAV 
1.04 40 TVVV 
0.97 64 SSSV 
0.94 Gallus gallus
(Chicken)P02450 羽毛角
蛋白41 RVVI 
1.15 76 GVPI 
1.12 34 RVVI 
1.10 Homo sapiens
(Human)Q9BYR7 头发角
蛋白19 ATTI 
1.10 40 PHTV 
1.06 30 RCGV 
1.03 46 LEPI 
1.01 64 CVPT 
0.99 Bos taurus
(Cattle)Q24JX8 头发角
蛋白80 LSGI 
1.23 19 ATTI 
1.10 64 YVPT 
1.10 30 RCGV 
1.03 86 TTFV 
1.00 Ovis aries
(Sheep)P02445 羊毛角
蛋白80 LESI 
1.12 18 ATTI 
1.10 83 INLT 
1.07 29 RCGV 
1.03 4 ACCA 
1.02 39 PHTV 
1.01 Capra hircus
(Goat)P02447 羊毛角
蛋白80 LSGI 
1.23 19 ATTI 
1.10 30 RCGV 
1.03 53 NSPV 
1.02 -
[1] YU J L, YU D W, CHECKLA D M, et al. Human hair keratins[J]. Journal of Investigative Dermatology,1993,101(1):56S−59S. doi: 10.1016/0022-202X(93)90501-8
[2] EHRLICH F, LACHNER J, HERMANN M, et al. Convergent evolution of cysteine-rich keratins in hard skin appendages of terrestrial vertebrates[J]. Molecular Biology and Evolution,2020,37(4):982−993. doi: 10.1093/molbev/msz279
[3] LI Q X. Progress in microbial degradation of feather waste[J]. Frontiers in Microbiology,2019,10:2717. doi: 10.3389/fmicb.2019.02717
[4] VIDMAR B, VODOVNIK M. Microbial keratinases: Enzymes with promising biotechnological applications[J]. Food Technology & Biotechnology,2018,56(3):312−328.
[5] GHAFFAR I, IMTIAZ A, HUSSAIN A, et al. Microbial production and industrial applications of keratinases: An overview[J]. International Microbiology,2018,21(4):163−174. doi: 10.1007/s10123-018-0022-1
[6] VERMA A, SINGH H, ANWAR S, et al. Microbial keratinases: Industrial enzymes with waste management potential[J]. Critical Reviews in Biotechnology,2017,37(4):476−491. doi: 10.1080/07388551.2016.1185388
[7] ABDEL-NABY M A, EL-REFAI H A, IBRAHIM M H A. Structural characterization, catalytic, kinetic and thermodynamic properties of keratinase from Bacillus pumilus FH9[J]. International Journal of Biological Macromolecules, 2017, 105(Pt 1): 973-980.
[8] CHOIŃSKA-PULIT A, ŁABA W, RODZIEWICZ A. Enhancement of pig bristles waste bioconversion by inoculum of keratinolytic bacteria during composting[J]. Waste Management,2019,84:269−276. doi: 10.1016/j.wasman.2018.11.052
[9] LI Z W, LIANG S, KE Y, et al. The feather degradation mechanisms of a new Streptomyces sp. isolate SCUT-3[J]. Communications Biology,2020,3(1):191−203. doi: 10.1038/s42003-020-0918-0
[10] SHARMA I, KANGO N. Production and characterization of keratinase by Ochrobactrum intermedium for feather keratin utilization[J]. International Journal of Biological Macromolecules,2021,166:1046−1056. doi: 10.1016/j.ijbiomac.2020.10.260
[11] SHAVANDI A, SILVA T H, BEKHIT A A, et al. Keratin: Dissolution, extraction and biomedical application[J]. Biomaterials Science,2017,5(9):1699−1735. doi: 10.1039/C7BM00411G
[12] NNOLIM N E, UDENIGWE C C, OKOH A I, et al. Microbial keratinase: Next generation green catalyst and prospective applications[J]. Frontiers in Microbiology,2020,11:580164. doi: 10.3389/fmicb.2020.580164
[13] UTOMO B, DJALALROSYIDI L E R, PUSPANINGSIH N N T, et al. Cleaning method by keratinase enzyme for improving quality edible bird nest[J]. Journal of Life Science and Biomedicine,2014,4(5):416−420.
[14] ODETALLAH N H, WANG J J, GARLICH J D, et al. Keratinase in starter diets improves growth of broiler chicks[J]. Poultry Science,2003,82(4):664−670. doi: 10.1093/ps/82.4.664
[15] BRANDELLI A, DAROIT D J, RIFFEL A. Biochemical features of microbial keratinases and their production and applications[J]. Applied Microbiology and Biotechnology,2010,85(6):1735−1750. doi: 10.1007/s00253-009-2398-5
[16] VASILEVA-TONKOVA E, GOUSTEROVA A, NESHEV G. Ecologically safe method for improved feather wastes biodegradation[J]. International Biodeterioration & Biodegradation,2009,63(8):1008−1012.
[17] 李雷, 冯红. 两株芽孢杆菌降解羽毛比较及抗氧化肽分离[J]. 应用与环境生物学报,2018,24(1):172−176. [LI L, FENG H. Comparison of feather degradation by two Bacillus strains and separation of antioxidant peptides[J]. Chinese Journal of Applied and Environmental Biology,2018,24(1):172−176. [18] LANGE L, HUANG Y, BUSK P K. Microbial decomposition of keratin in nature-a new hypothesis of industrial relevance[J]. Applied Microbiology and Biotechnology,2016,100(5):2083−2096. doi: 10.1007/s00253-015-7262-1
[19] ZHANG C, KIM S K. Research and application of marine microbial enzymes: Status and prospects[J]. Marine Drugs,2010,8(6):1920−1934. doi: 10.3390/md8061920
[20] BONUGLI-SANTOS R C, DOS SANTOS VASCONCELOS M R, PASSARINI M R, et al. Marine-derived fungi: Diversity of enzymes and biotechnological applications[J]. Frontiers in Microbiology,2015,6:269−283.
[21] JAMIR K, SESHAGIRIRAO K. Fluorescence quenching, structural and unfolding studies of a purified cysteine protease, ZCPG from Zingiber montanum rhizome[J]. International Journal of Biological Macromolecules,2018,106:277−283. doi: 10.1016/j.ijbiomac.2017.08.019
[22] 武翠玲, 宋英达, 高慧芳, 等. Salinivibrio sp.YH4胞外丝氨酸蛋白酶EYHS耐盐性及生物信息学分析[J]. 盐湖研究,2021,29(1):105−110. [WU C L, SONG Y D, GAO H F, et al. Salt-tolerance and bioinformatics analysis on the serine protease EYHS secreted by Salinivibrio sp. YH4[J]. Journal of Salt Lake Research,2021,29(1):105−110. [23] AHMAD S, KUMAR V, RAMANAND K B, et al. Probing protein stability and proteolytic resistance by loop scanning: A comprehensive mutational analysis[J]. Protein Science,2012,21(3):433−446. doi: 10.1002/pro.2029
[24] EMAMEH R Z, KAZOKAITĖ J, YAKHCHALI B. Bioinformatics analysis of extracellular subtilisin E from Bacillus subtilis[J]. Journal of Biomolecular Structure and Dynamics,2021,4:1−8.
[25] 蒋少龙, 蔡俊. 角蛋白酶及其应用研究进展[J]. 食品工业科技,2019,40(6):348−354, 360. [JIANG S L, CAI J. Research progress of keratinase and its application[J]. Science and Technology of Food Industry,2019,40(6):348−354, 360. [26] HE H L, GUO J, CHEN X L, et al. Structural and functional characterization of mature forms of metalloprotease E495 from Arctic sea-ice bacterium Pseudoalteromonas sp. SM495[J]. PLoS One,2012,7(4):e35442. doi: 10.1371/journal.pone.0035442
[27] LASKAR A, RODGER E J, CHATTERJEE A, et al. Modeling and structural analysis of evolutionarily diverse S8 family serine proteases[J]. Bioinformation,2011,7(5):239−245. doi: 10.6026/97320630007239
[28] FANG Z, ZHANG J, LIU B H, et al. Insight into the substrate specificity of keratinase KerSMD from Stenotrophomonas maltophilia by site-directed mutagenesis studies in the S1 pocket[J]. RSC Advances,2015,5:74953−74960. doi: 10.1039/C5RA12598G
[29] 李宁, 王柏柯, 杨生保, 等. 21种植物八氢番茄红素合成酶的生物信息学分析[J]. 新疆农业科学,2015,52(12):2157−2165. [LI N, WANG B K, YANG S B, et al. Bioinformatics analysis of PSY in 21 plant species[J]. Xinjiang Agricultural Sciences,2015,52(12):2157−2165. [30] 富玉竹, 李欣, 李晔, 等. 16种微生物蛋白酶的生物信息学分析[J]. 江苏农业科学,2020,48(4):65−72. [FU Y Z, LI X, LI Y, et al. Bioinformatics analysis of sixteen microbial proteases[J]. Jiangsu Agricultural Sciences,2020,48(4):65−72. [31] VALENCIA R, GONZÁLEZ V, UNDABARRENA A, et al. An integrative bioinformatic analysis for keratinase detection in marine-derived Streptomyces[J]. Marine Drugs,2021,19(6):286. doi: 10.3390/md19060286
[32] NGUYEN T T H, MYROLD D D, MUELLER R S. Distributions of extracellular peptidases across prokaryotic genomes reflect phylogeny and habitat[J]. Frontiers in Microbiology,2019,10:413. doi: 10.3389/fmicb.2019.00413
-
期刊类型引用(3)
1. 胡智恺,索一平,李爽,王雨婷,刘薇,史锦硕,杨霞,姜洁. 婴儿配方奶粉中克罗诺杆菌的快速检测. 中国酿造. 2023(08): 253-259 .  百度学术
百度学术
2. 丁伯乐,蔡为荣,闻志莹,岳丹伟,朱樱,李晶晶. 山药低聚糖制备分离及对五种益生菌的增殖作用. 食品与发酵工业. 2020(24): 74-79 .  百度学术
百度学术
3. 李婧,张柏林. 低聚葡萄糖对益生菌的增殖影响. 宁夏农林科技. 2019(12): 72-74+84 .  百度学术
百度学术
其他类型引用(3)





 下载:
下载:


 下载:
下载:



