Inhibitory Activity and Transcriptional Mechanism of PostbioYDFF against Pseudomonas aeruginosa
-
摘要: 在本研究中,通过分析后生元PostbioYDFF对铜绿假单胞菌(P. aeruginosa)生物膜的影响以及P. aeruginosa超微结构和转录组的变化,研究了其对P. aeruginosa的抗菌活性及其作用机制,同时,为了验证转录组数据,进行了qPCR验证。结果表明,后生元PostbioYDFF对铜绿假单胞菌具有显著的抑菌活性,形成的抑菌圈直径为28.9±0.3 mm,MIC值为100 μL/mL。此外,与对照组相比,后生元PostbioYDFF可以极显著抑制生物膜的形成,抑制率达到64.96%(P<0.01),可以显著清除已形成的生物膜,清除率达到30.66%(P<0.05)。还观察到后生元PostbioYDFF破坏了细菌结构并减弱了细菌运动性(游泳运动、集群运动和抽搐运动)。通过RNA-seq结合qPCR验证,与对照组相比,后生元PostbioYDFF处理后共发现503个显著差异的基因,其中237个显著上调的基因主要富集在核糖体、RNA降解、氧化磷酸化等通路,266个显著下调的基因主要富集在能量代谢、脂质代谢、双组分系统等通路。本研究为后生元PostbioYDFF防控铜绿假单胞菌提供了重要的理论依据,并指出了在食品保鲜中的潜在应用。Abstract: In the present study, the antibacterial activity of postbiotics PostbioYDFF against Pseudomonas aeruginosa (P. aeruginosa) and its mechanism of action were examined by analyzing its effects on biofilm formation and changes in the ultrastructure and transcriptome. Meanwhile, to validate transcriptome data, qPCR was conducted. The results showed that PostbioYDFF exhibited significant bacteriostatic activity against P. aeruginosa with a diameter of inhibition zone growth of 28.9±0.3 mm and the MIC value of 100 μL/mL. In addition, compared with the control group, PostbioYDFF was able to significantly inhibit biofilm formation by 64.96% (P<0.01) and disrupted established biofilms by 30.66% (P<0.05). It was also observed that PostbioYDFF disrupted the bacterial structure and attenuated bacterial motility (swimming, swarming and twitching). Through RNA-seq analysis combined with qPCR validation, a total of 503 significantly different genes were found after PostbioYDFF treatment as compared with the control group, including 237 significantly up-regulated genes which were mainly enriched in ribosomal, RNA degradation, oxidative phosphorylation and other pathways, and 266 significantly down-regulated genes which were mainly enriched in the energy metabolism, lipid metabolism, and two-component system and other pathways. This study provides an important theoretical basis for the prevention and control of P. aeruginosa by PostbioYDFF, and points out a potential application in food preservation.
-
Keywords:
- Pseudomonas aeruginosa /
- postbiotics /
- transcriptome /
- bacteriostasis /
- antiseptic
-
铜绿假单胞菌(Pseudomonas aeruginosa,P. aeruginosa)是一种需氧、能动、轻微弯曲的革兰氏阴性菌,广泛存在于各种环境中,包括空气、水、土壤和动植物组织。该菌可以造成多种感染,主要包括尿路感染、肺炎、胃肠道感染、皮炎、皮肤感染、菌血症、骨和关节感染以及其他感染,特别是容易造成严重烧伤患者和免疫功能低下者(即患有癌症或艾滋病)感染[1−2]。近些年来,由于过度使用抗生素而产生的抗生素耐药菌株的出现增加了免疫受损患者的发病率和死亡率,特别是那些对碳青霉烯类抗生素产生耐药性的铜绿假单胞菌成为当前医疗领域亟待解决的问题,为了有效应对这类细菌引起的机会性感染,迫切需要投入研究和开发新型抗菌剂[3]。另外,铜绿假单胞菌是食品工业中传染性很强的病原体之一,可存在于水、乳制品、肉类和植物源性食品中,该菌具有蛋白质水解、脂解和糖解的能力,不仅可以使蛋白质丰富的食品如肉类等发生变质,还可能通过增加食品中亚硝酸盐的含量导致人体中毒[4−5]。目前由铜绿假单胞菌污染引起的食物中毒及患者细菌感染等事件不断发生,食品和医疗安全问题日益凸显[6]。因此,寻找一种天然、安全、高效的食品级抗菌原料来替代或减少抗生素的使用,具有重大意义且时间紧迫。
乳酸菌(LAB)广泛应用于食品发酵,可分泌抑制病原菌生长的抗菌物质[7],Seval等[8]研究发现乳酸菌产生的细菌素对多种革兰氏阳性和革兰氏阴性菌具有拮抗作用,可以有效抑制牛奶、鲜肉和奶酪中的腐败菌,从而提高产品质量、延长保质期。这些由乳酸菌分泌或细菌裂解后释放的可能对宿主产生积极影响的代谢物或者菌体称为后生元[9]。后生元被定义为对宿主健康有益的无生命微生物和/或其成分的制剂[10],含有多种活性成分,包括磷壁酸、细胞外多醣等菌体成分,以及维生素、脂质、有机酸、短链脂肪酸、细菌素和氨基酸等活性代谢产物[11]。后生元具有免疫调节、抗菌、抗氧化、抗糖尿病、抗肿瘤和减少食物过敏等作用[12−13]。据研究报道,后生元的主要抑菌成分有细菌素、有机酸、抗菌肽等,对金黄色葡萄球菌、大肠杆菌、鼠伤寒沙门氏菌、铜绿假单胞菌等常见的致病菌具有较好的抑菌效果[14]。Lv等[15]研究表明细菌素plantaricin DL3具有较高的热稳定性(15 min,121 ℃)和pH稳定性(pH2.5~5.5),通过破坏铜绿假单胞菌细胞膜的完整性,导致细胞内蛋白质泄露,诱导细菌死亡。Da-hye等[16]研究发现乳酸片球菌(Pediococcus acidilactici)产生的细菌素(HW01)可有效抑制铜绿假单胞菌的生物膜形成并抑制毒力因子(如脓青素、蛋白酶和鼠李糖脂)的产生。Martins等[17]研究表明,四种乳酸菌CFSM对两种测试的铜绿假单胞菌菌株(9027™和27853™)均具有抗菌活性,在CFSM存在下铜绿假单胞菌形成的生物膜显著减少,生物膜抑制率最高可达到80%。Rezaei等[18]将铜绿假单胞菌用嗜酸乳杆菌的CFS处理24 h后,铜绿假单胞菌的生物膜去除率约为41%。尽管目前对后生元的抑菌作用已有广泛研究,但这些研究大多仅停留在表征其抑菌效果上,并未深入探讨其背后的抑菌机制。
本研究中,本团队成功开发了一种新型后生元原料,并命名为PostbioYDFF。本研究旨在通过综合分析PostbioYDFF对铜绿假单胞菌的抑菌活性、细胞形态变化、生物膜形成与清除能力、细菌运动性的影响,以及通过转录组学技术深入分析铜绿假单胞菌在后生元作用下的基因表达变化,以期系统阐明PostbioYDFF的抑菌机制。这些研究不仅为后生元在防控铜绿假单胞菌方面的应用提供了坚实的理论基础,而且通过转录组学分析揭示的抑菌机制,为后生元在更广泛抗菌领域的潜在应用开辟了新的视野。
1. 材料与方法
1.1 材料与仪器
铜绿假单胞菌(Pseudomonas aeruginosa,Pa)BNCC336458 河南省工业微生物菌种工程技术研究中心;发酵粘液乳杆菌Postbio-Q7、副干酪乳酪杆菌Postbio-P6、乳酸乳球菌乳亚种Postbio-F3 青岛农业大学益生菌与后生元基础研究与开发应用创新实验室乳酸菌菌种库;后生元PostbioYDFF 青岛元达生物科技有限公司;NB营养肉汤培养基、NB琼脂培养基 青岛海博生物技术有限公司;PBS缓冲液、结晶紫 北京索莱宝科技有限公司;氯化钠 国药集团化学试剂有限公司;乙酸 天津市富宇精细化工有限公司;RNAprep Pure Cell/Bacteria Kit 天根生化科技有限公司;USER酶、M-MulV反转录酶 美国NEB公司; Agencourt AMPure XP Kit 美国贝克曼库尔特有限公司;DNA聚合酶、dTTP、dUTP 美国Sigma Aldrich公司。
SWCJ-2FD超净工作台 上海沪净医疗器械有限公司;SCIENTZ-WSQ全自动生长曲线分析仪 宁波新芝生物科技股份有限公司;CLC111E CO恒温培养箱 MMM Medcenter Einrichtungen GmbH公司;Icount 11全自动菌落分析仪 杭州迅数科技有限公司;TU-1810分光光度计 普析通用公司;Cen trifuge 5810R高速离心机 艾本德公司;JSM-7500F扫描电子显微镜 株式会社日立制作所;E-Gel™ Power Snap2000恒琼脂糖凝胶电泳仪、Qubit2.0荧光计、Stratagene™ Mx3005PqPCR仪 赛默飞世尔科技有限公司;Q2000CPCR仪 杭州朗基科学仪器有限公司;Agilent 2100Agilent生化分析仪 安捷伦科技有限公司;NovaSeq 6000Illumina NovaSeq测序仪 美国Illumina公司。
1.2 实验方法
1.2.1 铜绿假单胞菌菌悬液的制备
取100 μL铜绿假单胞菌的菌液接种到10 mL NB营养肉汤培养基中,于37 ℃培养箱中培养18 h,用NaCl溶液进行梯度稀释,稀释完成后,取不同梯度的100 µL菌液置于PCA琼脂平板中进行涂布,涂布完成后将平板放入37 ℃培养箱中培养24 h。然后进行菌落计数,计算得到菌液浓度,选取菌液浓度为106~107 CFU/mL的梯度用于后续实验,所得到的即为菌悬液,置于4 ℃储藏,不得超过一周。以下实验没有特殊说明均采用此浓度。
1.2.2 后生元的制备
将发酵粘液乳杆菌Postbio-Q7、副干酪乳酪杆菌Postbio-P6和乳酸乳球菌乳亚种Postbio-F3,经37 ℃混合发酵48 h,然后97 ℃灭活2 h,得到后生元PostbioYDFF。将后生元PostbioYDFF在5000 r/min下离心10 min,以除去不溶性杂质。后生元PostbioYDF使用蒸馏水稀释到所需浓度。
1.2.3 牛津杯法检测后生元PostbioYDFF的抑菌作用
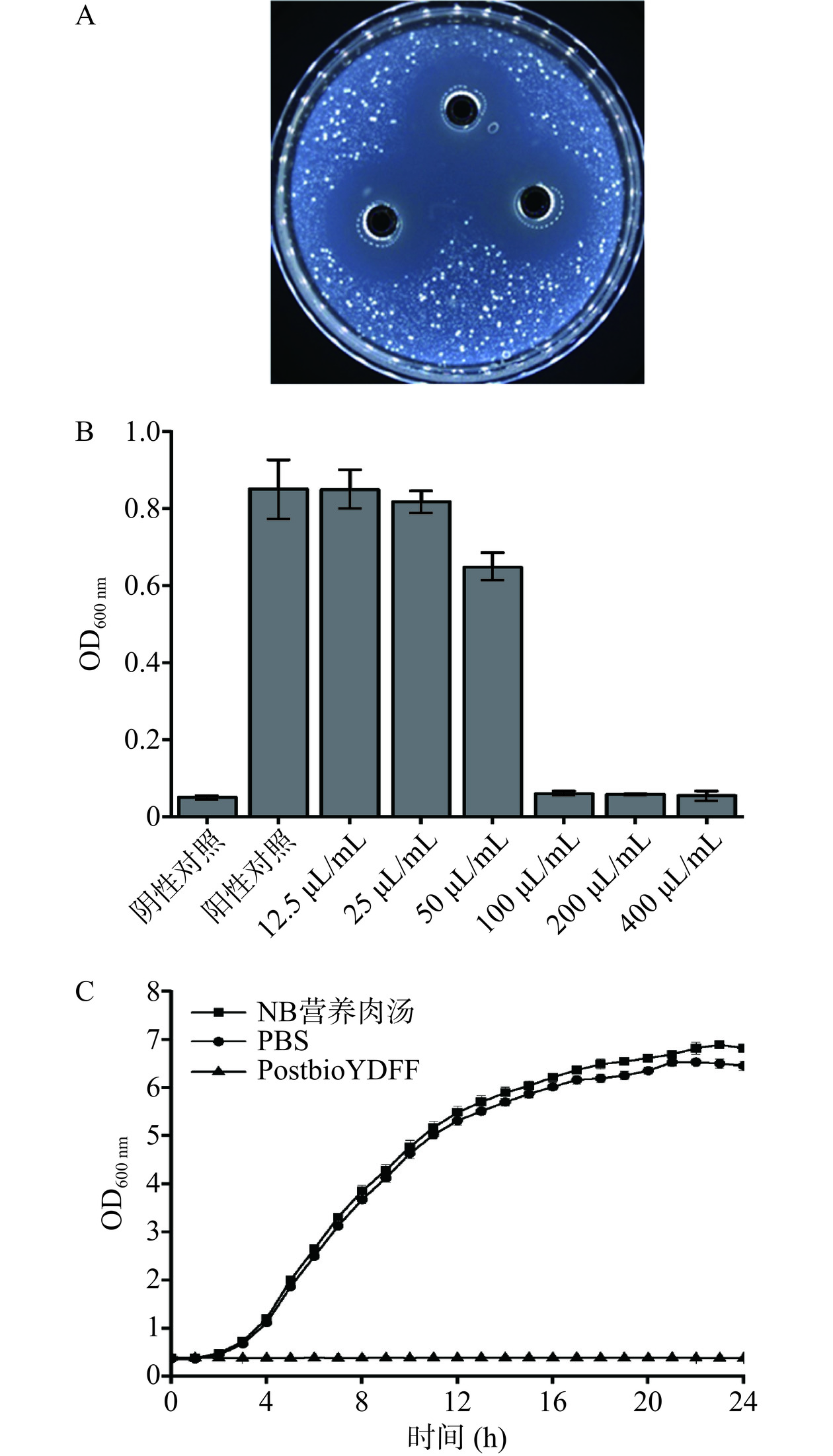
采用琼脂扩散法验证后生元PostbioYDFF对铜绿假单胞菌的抑菌活性。取铜绿假单胞菌菌悬液,按0.7%的比例接种到还未凝固的营养琼脂培养基中,充分混匀后,倾注到放置牛津杯的培养皿中,待培养基彻底凝固后,拿掉牛津杯,每孔加入150 μL后生元PostbioYDFF,每组三个平行,放入−4 ℃冰箱扩散4 h后,放入37 ℃培养箱中培养12 h,用全自动菌落计数器的抑菌圈测量功能进行抑菌圈直径测量。
1.2.4 最小抑菌浓度(MIC)的测定
采用倍比稀释法测定后生元PostbioYDFF对铜绿假单胞菌的MIC [19]。将100 μL后生元PostbioYDFF加入到96孔板中,并依次用100 μL NB培养基进行连续二倍稀释,使后生元PostbioYDFF的终浓度分别为400、200、100、50、25、12.5 μL/mL。随后将100 μL菌悬液(生理盐水稀释到OD600 nm=0.1)加入到稀释好的96孔板中。96孔板中包含阳性对照(100 μL NB培养基与100 μL菌悬液混合液)和阴性对照(培养基)。同时设置含不同浓度后生元PostbioYDFF的培养基为空白对照。将96孔板于37 ℃中孵育24 h后,使用酶标仪测定OD600 nm处的吸光度值,减去空白对照组的吸光度值得到细菌生长量。与阳性对照相比吸光度值出现显著变化的最小浓度为MIC。
1.2.5 生长曲线的测定
参考Ye等[20]的方法,将菌悬液用生理盐水稀释到OD600 nm=0.1~0.2,然后取100 μL菌悬液加入到48孔板中,再加入100 μL的后生元PostbioYDFF,混匀,对照组加入等量的PBS、NB培养基,最后每个孔各加入800 μL NB培养基,混匀,组成1 mL的培养体系。每组三个平行。在800 r/min 37 ℃下振荡培养24 h,测定时间间隔设置为1 h,使用微生物生长分析仪测定OD600 nm值,并绘制成图。
1.2.6 后生元PostbioYDFF对铜绿假单胞菌生物膜形成的影响
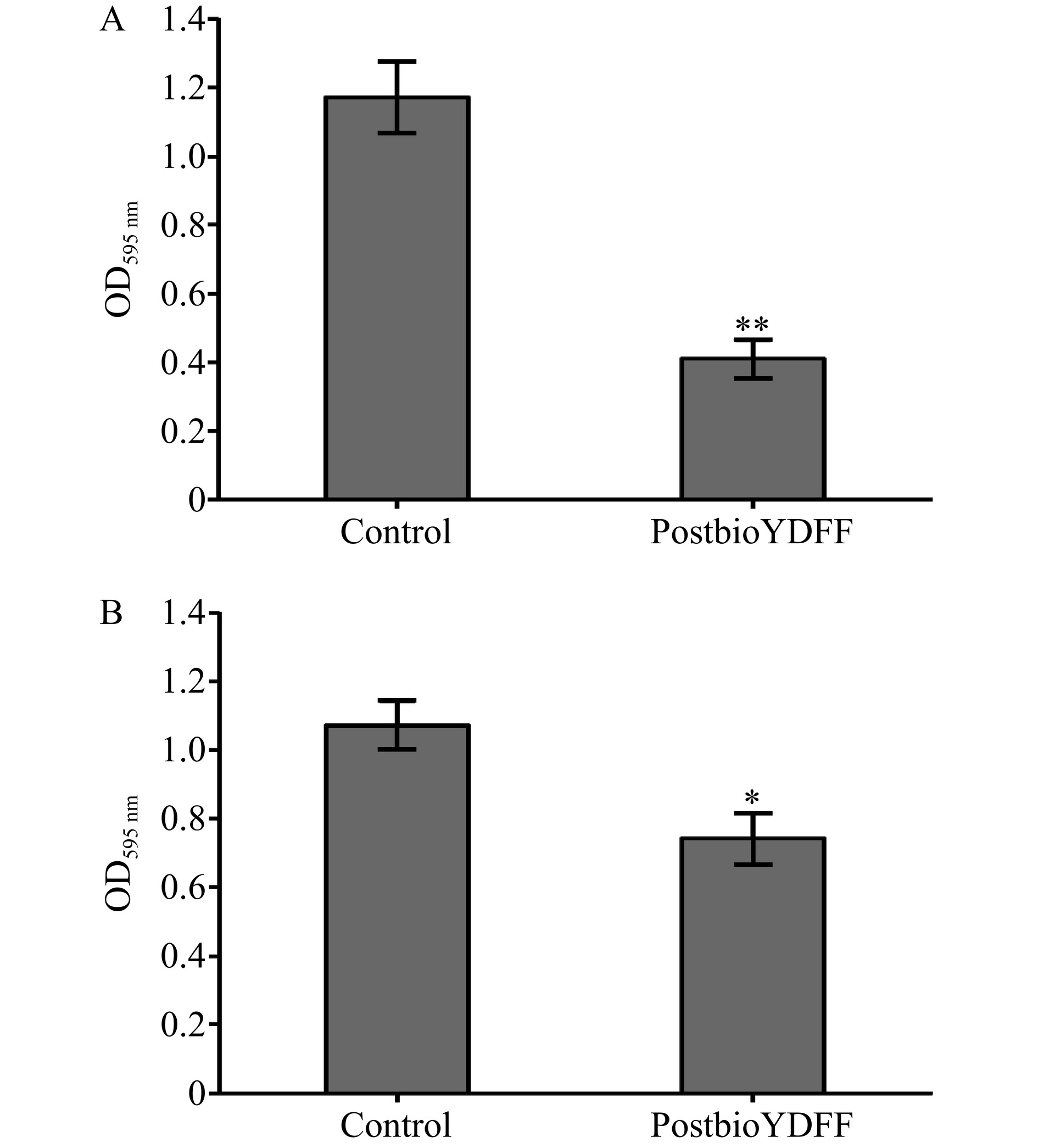
取100 μL菌悬液加入到96孔板中,再加入100 μL的后生元PostbioYDFF,混匀,对照组加入等量的PBS,放入37 ℃培养箱中培养24 h后,用 PBS 缓冲液洗涤3次。每组三个平行。每孔加入200 μL甲醇固定15 min后,弃甲醇,放置5 min后每孔加入200 μL 1%结晶紫溶液染色5 min,再用PBS缓冲液轻缓地洗涤3次,待干燥后,每孔加入200 μL 33%的乙酸溶液进行脱色。脱色完成后,使用酶标仪测OD595 nm值。按照以下公式计算后生元PostbioYDFF对铜绿假单胞菌生物膜形成的抑制率[20]。
式中:ODc为对照组OD值;ODt为后生元PostbioYDFF处理组的OD值。
1.2.7 后生元PostbioYDFF对铜绿假单胞菌生物膜清除的影响
取200 μL菌悬液加入到96孔板中,于37 ℃中培养24 h。形成成熟的生物膜后弃去培养液,加入200 μL的后生元PostbioYDFF,对照组加入等量的PBS,每组三个平行。于37 ℃中培养30 min后,用PBS缓冲液洗涤3次,使用结晶紫染色法观察生物膜剩余量,固定、染色和脱色同1.2.6节。脱色完成后,使用酶标仪测OD595 nm值。按照以下公式计算后生元PostbioYDFF对铜绿假单胞菌生物膜清除率[21−22]。
式中:ODc为对照组OD值;ODt为后生元PostbioYDFF处理组的OD值。
1.2.8 后生元PostbioYDFF对铜绿假单胞菌形态结构的影响
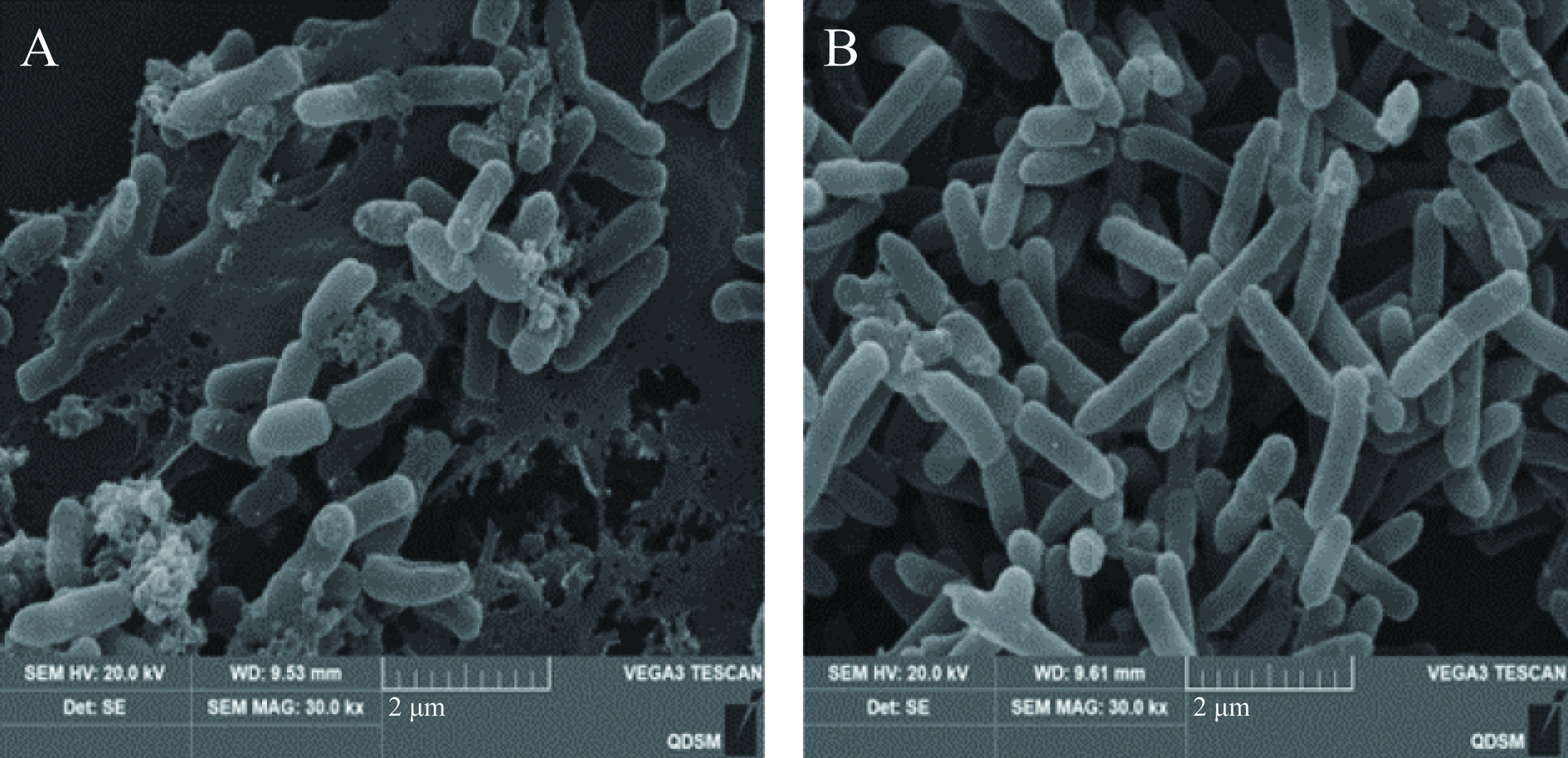
取2 mL菌悬液,在4 ℃下10000 r/min 离心10 min。弃上清液,用0.1 mol/L PBS缓冲液清洗菌体2~3次后复溶。取0.5 mL复溶后的菌悬液加入0.5 mL后生元PostbioYDFF,对照组加入等量PBS,混匀。37 ℃反应12 h后,4 ℃下10000 r/min离心10 min收集菌体,用PBS清洗2~3次,弃上清液,加入2.5%戊二醛溶液,置于4 ℃避光固定24 h。不同浓度的乙醇(30%、50%、70%、85%、95%和100%)对细胞进行梯度脱水。最后,将细胞铺在玻璃片上,在空气中干燥,进行喷金镀膜,使用扫描电子显微镜对铜绿假单胞菌的细胞形态进行观察[23]。
1.2.9 后生元PostbioYDFF对铜绿假单胞菌运动性的影响
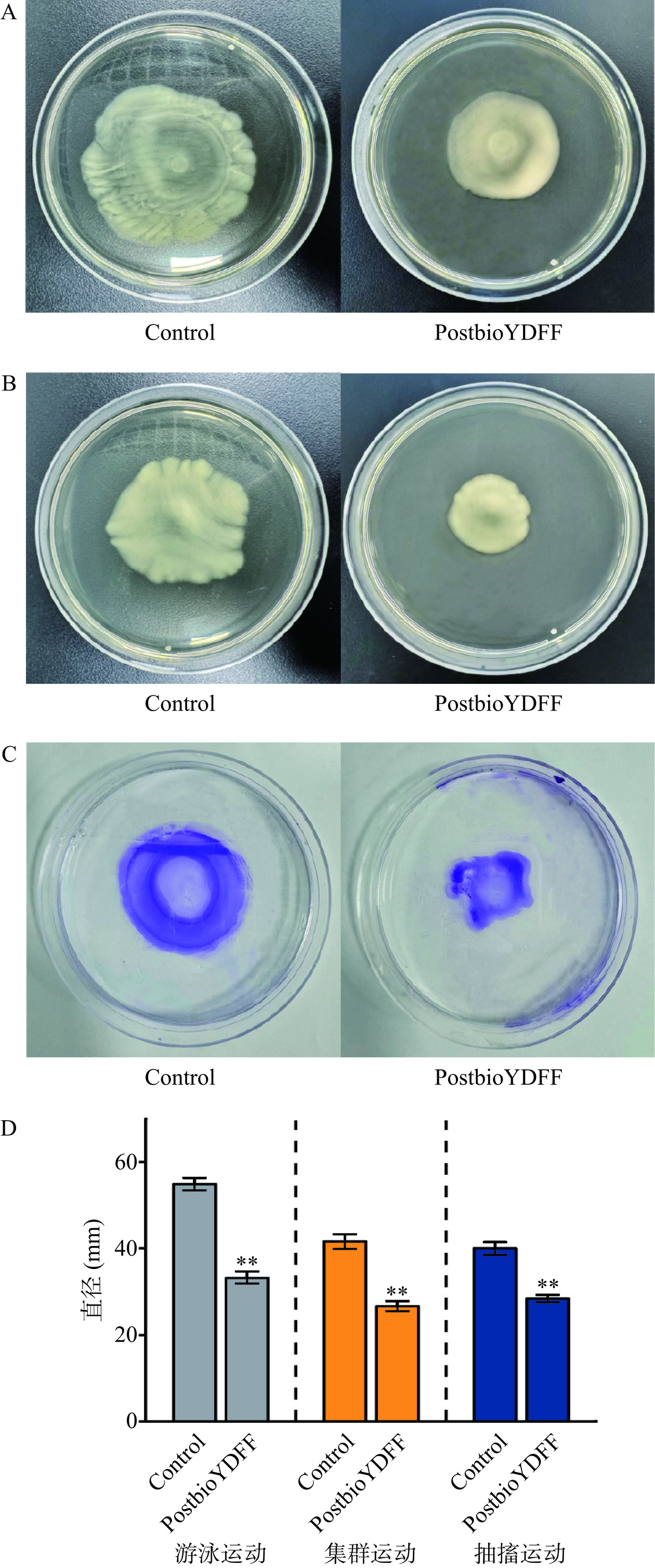
铜绿假单胞菌的游泳、集群和抽搐运动是按文献[20]的方法测定的。将后生元PostbioYDFF分别添加至未凝固的NB培养基中,培养基中琼脂浓度分别为0.3%、0.5%和1%,使后生元PostbioYDFF的终浓度为100 μL/mL,对照组加入等体积的PBS,每种处理重复三次。游泳运动检测:取2 μL菌悬液接种于含0.3%琼脂的NB培养基中心,置于37 ℃孵育24 h后测量菌落直径。集群运动检测:取2 μL菌悬液接种于含0.5%琼脂的NB培养基中心,置于37 ℃孵育24 h后测量菌落直径。抽搐运动检测:取2 μL菌悬液接种于含1%琼脂的NB培养基底层,置于37 ℃倒置孵育24 h后弃去培养基,用1%结晶紫染色1 min,流水洗去未结合的染料,测量抽搐运动形成的菌圈直径。
1.2.10 后生元PostbioYDFF对铜绿假单胞菌转录组学分析样品制备
取1 mL菌悬液,在4 ℃下10000 r/min 离心10 min。弃上清液,用0.1 mol/L PBS缓冲液清洗菌体2~3次后复溶。取0.5 mL复溶后的菌液加入0.5 mL后生元PostbioYDFF,对照组加入等量PBS,混匀。置于37 ℃水浴锅中反应30 min,在4 ℃下10000 r/min 离心10 min后,用0.1 mol/L PBS缓冲液洗涤菌体三次,按照RNAprep Pure Cell/Bacteria Kit RNAprep Pure培养细胞/细菌总RNA提取试剂盒说明和步骤提取纯化总RNA,将提取的RNA根据无RNase琼脂糖凝胶电泳(1%胶,180 V电压,电泳16 min)分析RNA降解和DNA污染。根据Agilent 2100 bioanalyzer精确检测RNA完整性和总量,提取到的RNA经检测合格后,通过探针去除Total RNA中的核糖体RNA(rRNA)富集mRNA。随后加入fragmentation buffer缓冲液将得到的mRNA随机打断成短片段,按照链特异性建库的方式建库。文库构建完成后,先使用Qubit2.0Fluorometer进行初步定量,稀释文库至1.5 ng/uL,随后使用Agilent2100bioanalyzer对文库的insertsize进行检测,insertsize符合预期后,qRT-PCR对文库有效浓度进行准确定量(文库有效浓度高于1.5 nmol/L),以保证文库质量。使用Illumina对构建的文库进行测序。根据基因的长度计算每个基因的FPKM(Fragments Per Kilobase of transcript per Million mapped fragments,每千碱基转录本长度每百万映射片段数),并计数映射到该基因的读数。通过GOseq R包软件实现差异表达基因的KEGG(Kyoto Encyclopedia of Genes and Genomes,京都基因与基因组百科全书)富集分析,具有小于0.05的校正的P值的term通过差异表达基因显著富集。
1.2.11 实时荧光定量PCR验证
为了验证RNA-seq结果的准确性,选择7个靶基因进行qPCR分析[24]。样品的前处理以及提取RNA同1.2.10小节中转录组学样品分析制备。相关的特异引物由北京擎科生物科技有限公司合成(表1)。使用逆转录试剂盒SynScript®Ⅲ RT SuperMix for qPCR进行逆转录扩增,反应体系(20 μL):RNA template 500 ng,5×gDNA Remover Mix 2 μL,5×SynScript®Ⅲ RT SuperMixMix 4 μL,RNase-free water加至20.0 μL。反应参数设置为:25 ℃ 10 min,50 ℃ 30 min,85 ℃ 5 min。扩增体系为(20 μL):ArtiCanCEO SYBR qPCR Mix 10 μL,10 μmol/L Primer F 0.8 μL,10 μmol/L Primer R 0.8 μL,cDNA 1 μL,ddH2O加至20 μL。扩增反应条件:95 ℃ 15 s;60 ℃ 20 s,72 ℃ 20 s,循环40次。以16S rRNA作为内参基因,通过对目的基因和内参基因的Ct值进行归一化处理,采用2−△△Ct法计算目的基因的表达水平。
表 1 实时荧光定量引物Table 1. Primers used for qPCR引物名称 上游引物序列(5'→3') 下游引物序列(5'→3') acs CGAGGACGGCTACTACTGGA GTAGACATAGATGCCCTGCCC acnA GACCGCAGGAACCTGAAACT ACTCGACCTCGTTGAGGGTA exaA CGATCACCTGGGAACAGGAC CTGGGCAGCTTGAATACCCA fleR GTGATCATCGACACCCTGCG ATCTGCGCGAGCTTGTAGC pmrA AAGGTACTCTCGCGCAACCA ACGGTGCGGATCAACTGG kdpB TCCGGGTGGGCAAGGAAT ATCACGTTGAGCACGTCGAG flgE ATCGGCAAGACTTCCTGGGT CGATGAGATTCACCAGCTCGC 16S rRNA AGTTGGGAGGAAGGGCAGTA GGATGCAGTTCCCAGGTTGA 1.3 数据处理
每个处理组都分别进行了三次独立试验,使用SPSS 18.0版,进行方差分析(ANOVA),数据以均值±标准差(SD)表示。对实验结果的数据统计使用Origin 8.0软件和GraphPad Prism 9.5.0软件,每组间两两比较采用t检验法。
2. 结果与分析
2.1 后生元PostbioYDFF对铜绿假单胞菌的抑菌能力
如图1A所示,后生元PostbioYDFF对铜绿假单胞菌的抑菌圈直径为28.9±0.3 mm。如图1B所示,与阳性对照相比后生元PostbioYDFF浓度为100 μL/mL时,为明显抑制细菌生长的最低浓度,抑制率为92.87%。因此,后生元PostbioYDFF对铜绿假单胞菌的最小抑菌浓度为100 μL/mL。如图1C所示,经过后生元PostbioYDFF处理的细菌生长受到明显抑制,生长缓慢甚至不生长;而未加后生元PostbioYDFF的对照组铜绿假单胞菌生长呈现出典型的S型生长曲线,细菌在2 h时进入生长对数期,生长迅速。上述结果说明,后生元PostbioYDFF可以抑制铜绿假单胞菌的生长,具有良好的抑菌活性。
2.2 后生元PostbioYDFF对铜绿假单胞菌生物膜的影响
通过结晶紫染色法测定了后生元PostbioYDFF对铜绿假单胞菌生物膜形成的抑制作用以及清除成熟的生物膜。如图2A所示,后生元PostbioYDFF对铜绿假单胞菌生物膜形成的抑制率为64.96%,具有极显著的抑制作用(P<0.01)。如图2B所示,后生元PostbioYDFF对铜绿假单胞菌成熟的生物膜清除率为30.66%,具有显著的清除作用(P<0.05)。由此可以推断,后生元PostbioYDFF能显著抑制铜绿假单胞菌生物膜的形成并可以清除已成熟的生物膜。
2.3 后生元PostbioYDFF对铜绿假单胞菌形态结构的影响
如图3所示,后生元 PostbioYDFF处理组细胞与对照组相比有显著的形态差异。经后生元PostbioYDFF处理的细胞变得不完整,铜绿假单胞菌的外部结构遭受严重损伤,细胞发生皱缩,光滑规则的表面变得松弛并伴有细胞质流出,细胞质边缘颗粒较多,细胞内容物出现浓缩现象(图3A)。而对照组细胞保持完好,表面光滑,没有明显的凹陷和断裂(图3B)。扫描电镜结果表明,后生元PostbioYDFF破坏了铜绿假单胞菌的菌体结构。
2.4 后生元PostbioYDFF对铜绿假单胞菌运动性的影响
如图4所示,后生元PostbioYDFF对铜绿假单胞菌的运动性(游泳运动、集群运动和抽搐运动)有明显影响。如图4A所示,以接种点为中心,形成同心圆状,与对照组相比(55.0±1.5 mm),后生元PostbioYDFF处理组的直径为33.2±1.7 mm,极显著减小(P<0.01)。如图4B所示,对照组(41.7±1.8 mm)呈现出不规则形状,有运动痕迹,后生元PostbioYDFF处理组的直径为26.7±1.3 mm,极显著减小(P<0.01)。如图4C所示,为铜绿假单胞菌在培养基底部形成的抽搐环,对照组以接种点为中心形成同心圆状,直径为40.1±1.7 mm,而后生元PostbioYDFF处理组(28.4±1.0 mm)呈现出不规则形状,直径极显著减小(P<0.01)。
2.5 后生元PostbioYDFF对铜绿假单胞菌基因转录表达的影响
2.5.1 PCA主成分分析
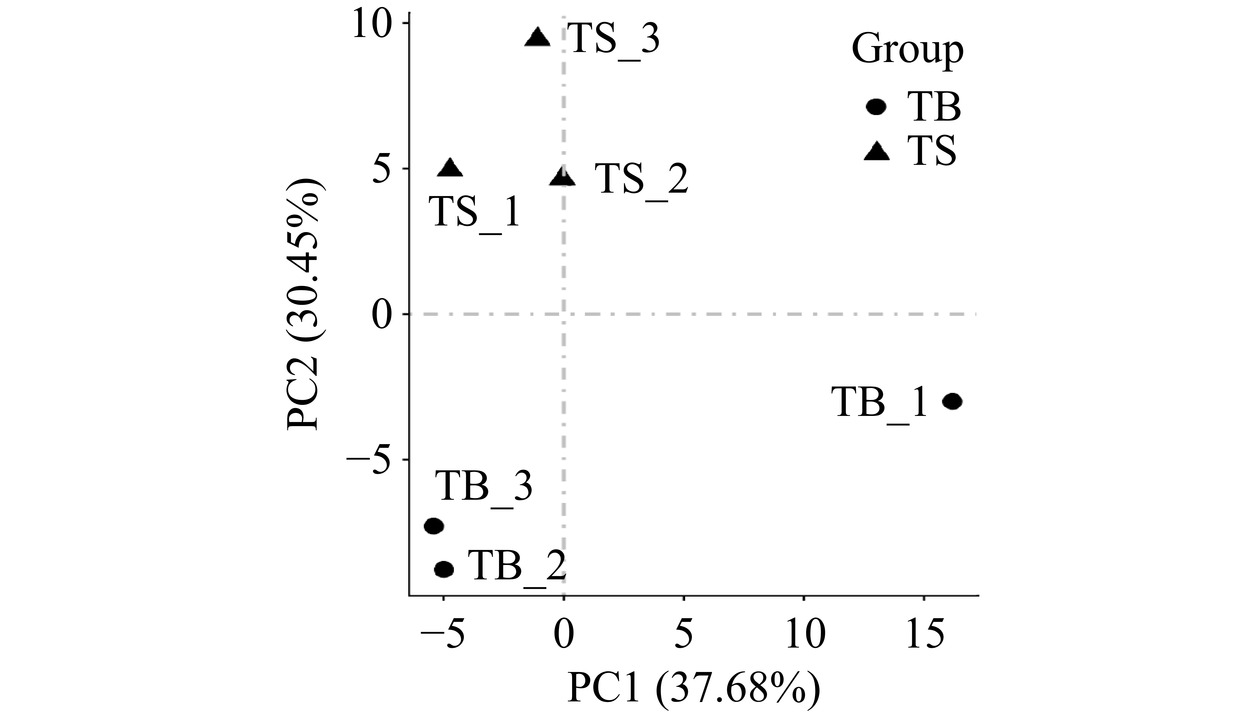
将上述RNA样品进行建库分析,每个样品进行三个生物学重复,后生元PostbioYDFF处理组(TB)和对照组(TS)样品的Q20值均大于96%,Q30均大于92%,GC含量在63%附近(表2),表明样品测序结果优良,数据准确度较高。PCA主成分分析如图5所示,经过后生元PostbioYDFF处理后,组内样本差异性小,而组间的差异性较大,表明后生元PostbioYDFF对铜绿假单胞菌基因的表达具有较大的影响。
表 2 数据预处理结果统计Table 2. Statistics of data preprocessing results样品名称 原始数据 过滤后数据 Q20(%) Q30(%) GC含量(%) TB_1 7939180 7870552 96.69 92.01 63.75 TB_2 7681576 7606790 96.91 92.48 62.73 TB_3 8008556 7915630 96.80 92.20 63.33 TS_1 7658326 7574666 97.28 93.30 63.64 TS_2 8007274 7904440 97.98 94.52 65.22 TS_3 7878928 7779418 97.43 93.62 63.88 2.5.2 差异表达基因分析
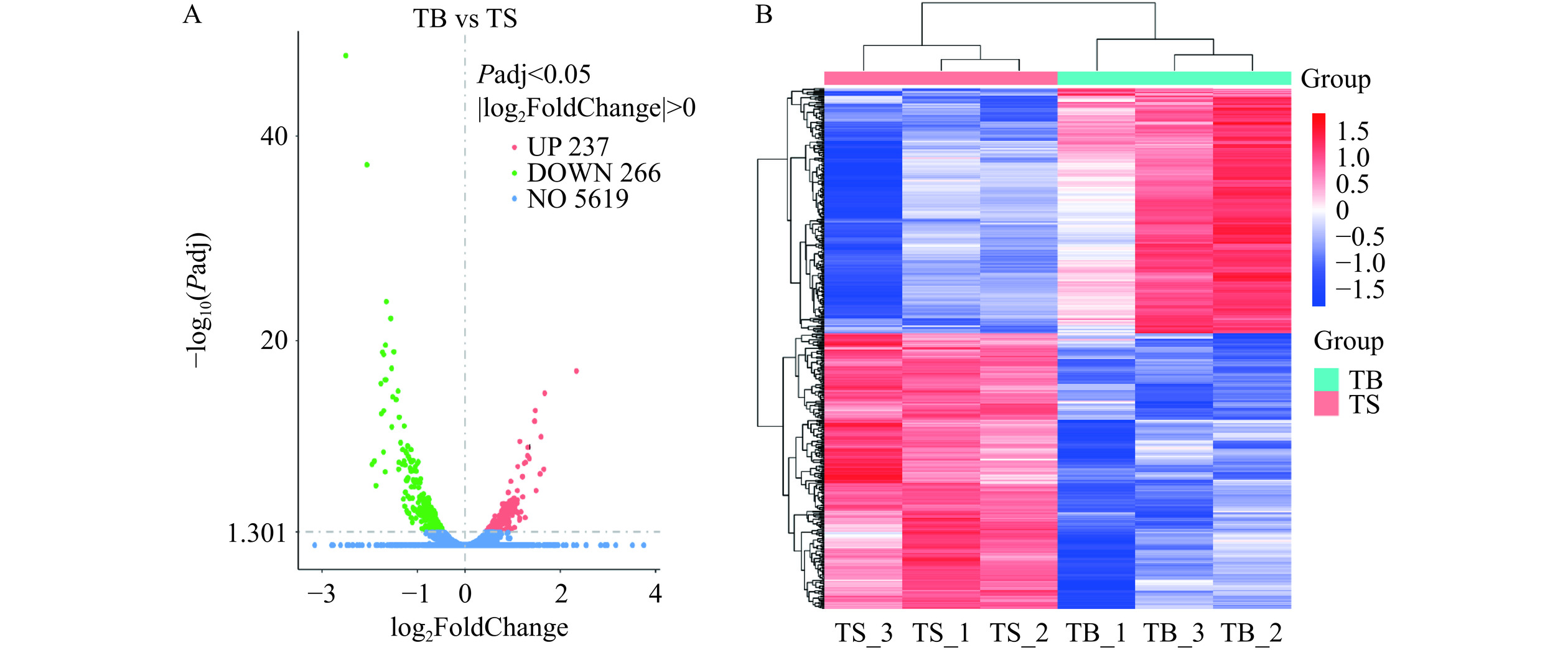
选择|log2(FoldChange)|>0和Padj<0.05作为筛选差异显著基因(DEGs)的标准[25],与对照组相比,经后生元PostbioYDFF处理后共筛选出503个DEGs,其中266个DEGs显著下调,237个DEGs显著上调(P<0.05),如图6A所示。基于两组样本的FPKM值,构建了热图以探究其间的基因表达模式。结果如图6B显示,平行样本间存在相似的DEGs,并且对照组与实验组之间的DEGs呈现出显著的差异性。
2.5.3 差异基因KEGG富集分析
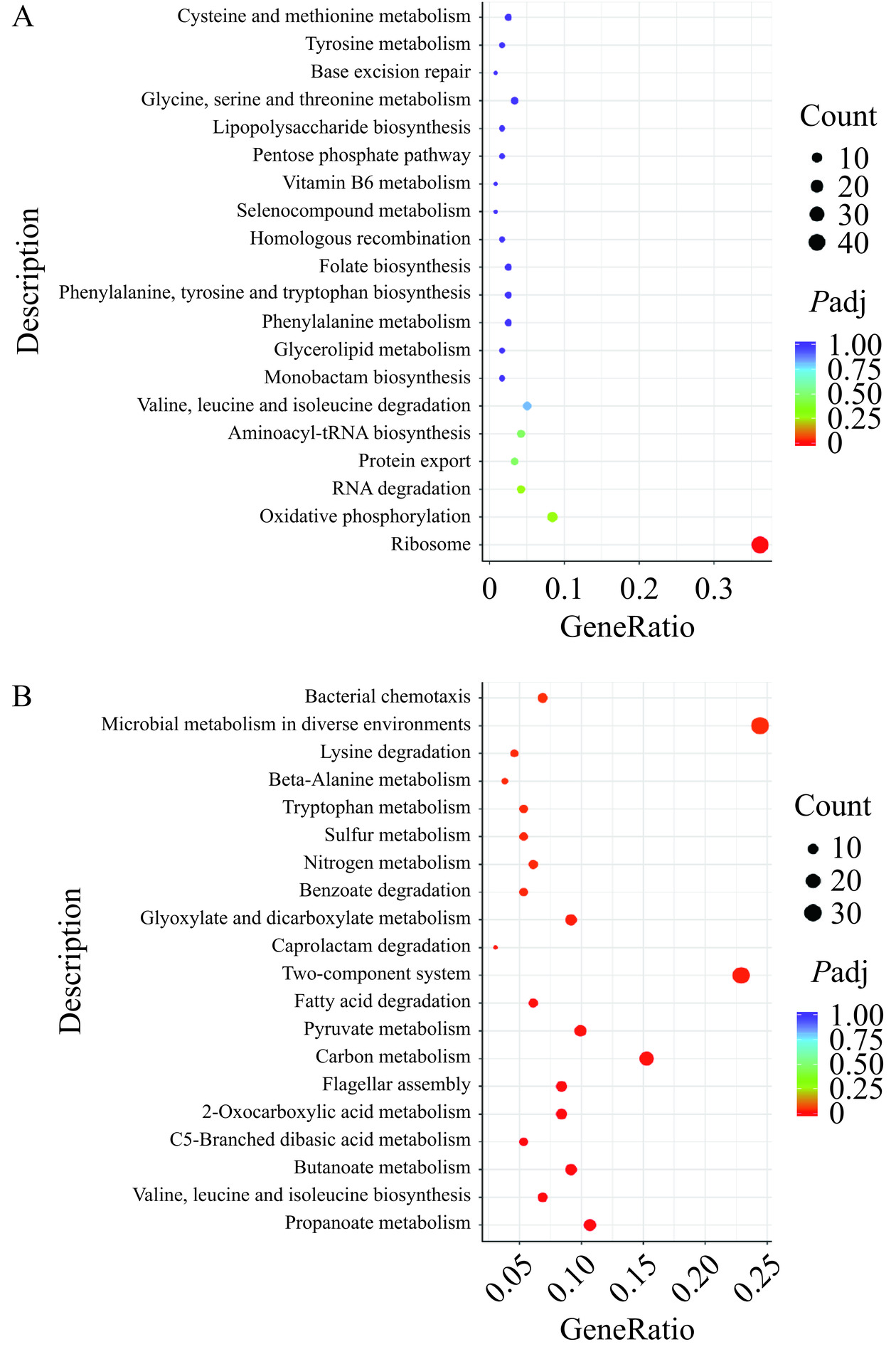
对后生元PostbioYDFF处理后的DEGs进行KEGG功能富集分析,选择富集结果top20的通路,结果如图7A所示,经后生元PostbioYDFF作用后P. aeruginosa上调DEGs主要富集在核糖体(Ribosome)、氧化磷酸化(Oxidative phosphorylation)、RNA降解(RNA degradation)、氨酰tRNA生物合成(Aminoacyl-tRNA biosynthesis)等59条通路;如图7B所示,下调DEGs主要富集在鞭毛组装(Flagellar assembly)、双组分系统(Two-component system)、丙酮酸代谢(Pyruvate metabolism)、脂肪酸降解(Fatty acid degradation)等61条通路。
在KEGG富集分析的结果中,与对照组相比,后生元PostbioYDFF处理组有大量DEGs富集在能量代谢中。作为微生物的重要代谢途径,能量代谢直接影响微生物的生长特性[26]。能量代谢是生物体维持生命活动和进行生物学转化的基础,能量代谢包括糖酵解(Glycolysis)、TCA循环(TCA cycle)、丙酮酸代谢等主要通路[27]。如表3所示,与对照组相比,后生元PostbioYDFF处理组编码糖酵解的关键基因除gap上调外,其他基因均显著下调(P<0.001)。其中acs下降了接近2倍,acs编码乙酰辅酶A合成酶(Acetyl-CoA synthetase),乙酰辅酶A合成酶负责催化乙酰辅酶A(Acetyl-CoA)的合成,乙酰辅酶A是生物体内许多重要代谢途径的中心分子,间接参与能量代谢,也是许多生物合成途径的起始或中间产物,包括脂肪酸合成、胆固醇合成、酮体生成等。与对照组相比,后生元PostbioYDFF处理组参与丙酮酸代谢通路的相关基因显著(P<0.001)下调,尤其是exaA下调了5倍。通常情况下,丙酮酸在有氧环境中通过丙酮酸脱氢酶复合物的催化作用转化为乙酰辅酶A(Acetyl-CoA),与草酰乙酸进行缩合反应,并经历一系列的氧化和脱羧反应,最终生成二氧化碳、水并释放出能量[28]。与对照组相比,后生元PostbioYDFF处理组参与TCA循环相关的基因RS11350、RS04820、acnA,在转录水平上均下调了一倍多。acnA基因编码乌头酸水合酶,催化柠檬酸异构化为异柠檬酸,再由异柠檬酸脱氢酶(Icd)进一步转化为α-酮戊二酸,有助于维持循环流动,利用这些碳源,通过TCA循环将其转化为能量和生物合成所需的前体物[29]。由此可以推断,后生元PostbioYDFF可能通过干扰糖酵解和抑制TCA循环与丙酮酸代谢来减少铜绿假单胞菌的有氧呼吸,导致铜绿假单胞菌的能量代谢途径受损,抑制了菌体的正常能量代谢过程,对细胞生长和代谢产生不利影响,从而发挥其抗菌活性。
表 3 后生元PostbioYDFF处理后铜绿假单胞菌与能量代谢有关的显著差异基因表达情况Table 3. Expression of significant difference genes related to energy metabolism in P. aeruginosa after postbiotic PostbioYDFF treatment基因编号 基因描述 TB vs TS
log2FCP-value 糖酵解 RS06190 exaA Ethanol dehydrogenase OS −2.50 3.27E-52 RS20885 nosZ propeller repeat −1.67 3.21E-23 RS13470 gap YegP family protein 1.14 4.49E-13 RS04640 acs Acetyl-coenzymeA synthetase N-terminus −0.94 1.27E-08 丙酮酸代谢 RS21100 ppc Phosphoenolpyruvate carboxylase −0.62 3.18E-05 RS04640 acs acetate--CoA ligase −0.94 1.27E-08 RS06190 exaA quinoprotein ethanol dehydrogenase −2.50 3.27E-52 RS05105 aldehyde dehydrogenase −1.65 1.11E-27 RS01755 acetyl-CoA C-acetyltransferase −1.19 1.85E-11 TCA 循环 RS11350 acetyl-CoA hydrolase/transferase family protein −0.55 6.46E-04 RS23310 acnA aconitate hydratase −0.54 1.74E-03 RS04820 class II fumarate hydratase −0.51 2.11E-03 注:TB表示后生元PostbioYDFF处理组;TS表示对照组;log2FC>0表示上调;log2FC<0表示下调;表4~表6同。 在KEGG富集分析的结果中,与对照组相比,后生元PostbioYDFF处理组有大量DEGs显著富集在脂肪酸降解通路中。如表4所示,与脂肪酸降解相关的7个显著差异基因(RS01755、RS01800、RS28740、RS10100、RS01760、RS12885、RS15790)表达下调,1个显著差异基因(RS18010)表达上调。脂肪酸除了能够作为信号分子穿越细胞膜影响细菌毒性,还能够影响生物膜的形成以及运动能力,同时脂肪酸还可以作为细菌营养物质参与细菌能量代谢和引发细胞应激反应[30]。脂肪酸的分解代谢通过β-氧化过程进行,并产生乙酰辅酶A,乙酰辅酶A进一步代谢以获得细胞生物合成的能量和前体。β氧化途径的每个循环由四个反应组成:酰基辅酶A脱氢酶,2-烯酰辅酶A水合酶,3-羟酰辅酶A脱氢酶和3-酮硫解酶[31]。在本研究中,后生元PostbioYDFF处理组的3-羟酰辅酶A脱氢酶(RS01760)和酰基辅酶A脱氢酶(RS12885)的表达均下调。由此可以推断,后生元PostbioYDFF抑制了铜绿假单胞菌的β氧化过程。
表 4 后生元PostbioYDFF处理后铜绿假单胞菌与脂质代谢合成有关的显著差异基因表达情况Table 4. Expression of significant difference genes related to lipid metabolism and synthesis in P. aeruginosa after postbiotic PostbioYDFF treatment基因编号 基因描述 TB vs TS log2FC P-value RS18010 acyl-CoA dehydrogenase 1.67 4.92E-18 RS01755 acetyl-CoA C-acetyltransferase −1.19 1.85E-11 RS01800 crotonase/enoyl-CoA hydratase family protein −1.39 5.05E-10 RS28740 acetyl-CoA C-acyltransferase −0.93 4.89E-09 RS10100 fadB fatty acid oxidation complex subunit alpha −1.07 3.55E-08 RS01760 3-hydroxyacyl-CoA dehydrogenase −0.87 1.90E-07 RS12885 acyl-CoA dehydrogenase −0.62 5.26E-05 RS15790 AMP-binding protein −0.54 1.49E-03 双组分系统(Two-component systems,TCSs)是细菌最重要的代谢系统之一。铜绿假单胞菌TCS是细菌信号转导的关键介质,在控制毒性因子的分泌和响应内部、外部刺激等多方面都发挥着重要作用[32]。TCS参与鞭毛蛋白的合成、组装和调节,例如fleR激活鞭毛基体、钩和丝蛋白的转录,因此对于细菌运动很重要[33]。如表5所示,与对照组相比,后生元PostbioYDFF处理组fleR的表达在转录水平下调了2倍。双组分系统还包括与群体感应相关的基因(pmrA),群体感应的细胞间通讯机制能够使细菌感知其所在微生物群落中细胞密度和物种组成的变化,并调控生物膜的形成。通过群体感应和生物膜的形成,细菌能够在复杂多变的微生物群落中生存和繁衍[34]。与对照组相比,后生元PostbioYDFF处理组pmrA的表达在转录水平下调了2.44倍。由此可以推断,后生元PostbioYDFF通过降低铜绿假单胞菌的运动水平,抑制生物膜的形成,引发群体感应系统从而削弱细菌行为来达到抑菌效果。
表 5 后生元PostbioYDFF处理后铜绿假单胞菌与双组分有关的显著差异基因表达情况Table 5. Expression of significant difference genes related to two components of P. aeruginosa after postbiotic PostbioYDFF treatment基因编号 基因描述 TB vs TS log2FC P-value RS19680 fleR Response regulator receiver domain −1.03 1.18E-08 RS00690 nirM Cytochrome C oxidase, cbb3-type, subunit III −1.76 6.58E-16 RS15705 kdpB potassium-transporting ATPase subunit −1.21 1.36E-12 RS25790 pmrA two-component system response regulator −1.22 7.45E-09 RS16865 dctP C4-dicarboxylate TRAP substrate-binding protein −1.24 7.02E-09 RS02990 bdlA biofilm dispersion protein −0.91 2.54E-07 RS18915 dctA C4-dicarboxylate transporter −1.10 5.37E-07 RS15710 kdpA potassium-transporting ATPase subunit −0.76 2.27E-06 RS16870 dctQ C4-dicarboxylate TRAP transporter small permease protein −1.17 3.35E-05 RS08465 ntrC two-component system response regulator −0.85 4.34E-05 RS15700 kdpC potassium-transporting ATPase subunit −0.91 6.18E-05 RS23460 opmH efflux transporter outer membrane subunit −0.63 1.78E-04 RS22785 aer2 aerotaxis transducer N-terminal domain −0.69 2.31E-03 细菌粘附是生物膜形成的初始阶段,抑制细菌粘附对于控制细菌生物膜的成熟至关重要[35]。铜绿假单胞菌的粘附与鞭毛及其游泳、集群和抽搐运动密切相关,鞭毛作为细菌主要运动器官,在细菌定植、运动、粘附、趋化性等生理活动中发挥重要作用,并通过分泌一些毒力蛋白参与细菌致病性[36−38]。此外,鞭毛相互作用能促进细菌生物膜的形成,并通过相邻菌落表面接触保持其结构[39]。如表6所示,与对照组相比,后生元PostbioYDFF处理组中与鞭毛装置相关的7个基因(flgE、fleR、flgG、flgH、fliO、fliF、flhA)表达显著(P<0.01)下调。由此可以推断,后生元PostbioYDFF通过抑制鞭毛组装相关基因的表达,从而导致鞭毛相关蛋白合成受阻,降低了其运动能力,减少了细菌初始表面附着,从而减少生物膜形成和清除成熟的生物膜,说明后生元PostbioYDFF可能干扰铜绿假单胞菌生物膜合成、粘附性以及运动性能,从而抑制其正常生长繁殖。这与后生元PostbioYDFF可以降低铜绿假单胞菌的运动性相一致。
表 6 后生元PostbioYDFF处理后铜绿假单胞菌与鞭毛装置有关的显著差异基因表达情况Table 6. Expression of significant difference genes related to flagellar apparatus of P. aeruginosa after postbiotic PostbioYDFF treatment基因编号 基因描述 TB vs TS log2FC P-value RS19775 flgE flagellar hook protein −1.15 3.66E-10 RS19680 fleR sigma-54-dependent response regulator −1.03 1.18E-08 RS19765 flgG flagellar basal-body rod protein −0.82 2.87E-06 RS19760 flgH flagellar basal body L-ring protein −0.78 3.30E-05 RS02820 fliO flagellar biosynthetic protein −0.83 1.06E-03 RS19670 fliF flagellar basal-body MS-ring/collar protein −0.50 1.39E-03 RS02785 flhA flagellar biosynthesis protein −0.52 2.24E-03 2.6 差异基因的qPCR验证
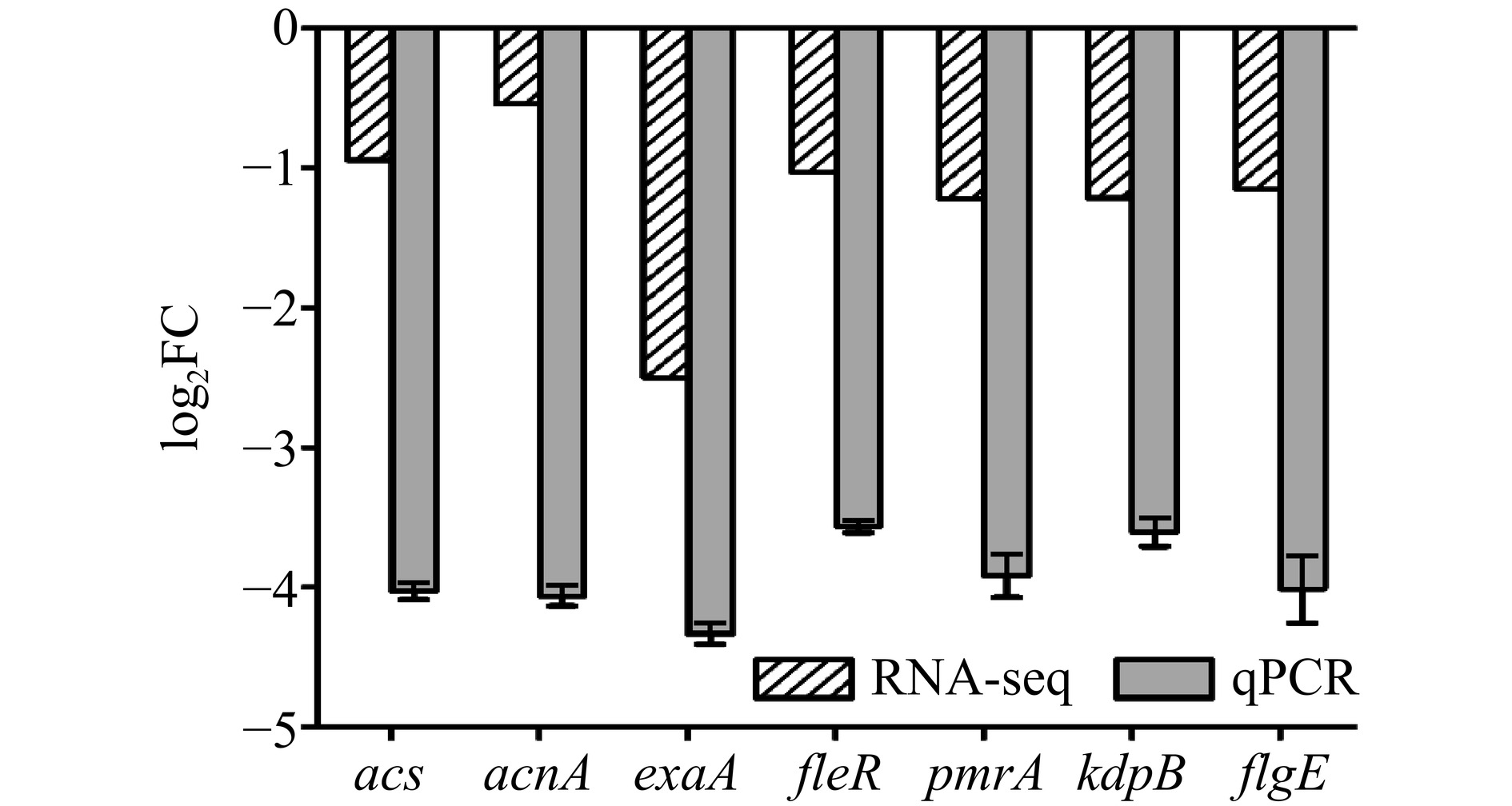
对RNA-seq分析得到的其中8个DEGs进行qPCR分析。如图8所示,DEGs表达趋势与测序结果一致,表明RNA-seq结果准确。
3. 结论
本研究的发现揭示了后生元PostbioYDFF对铜绿假单胞菌具有显著的抑制效果。具体而言,后生元PostbioYDFF处理形成了平均直径为28.9±0.3 mm的抑菌圈,能够抑制铜绿假单胞菌生物膜的形成,抑制率为64.96%,并能清除已经成熟的生物膜,清除率为30.66%。此外,后生元PostbioYDFF还能显著破坏菌体形态,导致细胞皱缩、松弛,并伴有细胞质的外泄,同时减弱了铜绿假单胞菌的运动性,包括游泳运动、集群运动和抽搐运动。通过转录组学分析,进一步发现后生元PostbioYDFF通过干扰细菌的能量代谢和β氧化过程,扰乱TCS系统,抑制鞭毛组装降低运动水平,并激活群体感应系统,从而在分子层面削弱了细菌的行为表现。这些发现为后生元PostbioYDFF作为食品防腐剂的可能性提供了理论基础,并为未来替代抗生素治疗提供了新的视角。尽管如此,仍有许多问题亟待深入研究:需要针对不同的食品体系来评估后生元PostbioYDFF对铜绿假单胞菌的实际抗菌效果,以及其对食品感官性质的潜在影响。此外,本研究仅触及了后生元PostbioYDFF影响铜绿假单胞菌相关基因表达的层面,而对于其上下游通路的具体作用机制尚需进一步的实验探索。这将有助于更全面地理解后生元PostbioYDFF的抑菌作用,为开发新型食品防腐剂和抗菌策略提供坚实的科学依据。
-
表 1 实时荧光定量引物
Table 1 Primers used for qPCR
引物名称 上游引物序列(5'→3') 下游引物序列(5'→3') acs CGAGGACGGCTACTACTGGA GTAGACATAGATGCCCTGCCC acnA GACCGCAGGAACCTGAAACT ACTCGACCTCGTTGAGGGTA exaA CGATCACCTGGGAACAGGAC CTGGGCAGCTTGAATACCCA fleR GTGATCATCGACACCCTGCG ATCTGCGCGAGCTTGTAGC pmrA AAGGTACTCTCGCGCAACCA ACGGTGCGGATCAACTGG kdpB TCCGGGTGGGCAAGGAAT ATCACGTTGAGCACGTCGAG flgE ATCGGCAAGACTTCCTGGGT CGATGAGATTCACCAGCTCGC 16S rRNA AGTTGGGAGGAAGGGCAGTA GGATGCAGTTCCCAGGTTGA 表 2 数据预处理结果统计
Table 2 Statistics of data preprocessing results
样品名称 原始数据 过滤后数据 Q20(%) Q30(%) GC含量(%) TB_1 7939180 7870552 96.69 92.01 63.75 TB_2 7681576 7606790 96.91 92.48 62.73 TB_3 8008556 7915630 96.80 92.20 63.33 TS_1 7658326 7574666 97.28 93.30 63.64 TS_2 8007274 7904440 97.98 94.52 65.22 TS_3 7878928 7779418 97.43 93.62 63.88 表 3 后生元PostbioYDFF处理后铜绿假单胞菌与能量代谢有关的显著差异基因表达情况
Table 3 Expression of significant difference genes related to energy metabolism in P. aeruginosa after postbiotic PostbioYDFF treatment
基因编号 基因描述 TB vs TS
log2FCP-value 糖酵解 RS06190 exaA Ethanol dehydrogenase OS −2.50 3.27E-52 RS20885 nosZ propeller repeat −1.67 3.21E-23 RS13470 gap YegP family protein 1.14 4.49E-13 RS04640 acs Acetyl-coenzymeA synthetase N-terminus −0.94 1.27E-08 丙酮酸代谢 RS21100 ppc Phosphoenolpyruvate carboxylase −0.62 3.18E-05 RS04640 acs acetate--CoA ligase −0.94 1.27E-08 RS06190 exaA quinoprotein ethanol dehydrogenase −2.50 3.27E-52 RS05105 aldehyde dehydrogenase −1.65 1.11E-27 RS01755 acetyl-CoA C-acetyltransferase −1.19 1.85E-11 TCA 循环 RS11350 acetyl-CoA hydrolase/transferase family protein −0.55 6.46E-04 RS23310 acnA aconitate hydratase −0.54 1.74E-03 RS04820 class II fumarate hydratase −0.51 2.11E-03 注:TB表示后生元PostbioYDFF处理组;TS表示对照组;log2FC>0表示上调;log2FC<0表示下调;表4~表6同。 表 4 后生元PostbioYDFF处理后铜绿假单胞菌与脂质代谢合成有关的显著差异基因表达情况
Table 4 Expression of significant difference genes related to lipid metabolism and synthesis in P. aeruginosa after postbiotic PostbioYDFF treatment
基因编号 基因描述 TB vs TS log2FC P-value RS18010 acyl-CoA dehydrogenase 1.67 4.92E-18 RS01755 acetyl-CoA C-acetyltransferase −1.19 1.85E-11 RS01800 crotonase/enoyl-CoA hydratase family protein −1.39 5.05E-10 RS28740 acetyl-CoA C-acyltransferase −0.93 4.89E-09 RS10100 fadB fatty acid oxidation complex subunit alpha −1.07 3.55E-08 RS01760 3-hydroxyacyl-CoA dehydrogenase −0.87 1.90E-07 RS12885 acyl-CoA dehydrogenase −0.62 5.26E-05 RS15790 AMP-binding protein −0.54 1.49E-03 表 5 后生元PostbioYDFF处理后铜绿假单胞菌与双组分有关的显著差异基因表达情况
Table 5 Expression of significant difference genes related to two components of P. aeruginosa after postbiotic PostbioYDFF treatment
基因编号 基因描述 TB vs TS log2FC P-value RS19680 fleR Response regulator receiver domain −1.03 1.18E-08 RS00690 nirM Cytochrome C oxidase, cbb3-type, subunit III −1.76 6.58E-16 RS15705 kdpB potassium-transporting ATPase subunit −1.21 1.36E-12 RS25790 pmrA two-component system response regulator −1.22 7.45E-09 RS16865 dctP C4-dicarboxylate TRAP substrate-binding protein −1.24 7.02E-09 RS02990 bdlA biofilm dispersion protein −0.91 2.54E-07 RS18915 dctA C4-dicarboxylate transporter −1.10 5.37E-07 RS15710 kdpA potassium-transporting ATPase subunit −0.76 2.27E-06 RS16870 dctQ C4-dicarboxylate TRAP transporter small permease protein −1.17 3.35E-05 RS08465 ntrC two-component system response regulator −0.85 4.34E-05 RS15700 kdpC potassium-transporting ATPase subunit −0.91 6.18E-05 RS23460 opmH efflux transporter outer membrane subunit −0.63 1.78E-04 RS22785 aer2 aerotaxis transducer N-terminal domain −0.69 2.31E-03 表 6 后生元PostbioYDFF处理后铜绿假单胞菌与鞭毛装置有关的显著差异基因表达情况
Table 6 Expression of significant difference genes related to flagellar apparatus of P. aeruginosa after postbiotic PostbioYDFF treatment
基因编号 基因描述 TB vs TS log2FC P-value RS19775 flgE flagellar hook protein −1.15 3.66E-10 RS19680 fleR sigma-54-dependent response regulator −1.03 1.18E-08 RS19765 flgG flagellar basal-body rod protein −0.82 2.87E-06 RS19760 flgH flagellar basal body L-ring protein −0.78 3.30E-05 RS02820 fliO flagellar biosynthetic protein −0.83 1.06E-03 RS19670 fliF flagellar basal-body MS-ring/collar protein −0.50 1.39E-03 RS02785 flhA flagellar biosynthesis protein −0.52 2.24E-03 -
[1] ALIMI B A, LAWAL R, ODETUNDE O N. Food safety and microbiological hazards associated with retail meat at butchery outlets in north-central Nigeria[J]. Food Control,2022,139:109061. doi: 10.1016/j.foodcont.2022.109061
[2] AZAM M W, KHAN A U. Updates on the pathogenicity status of Pseudomonas aeruginosa[J]. Drug Discovery Today,2019,24(1):350−359. doi: 10.1016/j.drudis.2018.07.003
[3] 杜峰, 谭文彬. 铜绿假单胞菌耐药机制的研究进展[J]. 中国病原生物学杂志,2023,18(10):1231−1234,1241. [DU Feng, TAN Wenbin. Advances in the study of drug resistance mechanism of Pseudomonas aeruginosa[J]. Journal of Pathogen Biology,2023,18(10):1231−1234,1241.] DU Feng, TAN Wenbin. Advances in the study of drug resistance mechanism of Pseudomonas aeruginosa[J]. Journal of Pathogen Biology, 2023, 18(10): 1231−1234,1241.
[4] LIANG J L, HUANG T Y, LI X J, et al. Germicidal effect of intense pulsed light on Pseudomonas aeruginosa in food processing[J]. Frontiers in Microbiology,2023,14:1247364. doi: 10.3389/fmicb.2023.1247364
[5] TENG X, ZHANG M, MUJUMDAR A S, et al. Inhibition of nitrite in prepared dish of Brassica chinensis L. during storage via non-extractable phenols in hawthorn pomace:A comparison of different extraction methods[J]. Food Chemistry,2022,393:133344. doi: 10.1016/j.foodchem.2022.133344
[6] 孙瑶, 乔梦伟, 刘诗宇, 等. 乳杆菌对致病假单胞菌的抑制作用研究进展[J]. 中国生物工程杂志,2021,41(8):103−109. [SUN Yao, QIAO Mengwei, LIU Shiyu, et al. Research progress on the inhibitory effect of Lactobacillus on pathogenic pseudomonas[J]. China Biotechnology,2021,41(8):103−109.] SUN Yao, QIAO Mengwei, LIU Shiyu, et al. Research progress on the inhibitory effect of Lactobacillus on pathogenic pseudomonas[J]. China Biotechnology, 2021, 41(8): 103−109.
[7] HOSSEN T S, KYEONGJUN K, MD A, et al. Korean kimchi-derived lactic acid bacteria inhibit foodborne pathogenic biofilm growth on seafood and food processing surface materials[J]. Food Control,2021,129:108276. doi: 10.1016/j.foodcont.2021.108276
[8] SEVAL A H, BUKET K. Antimicrobial activity of a bacteriocin produced by Enterococcus faecalis KT11 against some pathogens and antibiotic-resistant bacteria[J]. Korean Journal for Food Science of Animal Resources,2018,38(5):1064−1079. doi: 10.5851/kosfa.2018.e40
[9] IQBAL H M, RAHAMAN M M F, KUMAR R P, et al. Listeria monocytogenes biofilm inhibition on food contact surfaces by application of postbiotics from Lactobacillus curvatus B.67 and Lactobacillus plantarum M.2[J]. Food Research International,2021,148:110595−110595. doi: 10.1016/j.foodres.2021.110595
[10] SEPPO S, CARMEN C M, AKIHITO E, et al. The international scientific association of probiotics and prebiotics (ISAPP) consensus statement on the definition and scope of postbiotics[J]. Nature Reviews. Gastroenterology & Hepatology,2021,18(9):649−667.
[11] COLOMBO P T, GOMES C A, ELIENE P, et al. Postbiotics:An overview of concepts, inactivation technologies, health effects, and driver trends[J]. Trends in Food Science & Technology,2023,138:199−214.
[12] SAMIR D M, LONGXIN Q, A. T M, et al. Health benefits of postbiotics produced by E. coli Nissle 1917 in functional yogurt enriched with cape gooseberry (Physalis peruviana L.)[J]. Fermentation,2022,8(3):128−128. doi: 10.3390/fermentation8030128
[13] HOSSEN T S, JUN-HA P, KIM K, et al. Antibiofilm efficacy of Leuconostoc mesenteroides J.27-derived postbiotic and food-grade essential oils against Vibrio parahaemolyticus, Pseudomonas aeruginosa, and Escherichia coli alone and in combination, and their application as a green preservative in the seafood industry[J]. Food Research International,2022,156:111163−111163. doi: 10.1016/j.foodres.2022.111163
[14] PUCCETTI M, XROUDAKI, S, RICCI M, et al. Postbiotic-enabled targeting of the host-microbiota-pathogen interface:Hints of antibiotic decline?[J]. Pharmaceutics,2020,12(7):624. doi: 10.3390/pharmaceutics12070624
[15] LV X R, YANG L, YU J, et al. Purification, characterization, and action mechanism of Plantaricin DL3, a novel bacteriocin against Pseudomonas aeruginosa produced by Lactobacillus plantarum DL3 from Chinese Suan-Tsai[J]. European Food Research and Technology,2018,244(2):323−331.
[16] DA-HYE L, SUN K B, SEOK-SEONG K. Bacteriocin of Pediococcus acidilactici HW01 inhibits biofilm formation and virulence factor production by Pseudomonas aeruginosa[J]. Probiotics and Antimicrobial Proteins,2020,12(1):73−81. doi: 10.1007/s12602-019-09623-9
[17] MARTINS D M, PAULA T A, ELISABETH N, et al. Cell-free supernatant of probiotic bacteria exerted antibiofilm and antibacterial activities against Pseudomonas aeruginosa:A novel biotic therapy[J]. Frontiers in Pharmacology,2023,14:1152588. doi: 10.3389/fphar.2023.1152588
[18] REZAEI Z, KHANZADI S, SALARI A. Biofilm formation and antagonistic activity of Lacticaseibacillus rhamnosus (PTCC1712) and Lactiplantibacillus plantarum (PTCC1745)[J]. Amb Express,2021,11:1−7. doi: 10.1186/s13568-020-01157-6
[19] WIJESUNDARA N M, RUPASINGHE H P V. Bactericidal and anti-bofilm activity of ethanol extracts derived from selected medicinal plants against Streptococcus pyogenes[J]. Molecules,2019,24(6):1165. doi: 10.3390/molecules24061165
[20] YE Zuoji, YE Liumei, LI Dingbin, et al. Effects of daphnetin on biofilm formation and motility of Pseudomonas aeruginosa[J]. Frontiers in Cellular and Infection Microbiology,2022,12:1033540. doi: 10.3389/fcimb.2022.1033540
[21] 高毅敏, 李娜, 李新鸣, 等. 血链球菌cpnp培养上清液抑制铜绿假单胞菌生长和生物膜的作用[J]. 中国微生态学杂志,2024,36(6):637−646,656. [GAO Yimin, LI Na, LI Xinming, et al. Inhibition of growth and biofilm of Pseudomonas aeruginosa by Streptococcus sanguinis cpnp culture supernatant[J]. Chinese Journal of Microecology,2024,36(6):637−646,656.] GAO Yimin, LI Na, LI Xinming, et al. Inhibition of growth and biofilm of Pseudomonas aeruginosa by Streptococcus sanguinis cpnp culture supernatant[J]. Chinese Journal of Microecology, 2024, 36(6): 637−646,656.
[22] 王嘉康, 唐浩国, 陈静, 等. 水飞蓟素对S. aureus ATCC25923生物膜及其毒力因子的影响[J]. 食品与机械,2024,40(3):26−32,43. [WANG Jiakang, TANG Haoguo, CHEN Jing, et al. Effects of silymarin on S. aureus ATCC25923 biofilm and its virulence factors[J]. Food and Machinery,2024,40(3):26−32,43.] WANG Jiakang, TANG Haoguo, CHEN Jing, et al. Effects of silymarin on S. aureus ATCC25923 biofilm and its virulence factors[J]. Food and Machinery, 2024, 40(3): 26−32,43.
[23] YA W N, MA M, ZHANG Y J, et al. Antibacterial mechanism of sucrose laurate against Bacillus cereus by attacking multiple targets and its application in milk beverage[J]. Food Research International,2022,154:111018−111018. doi: 10.1016/j.foodres.2022.111018
[24] 林梦娇, 陈亚东, 刘洋, 等. 半滑舌鳎vstm2a基因的响应细菌感染的表达模式和功能的研究[J]. 上海海洋大学学报,2024,33(6):1299−1312. [LIN Mengjiao, CHEN Yadong, LIU Yang, et al. The expression pattern and function of vstm2a gene in response to bacterial infection in Cynoglossus semilaevis[J]. Journal of Shanghai Ocean University,2024,33(6):1299−1312.] LIN Mengjiao, CHEN Yadong, LIU Yang, et al. The expression pattern and function of vstm2a gene in response to bacterial infection in Cynoglossus semilaevis[J]. Journal of Shanghai Ocean University, 2024, 33(6): 1299−1312.
[25] JIAHANG Z, ZHIWEI Z, WEN L, et al. Transcriptome analysis revealed a positive role of ethephon on chlorophyll metabolism of Zoysia japonica under cold stress[J]. Plants,2022,11(3):442−442. doi: 10.3390/plants11030442
[26] CRISTIANO-FAJARDO S A, FLORES C, FLORES N, et al. Glucose limitation and glucose uptake rate determines metabolite production and sporulation in high cell density continuous cultures of Bacillus amyloliquefaciens 83[J]. Journal of Biotechnology,2019,299:57−65. doi: 10.1016/j.jbiotec.2019.04.027
[27] WU M, LU T, FU J P, et al. Antibacterial mechanism of protocatechuic acid against Yersinia enterocolitica and its application in pork[J]. Food Control,2022,133:108573. doi: 10.1016/j.foodcont.2021.108573
[28] DUPORT C, ZIGHA A, ROSENFELD E, et al. Control of enterotoxin gene expression in Bacillus cereus F4430/73 involves the redox-sensitive ResDE signal transduction system[J]. Journal of Bacteriology,2007,189(6):2581−2581. doi: 10.1128/JB.00066-07
[29] JESSICA A, SARA O, SUMANA V, et al. Studying acetylation of aconitase isozymes by genetic code expansion[J]. Frontiers in Chemistry,2022,10:862483−862483. doi: 10.3389/fchem.2022.862483
[30] IRMAK B T, DIDEM B, BARIŞ G, et al. Extremophilic Natrinema versiforme against Pseudomonas aeruginosa quorum sensing and biofilm[J]. Frontiers in Microbiology,2020,11:79. doi: 10.3389/fmicb.2020.00079
[31] JIA S, HONG H, YANG Q, et al. TMT-based proteomic analysis of the fish-borne spoiler Pseudomonas psychrophila subjected to chitosan oligosaccharides in fish juice system[J]. Food Microbiology,2020,90(prepublish):103494.
[32] MARIA S, REKHA A, KYU K K. Roles of two-component systems in Pseudomonas aeruginosa virulence[J]. International Journal of Molecular Sciences,2021,22(22):12152. doi: 10.3390/ijms222212152
[33] GELLATLY S L, BAINS M, BREIDENSTEIN E B M, et al. Novel roles for two-component regulatory systems in cytotoxicity and virulence-related properties in Pseudomonas aeruginosa[J]. AIMS Microbiology,2018,4(1):173−191. doi: 10.3934/microbiol.2018.1.173
[34] PAPENFORT K, BASSLER L. Quorum sensing signal-response systems in Gram-negative bacteria[J]. Nature Reviews. Microbiology,2016,14(9):576−588. doi: 10.1038/nrmicro.2016.89
[35] TSIRY R, QUENTIN L, PIERRE D, et al. The formation of biofilms by Pseudomonas aeruginosa:A review of the natural and synthetic compounds interfering with control mechanisms[J]. BioMed Research International,2015,2015:759348.
[36] TIAN M J, WU Z Y ZHANG R J, et al. A new mode of swimming in singly flagellated Pseudomonas aeruginosa[J]. Proceedings of the National Academy of Sciences of the United States of America,2022,119(14):e2120508119.
[37] PEJČIĆ M, STOJANOVIĆ-RADIĆ Z, GENČIĆ M, et al. Anti-virulence potential of basil and sage essential oils:Inhibition of biofilm formation, motility and pyocyanin production of Pseudomonas aeruginosa isolates[J]. Food and Chemical Toxicology,2020,141(prepublish):111431.
[38] 李交昆, 南美花, 吴学玲, 等. 细菌鞭毛在生理活动中的作用[J]. 生命科学,2018,30(6):673−679. [LI Jiaokun, NAN Meihua, WU Xueling, et al. The functions of bacterial flagella in physiological activity[J]. Chinese Bulletin of Life Sciences,2018,30(6):673−679.] LI Jiaokun, NAN Meihua, WU Xueling, et al. The functions of bacterial flagella in physiological activity[J]. Chinese Bulletin of Life Sciences, 2018, 30(6): 673−679.
[39] WASSERMANN T, MEINIKE J K, IVANYSHYN K, et al. The phenotypic evolution of Pseudomonas aeruginosa populations changes in the presence of subinhibitory concentrations of ciprofloxacin[J]. Microbiology (Reading, England),2016,162(5):865−875. doi: 10.1099/mic.0.000273





 下载:
下载:
 下载:
下载:



