Ancestral Sequence Reconstruction Enhances Thermal Stability of D-Allulose 3-Epimerase
-
摘要: 为解决现有D-阿洛酮糖3-差向异构酶(DAEase)热稳定性差的产业问题,本文采用系统发育指导的大数据挖掘、合理修饰和祖先序列重建策略(ASR),重建了具有不同催化结构域DAEase的祖先序列,构建了表达载体,通过重组表达与分子对接筛选出了DAEase A13并进行酶学性质表征,此外,还基于结构分析与分子动力学模拟揭示了DAEase A13热稳定性增强的分子机制。结果表明,基于ASR策略所构建的A13 70 ℃时半衰期可达8.4 h,其热稳定性较野生(WT)酶显著增强,最大转化率为31%,催化活性也略高于WT酶。立体结构模拟与分子动力学模拟揭示了ASR A13中大量氢键和疏水作用的增加维持了高温下酶分子结构的稳定性,是其热稳定性增强的主要因素。研究结果证实了ASR策略可以改造DAEase使其稳定性、活性和混杂性增强,可以为D-阿洛酮糖工业生产提供良好的生物催化剂。
-
关键词:
- 祖先序列重建 /
- D-阿洛酮糖 /
- D-阿洛酮糖3-差向异构酶 /
- 热稳定性
Abstract: To solve the problem of poor thermal stability of the current D-allulose 3-epimerase (DAEase), the ancestor sequences of DAEase with different catalytic domains were reconstructed by big data mining, reasonable modification and ancestor sequence reconstruction (ASR) strategy under the guidance of phylogenetic information. The expression vectors of the ancestor sequences were constructed, and DAEase A13 with significantly enhanced thermal stability was screened by recombinant expression and molecular docking, and its enzymatic properties were characterized. In addition, the molecular mechanism of thermal stability enhancement of DAEase A13 was revealed based on structural analysis and molecular dynamics. The results showed that the half-life of A13 constructed based on ASR strategy could reach 8.4 h at 70 ℃, indicating that its thermal stability was significantly enhanced compared with that of wild-type (WT) enzyme. The maximum conversion rate of A13 reached 31%, indicating that the catalytic activity of A13 was slightly higher than that of WT enzyme. The structural and molecular dynamics analysis revealed that the increase in hydrogen bonding and hydrophobic interaction in ASR A13 was the main factor responsible for maintaining the stability of the enzyme's molecular structure at high temperatures. The results showed that ASR strategy could modify DAEases to enhance the stability, activity or hybridity, which could provide superior biocatalyst sources for various industrial applications of functional sugars. -
祖先序列重建技术(Ancestral sequence reconstruction,ASR)是一种允许从同源野生型基因的集合推断“祖先”基因的方法,能够为解析生物酶结构与功能关系的演化过程提供新思路[1−3]。ASR通过使用已有蛋白质序列的数据集进行多重序列比对来构建系统发育树,基于精确的系统发育分析开发的算法来推断目标序列的祖先序列[2,4]。ASR产生的祖先生物酶通常具有更高的稳定性[5]或更广的底物谱[6],在一些情况下可以两者同时实现[7−9]。Joho等[8]成功应用ASR获得了热稳定性和催化活性显著提高的分解PET塑料的PET酶。Gumulya等[9]通过ASR技术重建了底物谱扩大且稳定性显著增强的新型P450酶。与传统方式相比,ASR具有多重优势,包括突变的随机性足够大且远离活性位点[10]、筛选范围更小[11−13]以及重构过程并不依赖目标酶的详细结构信息[14−16]。目前,尚未有将ASR应用于差向异构酶的改造的相关报道。
D-阿洛酮糖是一种公认安全的稀有糖,其相对甜度可以达到蔗糖的70%,但热量仅为0.2 kcal/g[17−18]。此外,D-阿洛酮糖具有多种生理作用,如抗肥胖、抗高血糖、抗糖尿病、抗氧化和神经保护作用[19−21]。因此,D-阿洛酮糖在食品和医药等行业具有重要的应用价值[22−23]。D-阿洛酮糖3-差向异构酶(DAEase)是基于Izumoring策略由D-果糖生物合成D-阿洛酮糖的关键酶,其催化最适温度为55~65 ℃[24−26]。目前所报道的DAEases大多具有较高的底物特异性和催化活性,但热稳定性较差,无法在实际生产的工业条件下应用[27]。Zhang等[28]筛选了来源于Treponema primitia ZAS-1的DAEase(TpDAE),它在70 ℃条件下催化500 g/L D-果糖转化D-阿洛酮糖的产率为137.5 g/L,转化率为27.5%,但孵育1 h后活性完全丧失。此外,来源于Clostridium cellulolyticum的DAEase在60 ℃条件下的半衰期仅为10 min[29],来源于Christensenellaceae.minuta的DAEase在50 ℃条件下半衰期为40 min[30]。因此,获得高催化活性和热稳定性的DAEase是当前该领域的研究热点。
本研究基于具有较高催化活性的TpDAE蛋白序列开展祖先序列重建策略研究,重建了具有不同催化结构域的祖先序列。此后,通过基因合成获得了序列重建的DAEase(A1和A13)基因序列,构建了重组表达质粒与大肠杆菌重组表达菌株,对重建后的DAEase进行纯化与表征,其中A13较野生(WT)酶热稳定性显著增强。基于结构分析与分子动力学模拟实验探索了DAEase稳定性和催化活性增强的内在机制。以上研究表明,祖先序列重建策略可以筛选出具有优良性能的差向异构酶,能为具有新特性的稀有糖生物转化酶的半理性工程设计提供新思路。
1. 材料与方法
1.1 材料与仪器
Escherichia coli JM109、Escherichia coli BL21 (DE3)、pET22b(+) 均为实验室保存;Nde I、Xho I 大连宝生物Takara公司;质粒小提试剂盒、小量DNA胶纯化回收试剂盒 Omega Bio-Tek公司;Luria-Bertani(LB)培养基:准确称取酵母浸粉5 g,蛋白胨10 g和NaCl 10 g,溶于1000 mL 蒸馏水中,121 ℃高压灭菌20 min;所有化学品均为分析级 均来自Sigma-Aldric公司或生工生物工程(上海)股份有限公司。
HYG-II型回旋式恒温调速摇床柜 上海欣蕊自动化设备有限公司;DYCZ-24D型核酸电泳仪 北京市六一仪器厂;Agilent 1260型高效液相色谱仪 上海莱睿科学仪器有限公司;Agilent 1260 Infinity蒸发光散射检测器 安捷伦科技有限公司;碳水化合物ES柱-w(5 μm,4.6×250 mm) 赛默飞世尔科技公司。
1.2 实验方法
1.2.1 D-阿洛酮糖3-差向异构酶的系统发育推断与祖先序列重建
本研究利用FireProtASR(https://loschmidt.chemi.muni.cz/fireprotasr)进行系统发育推断和祖先序列重建。将TpDAE的氨基酸序列输入FireProtASR进行分析,FireProtASR通过SwissProt和Catalytic Site Atlas对DAEase的氨基酸序列进行保守性评估,其Y6、E152、E158、D185、H211、R217和E246催化残基选为保守区域。FireProtASR首先利用PSI-BLAST对NCBI数据库的DAEase进行筛选以获取其同源性序列;其次通过ClustalΩ对同源序列进行多序列比对并利用IQTREE软件包推断其系统发育树;然后将多序列比对结果和建立的系统发育树输入RAxML算法进一步扩展其系统发育树;最后将系统发育树和多序列比对结果导入到使用Lazarus方法的PALM软件包中以重建其祖先序列。FireprotASR算法采用局部加权回归共识法处理误差,其进行50次自举以验证系统进化树,引导程序输出值的范围为5~100,大多数ASR节点的引导值大于70,表明节点的置信度较高。采用Discovery Studio 2019对重建的ASR DAEase进行分子对接实验。
1.2.2 A1和A13的克隆过表达和蛋白纯化
祖先序列重建后的DAEase及其后代的编码基因由金唯智生物科技有限公司合成,插入pET22b(+)载体的Nde I和Xho I限制性内切酶位点之间构建重组载体,测序验证后备用。将重组质粒转入E. coli BL21(DE3)中,接种于含有50 μg/mL卡那霉素的LB固体培养基筛选阳性转化子。
从平板上挑取长势良好的阳性转化子接种至含有50 μg/mL卡那霉素的200 mL LB培养基,37 ℃ 220 r/min条件下培养。当菌体密度(OD600)达到0.6~0.8时,加入0.2 mmol/L异丙基硫代半乳糖苷(IPTG)诱导其蛋白表达。在16 ℃ 110 r/min下进一步培养12 h,使DAEase缓慢表达。在4 ℃ 5000 r/min条件下离心15 min收集菌体,用0.85%生理盐水洗涤两次。
细胞在Lysis buffer(50 mmol/L Tris-HCl,10 mmol/L咪唑,500 mmol/L NaCl和1 mmol/L DTT,pH7.5)中超声破碎10 min,在4 ℃ 10000 r/min条件下离心30 min去除细胞碎片。将上清液与Ni-NTA树脂充分结合后加入重力柱,用10 mL Lysis buffer和Elution buffer(50 mmol/L Tris-HCl,500 mmol/L咪唑,500 mmol/L NaCl和1 mmol/L DTT,pH7.5)分别洗涤,Elution buffer洗脱后的流通液即为His标记的重组蛋白溶液。利用SDS-PAGE检验蛋白表达及纯化效果,并通过Nanodrop 2000测定纯化后的蛋白浓度,用于后续活性测定。
1.2.3 A13性质的表征
在40~80 ℃下反应确定温度对祖先序列重建后DAEase A13活性的影响。pH稳定性:在不同缓冲液(MES:pH5.5~7.0,PBS:7.0~8.0,Tris-HCl:8.0~9.0,CAPS:pH9.0~10.0)中孵育A13,通过1.2.4活性测定方法确定pH曲线以评估其pH稳定性。热稳定性:将纯化后的A13分别在50、60、70和80 ℃条件下孵育,在不同的时间间隔,从孵育混合物中提取适当的样品,采用1.2.4酶活性测定方法确定残余活性。半衰期:在最佳反应条件下,以未孵育处理的酶为空白对照,测定其残余活性。DAEase的失活符合一级动力学模型[31],所有反应均在三个独立实验中进行,其半衰期计算公式如下:
Kc=(2.0303/T)/lg(E0/E) t1/2=0.693/Kc 式中:Kc为衰减常数;E0为初始酶活性;E为T(温度)下DAEase的残余活性。
1.2.4 酶活性测定
反应液总体积为1.0 mL,含有1 μmol/L A13、50 g/L D-果糖和1 mmol/L CoCl2的Tris-HCl缓冲液(pH7.5)。将反应液在沸水中孵育10 min后终止反应,测定其活性。在此条件下,每分钟催化生成1 μmol D-阿洛酮糖的酶量被定义为一个酶活性单位。利用HPLC法测定A13在反应样品中产生的D-阿洛酮糖的量。
1.2.5 高效液相色谱(HPLC)法检测D-阿洛酮糖
测定酶促反应中由D-果糖转化为D-阿洛酮糖的量以测定酶的活性。使用碳水化合物ES柱-w(5 μm,4.6×250 mm)和1260 Infinity蒸发光散射检测器进行分离和检测,HPLC条件为:75%乙腈和25%去离子水作为流动相等度洗脱,进样量为10 μL,流速为1.0 mL/min,柱温为40 ℃,载气压力30 psi,漂移管温度为55 ℃[32]。测定重复三次。
1.2.6 分子动力学模拟
基于梭状芽孢杆菌(Clostridia bacterium)D-阿洛酮糖3-差向异构酶(PDB ID:7X7W)的晶体结构,使用SWISS-MODEL在线服务器(https://swissmodel.expasy.org/)建模,生成同源的TpDAE和A13的三维模型,并通过PyMoL(http://www.pymol.org)进行可视化分析。将预测的模型提交至SAVES在线服务器(https://saves.mbi.ucla.edu/),利用Ramachandran图对模型的3D结构参数进行评估。TpDAE和A13的氨基酸交互网络分析通过RING服务器(https://ring.biocomputingup.it/submit)进行,蛋白质表面疏水性分析使用在线网站Expasy(https://web.expasy.org/protscale)。
基于以上结构,开展分子动力学模拟分析,比较TpDAE及其祖先序列重建后A13的性质以阐明其热稳定性提高的分子机制。将系统的初始配置保持一致,使用AMBER 14软件包和AMBER99SB力场进行分子动力学(MD)模拟。最初的结构被放置在一个充满水分子的十二面体盒子,蛋白质分子与盒子边缘间隔为1 nm,利用Na+或Cl−中和系统的电荷。随后,为了消除潜在的不良接触,使整个系统能量最小化。在能量最小化后,整个系统在100 ps内从0逐渐加热到343 K(70 ℃)。最后,在Langevin算法控制的温度为343 K和压力为1.0 atm条件下进行30 ns的分子动力学模拟,时间步长为2 fs。
1.3 数据处理
所有实验数据均以3次实验结果的平均数±标准误差(mean±SE)表示。Origin 9.0软件进行绘图。
2. 结果与分析
2.1 D-阿洛酮糖3-差向异构酶的祖先序列重建
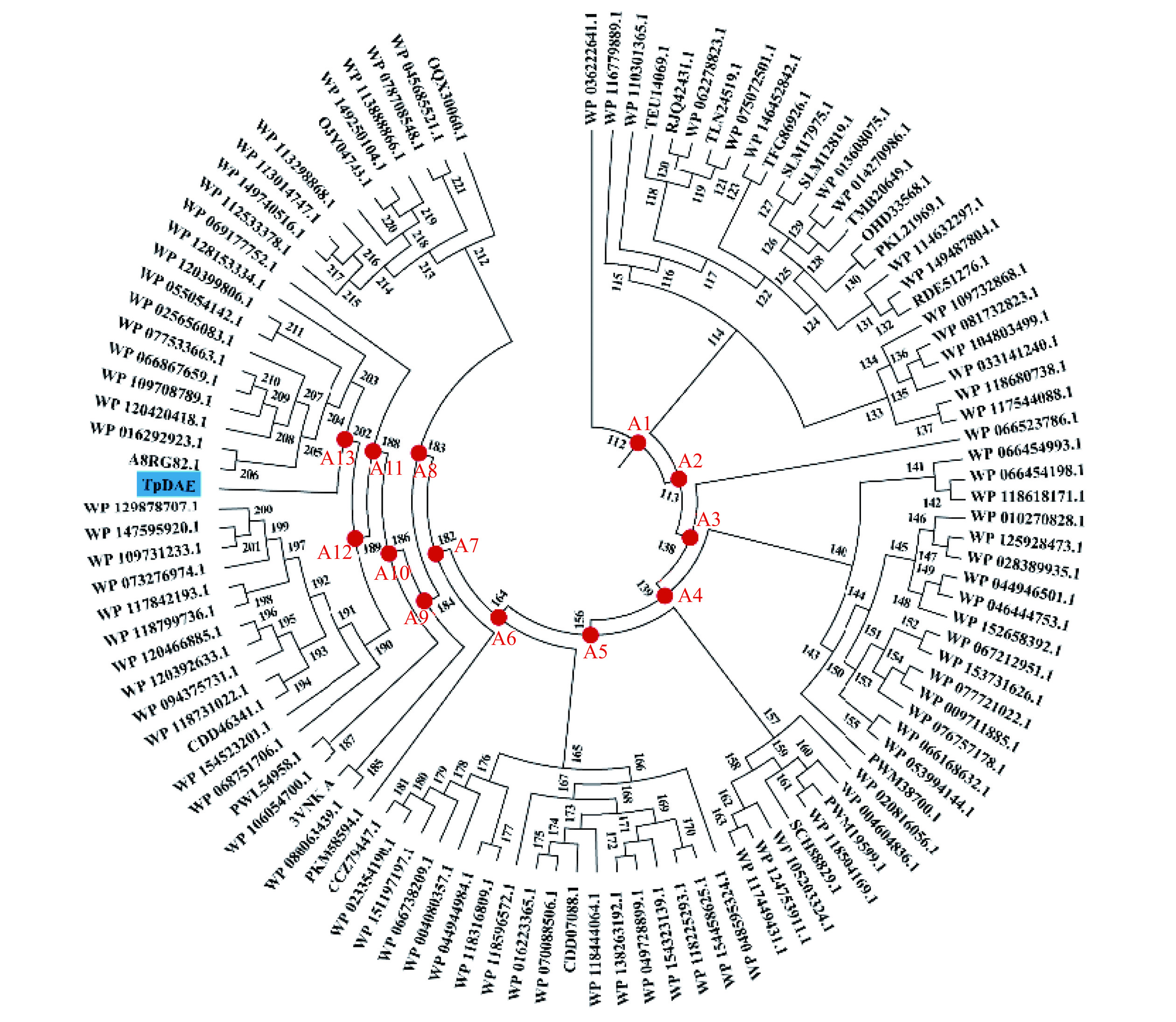
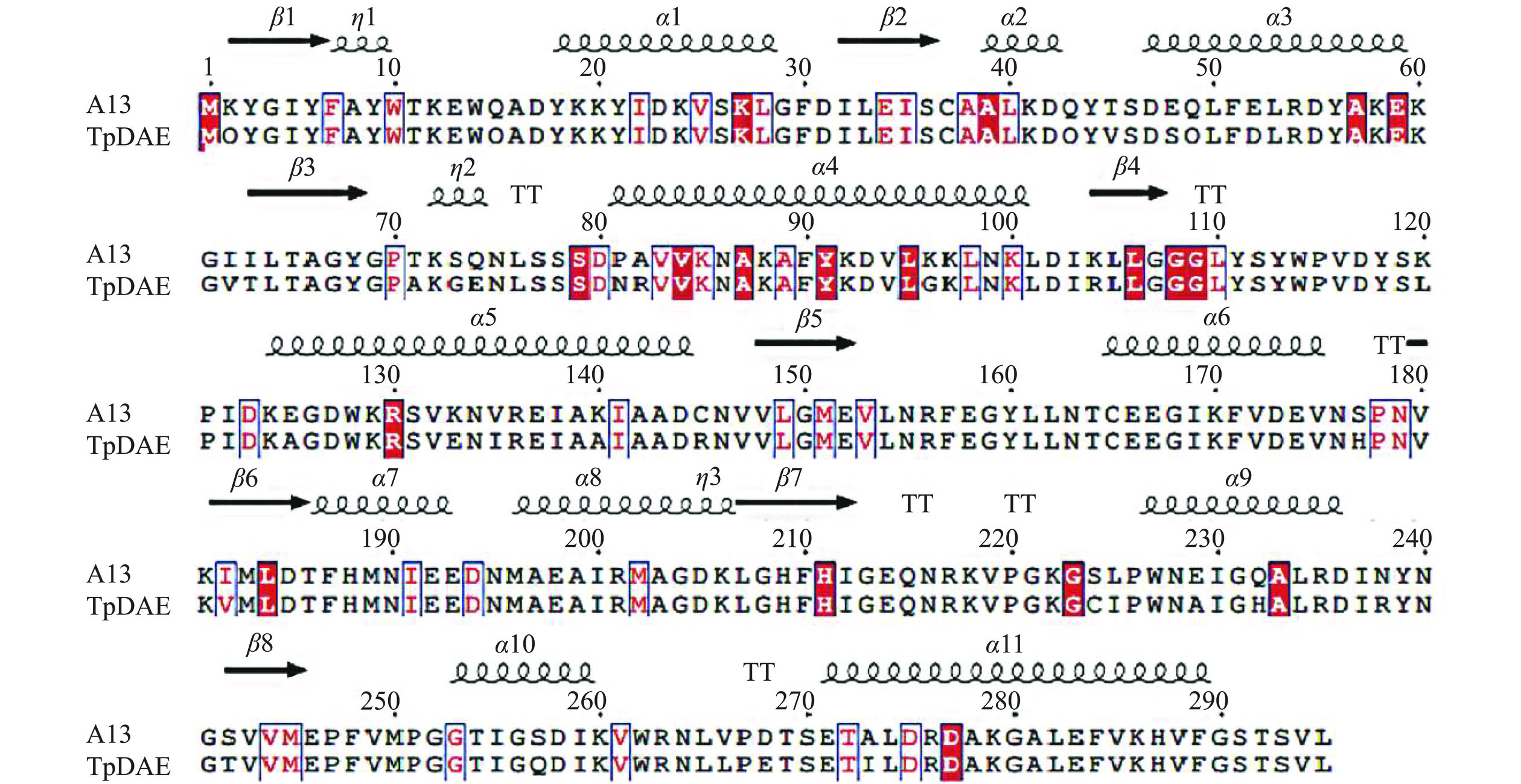
通过FireProtASR共检索出110个DAEase的同源序列,并在系统发育树中生成了110个ASR序列(图1)。祖先酶通常从系统发育树的节点中预测,将根节点的祖先DAEase指定为A1,一般认为A1是最好的祖先[9],其可能表现出最大的进化差异,因此本研究将A1作为候选。事实上,通过祖先序列重建不可能确定哪个序列在进化过程中是正确的,而只能推断最可能的祖先。在重建的基础上,对重建的节点ASR DAEase进行分子对接实验,以此作为预筛选过程后再对符合预期的ASR DAEase进行表征。分子对接实验中,A13的分子对接值(-CDOCKER_ENERGY)为8.15,与TpDAE的8.21接近,推测其具有与TpDAE相似的催化潜力,同样作为候选进行后续表征实验,祖先酶A13与野生酶TpDAE的序列差异共计30个位点,见表1。
表 1 祖先酶A13与野生酶TpDAE的序列差异Table 1. Sequence differences between A13 and TpDAE位点 2 45 48 52 61 62 71 73 74 81 82 96 104 120 125 A13 Q V S D V T A G E N R G R L A TpDAE K T E E I I T S Q P A K K K E 位点 133 135 140 145 177 182 224 225 229 232 238 242 257 268 273 A13 E I A R H V C I A H R T Q E I TpDAE K V K C S I S L E Q N S S D A 2.2 ASR DAEase的表达和纯化
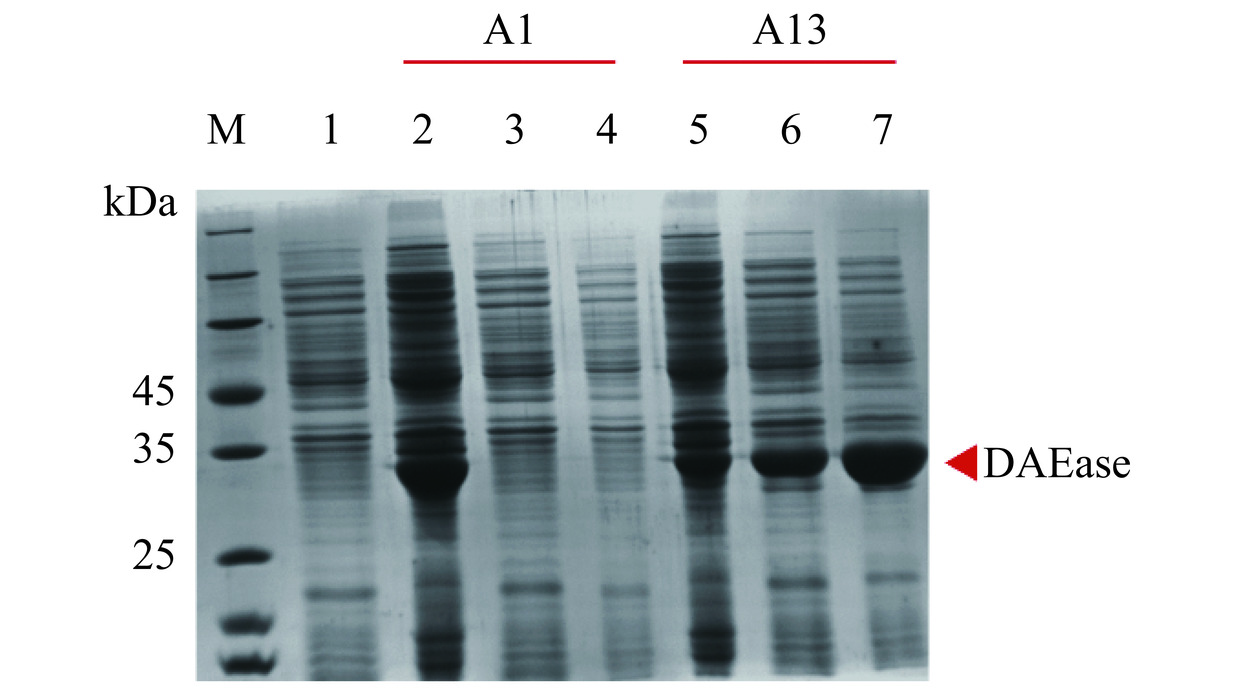
本研究利用pET22b(+)质粒在E. coli BL21(DE3)中成功表达了A1和A13,理论分子量均为33.56 kDa。重组蛋白在C端含有额外的His×6标签(HHHHHH),采用Ni-NTA亲和层析法纯化从菌体裂解物中纯化目标蛋白。A1在E. coli BL21(DE3)中虽然实现了重组表达,但几乎没有可溶性表达,这可能是由于重建后祖先酶具有特殊的折叠方式。A13在E. coli BL21(DE3)中成功实现了可溶性表达,纯化后的A13在SDS-PAGE凝胶上呈清晰的条带,表观分子质量约为35 kDa,但存在一些非特异性吸附杂蛋白(图2)。以上结果表明DAEase是可以直接从系统进化树中成功复活并实现可溶性表达的,因此选择具有可溶表达且具有高热稳定性潜力的A13进行各酶学性质的定量表征。
2.3 A13的酶学性质表征
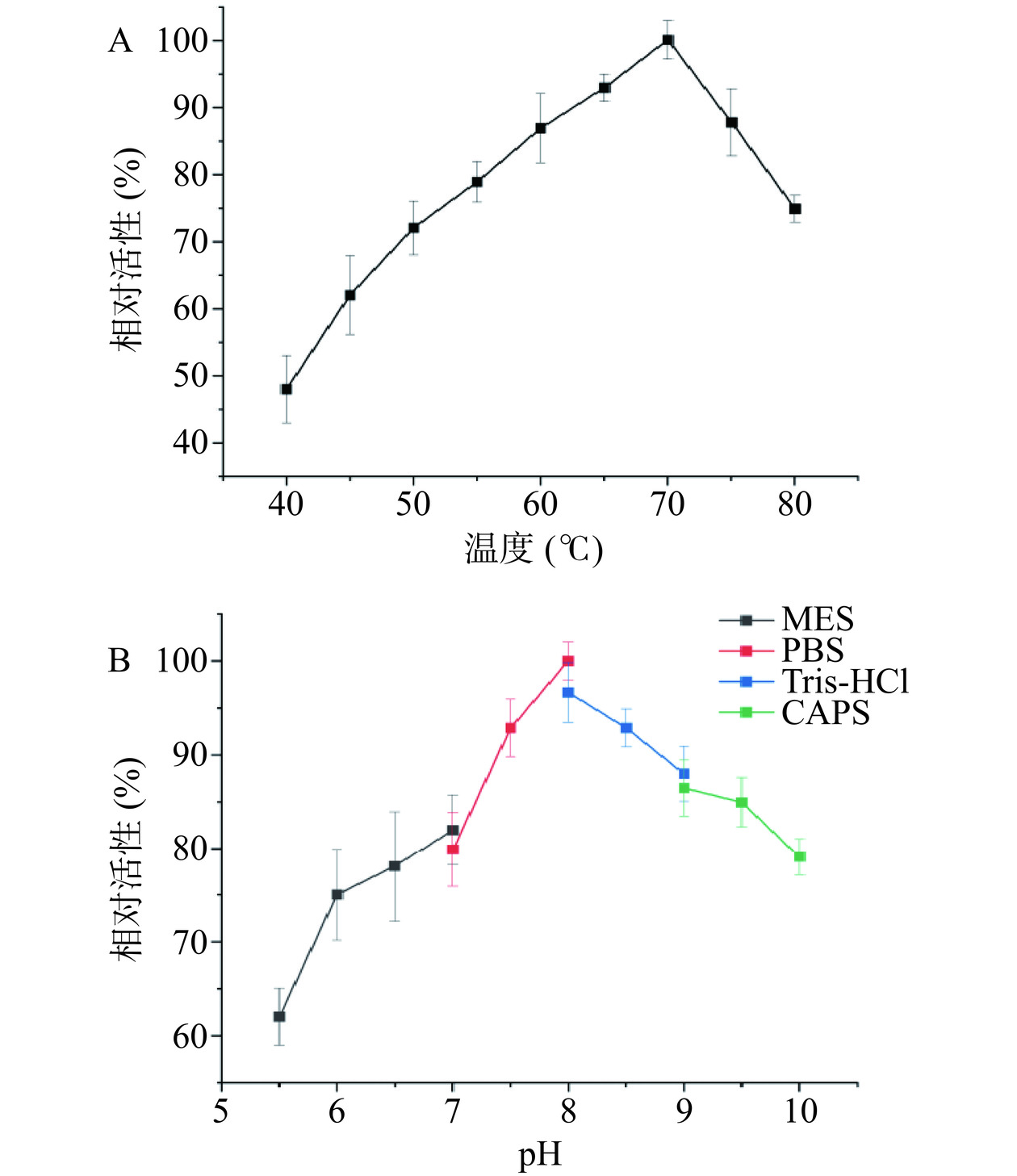
温度是影响酶催化反应的重要因素之一。一方面高温条件可以增加底物的溶解度,加速酶促反应进程,另一方面高温条件可以提高平衡转化率并降低微生物污染。包括稀有糖在内的工业生产通常需要生物酶制剂在相对较高的温度下能够保持较高的活性。本研究在40~80 ℃的温度范围内研究了温度对A13的活性影响,结果表明A13的最适温度为70 ℃(图3A),比活性为355.5 U/mg,相同条件下与TpDAE的最适温度和比活性基本一致[28]。A13在65~75 ℃之间具有很高活性,并在该温度范围内保持其最大活性的85%以上,在50~80 ℃的温度范围均显示超过70%相对活性,在此范围之外,相对活性急剧降低(图3A)。这些数据表明A13具有成为优质生物催化剂的潜力。图3B显示了pH对A13活性和稳定性的影响,在pH8.0的PBS缓冲液中,A13显示出最高活性,并在6.0~10.0的pH范围内超过70%相对活性。已报道的大多数DAEase在碱性条件下(pH7.5~9.0)表现出最佳活性,但酸性条件下碳水化合物的非酶褐变会减弱,可保证产品质量的稳定。A13在pH6.0时的相对活性为75%以上,在工业生产中具有很强的应用潜力。
2.4 A13的热稳定性与半衰期
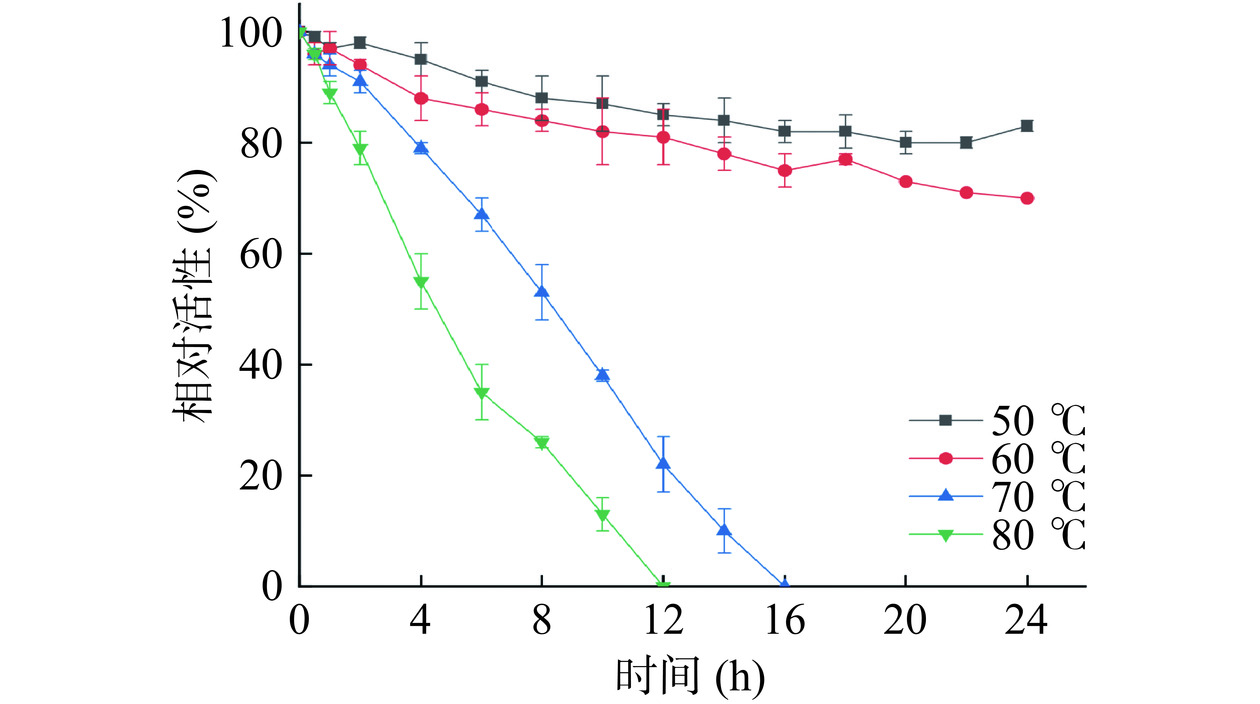
迄今为止报道的大多数DAEase的最适反应温度介于55~70 ℃之间,但在最适温度条件下的半衰期较短,难以满足工业生产需求[33]。如图4所示,A13在60 ℃以下是相对稳定的,在60 ℃孵育24 h后,其相对活性仍可保持在70%以上;在最适温度70 ℃条件下孵育4 h,可以保持80%的初始活性,表现出比现有的大多数DAEase具有更优异的热稳定性,这与ASR的预期结果一致。此外,本研究通过测定A13的半衰期对其热稳定性进行了表征,作为蛋白质耐热性指标,A13在其最适温度70 ℃条件下的半衰期为8.4 h,是TpDAE在相同温度下半衰期0.28 h的30倍,表明祖先序列重建后A13的热稳定性相较于WT酶显著增强。使用定向进化或蛋白质工程很难实现酶热稳定性的显著增强,但基于对酶分子非保守功能空间序列筛选重建的ASR在热稳定性增强方面具有广泛的应用前景[34−36]。
2.5 D-果糖向D-阿洛酮糖的生物转化
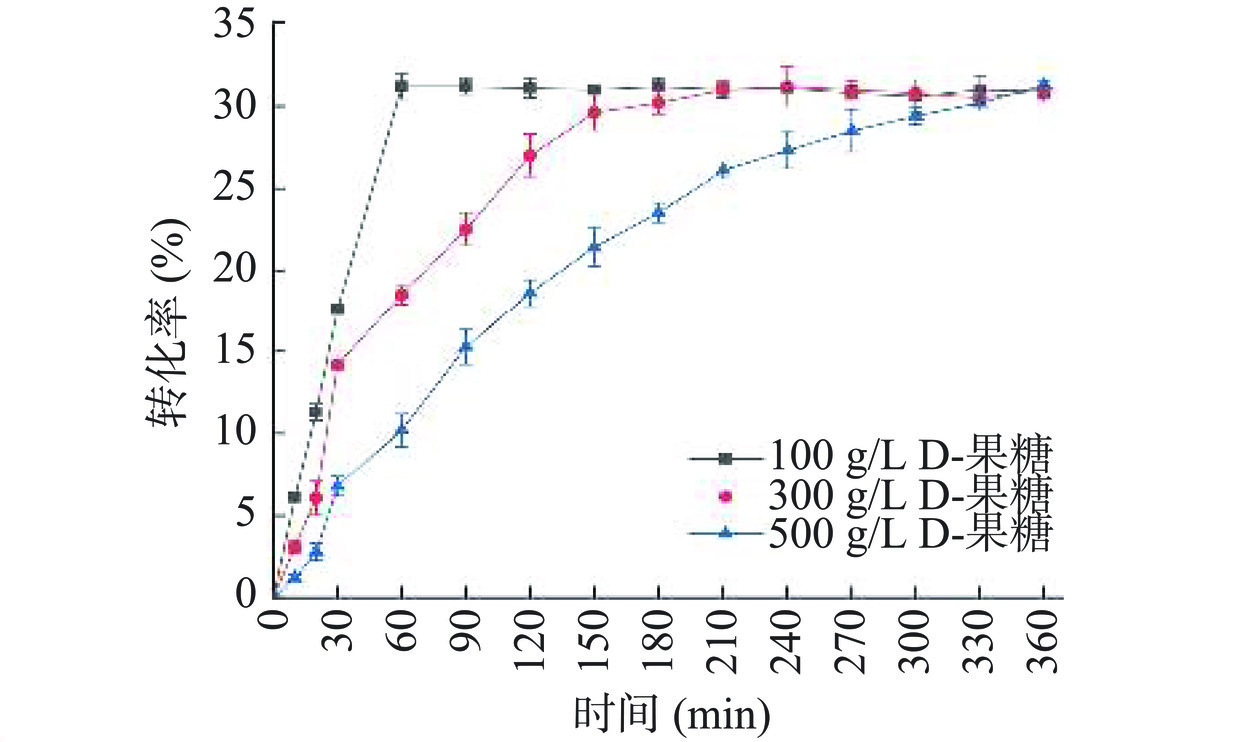
为了评估A13催化D-果糖生物转化为D-阿洛酮糖的能力,在500 mL含有1 μmol/L A13、D-果糖和1 mmol/L CoCl2的PBS缓冲液(50 mmol/L,pH8.0)中进行D-阿洛酮糖的酶促生产,D-果糖的浓度分别为100、300和500 g/L。如图5所示,其达到平衡所需的时间分别为60、210和360 min,最大转化率为31%(155 g/L),而TpDAE在相同条件下达到平衡所需的时间分别为60、180和360 min,最大转化率为27.5%,表明祖先序列重建后A13具有良好的催化活性。
2.6 TpDAE和A13的结构分析及分子动力学模拟
TpDAE的热稳定性较差,在70 ℃时半衰期仅为0.28 h[8],而祖先序列重建后A13的半衰期大幅提高,本研究通过立体结构分析及分子动力学模拟实验解析了A13的耐热性分子机制,模型质量分析全局得分为0.88,模型合理。基于RING服务器[37]分析的氨基酸交互网络结果显示TpDAE和A13的序列一致性为89.5%(图6),A13和TpDAE相互作用网络中涉及的氨基酸数均为295个,但涉及到的氨基酸残基以及作用方式存在很大差异。A13含有562个氢键和22个π-π堆叠,远超过TpDAE中的359个氢键和15个π-π堆叠,在高温下数量更多的氢键和π-π堆叠具有更强的分子间作用力,可以保持酶分子固有结构[38−39],因而A13较TpDAE具有良好的热稳定性。
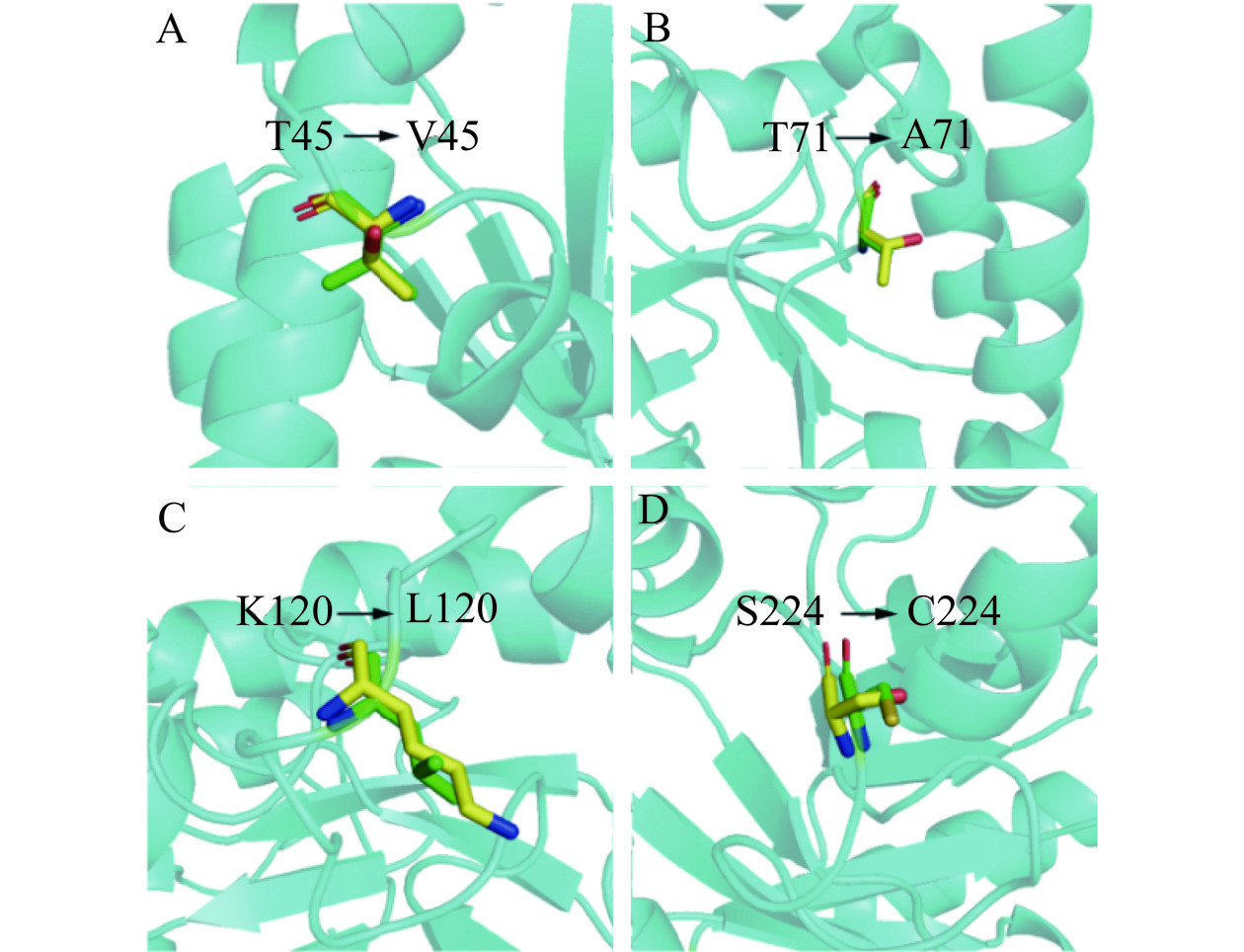
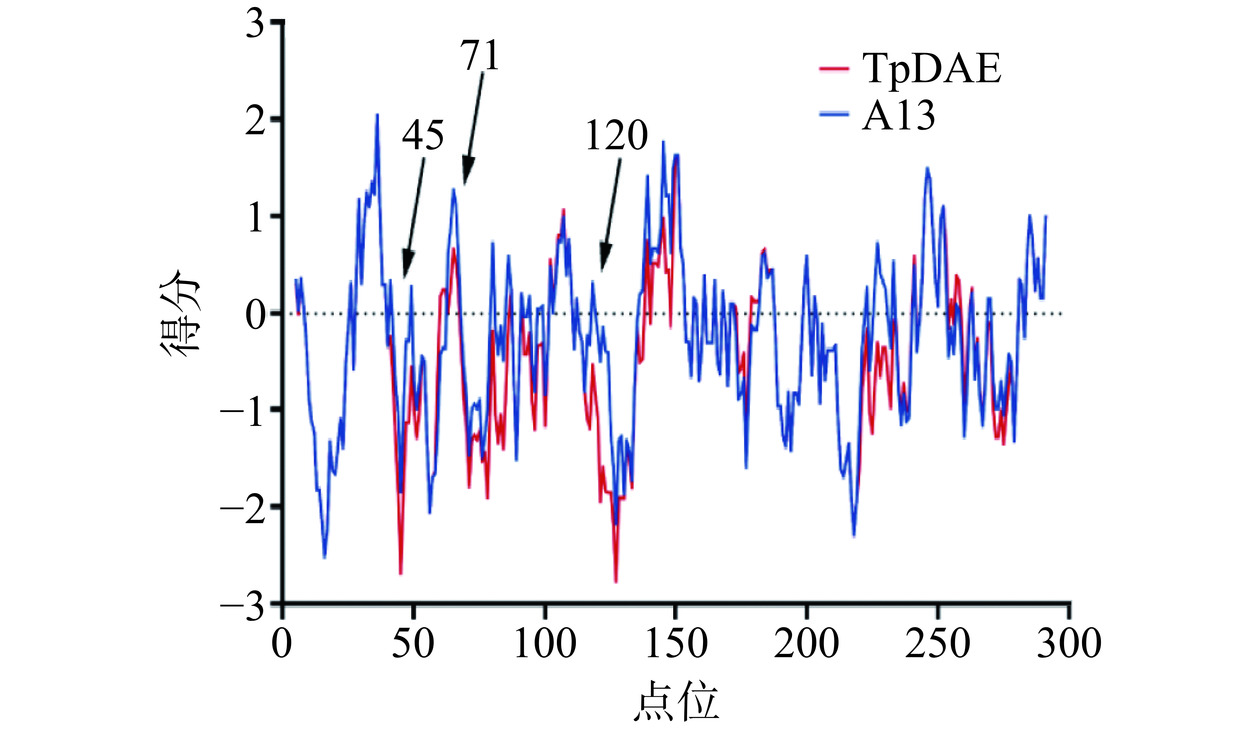
通过分析TpDAE和A13的结构发现,与TpDAE相比A13的loop结构中对热稳定性起到贡献的氨基酸残基数量更多。在TpDAE的224位的Ser变为A13中的Cys(图7D),推测Cys224参与蛋白质内二硫键的形成,通过减小蛋白质未折叠态的熵值来维持蛋白质的稳定[40]。在45号位、71号位和120号位的亲水性的Thr和Lys分别变为了疏水性的Val、Ala和Leu,这些疏水基团使得疏水相互作用增强,因而蛋白质抵抗热变性的能力就越强[41](图7A,B,C)。TpDAE和A13的蛋白质表面疏水性分析结果显示A13在45、71和120位附近的表面疏水性同TpDAE相比明显增加,且整体结构的疏水性也得到了增强(图8)。
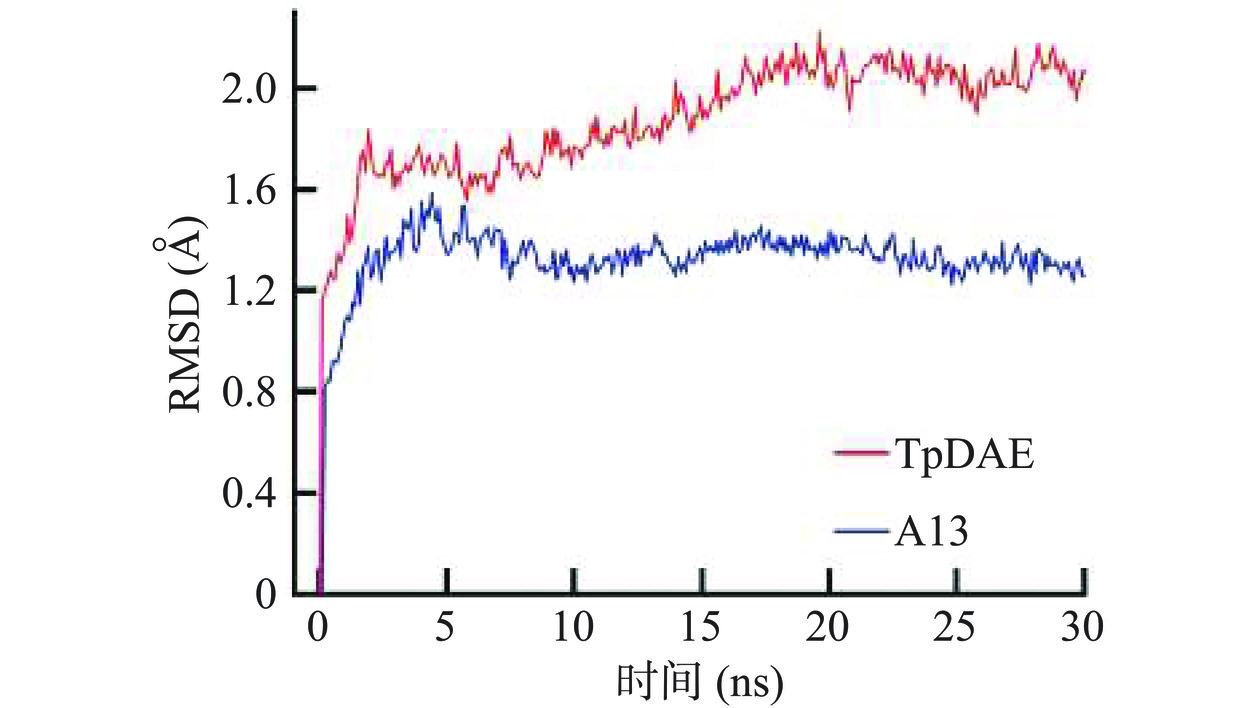
为了研究在高温催化条件下的A13与TpDAE构象变化,在70 ℃条件下对A13和TpDAE进行了30 ns的分子动力学模拟(图9)。均方根偏差(RMSD)是估计蛋白质构象热波动的一个重要参数,蛋白质的热稳定性与其RMSD值呈负相关,而RMSD值与蛋白质刚性呈正相关[42]。在前3 ns,TpDAE和A13的RMSD值迅速增加至1.8 Å和1.3 Å,此后A13的RMSD值稳定在1.2~1.6 Å的范围内直至模拟结束(t=30 ns)。在20 ns时,TpDAE的RMSD值达到了最高值2.1 Å,而在相同条件下,A13的RMSD值仍稳定在1.3 Å。分子模拟轨迹显示,在70 ℃条件下A13的平均RMSD值变化约为0.5 Å,变化平缓,表明其整体构象稳定,活性位点结构紧凑,热稳定性较好;TpDAE的平均RMSD值变化约为0.9 Å,变化幅度较大,表明其结构更为疏松,推测高温可能会导致其活性位点形变,使其酶活性降低甚至丧失。Wei等[43]通过综合计算辅助重新设计来提高用于D-阿洛糖合成的L-鼠李糖异构酶的热稳定性,所得组合突变体M2-4在75 ℃下的半衰期延长了5.7倍,分子动力学模拟研究表明在348 K时,M2-4的RMSD值变化较小,超过100 ns时的平均RMSD为0.17 Å,整体构象稳定,相比之下,WT的RMSD曲线在开始时急剧增加,60 ns后波动稳定,总体构象波动较大,平均RMSD为0.23 Å,引入M2-4的变异使柔性区域硬化,导致在高温下稳定折叠。本文分子动力学模拟结果解释了A13在高温下能表现出更强的稳定性催化性能的分子机制,证明了祖先序列重建策略的可靠性。
3. 结论
传统高性能的酶基因的挖掘受到筛选方式和样品发掘等因素的限制。ASR与其他工程方法不同,它基于对非保守功能空间的筛选来生成新序列,在给定准确的序列比对的前提下,许多输出都具有很高的功能性。本研究通过祖先序列重建获取的DAEase A13是一种很有前途的工业生物催化剂,其表现出与TpDAE相似的表达水平和催化活性,但热稳定性显著提高,证明了ASR策略的有效性与可行性,它可以改造出具有良好生物催化性能的酶分子。此外,祖先序列重建可能有助于识别调节酶活性的关键位点以避免改造的盲目性和实验工作量。本研究利用ASR充实了对新型DAEase生物催化剂的挖掘工作,实现了在DAEase新型酶基因挖掘技术上的探索。
-
表 1 祖先酶A13与野生酶TpDAE的序列差异
Table 1 Sequence differences between A13 and TpDAE
位点 2 45 48 52 61 62 71 73 74 81 82 96 104 120 125 A13 Q V S D V T A G E N R G R L A TpDAE K T E E I I T S Q P A K K K E 位点 133 135 140 145 177 182 224 225 229 232 238 242 257 268 273 A13 E I A R H V C I A H R T Q E I TpDAE K V K C S I S L E Q N S S D A -
[1] GUMULYA Y, GILLAM E M. Exploring the past and the future of protein evolution with ancestral sequence reconstruction:The 'retro' approach to protein engineering[J]. Biochemical Journal,2017,474(1):1−19. doi: 10.1042/BCJ20160507
[2] MERKL R, STERNER R. Ancestral protein reconstruction:techniques and applications[J]. Biological Chemistry,2016,397(1):1−21. doi: 10.1515/hsz-2015-0158
[3] SELBERG A G A, GAUCHER E A, LIBERLES D A. Ancestral sequence reconstruction:From chemical paleogenetics to maximum likelihood algorithms and beyond[J]. Journal of Molecular Evolution,2021,89(3):157−164. doi: 10.1007/s00239-021-09993-1
[4] RISSO V A, SANCHEZ-RUIZ J M, OZKAN S B. Biotechnological and protein-engineering implications of ancestral protein resurrection[J]. Current Opinion in Structural Biology,2018,51:106−115. doi: 10.1016/j.sbi.2018.02.007
[5] WHITFIELD J H, ZHANG W H, HERDE M K, et al. Construction of a robust and sensitive arginine biosensor through ancestral protein reconstruction[J]. Protein Science,2015,24(9):1412−1422. doi: 10.1002/pro.2721
[6] WILDING M, PEAT T S, KALYAANAMOORTHY S, et al. Reverse engineering:Transaminase biocatalyst development using ancestral sequence reconstruction[J]. Green Chemistry,2017,19:5375−5380. doi: 10.1039/C7GC02343J
[7] BABKOVA P, SEBESTOVA E, BREZOVSKY J, et al. Ancestral haloalkane dehalogenases show robustness and unique substrate specificity[J]. Chembiochemistry,2017,18(14):1448−1456. doi: 10.1002/cbic.201700197
[8] JOHO Y, VONGSOUTHI V, SPENCE M A, et al. Ancestral sequence reconstruction identifies structural changes underlying the evolution of Ideonella sakaiensis petase and variants with improved stability and activity[J]. Biochemistry,2023,62(2):437−450. doi: 10.1021/acs.biochem.2c00323
[9] GUMULYA Y, BAEK J M, WUN S J, et al. Engineering highly functional thermostable proteins using ancestral sequence reconstruction[J]. Nature Catalysis,2018,1:878−888. doi: 10.1038/s41929-018-0159-5
[10] SPENCE M A, KACZMARSKI J A, SAUNDERS J W, et al. Ancestral sequence reconstruction for protein engineers[J]. Current Opinion in Structural Biology,2021,69:131−141. doi: 10.1016/j.sbi.2021.04.001
[11] FOLEY G, MORA A, ROSS C M, et al. Engineering indel and substitution variants of diverse and ancient enzymes using graphical representation of ancestral sequence predictions (GRASP)[J]. PLoS Computational Biology,2022,18(10):e1010633. doi: 10.1371/journal.pcbi.1010633
[12] GOLDENZWEIG A, GOLDSMITH M, HILL S E, et al. Automated structure and sequence-based design of proteins for high bacterial expression and stability[J]. Molecular Cell,2016,63(2):337−346. doi: 10.1016/j.molcel.2016.06.012
[13] YANG Z. PAML 4:Phylogenetic analysis by maximum likelihood[J]. Molecular Biology and Evolution,2007,24(8):1586−1591. doi: 10.1093/molbev/msm088
[14] CHEN X, DOU Z, LUO T, et al. Directed reconstruction of a novel ancestral alcohol dehydrogenase featuring shifted pH-profile, enhanced thermostability and expanded substrate spectrum[J]. Bioresource Technology,2022,363:127886. doi: 10.1016/j.biortech.2022.127886
[15] PRAMANIK S, CONTRERAS F, DAVARI M D, et al. Protein engineering by efficient sequence space exploration through combination of directed evolution and computational design methodologies[J]. Protein Engineering,2021:153−176.
[16] YU H, DALBY P A. Coupled molecular dynamics mediate long- and short-range epistasis between mutations that affect stability and aggregation kinetics[J]. Proceedings of the National Academy of Sciences of the United States of America,2018,115(47):e11043−e11052.
[17] FUKADA K, ISHII T, TANAKA K, et al. Crystal structure, solubility, and mutarotation of the rare monosaccharide D-psicose[J]. Bulletin of the Chemical Society of Japan,2010,83:1193−1197. doi: 10.1246/bcsj.20100148
[18] MU W, ZHANG W, FENG Y, et al. Recent advances on applications and biotechnological production of D-psicose[J]. Applied Microbiology and Biotechnology,2012,94(6):1461−1467. doi: 10.1007/s00253-012-4093-1
[19] SHINTANI T, YAMADA T, HAYASHI N, et al. Rare sugar syrup containing D-allulose but not high-fructose corn syrup maintains glucose tolerance and insulin sensitivity partly via hepatic glucokinase translocation in wistar rats[J]. Journal of Agricultural and Food Chemistry,2017,65(13):2888−2894. doi: 10.1021/acs.jafc.6b05627
[20] ZHANG W, FANG D, XING Q, et al. Characterization of a novel metal-dependent D-psicose 3-epimerase from Clostridium scindens 35704[J]. PLoS One,2013,8(4):e62987. doi: 10.1371/journal.pone.0062987
[21] ZHANG W, YU S, ZHANG T, et al. Recent advances in D-allulose:Physiological functionalities, applications, and biological production[J]. Trends in Food Science and Technology,2016,54:127−137. doi: 10.1016/j.jpgs.2016.06.004
[22] KIM S E, KIM S J, KIM H J, et al. D-Psicose, a sugar substitute, suppresses body fat deposition by altering networks of inflammatory response and lipid metabolism in C57BL/6J-ob/ob mice[J]. Journal of Functional Foods,2017,28:265−274. doi: 10.1016/j.jff.2016.11.029
[23] SUNA S, YAMAGUCHI F, KIMURA S, et al. Preventive effect of D-psicose, one of rare ketohexoses, on di-(2-ethylhexyl) phthalate (DEHP)-induced testicular injury in rat[J]. Toxicology Letters,2007,173(2):107−117. doi: 10.1016/j.toxlet.2007.06.015
[24] IZUMORI K. Bioproduction strategies for rare hexose sugars[J]. Naturwissenschaften,2002,89(3):120−124. doi: 10.1007/s00114-002-0297-z
[25] KIM H J, HYUN E K, KIM Y S, et al. Characterization of an agrobacterium tumefaciens D-psicose 3-epimerase that converts D-fructose to D-psicose[J]. Applied and Environmental Microbiology,2006,72(2):981−985. doi: 10.1128/AEM.72.2.981-985.2006
[26] CHEN Z, GAO X D, LI Z. Recent advances regarding the physiological functions and biosynthesis of D-allulose[J]. Frontiers in Microbiology,2022,13:881037. doi: 10.3389/fmicb.2022.881037
[27] ZHANG W, WEI M, SUN X, et al. Fine-tuning of carbon flux and artificial promoters in Bacillus subtilis enables high-level biosynthesis of D-allulose[J]. Journal of Agricultural and Food Chemistry,2022,70(43):13935−13944. doi: 10.1021/acs.jafc.2c05585
[28] ZHANG W, ZHANG T, JIANG B, et al. Biochemical characterization of a D-psicose 3-epimerase from Treponema primitia ZAS-1 and its application on enzymatic production of D-psicose[J]. Journal of the Science of Food and Agriculture,2016,96(1):49−56. doi: 10.1002/jsfa.7187
[29] MU W, CHU F, XING Q, et al. Cloning, expression, and characterization of a D-psicose 3-epimerase from Clostridium cellulolyticum H10[J]. Journal of Agricultural and Food Chemistry,2011,59:7785−7792. doi: 10.1021/jf201356q
[30] WANG Y, RAVIKUMAR Y, ZHANG G, et al. Biocatalytic synthesis of D-allulose using novel D-tagatose 3-epimerase from Christensenella minuta[J]. Frontiers in Chemistry,2020,8:622325. doi: 10.3389/fchem.2020.622325
[31] SAKODA M, HIROMI K. Determination of the best-fit values of kinetic parameters of the Michaelis-Menten equation by the method of least squares with the Taylor expansion[J]. The Journal of Biochemistry,1976,80(3):547−555. doi: 10.1093/oxfordjournals.jbchem.a131310
[32] QI H, WANG T, LI H, et al. Sequence- and structure-based mining of thermostable D-allulose 3-epimerase and computer-guided protein engineering to improve enzyme activity[J]. Journal of Agricultural and Food Chemistry,2023,71(47):18431−18442. doi: 10.1021/acs.jafc.3c07204
[33] PATEL S N, KAUSHAL G, SINGH S P. A novel D-allulose 3-epimerase gene from the metagenome of a thermal aquatic habitat and D-allulose production by Bacillus subtilis whole-cell catalysis[J]. Applied and Environmental Microbiology,2020,86(5):e02605−19.
[34] CHEN S, XU Z, DING B, et al. Big data mining, rational modification, and ancestral sequence reconstruction inferred multiple xylose isomerases for biorefinery[J]. Science Advances,2023,9(5):1−16.
[35] THOMSON R E S, CARRERA-PACHECO S E, GILLAM E M J. Engineering functional thermostable proteins using ancestral sequence reconstruction[J]. Journal of Biological Chemistry,2022:102435.
[36] LIVADA J, VARGAS A M, MARTINEZ C A, et al. Ancestral sequence reconstruction enhances gene mining efforts for industrial ene reductases by expanding enzyme panels with thermostable catalysts[J]. ACS Catalysis,2023,13(4):2576−2585. doi: 10.1021/acscatal.2c03859
[37] PIOVESAN D, MINERVINI G, TOSATTO S C. The RING 2.0 web server for high quality residue interaction networks[J]. Nucleic Acids Research,2016,44(W1):W367−374. doi: 10.1093/nar/gkw315
[38] TOMPA D R, GROMIHA M M, SARABOJI K. Contribution of main chain and side chain atoms and their locations to the stability of thermophilic proteins[J]. Journal of Molecular Graphics & Modelling,2016,64:85−93.
[39] WANG Q, CEN Z, ZHAO J. The survival mechanisms of thermophiles at high temperatures:An angle of omics[J]. Physiology (Bethesda),2015,30(2):97−106. doi: 10.1152/physiol.00066.2013
[40] HARADA T, KURIMOTO E, TOKUHIRO K, et al. Disulfide bond formation in refolding of thermophilic fungal protein disulfide isomerase[J]. Journal of Bioscience and Bioengineering,2001,91(6):596−598. doi: 10.1016/S1389-1723(01)80180-8
[41] DIAS C L, ALA-NISSILA T, WONG-EKKABUT J, et al. The hydrophobic effect and its role in cold denaturation[J]. Cryobiology,2010,60(1):91−99. doi: 10.1016/j.cryobiol.2009.07.005
[42] BADIEYAN S, BEVAN D R, ZHANG C. Study and design of stability in GH5 cellulases[J]. Biotechnology and Bioengineering,2012,109(1):31−44. doi: 10.1002/bit.23280
[43] WEI M, GAO X, ZHANG W, et al. Enhanced thermostability of an L-rhamnose isomerase for D-allose synthesis by computation-based rational redesign of flexible regions[J]. Journal of Agricultural and Food Chemistry,2023,71(42):15713−15722. doi: 10.1021/acs.jafc.3c05736





 下载:
下载:
 下载:
下载:



