Effects of Naringenin and Its Glycosides on Functional Properties of Oat Protein and Their Interaction Mechanism
-
摘要: 为探究柚皮素及其糖苷衍生物(柚皮苷和芸香柚皮苷)对燕麦蛋白的起泡性、乳化性、及界面张力的影响差异及相互作用机制。本文联合荧光光谱和圆二色谱明晰了燕麦蛋白-黄酮复合体系的相互作用机制及蛋白结构变化,最后通过分子模拟技术将复合体系的相互作用可视化。结果表明,柚皮素、柚皮苷及芸香柚皮苷显著提升了燕麦蛋白的起泡、乳化性能,降低了燕麦蛋白的界面张力,且糖苷类黄酮的效果更佳,即柚皮苷>芸香柚皮苷>柚皮素。荧光及分子模拟结果共同表明三种黄酮与燕麦蛋白主要通过氢键作用和范德华力发生相互作用,常温下,结合能力大小分别为芸香柚皮苷(11.05×104 L/mol)>柚皮素(6.25×104 L/mol)>柚皮苷(0.23×104 L/mol)。三种黄酮改变燕麦蛋白的三级结构(荧光光谱红移)、二级结构(α-螺旋降低,β-折叠和无规则卷曲增加)、降低了表面疏水性,使得燕麦蛋白功能性质提升(起泡、乳化)。本研究可为选择合适的黄酮应用在燕麦蛋白基产品中提供理论参考。Abstract: To investigate the effect of naringenin and its glycoside (naringin and narirutin) on foaming, emulsifying and interfacial tension of oat protein and their interaction mechanism, in this study, fluorescence spectra and circular dichroism were used to elucidate the interaction mechanism and protein structure changes of oat protein-flavonoid complex system. The interaction of oat protein-flavonoid complex system was visualized by molecular simulation technique. The results showed that naringenin, naringin, and narirutin significantly improved the foaming and emulsifying properties of oat protein, and reduced the interfacial tension of oat protein, and the effect of glucoside flavonoids was better, namely naringin>narirutin>naringenin. The results of fluorescence and molecular simulation showed that the three flavonoids interact with oat protein mainly through hydrogen bonding and van der Waals force, and the binding constants at room temperature were narirutin (11.05×104 L/mol)>naringenin (6.25×104 L/mol)>naringin (0.23×104 L/mol). The three flavonoids changed the tertiary structure (fluorescence spectral redshift), secondary structure (α-helix decreased, β-folding and random curling increased), and decreased surface hydrophobicity of oat protein, which resulted the and functional properties (foaming and emulsification) of oat protein improved. These results can provide a theoretical reference for selecting suitable flavonoids for oat protein-based products.
-
Keywords:
- oat protein /
- naringenin /
- flavonoid glycosides /
- interactions /
- functional properties
-
燕麦蛋白被认为是一种优质谷物蛋白,其蛋白质含量约15%~20%,显著高于其他大多数谷物,在食品领域具有巨大的开发潜力[1]。从其组成上看,燕麦蛋白由70%~80%的球蛋白、4%~14%的醇溶蛋白、1%~12%的白蛋白和10%的谷蛋白组成[2]。燕麦蛋白具有优越的氨基酸组成,其中赖氨酸和苏氨酸等必需氨基酸显著高于其他谷物蛋白[3]。此外,燕麦蛋白具有较高的消化率,其消化率可达86.58%,可与大豆蛋白的87.67%相媲美[4]。然而,天然燕麦蛋白的功能性质较差,如溶解性、乳化性、起泡性等,在实际应用过程中具有较大的局限性[5−6]。已有多种改性技术对燕麦蛋白进行改性,如酶解、糖基化、乙酰化、琥珀酰化和脱酰胺化等,用于提升其溶解性、乳化性、起泡性等以扩宽其应用范围[7−10]。然而,这些方法在食品工业化应用方面均存在一定的局限性[11]。目前研究发现,采用多酚与蛋白的天然亲和相互作用来改善蛋白功能性质是一种简单、天然、绿色的改性技术[12]。
黄酮类化合物是一种多酚类化合物,存在于蔬菜、水果、坚果等植物性食品中,主要以糖苷的形式存在,具有抗氧化、抗肿瘤和抗炎以及降低慢性疾病的风险等多种有益的生理活性功能[13]。随着人们健康需求的增加,对具有功能性复合食品体系的需求也随之增加,功能性黄酮与膳食蛋白间相互作用的相关研究也备受关注。黄酮化合物中的羟基会与蛋白质的羟基、酰胺基及羧基发生相互作用,使得蛋白-黄酮复合物结构显示出较强的抗氧化能力。黄酮类化合物还能增加蛋白质的溶解度,原因是黄酮和蛋白的相互作用会增强蛋白的表面电荷,通过静电斥力提高体系的分散能力[14]。黄酮与蛋白的相互作用后还会改变蛋白质的空间结构,进而影响蛋白质的溶解性[15]。黄酮-蛋白的相互作用还能降低蛋白的界面吸附能力、表面疏水性以及改变蛋白的二级结构,进而影响其乳化和界面性能[16]。然而,缺乏黄酮化合物对燕麦蛋白功能性质影响的研究,其相互作用机制也尚不明晰。
柚皮素、柚皮苷和芸香柚皮苷是一类具有黄烷酮结构的黄酮类化合物,是许多柑橘类水果的抗氧化成分,主要存在于葡萄柚、橙子、橘子和柠檬中。柚皮苷和芸香柚皮苷为柚皮素的二糖糖苷化合物,由柚皮素A环7位C与鼠李糖和葡萄糖连接形成。柚皮苷由鼠李糖和葡萄糖通过α-1,2糖苷键连接,而芸香柚皮苷则由鼠李糖和葡萄糖通过α-1,6糖苷键连接[17]。除了结构上的差异外,三种黄酮在一些神经性疾病上也展现出不同的药理活性[18]。研究表明柚皮素、柚皮苷和芸香柚皮苷能提升人血清白蛋白的亲和力,且它们的结构差异对蛋白亲和力起重要作用[19]。然而,目前关于柚皮素、柚皮苷和芸香柚皮苷之间的结构差异是否会对蛋白质的结构及功能性质产生不同影响也尚不清楚。
因此,本研究以燕麦蛋白和柚皮素及柚皮苷、芸香柚皮苷为代表性研究对象,通过多光谱学和分子对接模拟技术探究柚皮素的糖苷结构对燕麦蛋白的结构、起泡、乳化及界面张力等性质的影响,探明其相互作用机制,以期为黄酮类化合物应用于燕麦蛋白产品提供理论参考。
1. 材料与方法
1.1 材料与仪器
燕麦蛋白(Oat protein,OPI,蛋白纯度≥80%) 购自陕西宝禾生物科技有限公司;柚皮素(纯度≥98%)、柚皮苷(纯度≥98%)、8-苯胺-1-萘磺酸(ANS,纯度≥95%)、十二烷基硫酸钠(SDS)、Bradford试剂盒 均购自于中国上海阿拉丁化学试剂有限公司;芸香柚皮苷(纯度≥98%) 购于上海源叶生物科技有限公司;其他试剂均为分析级。
U-T1810紫外分光光度计 北京普析通用仪器有限责任公司;F-7000荧光光谱仪 日本日立公司;T25-DS25高速分散机 德国IKA公司;ESr-O界面流变扩张测量仪 德国Data physics公司;MOS-450圆二色谱仪 法国Bio-Logic SAS公司。
1.2 实验方法
1.2.1 燕麦蛋白和黄酮溶液的制备
称取适量的OPI溶于超纯水中并将其pH调节至8.0,于95 ℃下加热搅拌1 h,冷却后的OPI溶液在8000 r/min条件下离心20 min,取上清液,调节pH至7.0。使用Bradford试剂盒测定OPI上清液中蛋白含量,并采用超纯水稀释其质量浓度至10 mg/mL,并作为蛋白母液使用。将柚皮素、柚皮苷、芸香柚皮苷分别溶解在无水乙醇中制备1 mmol/L黄酮母液,调节pH至7.0。后续试验过程中分别采用超纯水和无水乙醇稀释OPI和三种黄酮母液至测试所需浓度。
1.2.2 起泡性的测定
使用高速分散机在12000 r/min条件下剪切OPI和OPI-黄酮复合物(OPI质量浓度为5 mg/mL)120 s生成泡沫。观察并记录初始气泡(V0)的体积,静置10 min后,再次记录气泡体积(V10)[20]。采用公式(1)和(2)计算起泡能力和泡沫稳定性。
起泡能力(%)=V0−VFVF×100 (1) 泡沫稳定性(%)=V10V0×100 (2) 式中,VF为剪切前的溶液体积,V0为剪切后的初始泡沫体积,V10为10 min后的泡沫体积。
1.2.3 乳化特性测定
将OPI和OPI-黄酮复合物溶液(OPI质量浓度为5.0 mg/mL)与大豆油按体积比16:4混合,采用高速分散机在12000 r/min下混合60 s形成乳液。使用0.1%质量分数SDS,按体积比1:100稀释获得的乳液,采用紫外分光光度计,在500 nm处测定乳液在0 min和10 min时的紫外吸收。采用公式(3)和(4)计算得燕麦蛋白乳化活性和乳化稳定性[21]。
乳化活性(m2/g)=2×2.303×A0×DF10000×(1−φ)×L×C (3) 乳化稳定性(%)=A10A0×100 (4) 式中,DF为稀释比(100);C为蛋白质质量浓度(mg/mL);L为光程长度(1 cm),φ为油的体积百分比(0.25);A0和A10为剪切后0 min和10 min时的吸光值。
1.2.4 界面张力测定
采用悬滴法分析三种黄酮(0.10 mg/mL)对OPI(0.30 mg/mL)在油水界面的吸附影响[22]。采用界面流变扩张测量仪测定OPI和OPI-黄酮复合物在油-水界面上的吸附。具体操作如下:将连接到电动注射控制单元的不锈钢针(外径1.65 mm,内径1.19 mm)插入到含有大豆油(0.8719 g/cm3)的矩形玻璃皿中,然后通过电动控制单元的注射器注射OPI或OPI-黄酮复合物溶液。在针尖处形成14 μL液滴后,使用不锈钢针在水-油界面处吸附120 min。通过CCD摄像机系统连续记录和数字化液滴的图像。通过图像处理软件SCA20和Young-Laplace方程(5)和(6)获取和计算界面张力:
界面张力(mN/m)=Δρ⋅gC (5) 1x⋅ddx(x⋅sinθ)=2b−C⋅Z (6) 式中,Δρ表示两相之间的密度差(g/cm3);g表示重力加速度(N/kg);C表示毛细管常数;b表示液滴固定点P的曲率半径(cm);x和Z分别表示液滴外部轮廓的平面上任何点的垂直和水平坐标;θ表示任意点的接触角(°)。
1.2.5 荧光光谱测定
蛋白的自猝灭效应[23]和配体的内滤效应[24]会影响荧光实验的准确性,因此需要选择合适的蛋白和黄酮浓度来进行荧光实验。采用紫外-可见光谱仪扫描柚皮素、柚皮苷、芸香柚皮苷在200~400 nm处的紫外光谱。通过三种黄酮的紫外吸收情况选择合适的黄酮浓度,尽可能地降低甚至避免三种黄酮在后续荧光实验中产生的内滤效应。具体操作如下:将0.1 mL不同浓度黄酮(0.10、0.15、0.20、0.25、0.30 mmol/L)加入到3 mL的超纯水中,充分复合后扫描其紫外光谱,评估黄酮配体的内滤效应。采用荧光光谱仪扫描不同质量浓度的OPI溶液(0.10、0.15、0.20、0.25、0.30、0.35、0.40、0.45 mg/mL)在280 nm激发波长处的荧光强度,通过浓度与荧光强度的线性关系选择合适的OPI浓度。
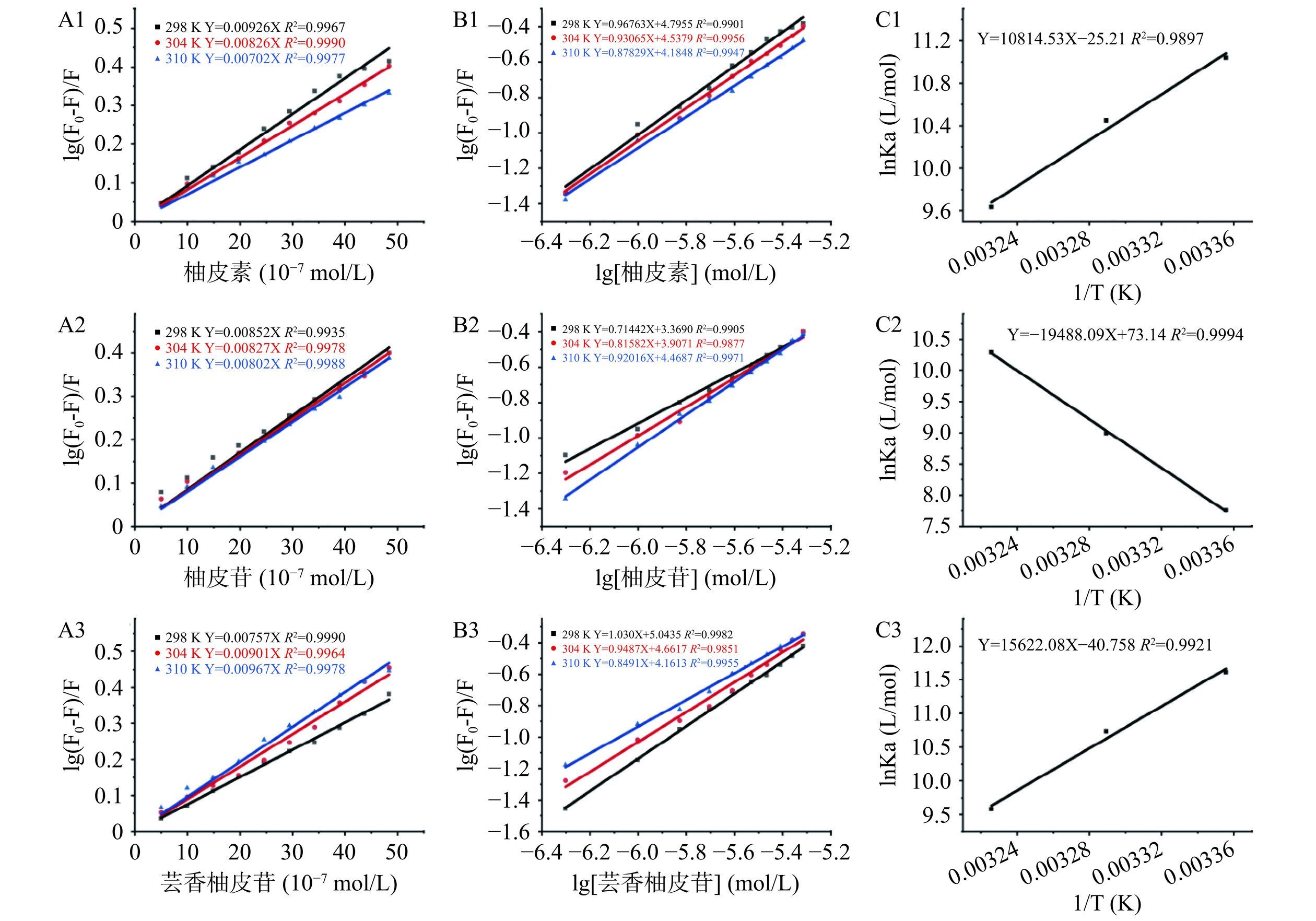
向3 mL优选浓度的OPI溶液中添加不同浓度的黄酮,随后扫描OPI的内源荧光光谱。激发波长固定在280 nm,收集发射波长为295~500 nm的荧光光谱,激发和发射狭缝宽度均为2.5 nm。分别于298、304和310 K的温度条件下收集OPI-黄酮复合物和OPI的荧光光谱信息[25]。根据Stern-Volmer方程(7)判断OPI与黄酮的荧光猝灭机制,再根据双对数方程(8)计算OPI与黄酮的结合常数(Ka)与结合位点数(n)。
F0−FcFc=Kqτ0[Q]=KSV[Q] (7) lgF0−FcFc=lgKa+nlg[Q] (8) 式中,F0和Fc分别为OPI和黄酮-OPI的荧光强度;[Q]为黄酮浓度(μmol/L);KSV是Stern-Volmer猝灭常数;Kq (KSV/τ0)为生物分子猝灭速率常数;τ0(10−8 s)为无猝灭剂时荧光团的平均寿命。
通过热力学参数确定OPI与三种黄酮的相互作用驱动力,由Van’t Hoff方程(9)和方程(10)计算二者结合的焓变(ΔH)、熵变(ΔS)和吉布斯自由能变(ΔG)。
lnKa=-ΔHRT+ΔSR (9) ΔG=ΔH−TΔS (10) 式中,Ka为对应温度下的结合常数;T、R分别为实验温度(298、304、310 K)和气体常数(8.314 J·mol−1·K−1)。
1.2.6 圆二色谱测定
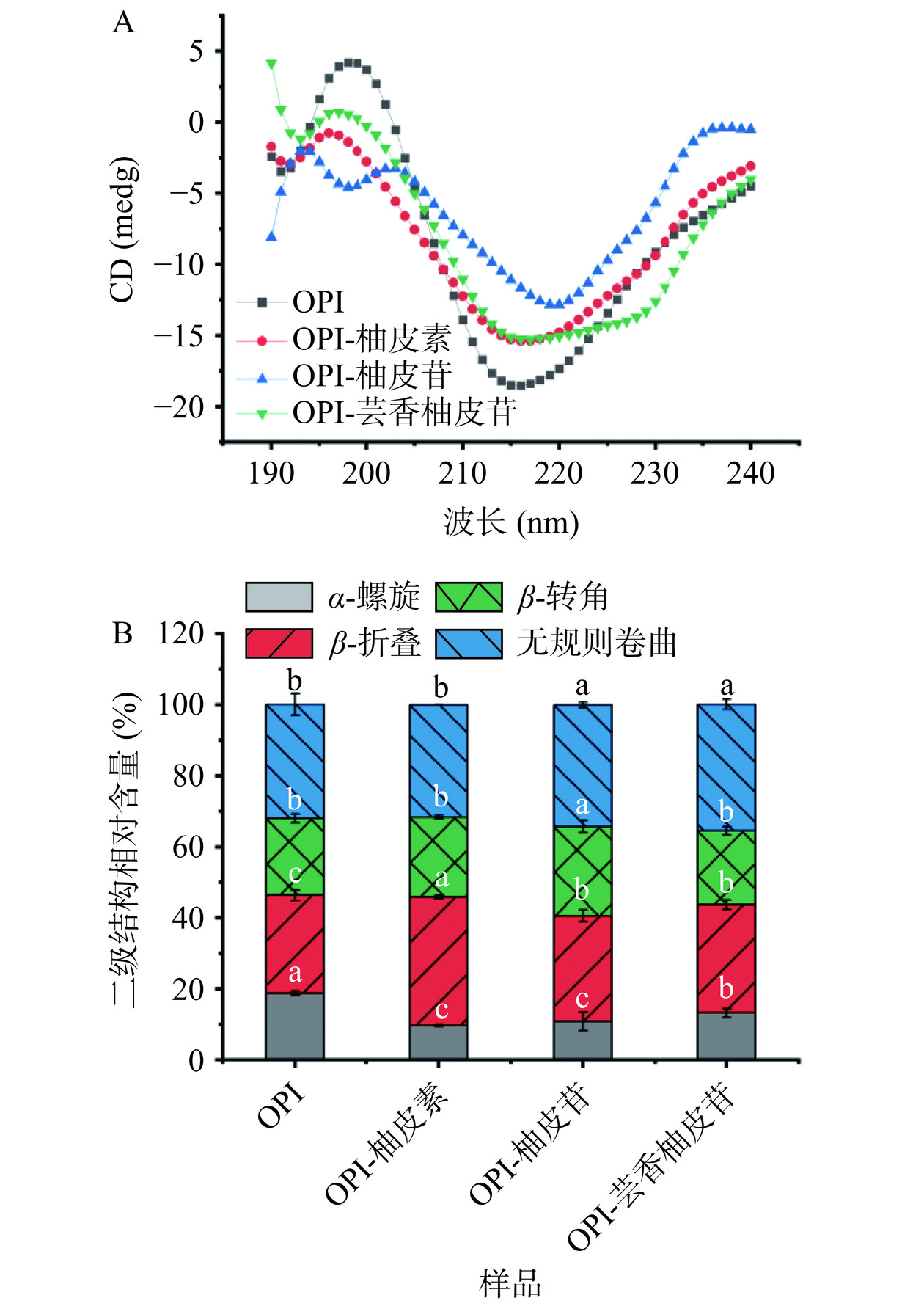
采用圆二色谱仪在190~240 nm范围内测定三种黄酮(2.46 μmol/L)对OPI(0.30 mg/mL)的圆二色谱吸收影响。根据获得的圆二色谱吸收信息,利用CONTIN程序计算OPI和OPI-黄酮复合物的α-螺旋、β-折叠、β-转角和无规则卷曲的含量[26]。
1.2.7 表面疏水性测定
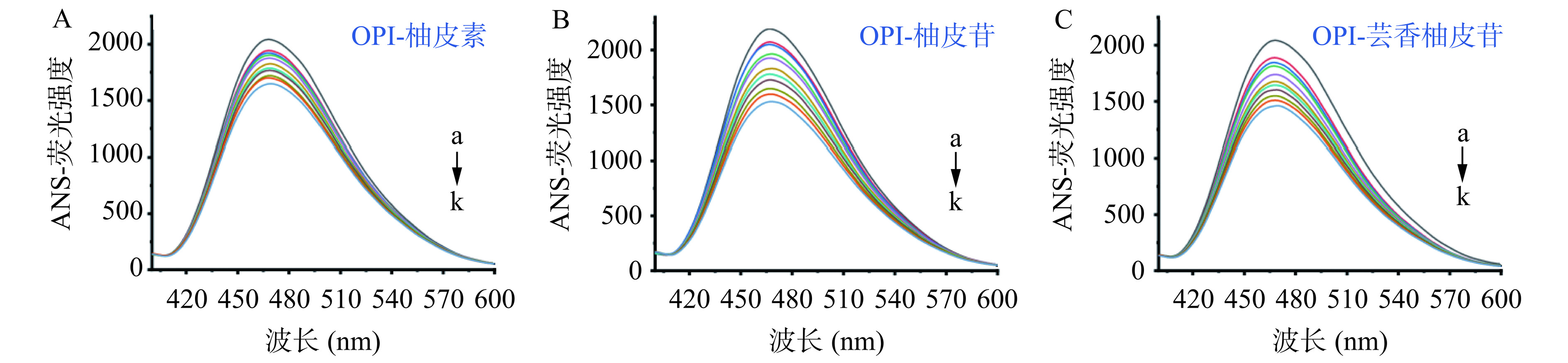
向3 mL 0.30 mg/mL OPI溶液中加入16 μL 8 mmol/L的ANS,然后在OPI-ANS溶液中逐渐加入10 μL相应的黄酮(0.15 mmol/L)溶液,逐次添加10次。扣除黄酮对应的背景荧光,通过ANS-OPI的荧光强度变化表征OPI的表面疏水性。设置的实验参数分别为:激发波长为380 nm,温度为298 K,激发和发射狭缝宽度分别为5.0 nm和2.5 nm,收集波长范围为400~600 nm的ANS荧光光谱[27]。
1.2.8 燕麦蛋白同源模型构建及分子对接模拟
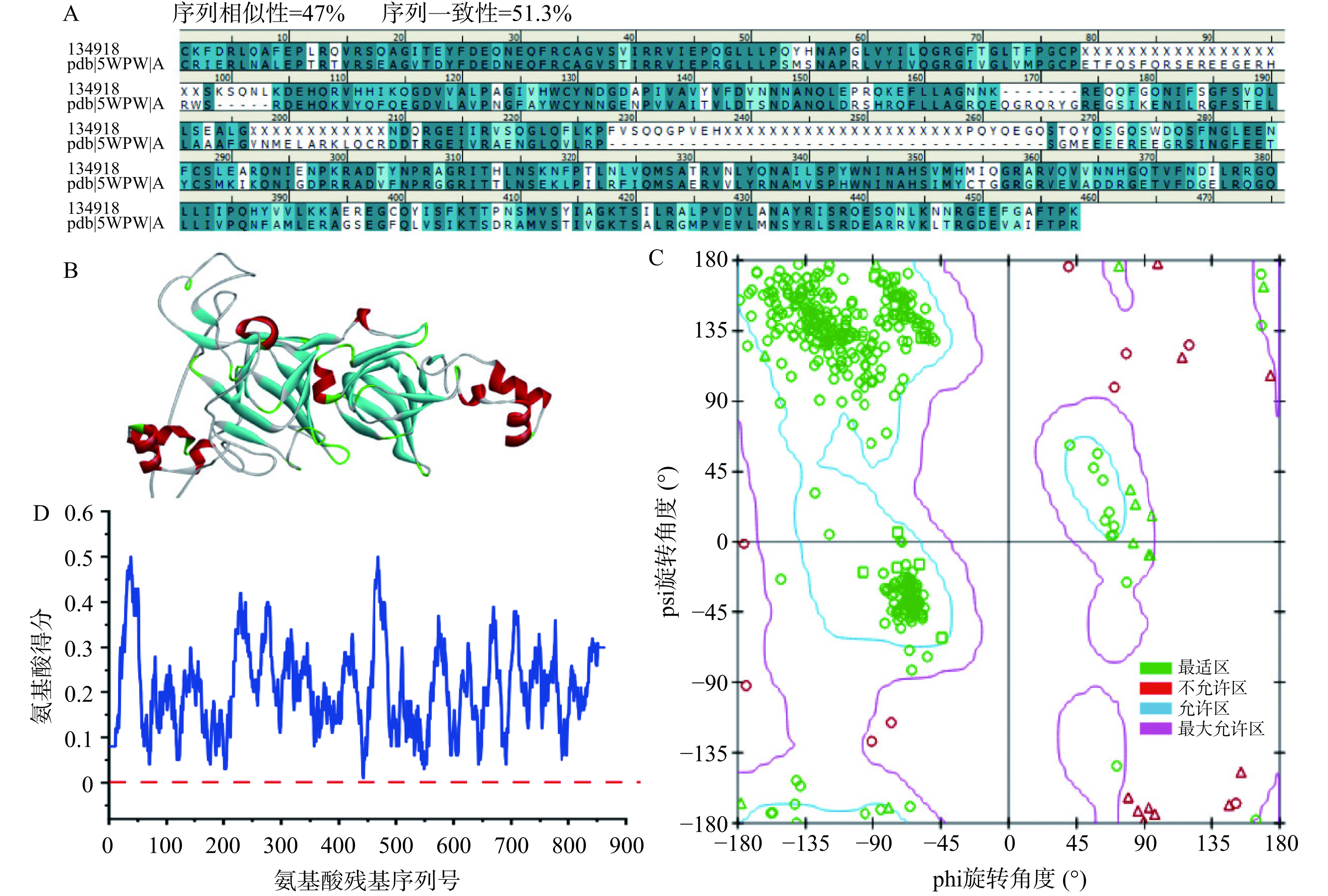
由于目前OPI的三维结构尚未被解析,采用同源建模方法构建OPI的三维结构。12S球蛋白(GI号:134918)是OPI的主成分蛋白[28],因此,在本研究中将12S球蛋白作为模型构建燕麦蛋白主要3D模型。12S球蛋白是分子量为320 kDa的六聚体,由54 kDa的亚基组成,每个亚基通过二硫键连接的酸性亚基(32 kDa)和碱性亚基(22 kDa)组成[29]。利用BLAST局部搜索算法对国家生物技术信息中心数据库(NCBI)进行搜索比对,寻找与OPI具有最高同源性的蛋白(PDB:5WPW)。从NCBI数据库中获得OPI(GI:134918)和模板(5WPW)的一级结构。燕麦蛋白3D结构的同源模型由Discovery Studio(DS)2019软件构建,并通过Ramachandran plot图、Profile-3D评分、X-ray进行模型质量评估。利用DS中CDOCKER对接算法分析黄酮与OPI的结合机制。从PubChem数据库获取三种黄酮分子的3D结构:柚皮素(CID:932),柚皮苷(CID:442428),芸香柚皮苷(CID:442431)。将同源模型导入到DS中并进行能量最低优化,黄酮分子结构导入ChemBioOffice 2020软件中进行能量最低优化。优化后的黄酮结构导入DS中,将蛋白定义为受体、黄酮定义为配体,并对二者赋予CHARMm力场,最后进行分子对接,具体对接参数参考Dai等[26]的研究,通过对比对接后模型的能量,优选最佳的对接构象,对最佳对接构象进行蛋白-黄酮相互作用的可视化。
1.3 数据处理
每组样品重复测定三次,结果以均值±标准差表示,使用SPSS 25.0软件分析数据的显著性,P<0.05为显著水平,在图中用不同字母表示样品间存在显著性差异;通过Origin 2022软件绘图。
2. 结果与分析
2.1 3种黄酮对燕麦蛋白的起泡与乳化性能影响
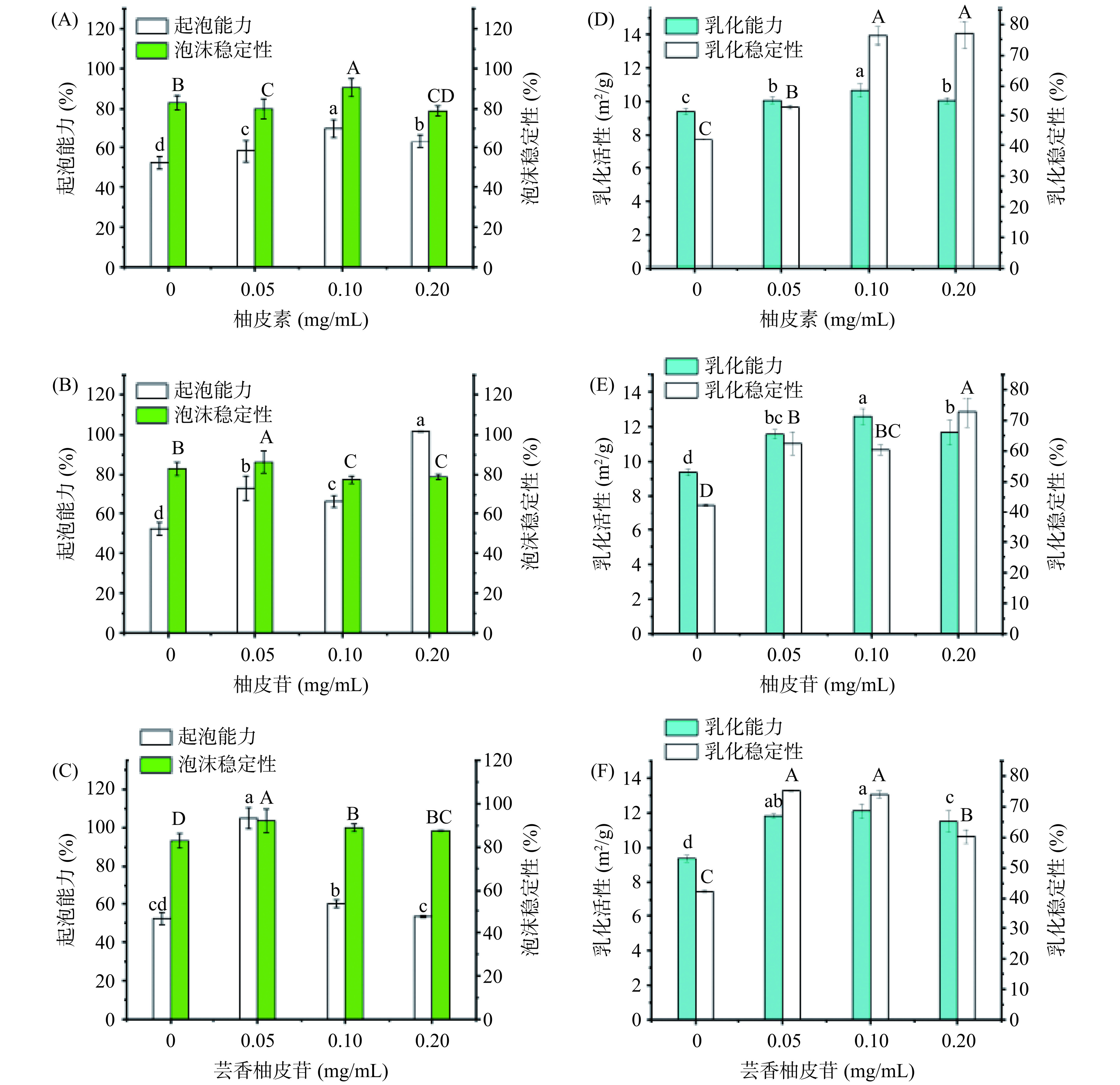
蛋白质具有双亲性,可以快速吸附到空气/水界面,降低表面张力,并在气泡周围形成粘弹性界面膜,支持泡沫的形成和稳定[30],蛋白质的起泡能力对创制新型功能食品具有重要意义。图1A~1C显示了三种黄酮对OPI起泡性能的影响。由图1A可知,OPI的起泡性能力为52.2%,当添加质量浓度为0.05、0.10和0.20 mg/mL的柚皮素后,起泡能力分别提高到了58.3%、70.0%和63.3%。这说明柚皮素能提升OPI的起泡性,但柚皮素浓度达到一定阈值后,OPI起泡性会被抑制。由图1B可知,柚皮苷也提高了OPI的起泡能力,添加相应浓度的柚皮苷后,OPI的起泡性能分别提高到了73.3%、66.7%和101.7%,起泡性随柚皮苷浓度出现了不规律波动。这说明黄酮物质对蛋白质的起泡性的影响是多样性,与具体的反应体系相关。曹艳芸[31]也发现了乳清蛋白-表儿茶素没食子酸(EGCG)体系的起泡性随EGCG浓度变化出现了类似的不规律变化。由图1C可知,高浓度(0.20 mg/mL)的芸香柚皮苷对OPI的起泡能力(53.3%)没有显著影响(P>0.05),但在低浓度(0.05 mg/mL)时对OPI的起泡能力有显著提高(105%)。以上结果表明三种黄酮能对泡沫特性起改善作用,改善效果则可能与黄酮的浓度及结构相关。总体而言,三种黄酮对起泡特性的效果为:柚皮苷>芸香柚皮苷>柚皮素,糖苷类黄酮改善效果更佳。OPI起泡性能的改善可能归因于三种黄酮诱导展开了部分蛋白质的空间结构[32],增强了蛋白结构的柔韧性,这使得蛋白更容易被吸附到空气-水界面上。此外,添加相应浓度的黄酮后,蛋白质泡沫稳定性的变化趋势与泡沫特性不同。两种糖苷化合物(柚皮苷、芸香柚皮苷)在0.05 mg/mL添加量时对OPI的泡沫稳定性有提升作用,而苷元柚皮素则在0.10 mg/mL添加量时能提升OPI的泡沫稳定性。但当三种黄酮含量增加到一定阈值(0.20 mg/mL)时,其泡沫稳定性的改善作用均受到抑制。稳定性的提升可能是由于三种黄酮与OPI相互作用改变了OPI的界面特性,形成更稳定的界面膜[33]。这些可能的结构变化对燕麦蛋白起泡性的影响在后续小节展开讨论。
![]() 图 1 柚皮素、柚皮苷、芸香柚皮苷对燕麦蛋白起泡和乳化性能的影响注:图中不同字母表示差异显著P<0.05;图6同。Figure 1. Effect of foaming and emulsifying properties of OPI with naringenin, naringin, and narirutin
图 1 柚皮素、柚皮苷、芸香柚皮苷对燕麦蛋白起泡和乳化性能的影响注:图中不同字母表示差异显著P<0.05;图6同。Figure 1. Effect of foaming and emulsifying properties of OPI with naringenin, naringin, and narirutin乳化剂的制造、蛋白乳饮料等食品的制备主要依赖于蛋白的乳化能力。蛋白可以通过快速扩散到油水界面,降低界面张力,达到稳定效果[22]。食品蛋白质的乳化能力通常可用乳化活性指数和乳化稳定性指数来表征。由图1D~1F可知,在0.10 mg/mL黄酮添加量时的OPI的乳化活性达到最佳:OPI-柚皮苷(12.58 m2/g)>OPI-芸香柚皮苷(12.11 m2/g)>OPI-柚皮素(10.69 m2/g)>OPI(9.35 m2/g)。OPI-黄酮复合物的乳化活性显著高于OPI(P<0.05),这可能是黄酮与蛋白的相互作用增加了蛋白结构的柔性,形成了更为稳定的界面膜,从而提升了蛋白的乳化活性[34]。OPI-黄酮复合物的乳化稳定性也明显优于OPI。乳化稳定性的提高可能是黄酮导致乳状液中吸附在油滴表面的蛋白质亲水性增强,油滴在水相中分布更稳定,难聚集成大颗粒,从而提高了乳液的稳定性[35]。
2.2 3种黄酮对燕麦蛋白的界面张力影响
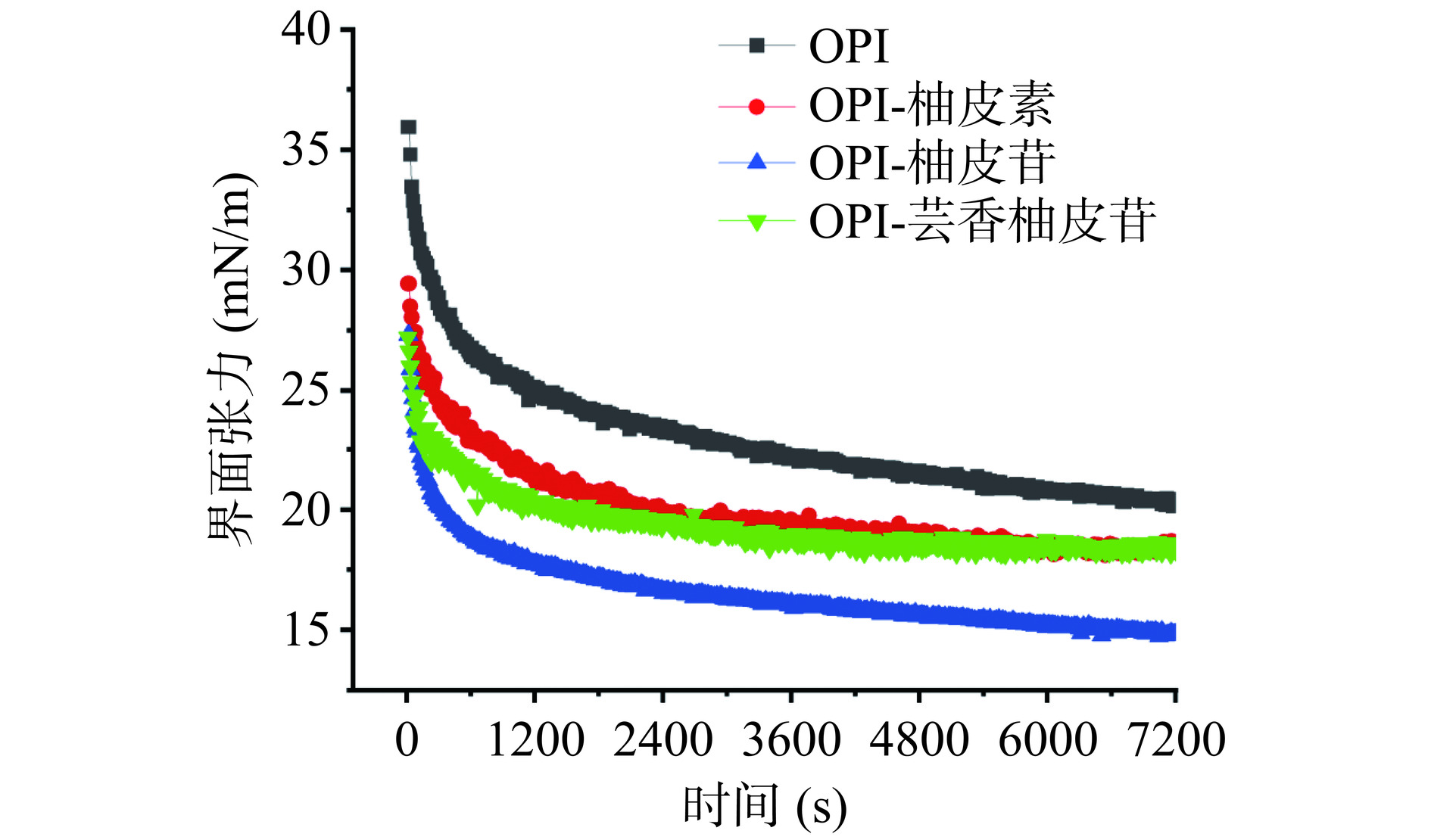
图2展示了OPI和OPI-黄酮复合物在油水界面的界面张力随时间的变化。OPI的界面张力随着时间的增加逐渐下降并趋于稳定,7200 s后,OPI溶液的界面张力为20.38 mN/m。加入柚皮素、柚皮苷、芸香柚皮苷后,OPI的界面张力分别降至18.49、14.38和18.30 mN/m。这一结果可能是黄酮-蛋白复合物的形成导致燕麦蛋白构象发生变化,降低了蛋白质的表面疏水性,提高了油水界面的吸附速度和吸附容量[36]。蛋白-黄酮间的相互作用可能抑制了蛋白的聚集,乳液表面张力降低,最终导致乳化活性和乳化稳定性升高。三种黄酮对OPI的界面张力有不同的影响,柚皮苷的影响最大,这一结果趋势与乳化能力结果一致。
2.3 燕麦蛋白和3种黄酮荧光浓度优化
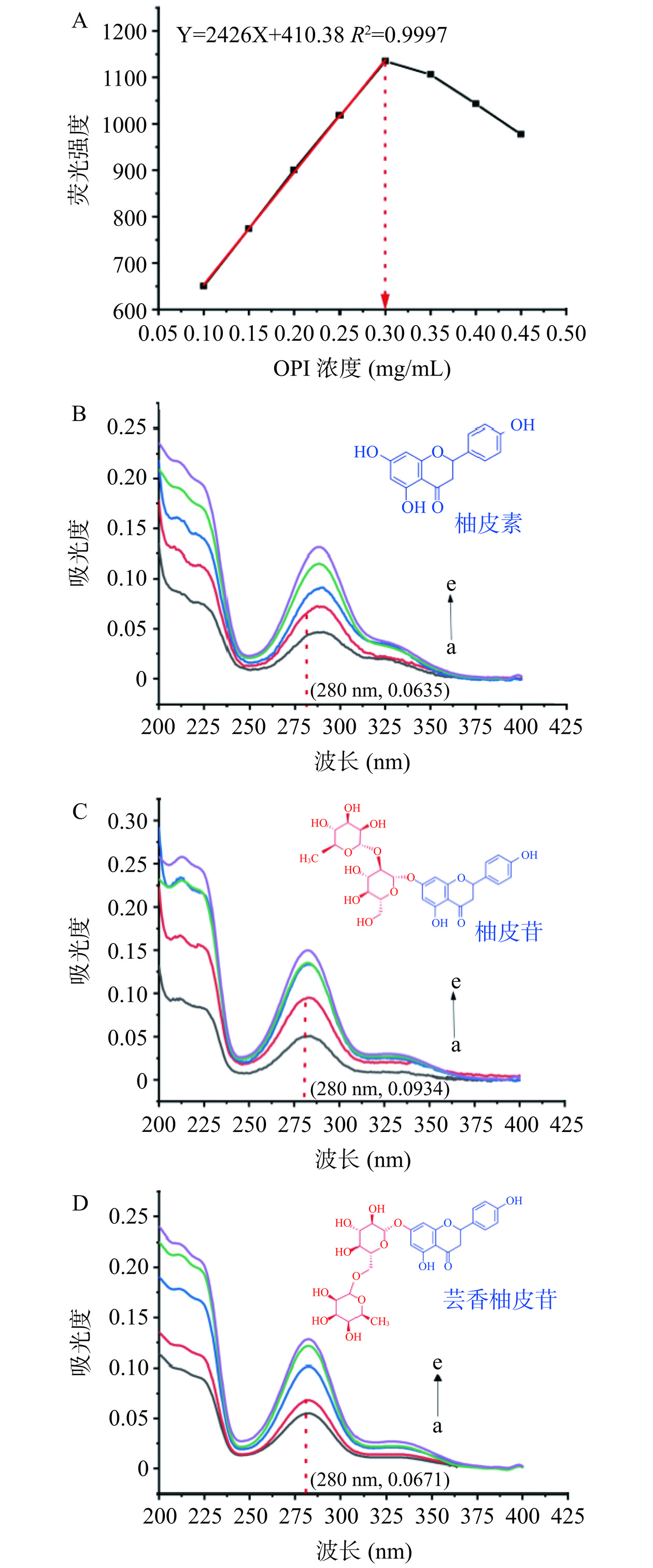
内滤效应包括配体小分子的内滤效应和蛋白质自身的内滤效应(自猝灭效应)。在高浓度的蛋白溶液中,激发光束被样品衰减导致荧光强度下降,这种衰减称为内滤效应[37]。内滤效应是荧光测量中常见的问题,可能会导致荧光光谱失真和光谱信号丢失,因此在测量过程中需消除或避免内滤效应。避免蛋白自身内滤效应最好的方法是测定不同浓度下的蛋白荧光强度,采用的蛋白浓度在浓度的线性区间内。如图3A所示,OPI的荧光强度在0.10~0.30 mg/mL浓度内有良好的线性关系(R2=0.9997)。因此,选用0.30 mg/mL的OPI进行荧光实验。在荧光实验中,OPI会受到激发而发射荧光,因此在测定黄酮对OPI的荧光影响时,也可能产生巨大的内滤效应。在荧光测定实验中,可通过校正方程和控制黄酮的浓度来消除或避免黄酮引起的内滤效应。虽然有许多荧光内滤效应可以采用校正方程,但都是有许多前提条件,最好控制荧光内滤效应的方法是最小化其影响,如控制配体在激发波长和发射波长处其吸收低于0.1 cm−1[38]。如图3B~3D所示,浓度为4.84 μmol/L的柚皮素、柚皮苷、芸香柚皮苷在280 nm激发波长处的紫外吸收相当小,分别为0.0635、00934、0.0671,且最大发射波长340 nm处的吸收几乎没有。非常微弱的紫外吸收对荧光猝灭数据的影响可以忽略不计[24]。因此,实验荧光使用的三种黄酮浓度为4.84 μmol/L。
2.4 3种黄酮对燕麦蛋白的荧光光谱影响
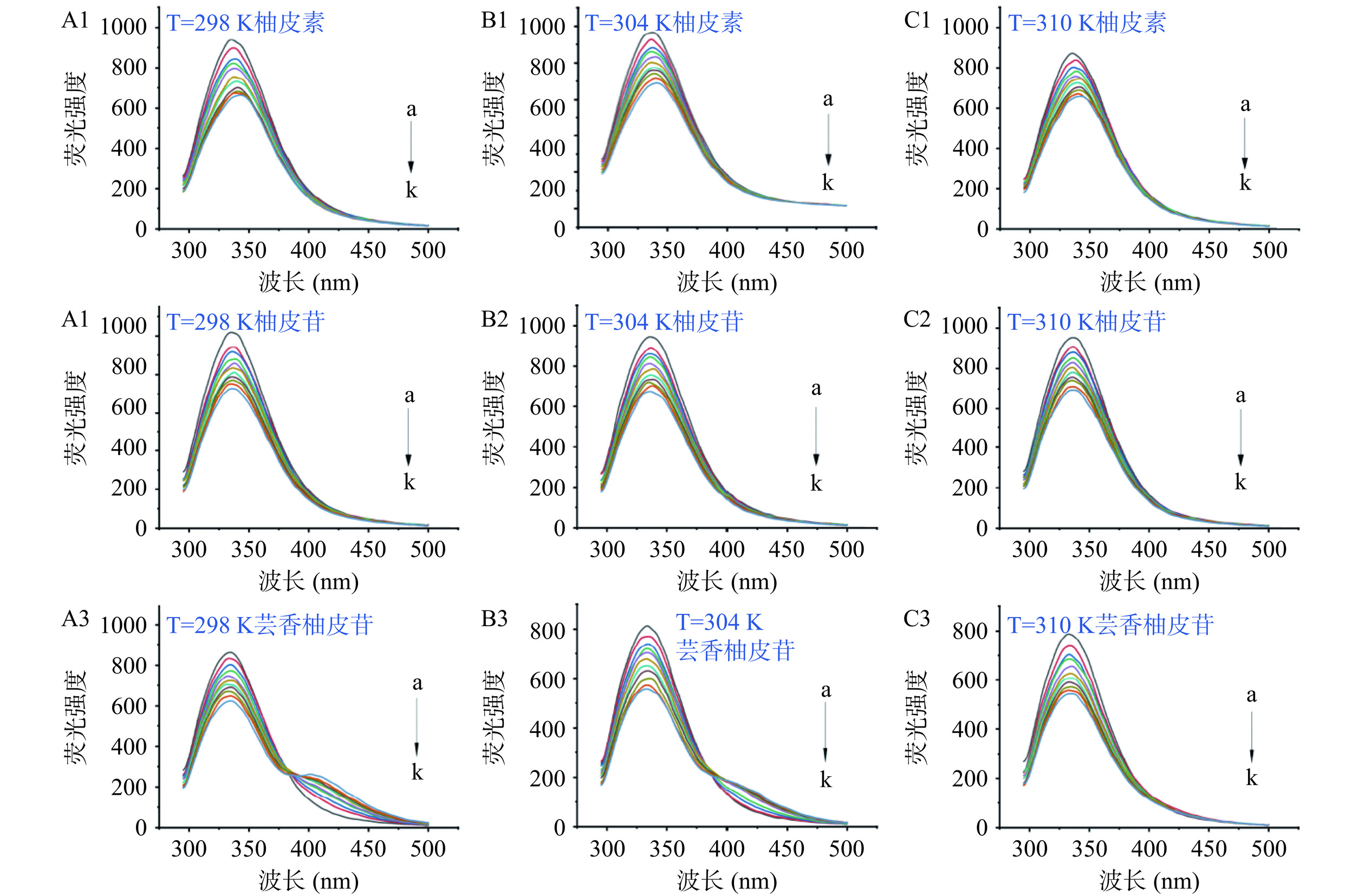
荧光分析可以提供关于蛋白质和小分子之间相互作用的关键信息,因为蛋白质的内源荧光对微环境的变化非常敏感[37]。图4A1~C3为OPI-黄酮复合物在不同温度下的荧光光谱,OPI经280 nm激发后,在约340 nm处出现最大发射峰,加入三种黄酮后,OPI的最大发射峰发生红移。此外OPI的最大荧光强度也随着黄酮浓度的增加逐渐降低,这表明柚皮素、柚皮苷和芸香柚皮苷可以猝灭OPI的内源荧光。三种黄酮对OPI的内源荧光的猝灭能力不同,在298 K和最大荧光浓度(4.839 μmol/L)条件下,三种黄酮的荧光猝灭效果为柚皮素(29.30%)>柚皮苷(28.61%)>芸香柚皮苷(27.57%)。这表明黄酮的结构差异会影响黄酮的荧光猝灭能力。Cao等[39]也观察到黄酮猝灭蛋白质荧光的能力与其结构有关。
2.5 3种黄酮与燕麦蛋白的猝灭机制及结合参数
荧光猝灭机制主要分为动态猝灭、静态猝灭以及内滤效应三种[40]。内滤效应已在上述实验中排除,采用Stern-Volmer方程确定三种黄酮与OPI的荧光猝灭机制。图5A1~A3描述了三种黄酮在不同温度下与OPI相互作用的Stern-Volmer曲线及猝灭常数(Ksv)。三种黄酮的Stern-Volmer方程均具有良好的线性关系,说明三种黄酮与OPI的猝灭机制为动态或静态机制中的一种。柚皮素和柚皮苷的Ksv值随着温度的升高而降低,而芸香柚皮苷的Ksv值则随着温度的升高而升高。Ksv降低可能是温度高时,形成复合物不稳定所导致的。如表1所示,在298 K时,OPI-柚皮素-复合体系的猝灭速率Kq(KSV/τ0)值为9.26×1012 L/mol/s,OPI-柚皮苷复合体系为8.52×1012 L/mol/s,OPI-芸香柚皮苷复合体系为7.57×1012 L/mol/s。这些数值均显著大于生物大分子的最大碰撞猝灭常数(2×1010 L/mol/s),说明三种黄酮的猝灭机制都是静态猝灭[41]。
表 1 不同温度下燕麦蛋白与三种黄酮反应的荧光猝灭常数及热力学参数Table 1. Fluorescence quenching constants and thermodynamic parameters of OPI reaction with three flavonoids at different temperatures样品 T(K) Ksv(104 L/mol) Kq(1012 L/mol/s) Ka(104 L/mol) n ΔH(kJ/mol) ΔS(J/mol) ΔG(kJ/mol) 298 9.26±0.002b 9.26±0.002b 6.25±0.02b 0.97±0.03 −27.36 OPI-柚皮素 304 8.26±0.01e 8.26±0.01e 3.53±0.01d 0.93±0.02 −25.19±9.16 −209.46±30.14 −26.46 310 7.02±0.001h 7.02±0.001h 1.53±0.01f 0.88±0.02 −24.83 298 8.52±0.002d 8.52±0.002d 0.23±0.01i 0.71±0.02 −19.22 OPI-柚皮苷 304 8.27±0.001e 8.27±0.001e 0.81±0.02h 0.82±0.03 −162.02±3.85 −608.06±12.65 −22.74 310 8.02±0.09f 8.02±0.09f 2.94±0.01e 0.92±0.02 −26.52 298 7.57±0.08g 7.57±0.08g 11.05±0.01a 1.03±0.02 −28.77 OPI-芸香柚皮苷 304 9.01±0.002c 9.01±0.002c 4.59±0.02c 0.95±0.04 −129.88±11.57 −338.87±38.08 −27.13 310 9.67±0.002a 9.67±0.002a 1.45±0.01fg 0.85±0.02 −24.70 注:T代表温度;Ksv、Kq、Ka、n分别代表荧光猝灭常数、猝灭速率、结合常数及结合位点数;ΔH、ΔS和ΔG则分别代表焓变、熵变和吉布斯自由能变;不同小写字母表示同一列中Ksv、Kq和Ka的值有显著差异(P<0.05)。 在静态猝灭的前提下,常通过双对数方程来计算结合常数(Ka)和结合位点数(n)。如图5B1~B3所示,在298、304、310 K温度下,三种黄酮与OPI的双对数方B程线性拟合良好。OPI与柚皮素和芸香柚皮苷的Ka随温度升高而降低,而OPI与柚皮苷的Ka则随温度升高而增加。在298 K时,从三种黄酮-OPI复合物的Ka可以得出OPI与三种黄酮的结合能力的强弱关系:OPI-芸香柚皮苷>OPI-柚皮素>OPI-柚皮苷。就柚皮苷而言,虽然与芸香柚皮苷一样含有许多羟基,但其Ka却显著小于芸香柚皮苷(P<0.05),这可能与它们连接的糖基片段类型有关[42]。如图5C1~C3,OPI与柚皮素、柚皮苷、芸香柚皮苷相互作用的 Van’t Hoff 方程分别为 Y=10814.53X−25.21(R2=0.9897)、Y=−19488.09X+73.14(R2=0.9994)、Y=15622.08X−40.76(R2=0.9921),热力学参数ΔH和ΔS可由方程的斜率和截距计算得出。蛋白质和小分子的结合常涉及一些非共价键作用力,如氢键、疏水相互作用、范德华力、静电相互作用力。ΔH和ΔS的大小和正负特性决定了蛋白-配体质复合物的结合方式以及作用在配体和蛋白质上的主要作用力类型。通常,当ΔH<0,ΔS<0时,氢键相互作用和范德华力占主导地位;当ΔH>0、ΔS>0时,疏水相互作用是关键驱动力;当ΔH<0,ΔS>0时,静电相互作用为主要作用力[43]。如表1所示,ΔG为负值表明OPI-黄酮复合物的形成为自发结合过程,ΔH、ΔS为负值说明三种黄酮与OPI形成复合物的形成是由焓驱动的,且氢键和范德华力是关键驱动力[44]。三种复合物的n值都大致约为1,说明OPI与三种黄酮以1:1的摩尔比例形成复合物。
2.6 3种黄酮对燕麦蛋白的二级结构影响
圆二色谱主要反映蛋白质二级结构的变化。如图6A,OPI的圆二色谱在216 nm附近出现一个负椭圆峰,这是二级结构β-折叠的特征峰。当与柚皮素、柚皮苷、芸香柚皮苷结合后,负峰强度逐渐减小,说明三种黄酮的加入影响了OPI的二级结构。如图6B,OPI的初始二级结构相对含量为18.8%的α-螺旋,27.6%的β-折叠,21.6%的β-转角,32.1%的无规则卷曲。当与柚皮素、柚皮苷、芸香柚皮苷结合后,OPI的α-螺旋相对含量分别从18.8%下降到9.7%、10.9%和13.2%,β-折叠相对含量则分别从27.6%增加到36.2%、29.7%和30.5%,而β-转角相对含量变化不显著。柚皮素对OPI的无规卷曲相对含量影响不显著,而与其糖苷化合物(柚皮苷、芸香柚皮苷)结合后,无规卷曲相对含量则从32.1%增至到34.3%和35.6%。以上变化说明三种黄酮改变了OPI的二级结构,且对OPI结构的影响程度与黄酮的结构差异有关。推测这些变化可能是由于OPI与三种黄酮的相互作用导致蛋白质多肽链的展开并重排,并破坏了维持OPI二级结构的氢键,从而使OPI的结构变得更疏松[45]。这些燕麦蛋白二级结构的变化可能是导致其功能性质(乳化性、起泡性)增强的重要原因,正如结果与分析中的2.1所示。
2.7 3种黄酮对燕麦蛋白的表面疏水性影响
蛋白质的表面疏水性与其界面特性密切相关。如图7A~C所示,OPI的表面疏水性随着黄酮浓度的增加而逐渐降低。这可能是黄酮分子与OPI表面的疏水性基团结合,从而阻止ANS与OPI的疏水性表面结合,ANS和OPI的结合位点减少,疏水性降低[46]。另外一种可能是黄酮的复合改变了OPI的结构,从而改变了OPI表面疏水氨基酸残基的分布[47]。Meng等[48]发现,黄酮结构上的羰基、羟基等极性基团可以结合在鹰嘴豆蛋白上,使二者结合后蛋白结构发生变化,增强了其表面亲水性。这些燕麦蛋白表面疏水性的降低可能也是导致其功能性质(乳化性、起泡性)增强的重要原因,正如结果与分析中的2.1所示。
2.8 燕麦蛋白同源模型质量评估及分子对接模拟
蛋白质结构预测需要模型质量估计,模型的正确性决定了它在实际场景中的实用程度。图8A为OPI(GI号:134918)与同源模板5WPW的氨基酸序列比对,二者序列相似性为47%、序列一致性为51.3%。蛋白三维结构往往比较保守,当与模板同源性达到40%时,可以利用模板文件构建蛋白的三维结构[49],构建的3D结构如图8B所示。X-ray数据显示同源模型中有96.5%的氨基酸处于合适的位置,绝大多数氨基酸序列是正确的,这表明所建立的模型是可信的。Ramachandran plot是蛋白质或肽空间结构中肽键内α碳原子和羰基碳原子间的键的旋转度(psi)对α碳原子和氮原子之间键的旋转度(phi)的作图,主要用来指明蛋白质或肽类中氨基酸残基的允许和不允许的构象[50],可用于表征同源建模结果的合理性。如图8C所示,同源模型的绝大部分氨基酸(绿色)位于蓝色圈内,说明模型是合理的。Profile-3D主要用于评估所构建的同源模型蛋白的3D结构与氨基酸序列的兼容性,其分析的重点是氨基酸侧链构象的合理性[51]。如图8D所示,同源模型的氨基酸Profile-3D评分结果显示,绝大多数氨基酸(89.1%)得分大于0.1,说明所建立的同源模型是可信的。综上所述,构建的OPI的3D结构可用于分子对接模拟。
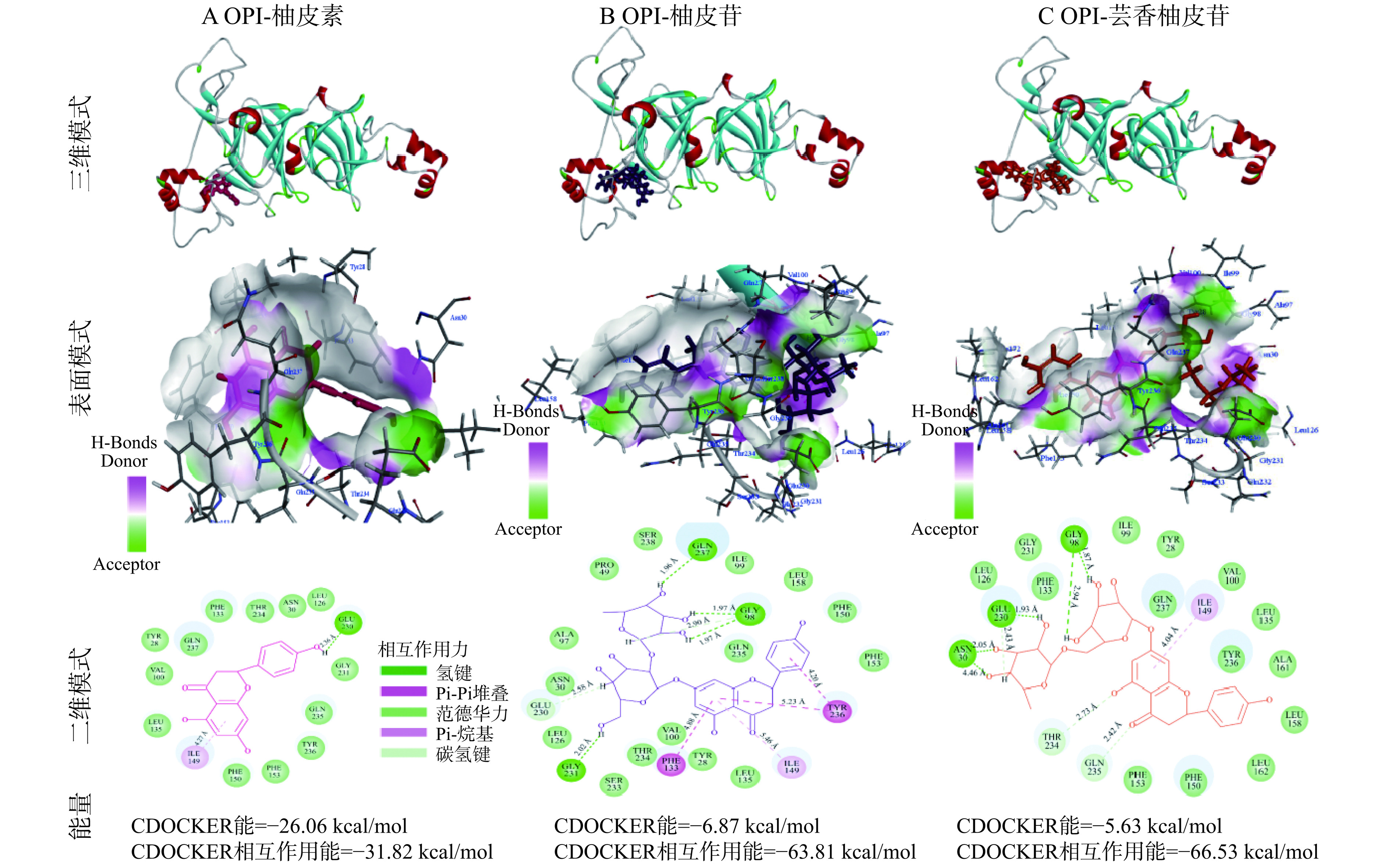
分子对接可以可视化OPI与三种黄酮类化合物之间的相互作用。根据CDOCKER模拟程序,具有最低能量分数的构型与最稳定的分子构象相关。因此,本研究选取具有最低CDOCKER能和CDOCKER相互作用能的OPI-黄酮复合物最佳构象来进一步分析蛋白-黄酮相互作用。三种黄酮与OPI对接后的结合位置、相互作用氨基酸和作用力如图9A~C所示。根据能量最低原则,OPI-柚皮素、OPI-柚皮苷、OPI-芸香柚皮苷最低CDOCKER能分别为−29.06、−6.87、−5.63 kcal/mol,最低CDOCKER相互作用能分别−31.82、−63.81、−66.53 kcal/mol。这说明三种黄酮的构象与OPI具有最高的结合可能性。蛋白质氢键的三维、表面、二维相互作用为黄酮与蛋白质的结合提供了更加直观的可视化效果。对二维相互作用原理图的分析如下:
如图9A所示,柚皮素与OPI之间主导的作用力是氢键和范德华力。OPI的Glu 230与柚皮素B环上的4′-羟基发生氢键相互作用,作用距离为2.46 Å。OPI的Ile 49与柚皮素A环上的羰基通过疏水作用相连,作用距离为4.27 Å。柚皮素还通过范德华力与OPI的多个氨基酸作用,如Leu 135、Val 100、Tyr 28、Gln 237、Phe 133、Thr 234、Asn 30、Leu 126、Gly 231、Gln 235、Tyr 236、Phe 153、Phe 150。
如图9B所示,柚皮苷与OPI的相互作用结果显示,氢键是二者相互作用的主要驱动力。OPI的Gly 231与柚皮苷葡萄糖吡喃基环上的羟基发生氢键相互作用,Gln 237和Gly 98则分别与柚皮苷鼠李糖吡喃基环上的羟基通过氢键相互作用。OPI的Phe 133、Ile 149和Try 236与柚皮苷A环上的羰基通过Pi-Pi堆叠疏水作用力相连,Try 236与柚皮苷B环上的羰基通过Pi-烷基疏水作用力相连。Leu 126、Glu 230、Asn 30、Val 97、Pro 49、Ser 238、Ile 99、Gln 235、Leu 158、Phe 150、Phe 153、Leu 135、Tyr 28、Val 100、Thy 234、Ser 233等氨基酸残基则通过范德华力与柚皮苷相互作用。
如图9C所示,芸香柚皮苷与OPI之间的主要相互作用力也是氢键。OPI的Asn 30和Glu 230与芸香柚皮苷鼠李糖吡喃基环上的羟基发生氢键相互作用,OPI的Gly 98与芸香柚皮苷的葡萄糖吡喃基环上的羟基通过氢键作用。OPI的Ile 149与芸香柚皮苷A环上的羰基通过Pi-烷基疏水相互作用相连。综上,OPI与三种黄酮之间存在不同的作用力(氢键、疏水相互作用、范德华力等),但OPI与三种黄酮的相互作用力都以氢键和范德华力为主,这与荧光热力学结果一致。
3. 结论
本研究通过多种光谱学联合分子模拟技术对比研究了三种结构相似的代表性黄酮(柚皮素、柚皮苷及芸香柚皮苷)与燕麦蛋白的相互作用机理,并探讨了三种黄酮的结构差异对燕麦蛋白的乳化、起泡、界面及结构性质的影响。结果表明,柚皮素、柚皮苷及芸香柚皮苷与蛋白结合后,显著提升了燕麦蛋白的起泡、乳化性能,降低了燕麦蛋白的界面张力,且糖苷类黄酮的效果更佳,即柚皮苷>芸香柚皮苷>柚皮素。荧光及分子模拟结果共同表明三种黄酮与燕麦蛋白主要通过氢键作用和范德华力发生相互作用,且常温下结合能力大小分别为芸香柚皮苷(11.05×104 L/mol)>柚皮素(6.25×104 L/mol)>柚皮苷(0.23×104 L/mol)。三种黄酮与燕麦蛋白的结合显著改变蛋白的三级结构、二级结构(α-螺旋降低,β-折叠和无规则卷曲增加)、降低了表面疏水性。本研究对燕麦蛋白和黄酮化合物的利用具有实际意义,并为燕麦蛋白的功能性质改善提供了理论基础。
-
图 1 柚皮素、柚皮苷、芸香柚皮苷对燕麦蛋白起泡和乳化性能的影响
注:图中不同字母表示差异显著P<0.05;图6同。
Figure 1. Effect of foaming and emulsifying properties of OPI with naringenin, naringin, and narirutin
表 1 不同温度下燕麦蛋白与三种黄酮反应的荧光猝灭常数及热力学参数
Table 1 Fluorescence quenching constants and thermodynamic parameters of OPI reaction with three flavonoids at different temperatures
样品 T(K) Ksv(104 L/mol) Kq(1012 L/mol/s) Ka(104 L/mol) n ΔH(kJ/mol) ΔS(J/mol) ΔG(kJ/mol) 298 9.26±0.002b 9.26±0.002b 6.25±0.02b 0.97±0.03 −27.36 OPI-柚皮素 304 8.26±0.01e 8.26±0.01e 3.53±0.01d 0.93±0.02 −25.19±9.16 −209.46±30.14 −26.46 310 7.02±0.001h 7.02±0.001h 1.53±0.01f 0.88±0.02 −24.83 298 8.52±0.002d 8.52±0.002d 0.23±0.01i 0.71±0.02 −19.22 OPI-柚皮苷 304 8.27±0.001e 8.27±0.001e 0.81±0.02h 0.82±0.03 −162.02±3.85 −608.06±12.65 −22.74 310 8.02±0.09f 8.02±0.09f 2.94±0.01e 0.92±0.02 −26.52 298 7.57±0.08g 7.57±0.08g 11.05±0.01a 1.03±0.02 −28.77 OPI-芸香柚皮苷 304 9.01±0.002c 9.01±0.002c 4.59±0.02c 0.95±0.04 −129.88±11.57 −338.87±38.08 −27.13 310 9.67±0.002a 9.67±0.002a 1.45±0.01fg 0.85±0.02 −24.70 注:T代表温度;Ksv、Kq、Ka、n分别代表荧光猝灭常数、猝灭速率、结合常数及结合位点数;ΔH、ΔS和ΔG则分别代表焓变、熵变和吉布斯自由能变;不同小写字母表示同一列中Ksv、Kq和Ka的值有显著差异(P<0.05)。 -
[1] SPAEN J, SILVA J V C. Oat proteins:Review of extraction methods and techno-functionality for liquid and semi-solid applications[J]. LWT-Food Science and Technology,2021,147:111478. doi: 10.1016/j.lwt.2021.111478
[2] KLOSE C, ARENDT E K. Proteins in oats; their synthesis and changes during germination:A review[J]. Critical Reviews in Food Science and Nutrition,2012,52(7):629−639. doi: 10.1080/10408398.2010.504902
[3] NIETO-NIETO T V, WANG Y X, OZIMEK L, et al. Effects of partial hydrolysis on structure and gelling properties of oat globular proteins[J]. Food Research International,2014,55:418−425. doi: 10.1016/j.foodres.2013.11.038
[4] HE Y Q, MIN C, SHEN L L, et al. Ultrasound pretreatment increases the bioavailability of dietary proteins by dissociating protein structure and composition[J]. Food Biophysics,2020,15(4):409−415. doi: 10.1007/s11483-020-09634-y
[5] KUMAR L, SEHRAWAT R, KONG Y Z. Oat proteins:A perspective on functional properties[J]. LWT-Food Science and Technology,2021,152:112307. doi: 10.1016/j.lwt.2021.112307
[6] PATRA T, AXEL C, RINNAN A, et al. The physicochemical stability of oat-based drinks[J]. Journal of Cereal Science,2022,104:103422. doi: 10.1016/j.jcs.2022.103422
[7] BOUKID F. Oat proteins as emerging ingredients for food formulation:where we stand?[J]. European Food Research and Technology,2021,247(3):535−544. doi: 10.1007/s00217-020-03661-2
[8] IMMONEN M, MYLLYVIITA J, SONTAG-STROHM T, et al. Oat protein concentrates with improved solubility produced by an enzyme-aided ultrafiltration extraction method[J]. Foods,2021,10(12):3050. doi: 10.3390/foods10123050
[9] JIANG Z M, LI T Q, MA L, et al. Comparison of interaction between three similar chalconoids and α-actalbumin:Impact on structure and functionality of α-lactalbumin[J]. Food Research International,2020,131:109006. doi: 10.1016/j.foodres.2020.109006
[10] ZHONG L, MA N, WU Y L, et al. Characterization and functional evaluation of oat protein isolate-pleurotus ostreatus beta-glucan conjugates formed via Maillard reaction[J]. Food Hydrocolloids,2019,87:459−469. doi: 10.1016/j.foodhyd.2018.08.034
[11] KUTZLI I, WEISS J, GIBIS M. Glycation of plant proteins via Maillard reaction:Reaction chemistry, technofunctional properties, and potential food application[J]. Foods,2021,10(2):376. doi: 10.3390/foods10020376
[12] CHENG J J, DUDU O E, ZHANG J J Q, et al. Impact of binding interaction modes between whey protein concentrate and quercetin on protein structural and functional characteristics[J]. Food Hydrocolloids,2023,142:108787. doi: 10.1016/j.foodhyd.2023.108787
[13] ZHOU W N, PENG C Y, WANG D S, et al. Interaction mechanism between OVA and flavonoids with different hydroxyl groups on B-ring and effect on antioxidant activity[J]. Foods,2022,11(9):1302. doi: 10.3390/foods11091302
[14] CAO H, LIU X J, AULRIH P N, et al. Plasma protein binding of dietary polyphenols to human serum albumin:A high performance affinity chromatography approach[J]. Food Chemistry,2018,270:257−263.
[15] PU P, ZHENG X, JIAO L, et al. Six flavonoids inhibit the antigenicity of β-lactoglobulin by noncovalent interactions:A spectroscopic and molecular docking study[J]. Food Chemistry,2021,339(1):128106.
[16] RAMOS-PINEDA A M, CARPENTER G H, GARCÍA-ESTÉVEZ I, et al. Influence of chemical species on polyphenol-protein interactions related to wine astringency[J]. Journal of Agricultural and Food Chemistry,2020,68(10):2948−2954. doi: 10.1021/acs.jafc.9b00527
[17] CHO S C, SHAW S Y. Comparison of the inhibition effects of naringenin and its glycosides on LPS-induced inflammation in RAW 264.7 macrophages[J]. Molecular Biology Reports,2024,51(1):56. doi: 10.1007/s11033-023-09147-0
[18] EMRAN T B, ISLAM F, NATH N, et al. Naringin and naringenin polyphenols in neurological diseases:Understandings from a therapeutic viewpoint[J]. Life-Basel,2023,13(1):99.
[19] CAO H, CHEN L S, XIAO J B. Binding citrus flavanones to human serum albumin:Effect of structure on affinity[J]. Molecular Biology Reports,2011,38(4):2257−2262. doi: 10.1007/s11033-010-0356-z
[20] HAO L L, SUN J W, PEI M Q, et al. Impact of non-covalent bound polyphenols on conformational, functional properties and in vitro digestibility of pea protein[J]. Food Chemistry,2022,383:132623. doi: 10.1016/j.foodchem.2022.132623
[21] PAN Y, XIE Q T, ZHU J X, et al. Study on the fabrication and in vitro digestion behavior of curcumin-loaded emulsions stabilized by succinylated whey protein hydrolysates[J]. Food Chemistry,2019,287:76−84. doi: 10.1016/j.foodchem.2019.02.047
[22] ZHU L J, XU Q Y, LIU X Y, et al. Oil-water interfacial behavior of soy β-conglycinin-soyasaponin mixtures and their effect on emulsion stability[J]. Food Hydrocolloids,2020,101:105531. doi: 10.1016/j.foodhyd.2019.105531
[23] CONDICT L, KASAPIS S. Critical issues encountered in the analysis of protein-phenolic binding interactions via fluorescence spectroscopy[J]. Food Hydrocolloids,2022,124:107219. doi: 10.1016/j.foodhyd.2021.107219
[24] VAN DE WEERT M. Fluorescence quenching to study protein-ligand binding:Common errors[J]. Journal of Fluorescence,2010,20(2):625−629. doi: 10.1007/s10895-009-0572-x
[25] LI X, DAI T T, HU P, et al. Characterization the non-covalent interactions between beta lactoglobulin and selected phenolic acids[J]. Food Hydrocolloids,2020,105:105761. doi: 10.1016/j.foodhyd.2020.105761
[26] DAI T T, CHEN J, MCCLEMENTS D J, et al. Protein-polyphenol interactions enhance the antioxidant capacity of phenolics:Analysis of rice glutelin-procyanidin dimer interactions[J]. Food & Function,2019,10(2):765−774.
[27] DAI T T, CHEN J, LI Q, et al. Investigation the interaction between procyanidin dimer and alpha-amylase:Spectroscopic analyses and molecular docking simulation[J]. International Journal of Biological Macromolecules,2018,113:427−433. doi: 10.1016/j.ijbiomac.2018.01.189
[28] CHANG Y W, ALLI I, KONISHI Y, et al. Characterization of protein fractions from chickpea (Cicer arietinum L.) and oat (Avena sativa L.) seeds using proteomic techniques[J]. Food Research International,2011,44(9):3094−3104.
[29] GUAN X, YAO H Y, CHEN Z X, et al. Some functional properties of oat bran protein concentrate modified by trypsin[J]. Food Chemistry,2007,101(1):163−170. doi: 10.1016/j.foodchem.2006.01.011
[30] HILL C, EASTOE J. Foams:From nature to industry[J]. Advances in Colloid Interface Science,2017,247:496−513. doi: 10.1016/j.cis.2017.05.013
[31] 曹艳芸. 乳清蛋白与多酚在中性pH条件下的相互作用对蛋白功能性质的影响研究[D]. 无锡:江南大学, 2017. [CAO Y Y. Functionality modification of whey proteins induced by the binding with phytophenolics at neutral pH[D]. Wuxi:Jiangnan University, 2017.] CAO Y Y. Functionality modification of whey proteins induced by the binding with phytophenolics at neutral pH[D]. Wuxi: Jiangnan University, 2017.
[32] WANG X, GU L P, SU Y J, et al. Microwave technology as a new strategy to induce structural transition and foaming properties improvement of egg white powder[J]. Food Hydrocolloids,2020,101:105530. doi: 10.1016/j.foodhyd.2019.105530
[33] ZHAN F C, LI J, WANG Y T, et al. Bulk, foam, and interfacial properties of tannic acid/sodium caseinate nanocomplexes[J]. Journal of Agricultural and Food Chemistry,2018,66(26):6832−6839. doi: 10.1021/acs.jafc.8b00503
[34] LIU F G, MA C C, GAO Y X, et al. Food-grade covalent complexes and their application as nutraceutical delivery systems:A review[J]. Comprehensive Reviews in Food Science and Food Safety,2017,16(1):76−95. doi: 10.1111/1541-4337.12229
[35] JAUREGI P, GUO Y C, ADELOYE J B. Whey proteins-polyphenols interactions can be exploited to reduce astringency or increase solubility and stability of bioactives in foods[J]. Food Research International,2021,141:110019. doi: 10.1016/j.foodres.2020.110019
[36] YE J P, DENG L P, WANG Y R, et al. Impact of rutin on the foaming properties of soybean protein:Formation and characterization of flavonoid-protein complexes[J]. Food Chemistry,2021,362:130238. doi: 10.1016/j.foodchem.2021.130238
[37] LI C H, DAI T T, CHEN J, et al. Protein-polyphenol functional ingredients:The foaming properties of lactoferrin are enhanced by forming complexes with procyanidin[J]. Food Chemistry,2021,339:128145. doi: 10.1016/j.foodchem.2020.128145
[38] 戴涛涛. 蛋白-多酚复合物相互作用及其对蛋白性质的影响[D]. 南昌:南昌大学, 2020. [DAI T T. Protein-polyphenol complexes interaction and their effects on protein properties[D]. Nanchang:Nanchang University, 2020.] DAI T T. Protein-polyphenol complexes interaction and their effects on protein properties[D]. Nanchang: Nanchang University, 2020.
[39] CAO H, JING X H, WU D H, et al. Methylation of genistein and kaempferol improves their affinities for proteins[J]. International Journal of Food Sciences and Nutrition,2013,64(4):437−443. doi: 10.3109/09637486.2012.759186
[40] VAN DE WEERT M, STELLA L. Fluorescence quenching and ligand binding:A critical discussion of a popular methodology[J]. Journal of Molecular Structure,2011,998(1-3):144−150. doi: 10.1016/j.molstruc.2011.05.023
[41] TIAN Y, XU G, CAO W W, et al. Interaction between pH-shifted β-conglycinin and flavonoids hesperetin/hesperidin:Characterization of nanocomplexes and binding mechanism[J]. LWT-Food Science and Technology,2021,140:110698. doi: 10.1016/j.lwt.2020.110698
[42] XIAO J B, CAO H, WANG Y F, et al. Glycosylation of dietary flavonoids decreases the affinities for plasma protein[J]. Journal of Agricultural and Food Chemistry,2009,57(15):6642−6648. doi: 10.1021/jf901456u
[43] XU J H, HAO M H, SUN Q F, et al. Comparative studies of interaction of beta-lactoglobulin with three polyphenols[J]. International Journal of Biological Macromolecules,2019,136:804−812. doi: 10.1016/j.ijbiomac.2019.06.053
[44] ZENG L, DING H F, HU X, et al. Galangin inhibits α-glucosidase activity and formation of non-enzymatic glycation products[J]. Food Chemistry,2019,271:70−79. doi: 10.1016/j.foodchem.2018.07.148
[45] BI H N, TANG L, GAO X, et al. Spectroscopic analysis on the binding interaction between tetracycline hydrochloride and bovine proteins β-casein, α-lactalbumin[J]. Journal of Luminescence,2016,178:72−83. doi: 10.1016/j.jlumin.2016.05.048
[46] SHPIGELMAN A, SHOHAM Y, ISRAELI-LEV G, et al. β-Lactoglobulin-naringenin complexes:Nano-vehicles for the delivery of a hydrophobic nutraceutical[J]. Food Hydrocolloids,2014,40:214−224. doi: 10.1016/j.foodhyd.2014.02.023
[47] LI T, HU P, DAI T T, et al. Comparing the binding interaction between β-lactoglobulin and flavonoids with different structure by multi-spectroscopy analysis and molecular docking[J]. Spectrochimica Acta Part a-Molecular and Biomolecular Spectroscopy,2018,201:197−206. doi: 10.1016/j.saa.2018.05.011
[48] MENG Y, WEI Z H, XUE C H. Deciphering the interaction mechanism and binding mode between chickpea protein isolate and flavonoids based on experimental studies and molecular simulation[J]. Food Chemistry,2023,429:136848. doi: 10.1016/j.foodchem.2023.136848
[49] BAKER D, SALI A. Protein structure prediction and structural genomics[J]. Science,2001,294(5540):93−96. doi: 10.1126/science.1065659
[50] 乔晶, 崔晟榕, 石宏武, 等. 罗汉果环阿屯醇合酶的同源建模、分子对接及催化环化的机理推测[J]. 生物技术通报,2019,35:101−108. [QIAO J, CUI C R, SHI H W, et al. Homology modeling and molecular docking of cycloartenol synthase in siraitia grosvenorii and speculated mechanism of catalytic cyclization[J]. Biotechnology Bulletin,2019,35:101−108.] QIAO J, CUI C R, SHI H W, et al. Homology modeling and molecular docking of cycloartenol synthase in siraitia grosvenorii and speculated mechanism of catalytic cyclization[J]. Biotechnology Bulletin, 2019, 35: 101−108.
[51] 吴玲, 何佩勋, 孙磊, 等. G蛋白偶联受体81同源建模及其与二羟基苯甲酸对接研究[J]. 化学研究与应用,2017,29:841−846. [WU L, HE P X, SUN L, et al. Homology modeling of G-coupled protein receptor 81 and docking simulation with dihydroxy-benzoic acid agonists[J]. Chemical Research and Application,2017,29:841−846.] doi: 10.3969/j.issn.1004-1656.2017.06.015 WU L, HE P X, SUN L, et al. Homology modeling of G-coupled protein receptor 81 and docking simulation with dihydroxy-benzoic acid agonists[J]. Chemical Research and Application, 2017, 29: 841−846. doi: 10.3969/j.issn.1004-1656.2017.06.015





 下载:
下载:
 下载:
下载:



