Advances in Microbial Genome Reduction and Optimization Based on Synthetic Biology
-
摘要: 微生物菌株的改造优化是现代工业生物技术的核心,其中工业微生物底盘细胞的构建已成为合成生物学的研究热点之一。基于合成生物学“设计-构建-测试-学习”策略,对微生物底盘细胞理想设计和改造是优良细胞工厂构建的重要手段,本文对合成生物学和微生物底盘细胞的改造优化策略进行了相关概述,同时总结了基于合成生物学各工业微生物底盘细胞的构建及应用,最后对在构建底盘细胞时所遇到的障碍及未来的前景进行了展望,以推动具有优良性状的微生物细胞工厂的构建。Abstract: The engineering and optimization of microbial strains form the core of modern industrial biotechnology, and the foundation of industrial microbial chassis cells has emerged as a hotspot in the field of synthetic biology. Guided by the "design-build-test-learn" approach, designing and engineering microbial chassis cells have become the critical strategy to develop excellent cell factories. This study presents a comprehensive review on strategies for modification and optimization of microbial chassis cells in synthetic biology. Meanwhile, this study summarized the applications and platforms benefited from industrial microbial chassis cells based on biology synthesis. Finally, this study discussed challenges encountered during chassis cell construction and put forward the future prospects to pave the way for microbial cell factories with superior attributes.
-
合成生物学是以人工设计和改造基因组为核心的一门多学科交叉融合的前沿学科,在解决环境、食品、能源、医疗健康等方面的问题不断取得突破[1]。利用合成生物学相关技术可对微生物细胞进行遗传及代谢途径改造,为构建具有优良性状的底盘细胞提供可能,进一步为提高工业微生物的生产性能提供一个理想的平台。
微生物底盘细胞是利用细胞平台,置入功能化的生物系统,使该细胞具备所需要的功能。微生物基因组复杂程度高,且常通过基因水平转移等方式吸收外源DNA序列,并将其保留在自身基因组中,从而增加了基因组容量,造成微生物的代谢负担。因此,通过删除非必需代谢途径的相关基因,可以降低细胞的复杂性。在特定条件下,这种操作能够保持与亲本菌株相同的生长行为和健壮性,从而优化细胞的代谢网络,提高细胞的遗传稳定性等方面的性能[2];同时,通过降低转录成本、消除竞争功能和限制不必要的调控相互作用,可进一步提高底盘微生物性能。目前基于合成生物学技术改造微生物底盘细胞的性能已成为研究重点,本文简述了合成生物学和各工业微生物底盘细胞的构建与优化策略,并对各微生物底盘细胞在合成生物学中的应用和所面临的问题进行了概述和展望,以期为深入微生物底盘细胞构建的研究提供参考。
1. 合成生物学制造过程及菌株开发
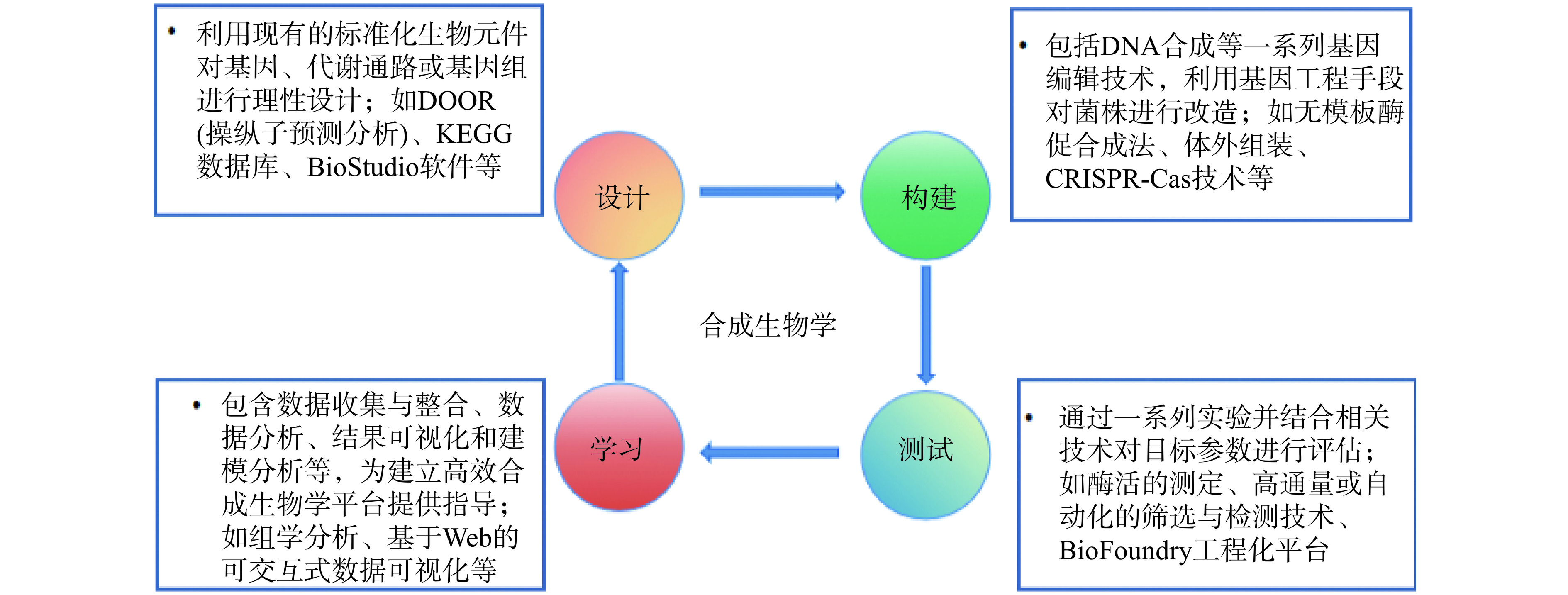
合成生物学是在工程学指导下,通过人工设计构建生物功能元件、装置和系统,对细胞进行遗传学改造,使其拥有满足人类需求的生物功能,其主要通过“设计-构建-测试-学习”(DBTL)循环来进行细胞的构建和优化[1](见图1)。
合成生物学制造产品是从原料到菌种再到产品的设计和优化。其基本过程可以概括为:首先根据产品特点设计生产的代谢通路,选择一个性状优良的底盘细胞作为宿主,然后通过基因编辑技术构建出合理的代谢途径,再通过一系列手段增强菌株耐受性及优化代谢通量,最后进行发酵、产品回收和纯化等操作,实现从实验室规模到商业化生产的放大。相较于化学合成,合成生物学在生物发酵方面体现了一定的优势,它是利用基因编辑技术,菌株培育及筛选等方面对生物原料进行加工合成,且其菌种是经合理设计并优化的,可通过操纵代谢途径上的基因使目标产物产量达到最大化。
合成生物学制造的整个生产链可概括为原料选择、底盘细胞的构建优化以及产品生产三个部分,其中底盘细胞的构建优化是合成生物学的核心。以合成生物学为理论指导,通过设计最优代谢途径、模块组装适配等调控代谢通量,从而选育出具有“节能、降耗、抗逆”等优良特性的底盘细胞[2]。目前合成生物学所面临的最大挑战是如何根据终产物和底盘细胞的代谢特点合理设计产品合成路径,从而构建出具有特定功能的人工制造体系,未来仍需不断的技术优化才能获得较为理想的细胞工厂。
2. 基于合成生物学底盘细胞构建优化的策略
通过合成生物学的技术手段和策略对微生物进行设计改造,可以使底盘生物实现特定的生物学功能,从而以高效安全的方式合成具有较高价值的生物产品。以下主要总结了利用合成生物学策略在构建理想微生物底盘细胞时的应用,从而为构建高效的细胞工厂奠定理论基础。
2.1 基因组精简
2.1.1 基因组精简的技术路线
基因组精简是构建微生物底盘细胞的策略之一,适度的基因组缩减可以改造细胞的遗传稳定性,优化细胞代谢途径,改善细胞对能量的利用率等。微生物底盘细胞的构建目前主要采用“自底向上”和“自顶向下”的策略[2−3]。
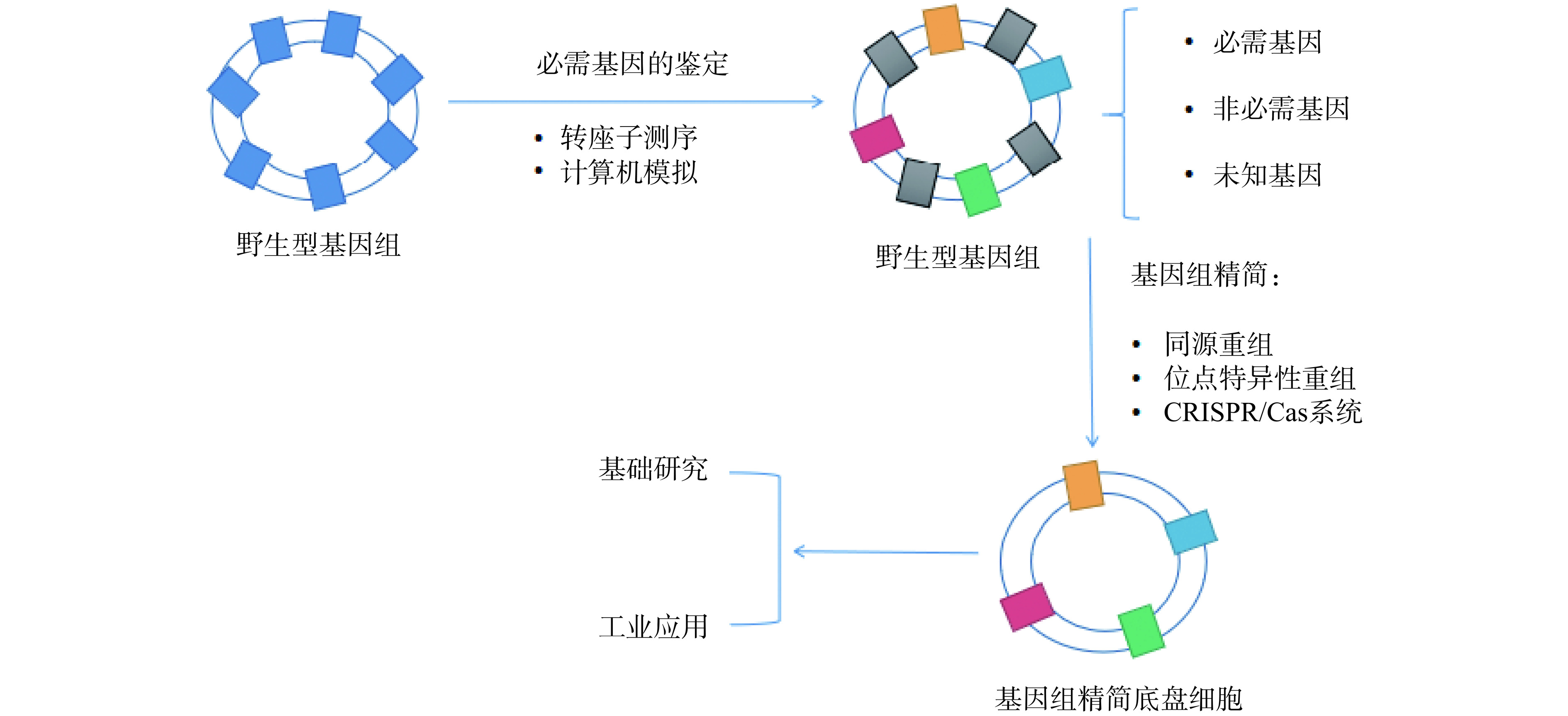
自底向上路线是通过理性设计,采用从头合成路线,将人工合成的标准化元件、功能基因模块逐步组装起来以合成最小基因组[2],但这种水平的工程需要对所有的生物过程和相互作用,以及基因组稳定性有一个全面的认识,因此该方法在对菌株进行改造时具有一定的局限性。与自底向上路线相比,自顶向下路线更有望获得有效和易处理的底盘细胞。自顶向下路线是对遗传背景已知的基因组进行大片段删减操作,尽可能去除非必需基因DNA序列,逐步缩减基因组的规模以构建最小化基因组细胞[2](图2)。其主要优点是可操作性强,由删除引起的任何不良变化都可以通过恢复到最近一次删除之前来进行补救。目前该方法已应用于各种生物产品的生产,如氨基酸,维生素,生物能源等[4]。
2.1.2 必需基因的鉴定
在利用自顶向下路线对基因组改造时首先需要鉴别基因的必需性,通过识别必需基因和非必需基因确定核酸序列片段中需要删除的靶点[5]。必需基因主要参与维持细胞的基本生命活动,DNA的复制、传递遗传信息等重要生理功能[6],因此必需基因与细胞的存活密不可分,它的缺失通常会导致细胞死亡。基因的必需性通常与环境条件及科研工作者对其预期应用密切相关,如氨基酸合成基因在特定的最小培养基上生长时被认为是必需基因,而在营养丰富的复合培养基上不一定是必需的[3]。目前确定基因必需性主要有比较基因组学、基因失活和计算机模拟等方法,这三种方法对基因组设计和必需基因的确定各有利弊,通常会组合运用共同确定必需基因。
比较基因组学法是将目的菌株与数据库中近亲和远亲菌株必需基因的序列相似性进行比对,从而推测基因的必需性。如Mushegian等[7]通过比较远亲细菌的基因组,发现它们之间共有262个必需基因,而当更多的物种被加入到这个比较中时,基因组缩小到63个共同的核心基因,因此该方法的准确性与参照物种密切相关。目前基于近亲菌株的基因序列比对较为准确,而基于远缘物种的比较差别较大,因此在利用该方法鉴定必需基因时可同时利用在线软件预测非必需基因,从而使结果更为可靠。
基因失活是指将某基因失活后确定菌株的存亡从而鉴定出必需基因。利用该方法不仅可以确定必需基因,同时还能鉴定出部分非必需基因[8]。由于基因间的相互作用,多个基因的组合缺失可能导致对基因必需性的错误鉴定,因此该方法虽然准确率高,但工作量巨大,且可能错误地将同工酶基因鉴定为非必需基因,导致敲除效率低的同时也会影响鉴定结果[3,8]。基于以上因素可以考虑结合基因组序列比对法和代谢网络模型分析来提高筛选效率。
计算机模拟是利用基因组序列注释信息、构建相关基因组尺度代谢模型,结合代谢反应、相关理化因素等利用通量平衡分析评估代谢网络的理论性能[9]。目前已开发的计算程序和模型(如DEG、OGEE、CEG等数据库)一定程度上填补了确定必需基因的耗时和昂贵的实验方法的空白,但未来仍需进一步改进以作为独立的鉴定必需基因的工具[8−10]。表1总结了以上三种方法的优缺点。
表 1 必需基因的鉴定方法Table 1. Methods for identifying essential genes方法 定义 优点 缺点 参考文献 比较基因组学 将目的菌株与数据库中近缘菌株必需基因的序列相似性进行比对,从而推测基因的必需性 易确定所有物种生命活动所必需的基因 具相似功能的蛋白质不一定具有序列相似性,因此最小基因集的大小可能被低估,且存在基因的环境依赖性 [6] 基因失活 利用相关技术使基因不表达,必需基因的失活必将导致细胞死亡;常用技术有转座子测序(TraDIS、Inseq、TnSeq)、LoxTnSeq、CRISPRi-Seq等 可以快速识别基因组中的非必需基因,效率高,应用广泛 错误的将某些不含转座子的小基因鉴定为必需基因;存在缺失合成对致死情况 [8] 计算机模拟 利用基因组序列注释信息、构建基因组尺度代谢模型,结合代谢反应、相关理化因素以及对细胞生理表型行为的限制约束条件,利用通量平衡分析评估代谢网络的理论性能(如DEG、pDEG、OGEE、CEG等数据库) 数据库大,高效,应用广泛 预测结果的准确性与代谢模型的精度相关,在代谢网络模型精度及完善程度欠缺的情况下,预测结果的准确性受较大影响 [3,8−11] 2.1.3 靶向删除非必需基因
在删除非必需基因的过程中,主要是对原噬菌体、插入序列和基因组岛等无关基因簇进行舍弃,这些非必需基因簇已被证明是可以删除且不影响菌株生长特性的,因此通过删除非必需基因精简基因组可构建具有生长稳健性和遗传稳定性的微生物底盘,且有望使精简菌株过量生产目标产物,这也可提高细胞生理性能的预测性和可控性。其中基因编辑技术起着至关重要的作用,迄今对基因组缺失的常规方法主要有同源重组、位点特异性重组、CRISPR/Cas系统等。
同源重组是DNA分子之间同源区域通过交换实现序列重组的过程,常引入负筛选标记基因以提高无痕敲除突变菌株的效率[12]。如Reuß等[13]在枯草芽孢杆菌MGB469基础上,采用基于同源重组原理的无痕敲除技术获得了菌株MBG874,该菌株使得重组菌纤维素酶和蛋白激酶的产量显著提高,分别达到了亲本菌株的1.7和2.5倍;位点特异性重组是通过特异性酶识别相应位点并删除位点间基因片段,主要的重组系统有Flp/FRT和Cre/loxP[8]。如Kitagawa等[14]采用Cre/loxP重组系统删除了红球菌的七个基因组区域,占总基因组的10%,所得的突变株基因结构稳定,且能够消除阻碍异源基因表达的不必要的功能基因;CRISPR/Cas系统是用于抵抗存在于噬菌体或质粒的外源遗传元件入侵的一种防御机制[15],目前开发了多种CRISPR/Cas系统用于微生物基因组的编辑,主要包括CRISPR-Cas9、CRISPR-Cas9n和CRISPR-Cpf1/dCpf1等。如Huang等[16]利用该系统对大肠杆菌基因组简化,总共累积缺失基因组370 kb,突出其在合成生物学和代谢工程中的应用潜力。表2总结了上述方法的优缺点。
表 2 常用的基因编辑技术Table 2. Common genome editing techniques方法 定义 优点 缺点 参考文献 同源重组 DNA分子之间同源区域通过交换实现序列重组的过程 可实现无痕敲除 需引入负筛选标记,效率较低,易出现回复突变,不利于筛选出突变株 [12] 位点特异性
重组通过特异性酶识别相应位点并删除位点间基因片段;主要的重组系统有Flp/FRT和Cre/loxP 敲除效率高,
操作方便染色体中会残留重组位点,从而会干扰下一轮位点的特异性识别 [8,14] CRISPR/
Cas系统用于抵抗存在于噬菌体或质粒的外源遗传元件的入侵的一种防御机制,在sgRNA的引导下,cas蛋白对靶位点进行切割;主要包括CRISPR-Cas9/12、CRISPR-Cas9n和CRISPR-Cpf1/dCpf1等 操作简便,编辑效率高效且通用性广 存在较高的脱靶效应,具有一定的
细胞毒性[15,17] 2.2 动态调控
近年来,合成生物学的快速发展为微生物底盘细胞的构建优化提供了技术支持,在改善微生物生长性能、提高碳源利用率和生物分子生产等方面均有重要应用[18]。因此,未来如何提高人工合成途径与底盘细胞的适配性已成为研究热点。
动态调控是通过理性设计基因线路以精确调节细胞生长和目标产物生产之间的能量代谢平衡,目前已成为微调微生物细胞工厂代谢通量的重要策略[19−20]。许多科学家利用生物传感器、代谢调控开关等动态调节代谢合成途径中的基因,从而使目标产物的生产在不影响细胞的生长情况下达到最大化(见表3)。如刘龙课题组在枯草芽孢杆菌中创建了一种可编程和双功能丙酮酸响应遗传回路,用于动态激活和抑制中心代谢通量。通过使用这些遗传回路设计一个反馈回路控制系统,该系统依靠丙酮酸的细胞内浓度以平衡细胞生长和葡萄糖酸的合成,同时阻止副产物的形成,最终使葡萄糖酸滴度从207增加到802 mg/L,目前利用该系统动态控制中心代谢通量已成为构建理想微生物细胞工厂的一种有效工具[19]。尽管目前已开发出多种策略以动态调节基因的表达,但响应这些所设计的调节元件常常是针对某一特定生物大分子,因此未来如何扩大调节元件的范围使细胞可动态控制多条代谢途径将会成为面临的主要问题。
表 3 动态调控在构建底盘菌株中的应用Table 3. Application of dynamic regulation in the construction of chassis strains调控系统 诱导剂 菌株 产物 调控效果 参考文献 代谢物依赖型调控系统 乙酰磷酸 大肠杆菌 番茄红素 18倍 [24] 丙二酰辅酶A 大肠杆菌 脂肪酸 2.1倍 [25] 丙酮酸 枯草芽孢杆菌 葡萄糖酸 802 mg/L [19] 光调控系统 光 大肠杆菌 莽草酸 76 g/L [21] 光 大肠杆菌 异丁醇 4倍 [26] 温度调控系统 温控开关 大肠杆菌 聚羟基脂肪酸酯 − [22] 温度 酵母 番茄红素 1.12 g/L [27] 群体感应调控系统 AHLs 大肠杆菌 水杨酸、4-羟基香豆素 523.2 mg/L、11.3倍 [23] AHLs 大肠杆菌 葡萄糖二酸 2 g/L [27] 2.3 适应性进化工程
由于细胞代谢和调控网络的复杂性,尤其是对于多基因调控的复杂性状和遗传工具有限的生物系统而言,通过人工理性设计及基因扰动难以获得较好表型,因此基因组进化在微生物细胞工厂的构建中发挥着重要的作用。
微生物适应性进化工程通过模拟自然进化过程,构建大量遗传多样性的突变库,经过筛选可快速获得稳定性状的菌株,其优势在于可在较短时间内根据定向选择适应特定条件的耐受工业环境的有效细胞,从而提高微生物生产性能[28-29]。如:a.提高微生物生长速率。在微生物的代谢过程中常常因多基因的调控干扰了生理机能,降低了生长速率甚至造成生长缺陷,因此通过适应性进化可改善菌株的生长情况。通过将菌株置于一个特定环境中(高渗透压、高酸碱物质浓度下等),从中反复筛选生长良好的菌株,从而实现菌株进化。如Choe等[29]通过适应性进化来优化基因组减少的大肠杆菌的生长性能,并且一定程度上协调了代谢和生长,为未来构建大肠杆菌底盘提供了理论依据;b.增加耐受性。当细胞处于一定压力下(如生长环境和生物分子胁迫等),细胞即产生应激从而造成细胞生理变化,甚至导致代谢失调,因此在发酵过程中通过适应性进化可获得对某一底物或产物的耐受性。为提高菌株发酵后期的耐受性,Sun等[30]通过紫外诱变构建突变文库富集优势菌株,最终构建的大肠杆菌底盘菌株的耐受性明显增加,且2'-岩藻糖基乳糖的产量达到最高;c.平衡细胞生长和生产。在利用微生物生产目标产物的过程中,常常因为细胞生长与生产不平衡造成代谢分流或生长缺陷,因此通过适应性进化也可以平衡生长与生产,从而提高产物产量,进一步激发底盘菌株的生产能力。目前还开发了多种基因组规模的多位点快速进化工具,并结合计算机辅助进化系统实现了自动化连续进化技术,如在构建高效的酿酒酵母底盘细胞时开发了基因组重排进化技术(SCRaMbLE),这些技术的新兴发展可在单一细胞中实现多个不同性状的同时改造,缩短了改造时间,为提升微生物底盘细胞的生长速度和使其具有较强的环境耐受等较优生理特性提供了技术支撑[31-32]。
2.4 辅因子工程
辅因子NADH/NAD+和NADPH/NADP+比例在生物有机体中起着重要的作用,许多目标产物的生物合成都涉及到NAD(P)H氧化或NAD(P)+还原,它们的比例调节生物体代谢流的方向。因此,维持氧化还原平衡是增加目标产物生物合成途径代谢通量,及提高目标产物产量的重要手段之一。
目前主要有四种策略提高辅因子在生物代谢合成中的作用:重构辅因子的生物合成途径;提升胞内的辅因子代谢水平;平衡辅因子稳态及提高辅因子的偏好性。酚酸的生物合成需要辅因子NADPH和FAD(H)2,中科院周雍进团队在酿酒酵母中改造了中心代谢途径以提高NADPH,同时通过构建FAD(H)2合成途径增加FAD(H)2供应,最终显著提高酚酸产量,该研究为辅因子调控提供了理论指导[33];江南大学Li等[34]在谷氨酸棒杆菌产L-高丝氨酸的研究中,构建一个双通道糖酵解以平衡NADPH和NADH的供应,主要通过改造磷酸戊糖途径,引入来自不同菌编码NADH依赖性天冬氨酸脱氢酶aspB基因,将NADPH的消耗过程转化为消耗NADH,改造启动子和引入可利用NADP+作为辅因子的gapN基因等策略以提高高丝氨酸的产量和NADPH,最终作者证明了当NADH/NAD+和 NADPH/NADP+比例达到一定阈值后,菌株可通过调节糖酵解路径和GapN通道的通量来维持辅因子平衡,从而保证高糖酵解速率,促进目标产物的合成。通过开发和利用辅因子工程策略,同时还可利用高通量筛选辅助,进一步推动辅因子工程在代谢合成和底盘细胞构建中的应用。
2.5 蛋白骨架技术
蛋白骨架技术指通过改变酶与底物间的距离,使酶按照特定空间位置锚定在骨架上,从而提高酶与底物的结合效率,进而提高目标产物的合成速率[35]。该技术主要是通过调节酶的催化效率从而提高产物的合成效率,其开发无疑将成为合成生物学的一个研究热点。
在构建微生物细胞工厂中,参与代谢途径中反应的酶与底物间的距离以及相邻酶所处的位置均是影响代谢效率的重要因素,因此通过人工合成的蛋白质骨架技术可以提高催化效率,进而提高目标产物产量。Kang等[35]通过利用短肽标签RIAD-RIDD的相互作用在甲萘醌生物合成途径中进行组装,产生了具有不同催化效率的蛋白质纳米颗粒,且在大肠杆菌中将上游甲羟戊酸途径的最后一种酶与下游类胡萝卜素途径的第一种酶组装在一起,最终使类胡萝卜素的产量增加了5.7倍,同样的策略使工程酿酒酵母的番茄红素产量增加了58%;Ledesma等[36]利用四肽重复亲和蛋白(Tetrapeptide Repeat Affinity Proteins,TRAPs)作为生物催化的支架,构建了一个纳米级的多酶组装系统,然后对TRAPs进行编程以识别与酶融合的肽标签,此外,该支架蛋白还可通过相互作用选择性地隔离反应中涉及的辅因子,从而增加局部浓度,提高其催化效率。因此,利用蛋白支架将多酶进行近距离排列,可以触发底物通道效应,从而实现辅因子的高效利用和级联催化。然而未来如何利用蛋白支架实现酶的精确组装仍具有一定挑战性。
2.6 高通量筛选
高通量筛选为微生物细胞工厂的构建提供了一定的技术支持,主要由高容量化合物库、自动化操作、高灵敏度检测、高特异筛选模型、高效率数据处理5个子系统组合而成,具有高特异性、灵敏度和筛选效率高等特点[37]。
通过对微生物改造会产生巨大的筛选文库,且传统筛选效率较低,因此近年来开发的多种高通量筛选技术,如自动化液体处理平台、荧光激活细胞分选技术、液滴微流控分选技术和基于生物传感器筛选策略等为克服以上问题提供了技术支撑,且在高效筛选微生物底盘细胞方面有着重要应用。广东省科学院研究所Su等[38]带领的团队使用赖氨酸核糖开关构建了赖氨酸生物传感器,并利用该传感器开发出高通量筛选技术以增强嵌合天冬氨酸激酶在大肠杆菌中的性能,最终使L-苏氨酸产量得到大幅提高,为未来选育高产L-苏氨酸菌株提供了新的方法;Gao等[39]利用响应L-半胱氨酸的转录调控因子CcdR,开发了L-半胱氨酸生物传感器,将其与流式细胞分选技术偶联建立了L-半胱氨酸高通量筛选平台,最终L-半胱氨酸产量显著提高。因此,通过将高通量筛选平台和其他代谢工程策略(如构建启动子和RBS文库、适应性进化、辅因子工程等)相结合可以提高生物分子的生产,为进一步构建微生物细胞工厂奠定基础。
3. 常见工业微生物底盘的构建及应用
在合成生物学中底盘细胞是各微生物进一步改造优化的基础[40]。迄今已开展了对酿酒酵母、大肠杆菌、枯草芽孢杆菌、谷氨酸棒状杆菌、假单胞菌等工业微生物底盘细胞构建的研究。以下主要总结了目前利用合成生物学手段在构建代表性底盘细胞方面的研究进展。
3.1 酿酒酵母
酿酒酵母(Saccharomyces cerevisiae)具有生长繁殖快,遗传操作简单等优势[41]。目前,酿酒酵母已作为重要的模式生物和微生物细胞工厂广泛用于下游生产。基因遗传改造技术的开发将有利于推动酿酒酵母底盘细胞的构建,新兴的CRISPR/Cas系统在酿酒酵母中已被应用于生产各种产物(如有机酸、糖醇和氨基酸类等),通过对启动子、终止子等基因表达元件改造,将有助于提高菌株的遗传稳定性;在基因表达调控方面,通过提高基因拷贝数、密码子优化以及动态调控等策略,将能最大限度的提高终产物产量。以上的研究策略及手段将为酿酒酵母底盘细胞的改造与应用提供理论和技术支撑。
在精简基因组方面,中国科学院深圳先进技术研究院戴俊彪等开发了SGC(SCRaMbLE-based genome compaction)人工基因组高效简化策略,并以此方法成功删除了第十二号染色体左臂中超过一半的非必需基因,为第一个最小真核基因组的构建及理解真核生命的核心组成奠定了理论和技术基础[42]。在代谢途径设计优化方面,Xu等[43]先构建了甜茶苷和莱鲍迪苷从头生物合成途径,再通过基因敲除和融合不同的短肽标签等遗传操作消除其中的限速步骤,最后通过合成生物学手段和系统工程策略对细胞适应性的压力和不平衡的代谢网络进行改造优化,最终使甜茶苷和莱鲍迪苷的滴度分别达到了1368.6 mg/L和132.7 mg/L,为高糖饮食带来的健康问题提供了一种可行的糖替代品。目前对酿酒酵母的研究不仅集中在途径优化上,对辅因子的改造也日渐流行,有研究者对酿酒酵母系统设计三种辅助因子(FADH2、S-adenosyl-L-methion和NADPH)的生物合成、区域化和循环,从而高水平生产咖啡酸和阿魏酸,该策略为调节其他辅因子的代谢过程提供了依据[33]。
3.2 大肠杆菌
大肠杆菌(Escherichia coli)具有生长速度快和易于遗传操作等优点,因此它是被用作遗传和基因组工程的重要微生物细胞模型[44]。迄今已实现利用大肠杆菌底盘生产氨基酸、有机醇和天然产物等生物产品。
通过理性设计对基因组大规模精简,以减少不必要的能量和资源消耗,从而改善底盘细胞的遗传性能,如Ma等[45]在构建代谢工程底盘细胞时利用转座子介导的随机缺失(Transposon-Mediated Random Deletion,TMRD)对E. coli MG1655基因组精简,其细胞生长速率与野生型相当,而电穿孔效率至少提高了2个数量级,同时增强了细胞的环境适应性;此外,对底盘菌株生产生物分子的改造主要集中于合成途径的创建优化及细胞全局优化等方面[44],CRISPR/Cas基因编辑技术的新兴发展在构建底盘细胞时提高了基因编辑效率,通过对靶基因进行微调或干扰,最终可实现目标产物的高产。在对大肠杆菌底盘改造时,常利用动态调控微调基因以平衡细胞生长和生产,改变密码子偏好性和适应性进化等策略不断提高目标产物的生产和底盘菌株的稳定性。如Qi等[46]利用高通量筛选和定向进化等策略对代谢途径进行改造优化,先对来自大肠杆菌BL4(DE3)的21-羟基苯乙酸3-羟化酶(EcHpaB)建立了随机突变文库,提高了EcHpaB将酪醇转化为羟基酪醇的能力,且对突变株采用了定向进化,最终使羟基酪醇(HT)的产率达到7.43 g/L,该研究为高效生产HT提供了一种可行的技术手段。
3.3 枯草芽孢杆菌
枯草芽孢杆菌(Bacillus subtilis)是一种革兰氏阳性细菌,具有强大的蛋白表达系统。目前,构建该模式生物底盘细胞的研究已成为热点之一。迄今通过对枯草芽孢杆菌进行合成生物学和代谢工程的改造已使其适用于核苷类、抗生素和分泌蛋白酶等生物化学产品的生产[13]。
在枯草芽孢杆菌底盘细胞的构建中,动态调控、膜工程改造以及酶组装等策略为其提供了理论支撑,且通过CRISPR-Cas系统实现了多基因的精确调控,如Wu等[47]在枯草芽孢杆菌中创建了自主双调节系统以提高N-乙酰氨基葡萄糖的生产,通过对多个靶点动态控制可实现不同合成途径间的代谢平衡,从而有利于目标产物的合成。近年来新兴发展基因编辑技术也为枯草芽孢杆菌细胞工厂的构建提供了技术支持,如利用蛋白支架和动态调控策略与CRISPR-Cas系统相结合以高产核黄素、透明质酸和萜类化合物等,其中N-乙酰氨基葡萄糖的产量已达到130 g/L[48]。总之,通过遗传操作增加了所需产品的产量和细胞密度,从而利于下游产品的生产与应用。
3.4 谷氨酸棒状杆菌
谷氨酸棒状杆菌(Corynebacterium glutamicum)是一种革兰氏阳性细菌,因其无毒且遗传背景清晰而被广泛应用于食品、医药领域[49],如利用谷氨酸棒状杆菌生产氨基酸、有机酸和天然产物等多种产品。
近年来越来越多的遗传元件(如启动子、核糖体结合位点等)已经被挖掘、表征和应用,且经典基因编辑技术不断改进,如基于同源重组的单/双交换、位点特异性重组等加速了谷氨酸棒状杆菌的分子研究,在改善生长特性,增加生物分子产生等方面有重要意义。如Baumgart等[50]于2013年利用同源重组原理对谷氨酸棒杆菌基因组删除了三个噬菌体片段(CGP1、CGP2、CGP3),占总基因组的6%,获得了精简菌株MB001,其转化效率和质粒拷贝数均有所增加,生长情况和环境适应性均有所改善;由于微生物代谢网络机制的复杂性,对其采用单一调控可能会导致代谢失衡,因此近年新兴的动态调控策略一定程度上平衡了细胞的代谢流,如Wei等[51]通过耦合生长依赖型启动子及蛋白降解标签设计了双功能遗传开关(GABS),以动态平衡细胞生长与γ-氨基丁酸(GABA)间代谢通量的平衡;随着编辑系统不断更新、优化,郑平课题组开发了基于CRISPR-dCpf1的多基因表达调控技术,使多个目标基因实现快速表达调控,该系统可达到更加精准的编辑效果,且解决了细胞毒性问题,一定程度上还降低了脱靶效应[52]。此外,高通量筛选技术在筛选具有优良性状的谷氨酸棒状杆菌底盘中也有重要应用,如薛宁等[53]开发了一种基于L-苯丙氨酸的生物传感器,可以实现对L-苯丙氨酸浓度的准确测定,并与液滴微流控高通量筛选技术结合,从而实现L-苯丙氨酸高产菌株的快速筛选。这些技术为谷氨酸棒状杆菌底盘细胞的构建优化提供支持,实现了多种氨基酸的生物合成。
恶臭假单胞菌KT2440(Pseudomonas putida KT2440)常用于生产聚羟基脂肪酸(polyhydroxyalkanoate,PHA)等聚合物,在合成塑料、医疗产品中具有较高应用价值[54]。Cdvorak等[54]采用无痕敲除技术删除恶臭假单胞菌中的基因组岛构建了菌株KTU-U13,该菌株PHA产量和细胞干重显著提高,且改善了细胞对能量的利用率,为未来利用该菌株生产PHA创造了一个优良底盘;Fan等[55]利用CRISPR/Cas9系统减少恶臭假单胞菌7.7% 的基因组,构建了菌株NKU421,其ATP/ADP比率提高了11倍,同时PHA产量较亲本菌株提高了114.8%。因此,利用恶臭假单胞菌底盘细胞生产生物产品具有较大的应用前景。
乳酸乳球菌(Lactococcus lactis)被用作食品添加剂,在微生物细胞工厂中具有重要的作用[56]。Qiao等[56]通过缺失原噬菌体和基因组岛使乳酸乳球菌N8的基因组减少了6.9%,提高了乳链菌肽产量和转化效率;张雅婷[57]实现了菌株NZ9000占全基因组8.566%的非必需基因的删减,菌株显示出生长快,碳源利用能力强等优点;在最近的研究中通过删除原噬菌体相关片段构建出精简菌株N8-1,在各种胁迫环境下与野生型菌株对比发现,精简菌株N8-1在39 ℃时仍能保持生长,而野生型生长急剧下降[57]。目前关于乳酸乳球菌微生物底盘菌株的构建还具有许多局限性,如在面对极端环境压力时,如何利用底盘细胞实现有价值化合物的高产以及对乳酸乳球菌胁迫耐受机制的研究等,这将为未来更深层次的探索提供指引。
4. 结论
微生物野生型菌株由于基因组十分复杂,且异源合成途径的引入会产生代谢负担,因此将其作为理想宿主用于工业生产具有一定的局限性,且传统的发酵工业有诸多亟待解决的问题,如发酵菌株的不稳定性,发酵的生产周期长且发酵过程中有诸多副产物,从而会增加生产产品的成本,而构建微生物小基因组细胞工厂可以弥补传统发酵工业的弊端。
微生物底盘细胞的设计构建,是通过对复杂生命体的工程化重构,实现目标产品的可控和高效合成。
随着基因编辑效率的提高、脱靶效应的降低和新方法的不断更新,使构建理想的底盘细胞成为可能[1,58]。未来依然需加大对工业微生物的基因组学分析和探索高产性状的分子机制,最终对工业微生物的基因组改造实现可预测、可控制的调控。由于传统单一策略的代谢工程改造对于性状改善的空间有限,需要借助合成生物学的工程改造手段,例如可通过适应性实验室进化和动态调控策略对工业微生物的代谢途径进行调控,有效分配底盘细胞的代谢负担,同时还可结合高通量分析手段,缩短育种周期,从而获得工业生产所需的优良工业微生物。总之,尽管构建理想的底盘菌株还面临着许多挑战,但未来利用底盘细胞高产生物分子将具有更加广阔的应用前景。
-
表 1 必需基因的鉴定方法
Table 1 Methods for identifying essential genes
方法 定义 优点 缺点 参考文献 比较基因组学 将目的菌株与数据库中近缘菌株必需基因的序列相似性进行比对,从而推测基因的必需性 易确定所有物种生命活动所必需的基因 具相似功能的蛋白质不一定具有序列相似性,因此最小基因集的大小可能被低估,且存在基因的环境依赖性 [6] 基因失活 利用相关技术使基因不表达,必需基因的失活必将导致细胞死亡;常用技术有转座子测序(TraDIS、Inseq、TnSeq)、LoxTnSeq、CRISPRi-Seq等 可以快速识别基因组中的非必需基因,效率高,应用广泛 错误的将某些不含转座子的小基因鉴定为必需基因;存在缺失合成对致死情况 [8] 计算机模拟 利用基因组序列注释信息、构建基因组尺度代谢模型,结合代谢反应、相关理化因素以及对细胞生理表型行为的限制约束条件,利用通量平衡分析评估代谢网络的理论性能(如DEG、pDEG、OGEE、CEG等数据库) 数据库大,高效,应用广泛 预测结果的准确性与代谢模型的精度相关,在代谢网络模型精度及完善程度欠缺的情况下,预测结果的准确性受较大影响 [3,8−11] 表 2 常用的基因编辑技术
Table 2 Common genome editing techniques
方法 定义 优点 缺点 参考文献 同源重组 DNA分子之间同源区域通过交换实现序列重组的过程 可实现无痕敲除 需引入负筛选标记,效率较低,易出现回复突变,不利于筛选出突变株 [12] 位点特异性
重组通过特异性酶识别相应位点并删除位点间基因片段;主要的重组系统有Flp/FRT和Cre/loxP 敲除效率高,
操作方便染色体中会残留重组位点,从而会干扰下一轮位点的特异性识别 [8,14] CRISPR/
Cas系统用于抵抗存在于噬菌体或质粒的外源遗传元件的入侵的一种防御机制,在sgRNA的引导下,cas蛋白对靶位点进行切割;主要包括CRISPR-Cas9/12、CRISPR-Cas9n和CRISPR-Cpf1/dCpf1等 操作简便,编辑效率高效且通用性广 存在较高的脱靶效应,具有一定的
细胞毒性[15,17] 表 3 动态调控在构建底盘菌株中的应用
Table 3 Application of dynamic regulation in the construction of chassis strains
调控系统 诱导剂 菌株 产物 调控效果 参考文献 代谢物依赖型调控系统 乙酰磷酸 大肠杆菌 番茄红素 18倍 [24] 丙二酰辅酶A 大肠杆菌 脂肪酸 2.1倍 [25] 丙酮酸 枯草芽孢杆菌 葡萄糖酸 802 mg/L [19] 光调控系统 光 大肠杆菌 莽草酸 76 g/L [21] 光 大肠杆菌 异丁醇 4倍 [26] 温度调控系统 温控开关 大肠杆菌 聚羟基脂肪酸酯 − [22] 温度 酵母 番茄红素 1.12 g/L [27] 群体感应调控系统 AHLs 大肠杆菌 水杨酸、4-羟基香豆素 523.2 mg/L、11.3倍 [23] AHLs 大肠杆菌 葡萄糖二酸 2 g/L [27] -
[1] MA S, SU T Y, LU X M, et al. Bacterial genome reduction for optimal chassis of synthetic biology:A review[J]. Critical Reviews in Biotechnology,2023,28:1−14.
[2] WANG L, MARANAS C D. Mingenome:An in silico top-down approach for the synthesis of minimized genomes[J]. ACS Synthetic Biology,2018,7(2):462−473. doi: 10.1021/acssynbio.7b00296
[3] LEBLANC N, CHARLES T C. Bacterial genome reductions:Tools, applications, and challenges[J]. Frontiers in Genome Editing,2022,4:957289. doi: 10.3389/fgeed.2022.957289
[4] 林璐, 吕雪芹, 刘延峰, 等. 枯草芽孢杆菌底盘细胞的设计、构建与应用[J]. 合成生物学,2020,1(2):247−265. [LIN L, LÜ X Q, LIU Y F, et al. Design, advances in design, construction and applications of Bacillus subtilis chassis cells[J]. ACS Synthetic Biology,2020,1(2):247−265.] doi: 10.12211/2096-8280.2020-030 LIN L, LÜ X Q, LIU Y F, et al. Design, advances in design, construction and applications of Bacillus subtilis chassis cells[J]. ACS Synthetic Biology, 2020, 1(2): 247−265. doi: 10.12211/2096-8280.2020-030
[5] SOLANA J, GARROTE S E, GIL R. DELEAT:Gene essentiality prediction and deletion design for bacterial genome reduction[J]. BMC Bioinformatics,2021,22(1):444. doi: 10.1186/s12859-021-04348-5
[6] GOODALL E C A, ROBINSON A, JOHNSTON I G, et al. The essential genome of Escherichia coli K-12[J]. Cold Spring Harbor Laboratory,2018,9(1):e02096−17.
[7] MUSHEGIAN A R, KOONIN E V. A minimal gene set for cellular life derived by comparison of complete bacterial genomes[J]. Proceedings of the National Academy of Sciences of the United States of America,1996,93(19):10268−10273.
[8] SHAW D, MIRAVET V S, PINERO C L, et al. LoxTnSeq:Random transposon insertions combined with cre/lox recombination and counterselection to generate large random genome reductions[J]. Cold Spring Harbor Laboratory,2021,14(6):2403−2419.
[9] DONG C, JIN Y T, HUA H L, et al. Comprehensive review of the identification of essential genes using computational methods:Focusing on feature implementation and assessmen[J]. Briefings in Bioinformatics,2020,21(1):171−181.
[10] LIN Y, ZHANG F Z, XUE K, et al. Identifying bacterial essential genes based on a feature-integrated method[J]. IEEE/ACM Transactions on Computational Biology and Bioinformatics (TCBB),2019,16(4):1274−1279. doi: 10.1109/TCBB.2017.2669968
[11] LUO H, LIN Y, LIU T, et al. DEG 15, an update of the database of essential genes that includes built-in analysis tools[J]. Nucleic Acids Research,2021,49(D1):D677−D686. doi: 10.1093/nar/gkaa917
[12] ZHAO Z H, SHANG P, SAGE F, et al. Ligation-assisted homologous recombination enables precise genome editing by deploying both MMEJ and HDR[J]. Nucleic Acids Research,2022,50(11):e62. doi: 10.1093/nar/gkac118
[13] REUß D R, ALTENBUCHNER J, MADER U, et al. Large-scale reduction of the Bacillus subtilis genome:Consequences for the transcriptional network, resource allocation, and metabolism[J]. Genome Research,2017,27(2):289−299. doi: 10.1101/gr.215293.116
[14] KITAGAWA W, HATA M. Development of efficient genome-reduction tool based on cre/loxP system in Rhodococcus erythropolis[J]. Microorganisms,2023,11(2):268. doi: 10.3390/microorganisms11020268
[15] MAKAROVA K S, WOLF Y I, IRANZO J, et al. Evolutionary classification of CRISPR-Cas systems:A burst of class 2 and derived variants[J]. Nature Publishing Group,2020,18(2):67−83.
[16] HUANG C Y, GUO L W, WANG J G, et al. Efficient long fragment editing technique enables large-scale and scarless bacterial genome engineering[J]. Applied Microbiology and Biotechnology,2020,104(18):7943−7956. doi: 10.1007/s00253-020-10819-1
[17] SONG G X, ZHANG F, TIAN C H, et al. Discovery of potent and versatile CRISPR-Cas9 inhibitors engineered for chemically controllable genome editing[J]. Nucleic Acids Research,2022,50(5):2836−2853. doi: 10.1093/nar/gkac099
[18] CHAI M, DENG C, CHEN Q, et al. Synthetic biology toolkits and metabolic engineering applied in Corynebacterium glutamicum for biomanufacturing[J]. ACS Synthetic Biology,2021,10(12):3237−3250. doi: 10.1021/acssynbio.1c00355
[19] XU X H, LI X L, LIU Y F, et al. Pyruvate-responsive genetic circuits for dynamic control of central metabolism[J]. Nature Chemical Biology,2020,16(11):1261−1268. doi: 10.1038/s41589-020-0637-3
[20] ZHOU S H, YUAN S F, NAIR P H, et al. Development of a growth coupled and multi-layered dynamic regulation network balancing malonyl-CoA node to enhance (2S)-naringenin biosynthesis in Escherichia coli[J]. Metabolic Engineering,2021,67:41−52. doi: 10.1016/j.ymben.2021.05.007
[21] KOMERA I, GAO C, GUO L, et al. Bifunctional optogenetic switch for improving shikimic acid production in E. coli[J]. Biotechnology for Biofuels and Bioproducts,2022,15(1):13. doi: 10.1186/s13068-022-02111-3
[22] WANG X, HAN J N, ZHANG X, et al. Reversible thermal regulation for bifunctional dynamic control of gene expression in Escherichia coli[J]. Nature Communications,2021,12(1):1411. doi: 10.1038/s41467-021-21654-x
[23] GE C, YU Z, SHENG H K, et al. Redesigning regulatory components of quorum-sensing system for diverse metabolic control[J]. Nature Communications,2022,13(1):2182. doi: 10.1038/s41467-022-29933-x
[24] 叶健文, 陈江楠, 张旭, 等. 动态调控:一种高效的细胞工厂工程化代谢改造策略[J]. 生物技术通报,2020,36(6):1−12. [YE J W, CHEN J N, ZHANG X, et al. Dynamic regulation:An efficient metabolic modification strategy for cell factory engineering[J]. Biotechnology Bulletin,2020,36(6):1−12.] YE J W, CHEN J N, ZHANG X, et al. Dynamic regulation: An efficient metabolic modification strategy for cell factory engineering[J]. Biotechnology Bulletin, 2020, 36(6): 1−12.
[25] XU P, LI L Y, ZHANG F M, et al. Improving fatty acids production by engineering dynamic pathway regulation and metabolic control[J]. Proceedings of the National Academy of Sciences of the United States of America,2014,111(31):11299−11304.
[26] ZHAO E M, ZHANG Y F, MEHL J, et al. Optogenetic regulation of engineered cellular metabolism for microbial chemical production[J]. Nature,2018,555(7698):683−687. doi: 10.1038/nature26141
[27] 于政, 申晓林, 孙新晓, 等. 动态调控策略在代谢工程中的应用研究进展[J]. 合成生物学,2020,1(4):440−453. [YU Z, SHEN X L, SUN X X, et al. Progress in the application of dynamic regulation strategies in metabolic engineering[J]. Acs Synthetic Biology,2020,1(4):440−453.] doi: 10.12211/2096-8280.2020-029 YU Z, SHEN X L, SUN X X, et al. Progress in the application of dynamic regulation strategies in metabolic engineering[J]. Acs Synthetic Biology, 2020, 1(4): 440−453. doi: 10.12211/2096-8280.2020-029
[28] GIBSON B, DAHABIEH M, KROGERUS K, et al. Adaptive laboratory evolution of ale and lager yeasts for improved brewing efficiency and beer quality[J]. Annual Review of Food Science Technology,2020,11:23−44. doi: 10.1146/annurev-food-032519-051715
[29] CHOE D H, LEE J H, YOO M, et al. Adaptive laboratory evolution of a genome-reduced Escherichia coli[J]. Nature Communications,2019,10(1):935. doi: 10.1038/s41467-019-08888-6
[30] SUN X, PENG Z T, LI C, et al. Combinatorial metabolic engineering and tolerance evolving of Escherichia coli for high production of 2'-fucosyllactose[J]. Bioresource Technology,2023,372:128667. doi: 10.1016/j.biortech.2023.128667
[31] LIU W, LUO Z Q, WANG Y, et al. Rapid pathway prototyping and engineering using in vitro and in vivo synthetic genome SCRaMbLE-in methods[J]. Nature Communication,2018,9(1):1936. doi: 10.1038/s41467-018-04254-0
[32] RICHARDSON S M, MITCHELL L A, STRACQUADANIO G, et al. Design of a synthetic yeast genome[J]. Science,2017,355(6329):1040−1044. doi: 10.1126/science.aaf4557
[33] CHEN R B, GAO J Q, YU W, et al. Engineering cofactor supply and recycling to drive phenolic acid biosynthesis in yeast[J]. Nature Chemical Biology,2022,18(5):520−529. doi: 10.1038/s41589-022-01014-6
[34] LI N, LI L H, YU S Q, et al. Dual-channel glycolysis balances cofactor supply for l-homoserine biosynthesis in Corynebacterium glutamicum[J]. Bioresource Technology,2023,369:128473. doi: 10.1016/j.biortech.2022.128473
[35] KANG W, MA T, LIU M, et al. Modular enzyme assembly for enhanced cascade biocatalysis and metabolic flux[J]. Nature Communication,2019,10(1):4248. doi: 10.1038/s41467-019-12247-w
[36] LEDESMA F A, VELASCO L S, SANTIAGO A J, et al. Engineered repeat proteins as scaffolds to assemble multi-enzyme systems for efficient cell-free biosynthesis[J]. Nature Communication,2023,14(1):2587. doi: 10.1038/s41467-023-38304-z
[37] 方莲花, 王月华, 杜冠华. 高通量筛选技术在药物发现中的应用进展[J]. 中国药学杂志,2023,58(4):289−295. [FANG L H, WANG Y H, DU G H. Progress in the application of high-throughput screening techniques in drug discovery[J]. Chinese Journal of Pharmaceuticals,2023,58(4):289−295.] doi: 10.11669/cpj.2023.04.001 FANG L H, WANG Y H, DU G H. Progress in the application of high-throughput screening techniques in drug discovery[J]. Chinese Journal of Pharmaceuticals, 2023, 58(4): 289−295. doi: 10.11669/cpj.2023.04.001
[38] SU B, LAI P X, DENG M R, et al. Design of a dual-responding genetic circuit for high-throughput identification of L-threonine-overproducing Escherichia coli[J]. Bioresource Technology,2024,395:130407. doi: 10.1016/j.biortech.2024.130407
[39] GAO J S, DU M H, ZHAO J H, et al. Design of a genetically encoded biosensor to establish a high-throughput screening platform for L-cysteine overproduction[J]. Metabolic Engineering,2022,73:144−157. doi: 10.1016/j.ymben.2022.07.007
[40] YONEJI T, FUJITA H, MUKAI T, et al. Grand scale genome manipulation via chromosome swapping in Escherichia coli programmed by three one megabase chromosomes[J]. Nucleic Acids Research,2021,49(15):8407−8418. doi: 10.1093/nar/gkab298
[41] SANDERS J, HOFFMANN S A, GREEN A P, et al. New opportunities for genetic code expansion in synthetic yeast[J]. Current Opinion in Biotechnology,2022,75:102691. doi: 10.1016/j.copbio.2022.102691
[42] LUO Z Q, YU K, XIE S Q, et al. Compacting a synthetic yeast chromosome arm[J]. Genome Biology,2021,22(1):5. doi: 10.1186/s13059-020-02232-8
[43] XU Y M, WANG X L, ZHANG C Y, et al. De novo biosynthesis of rubusoside and rebaudiosides in engineered yeasts[J]. Nature Communications,2022,13(1):3040. doi: 10.1038/s41467-022-30826-2
[44] 于勇, 朱欣娜, 毕昌昊, 等. 大肠杆菌细胞工厂的创建技术[J]. 生物工程学报,2021,37(5):1564−1577. [YU Y, ZHU X N, BI C H, et al. The creation technology of Escherichia coli cell factory[J]. Journal of Bioengineering,2021,37(5):1564−1577.] YU Y, ZHU X N, BI C H, et al. The creation technology of Escherichia coli cell factory[J]. Journal of Bioengineering, 2021, 37(5): 1564−1577.
[45] MA S, SU T Y, LIU J M, et al. Reduction of the bacterial genome by transposon-mediated random deletion[J]. ACS Synthetic Biology,2022,11(2):668−677. doi: 10.1021/acssynbio.1c00353
[46] QI L N, LIU C X, PEPLOWSKI L, et al. Efficient production of hydroxytyrosol by directed evolution of HpaB in Escherichia coli[J]. Biochemical and Biophysical Research Communications,2023,663:16−24. doi: 10.1016/j.bbrc.2023.04.024
[47] WU Y K, CHEN T C, LIU Y F, et al. Design of a programmable biosensor-CRISPRi genetic circuits for dynamic and autonomous dual-control of metabolic flux in Bacillus subtilis[J]. Nucleic Acids Research,2020,48(2):996−1009. doi: 10.1093/nar/gkz1123
[48] 李寅. 合成生物制造[J]. 生物工程学报,2022,38(4):1267−1294. [LI Y. Synthetic biofabrication[J]. Journal of Bioengineering,2022,38(4):1267−1294.] LI Y. Synthetic biofabrication[J]. Journal of Bioengineering, 2022, 38(4): 1267−1294.
[49] BAUMGART M, UNTHAN S, KLOß R, et al. Corynebacterium glutamicum chassis C1*:Building and testing a novel platform host for synthetic biology and industrial biotechnology[J]. Acs Synthetic Biology,2017,7(1):132−144.
[50] BAUMGART M, UNTHAN S, RUCKERT C, et al. Construction of a prophage-free variant of Corynebacterium glutamicum ATCC 13032 for use as a platform strain for basic research and industrial biotechnology[J]. Applied and Environmental Microbiology,2013,79(19):6006−6015. doi: 10.1128/AEM.01634-13
[51] WEI L, ZHAO J H, WANG Y R, et al. Engineering of Corynebacterium glutamicum for high-level γ-aminobutyric acid production from glycerol by dynamic metabolic control[J]. Metabolic Engineering,2022,69:134−146. doi: 10.1016/j.ymben.2021.11.010
[52] LI M Y, CHEN J Z, WANG Y, et al. Efficient multiplex gene repression by CRISPR-dCpf1 in Corynebacterium glutamicum[J]. Front Bioeng Biotechnol,2020,8:357. doi: 10.3389/fbioe.2020.00357
[53] 薛宁, 王瑾, 李世新, 等. 多基因同步调控结合高通量筛选构建高产L-苯丙氨酸的谷氨酸棒杆菌工程菌株[J]. 生物技术通报,2023,39(9):268−280. [XUE N, WANG J, LI S X, et al. The engineering strains of Corynebacterium glutamicum with high yield of L-phenylalanine were constructed by multi-gene synchronous regulation combined with high-throughput screening[J]. Biotechnology Bulletin,2023,39(9):268−280.] XUE N, WANG J, LI S X, et al. The engineering strains of Corynebacterium glutamicum with high yield of L-phenylalanine were constructed by multi-gene synchronous regulation combined with high-throughput screening[J]. Biotechnology Bulletin, 2023, 39(9): 268−280.
[54] CDVORAK P, BAYER E A, LORENZO V D. Surface display of designer protein scaffolds on genome-reduced strains of Pseudomonas putida[J]. Acs Synthetic Biology,2020,9(10):2749−2764. doi: 10.1021/acssynbio.0c00276
[55] FAN X, ZHANG Y T, ZHAO F J, et al. Genome reduction enhances production of polyhydroxyalkanoate and alginate oligosaccharide in Pseudomonas mendocina[J]. Interational Journal of Biological Macromolecules,2020,163:2023−2031. doi: 10.1016/j.ijbiomac.2020.09.067
[56] QIAO W J, LIU F L, WAN X, et al. Genomic features and construction of streamlined genome chassis of nisin Z producer Lactococcus lactis N8[J]. Microorganisms,2021,10(1):47. doi: 10.3390/microorganisms10010047
[57] 张雅婷. 乳酸乳球菌基因组删减底盘构建与表型研究[D]. 天津:南开大学, 2021. [ZHANG Y T. Construction of genome deletion chassis and phenotypic study of Lactococcus lactis[D]. Tianjin:Nankai University, 2021.] ZHANG Y T. Construction of genome deletion chassis and phenotypic study of Lactococcus lactis[D]. Tianjin: Nankai University, 2021.
[58] 杨永富, 耿碧男, 宋皓月, 等. 合成生物学时代基于非模式细菌的工业底盘细胞研究现状与展望[J]. 生物工程学报,2021,37(3):874−910. [YANG Y F, GENG B N, SONG H Y, et al. Current situation and prospect of industrial chassis cell research based on non-model bacteria in the era of synthetic biology[J]. Journal of Bioengineering,2021,37(3):874−910.] YANG Y F, GENG B N, SONG H Y, et al. Current situation and prospect of industrial chassis cell research based on non-model bacteria in the era of synthetic biology[J]. Journal of Bioengineering, 2021, 37(3): 874−910.
-
期刊类型引用(0)
其他类型引用(4)





 下载:
下载:
 下载:
下载:



