Effect of the C-terminal Non-catalytic Domain on the Enzymatic Properties of Thermophilic α-Glucan Phosphorylase TsGP
-
摘要: 为获得异源表达水平与酶学性质良好的α-葡聚糖磷酸化酶并考察结构域对酶的影响,研究通过数据库挖掘和序列分析,获得了来源于超级嗜热古菌Thermococcus sp. EP1的新型α-葡聚糖磷酸化酶TsGP。利用AlphaFold2预测三维结构确定TsGP的C端为非催化结构域后,构建了C端截短的突变体ΔTsGP。在E. coli BL21(DE3)中对TsGP与ΔTsGP进行异源重组表达,并对其酶学性质进行了表征。结果表明,ΔTsGP在大肠杆菌中的表达水平较TsGP提高了3.76倍。在最适反应温度为70 ℃时,ΔTsGP的比酶活为23.87 U/mg,较TsGP提高1.3倍。在50~65 ℃的工业酶催化条件下,ΔTsGP的热稳定性与TsGP相当,且酶活力更高。此外,ΔTsGP与TsGP具有相同的底物特异性,最小底物为麦芽三糖,且随着底物链长的增加,比酶活逐渐提升。以麦芽七糖为底物,ΔTsGP的kcat值为37.09 s−1,比TsGP提高了1.21倍,但底物亲和力(Km=3.30 mmol/L)降低了2.77倍。研究通过结构分析与非催化结构域截短策略成功提高了TsGP在大肠杆菌中的表达水平和工业酶催化条件下的比酶活。这为αGP酶蛋白的高效表达与应用提供了借鉴,并为进一步通过工程改造优化其性能奠定了基础。
-
关键词:
- 嗜热α-葡聚糖磷酸化酶 /
- 葡萄糖-1-磷酸 /
- 非催化结构域 /
- 蛋白截短 /
- 酶学性质
Abstract: In order to obtain α-glucan phosphorylase with good heterologous expression level and enzymatic properties and to investigate the impact of structural domains on the enzyme, a novel α-glucan phosphorylase TsGP was screened from the hyperthermophilic archaeon Thermococcus sp. EP1 through database mining and sequence analysis. After confirming that the C-terminal of TsGP was a non-catalytic domain in the three-dimensional structure predicted by AlphaFold2, a C-terminal truncated mutant ΔTsGP was constructed. Heterologous recombinant expression of TsGP and ΔTsGP was performed in E.coli BL21(DE3), and their enzymatic properties were characterized. The results showed that the expression level of ΔTsGP in E.coli was 3.76 times higher than that of TsGP. At the optimal reaction temperature 70 ℃, the specific enzyme activity of ΔTsGP was 23.87 U/mg, which was 1.3 times higher than that of TsGP. Under 50~65 ℃ which was widely applied as industrial catalytic temperature, the thermal stability of ΔTsGP was equivalent to that of TsGP, and the enzyme activity was higher. In addition, ΔTsGP and TsGP had the same substrate specificity, their smallest substrate was maltotriose, and as the substrate chain length increased, their specific enzyme activity gradually increased. Using maltoheptaose as the substrate, the kcat value of ΔTsGP was 37.09 s−1, which was 1.21 times higher than that of TsGP, but its substrate affinity (Km=3.30 mmol/L) was reduced by 2.77 times. Through structural analysis and non-catalytic domain truncation strategies, the study successfully improved the expression level of TsGP in E. coli and the specific enzyme activity under industrial enzyme catalytic conditions. The study would provide guidance for the efficient expression and application of αGPase protein and lays the foundation for further optimizing the performance of this enzyme through engineering modification. -
α-葡聚糖磷酸化酶(αGP,EC2.4.1.1)广泛存在于微生物、植物和动物中,可以催化淀粉、糖原、麦芽糊精等α-1,4葡聚糖的可逆磷酸解反应[1]。在碳水化合物酶(CAZy)数据库中,αGP被归类于糖基转移酶GT_35家族[2],不同来源的αGP催化机制基本相同,均为吡哆醛-5-磷酸(PLP)依赖型糖基转移酶[1]。
αGP可用于催化无机磷与可溶性淀粉制备α-D-葡萄糖-1-磷酸(α-D-Glucose 1-phosphate,G-1-P),具有底物廉价易得、理论得率高等优点,是生产G-1-P的关键酶[3−4]。G-1-P作为一种活化形式的葡萄糖磷酸盐,是合成糖核苷酸[5]、寡糖[6]、糖脂[7]等复杂碳水化合物的重要前体。在食品及医药行业,G-1-P可作为营养强化剂促进肠道内钙的主动运输,增加骨骼密度,促进骨骼发育[8],也可作为肠外营养混合剂的稳定剂,避免无机磷引起的钙沉淀[9]。除G-1-P外,αGP也被广泛应用于甘油葡萄糖苷[10]、果糖[11]、甘露糖[12]、阿洛酮糖[13]、塔格糖[14]、甘露寡糖[15]、母乳寡糖[16]、肌醇[17]、甘露醇[18]、人工淀粉[19]、葡萄糖胺[20]等重要营养化学品的体外合成研究。
近年来,国内外学者在αGP的研究上取得了显著进展。与嗜温微生物来源的嗜温酶相比,嗜热微生物来源的αGP热稳定性强,可以通过热处理纯化,在高温下具有酶促反应速率更快、底物浓度更高、粘度更低、微生物污染风险更小等优点[21−22]。到目前为止,来源于嗜热细菌Dictyoglomus turgidum、Thermoanaerobacter tengcongensis、Clostridium thermocellum等[14,23−24]和嗜热古菌Thermococcus litoralis、Sulfolobus tokodaii、Sulfolobus solfataricus等[25−28]来源的嗜热αGP已有诸多研究报道。然而,嗜热αGP的异源表达与酶学性质存在一些问题亟待解决。首先,αGP的分子量较大,表达过程中非常容易形成无活性的包涵体,导致αGP的可溶性表达水平偏低,这限制了其在大规模生产中的应用[29]。其次,多数报道的嗜热αGP在70 ℃以上时具有最佳的酶活性,在工业反应条件下(50~65 ℃)酶活性相对较低[30],这影响了其在工业领域的应用效果。
针对以上问题,本文筛选获得了一种来源于嗜热古菌Thermococcus sp. EP1的α-葡聚糖磷酸化酶TsGP,并利用AlphaFold2预测了其三维结构,发现该酶具有C端非催化结构域,将该结构域对应的氨基酸序列进行截短,研究了C端非催化结构域截短对TsGP表达水平和酶学性质的影响,以期为αGP的高效表达及应用提供理论指导。
1. 材料与方法
1.1 材料与仪器
质粒克隆菌株E. coli DH5α、蛋白表达菌株E. coli BL21(DE3) 为本实验室保藏;α-葡聚糖磷酸化酶基因 由南京金斯瑞生物科技有限公司合成。在本研究中涉及到的菌株、质粒如表1所示;Phanta Max Super-Fidelity DNA Polymerase扩增试剂盒 南京诺维赞生物科技股份有限公司;质粒提取试剂盒 Omega Bio-Tek公司;BCA蛋白浓度测定试剂盒、氨苄青霉素(Amp)、异丙基-β-D-硫代吡喃半乳糖苷(IPTG)、琼脂粉、三羟甲基氨基甲烷(Tris) 北京索莱宝科技有限公司;葡萄糖含量测定试剂盒 芬兰Megazyme公司;麦芽糊精(DE值4~7)、麦芽二糖、麦芽三糖、麦芽四糖、麦芽五糖、麦芽六糖、麦芽七糖 分析纯,Sigma-Aldrich公司;无水乙醇、NaCl、K2HPO4、KH2PO4 分析纯,国药集团化学试剂有限公司;酵母粉、蛋白胨 英国OXOID公司;来源于Thermococcus kodakarensis KOD1的葡萄糖磷酸变位酶(Phosphoglucomutase,PGM)、来源于Thermotoga sp. 38H的6-磷酸葡萄糖磷酸酶(Glucose-6-phosphatase,G6PP) 本实验室制备;所用培养基为LB培养基。
表 1 本研究中使用的菌株、质粒Table 1. Strains and plasmids used in this study菌株/质粒 表型 来源 菌株 E. coli DH5α F- φ80 lacZΔM15 Δ(lacZYA-argF)U169 recA1 endA1 hsdR17(rk-, mk+) phoA,
supE44 λ- thi-1 gyrA96 relA1本实验室保存 E. coli BL21(DE3) F-ompT hsdS (rB-mB-)gal dcm 本实验室保存 E. coli BL21(DE3)/pET-21a(+)-AtGP E. coli BL21(DE3) with vector pET-21a(+)-AtGP 本研究 E. coli BL21(DE3)/pET-21a(+)-BtGP E. coli BL21(DE3) with vector pET-21a(+)-BtGP 本研究 E. coli BL21(DE3)/pET-21a(+)-DtGP E. coli BL21(DE3) with vector pET-21a(+)-DtGP 本研究 E. coli BL21(DE3)/pET-21a(+)-HsGP E. coli BL21(DE3) with vector pET-21a(+)-HsGP 本研究 E. coli BL21(DE3)/pET-21a(+)-PfGP E. coli BL21(DE3) with vector pET-21a(+)-PfGP 本研究 E. coli BL21(DE3)/pET-21a(+)-TaGP E. coli BL21(DE3) with vector pET-21a(+)-TaGP 本研究 E. coli BL21(DE3)/pET-21a(+)-TsGP E. coli BL21(DE3) with vector pET-21a(+)-TsGP 本研究 E. coli BL21(DE3)/pET-21a(+)-ΔTsGP E. coli BL21(DE3) with vector pET-21a(+)-ΔTsGP 本研究 质粒 pET-21a(+)-AtGP pET-21a(+) harboring AtGP 本研究 pET-21a(+)-BtGP pET-21a(+) harboing BtGP 本研究 pET-21a(+)-DtGP pET-21a(+) harboring DtGP 本研究 pET-21a(+)-HsGP pET-21a(+) harboring HsGP 本研究 pET-21a(+)-PfGP pET-21a(+) harboring PfGP 本研究 pET-21a(+)-TaGP pET-21a(+) harboring TaGP 本研究 pET-21a(+)-TsGP pET-21a(+) harboring TsGP 本研究 pET-21a(+)-ΔTsGP pET-21a(+) harboring ΔTsGP 本研究 T100 PCR仪 伯乐生命医学产品(上海)有限公司(Bio-Rad);SW-CJ-2FD超净工作台 苏州安泰空气科技有限公司;ScienTZ-11D超声波细胞粉碎机 宁波新芝生物科技股份有限公司;Sorvall Stratos高速冷冻离心机 Thermo Fisher Scientific公司;DHP-9082电热恒温培养箱 上海一恒科技有限公司;SQ-510C台式高压灭菌锅 重庆雅马拓科技科技有限公司。
1.2 实验方法
1.2.1 基因挖掘与表达
1.2.1.1 α-葡聚糖磷酸化酶的挖掘
以来源于Pyrococcus furiosus的α-葡聚糖磷酸化酶氨基酸序列为模板,于NCBI数据库中进行BLAST蛋白序列比对,选择不同菌种来源且序列相似性不同的氨基酸序列。通过Expasy网站预测酶的理化性质,利用软件MEGA 11构建系统发育树。筛选获得的序列由金唯智生物科技有限公司合成,进行密码子优化并克隆至pET-21a(+)表达载体的Nde Ⅰ和Xho Ⅰ酶切位点之间。
1.2.1.2 α-葡聚糖磷酸化酶表达与纯化
诱导表达:将含有α-葡聚糖磷酸化酶基因的表达质粒转化至E. coli BL21(DE3)感受态细胞,获得蛋白表达菌株。从平板挑取单菌落,接种至含有100 μg/mL氨苄青霉素的LB培养基中,在37 ℃恒温摇床中过夜培养,获得种子液。取1 mL种子液接种至50 mL含有100 μg/mL的氨苄青霉素的LB培养基中,在37 ℃恒温摇床中培养,当600 nm的吸光值为0.6~0.8时,加入终浓度为0.5 mmol/L的IPTG,在16 ℃恒温摇床中诱导表达16~20 h。离心收集菌体,用2 mL PBS(50 mmol/L,pH7.0)缓冲液重悬菌体,超声破碎仪破碎细胞获得细胞裂解液,离心获得粗酶上清液。
蛋白纯化:通过Ni-NTA亲和层析对粗酶上清液进行纯化。用含有50 mmol/L Tris-HCl、50 mmol/L NaCl、20 mmol/L(pH8.0)咪唑的结合缓冲液洗涤杂蛋白,用含有50 mmol/L Tris-HCl、120 mmol/L NaCl、500 mmol/L(pH8.0)咪唑的洗脱缓冲液洗脱蛋白获得纯酶液。将纯酶液移入截留分子量为30 kDa的超滤管内,在4 ℃、4 000 r/min下离心20 min进行脱盐处理,50 mmol/L PBS(pH7.0)缓冲液洗脱3次,使用SDS-PAGE凝胶电泳鉴定纯化结果,使用BCA蛋白浓度测定试剂盒测定蛋白浓度。
1.2.2 酶活测定
反应体系为200 μL,包含20 mg/mL 麦芽糊精、10 mmol/L PBS(pH7.0)缓冲液、5 mmol/L MgCl2、20 μg PGM、10 μg G6PP、5 μg αGP,用去离子水补充至200 μL,对照组加入失活的5 μg αGP,70 ℃反应30 min,加入1 μL 10%的硫酸溶液,沸水浴10 min终止反应。
将反应液稀释适当倍数,使用葡萄糖含量测定试剂盒测定反应液中葡萄糖含量,在上述条件下每分钟产生1 μmol葡萄糖所需的酶量定义为一个酶活力单位。
1.2.3 酶分子结构建模与结构域截短
使用Clustal W程序进行蛋白质序列多重比对,使用ESPript 3.0展示氨基酸序列的多重序列比对结果,并同时展示序列的二级结构。根据筛选的α-葡聚糖磷酸化酶的Uniprot ID号,从EMBL-EBI AlphaFold Protein Structure Database数据库获得α-GPs的三维结构,用PyMOL软件对其进行分析。根据分析结果,将TsGP的C端β-sheet结构域第700位至831位氨基酸进行截短。具体实验方法如下:
以α-葡聚糖磷酸化酶TsGP的表达载体pET-21a(+)-TsGP为模板,设计引物ΔTsGP_F:5’-TATCCTGCTGCACCGCCTCGAGCACCACCACCACCACCA-3’、ΔTsGP_R:5’-GGTGGTGCTCGAGGCGGTGCAGCAGGATATAGTTAGCCA-3’,PCR扩增将质粒pET-21a(+)-TsGP进行线性化,PCR反应体系为:16 μL ddH2O,25 μL 2X Buffer,2 μL ΔTsGP_F,2 μL ΔTsGP_R,2 μLdNTP,2 μL模板质粒,1 μL DNA聚合酶;PCR程序:95 ℃(15 s),56 ℃(15 s),72 ℃(4 min)共30个循环,72 ℃(5 min)。将PCR产物用Dpn I酶于37 ℃消化1 h,将消化产物转化至大肠杆菌E. coli DH5α感受态细胞,涂布于含有氨苄青霉素的LB抗性平板筛选重组菌株。挑取单克隆进行测序,验证成功后将重组质粒pET-21a(+)-ΔTsGP转入大肠杆菌E. coli BL21(DE3),获得α-葡聚糖磷酸化酶C端结构域截短的表达菌株E. coli BL21(DE3)/pET-21a(+)-ΔTsGP。
1.2.4 酶学性质
1.2.4.1 最适反应温度
把反应液分别放置在50、60、70、80和90 ℃下测定酶活,以最适温度下测定的酶活为100%,计算其他温度下的相对酶活力,确定最适反应温度。
1.2.4.2 温度稳定性
温度稳定性:将酶液在50、60、70和80 ℃下保温,在0、6、12和24 h取样测定酶活,以0 h时测定的酶活为100%,计算其余相对酶活力,研究酶的温度稳定性。
1.2.4.3 最适反应pH
最适反应pH:在最适温度条件下,于不同pH的反应体系测定酶活力(CH3COOH-CH3COONa:pH4.0~6.0;K2HPO4-KH2PO4:pH6.0~8.0;Tris-HCl:pH7.0~8.0;Glycine-NaOH:pH9.0~11.0),以最适pH下的酶活为100%,计算其他pH条件下的相对酶活力,确定最适反应pH。
1.2.4.4 金属离子
金属离子对酶反应的影响:在最适温度和最适pH条件下,加入终浓度为1 mmol/L的Mg2+、Ba2+、Ca2+、Cu2+、Mn2+、Ni2+和Zn2+,对照组不加金属离子。将对照组酶活设为100%,确定金属离子存在下的相对酶活力。
1.2.4.5 底物特异性
底物特异性:在最适条件下,以麦芽糖、麦芽三糖、麦芽四糖、麦芽五糖、麦芽六糖和麦芽七糖为底物,测定酶活,确定酶的最适底物。
1.2.4.6 动力学参数
动力学参数:以不同浓度的麦芽七糖为底物,测定最适条件下的酶活力,使用Graphpad Prism 9.3.0软件的非线性拟合计算酶的动力学参数kcat与Km值。
1.2.5 分子动力学模拟
利用AlphaFold2[31]建模获得α-葡聚糖磷酸化酶的结构,使用CHARMM-GUI建立野生型和截短酶的模拟体系,将酶放置于带有周期性边界的立方体水盒子中,并加入钠离子作为抗衡离子使体系保持电中性。分子动力学模拟均采用NAMD 3.0软件包[32],分别使用CHARMM36m蛋白质力场和TIP3P水模型中的参数描述α-葡聚糖磷酸化酶和水分子。使用SHAKE/RATTLE算法约束与氢相连的共价键;采用PME方法计算长程静电相互作用,范德华和短程静电相互作用的截断半径为12 Å;对运动方程积分步长为2 fs。为了研究温度对蛋白结构的影响,分子动力学模拟的温度分别设置为25和70 ℃,并使用Langevin动力学方法进行控温。使用Langevin活塞方法将压力控制在1.0 bar。每个模拟体系在经历1000步的初始能量最小化后,进行了300 ns的平衡模拟,储存数据的频率为100 ps。使用VMD软件进行轨迹的可视化分析。
1.3 数据处理
所有试验重复3次,使用软件Graphpad Prism 9.3.0进行数据处理。使用Expasy网站ProtParam预测蛋白质性质。使用Clustal程序进行多重序列比对,MEGA11软件绘制进化树,ESPript 3.0展示蛋白质二级结构,PyMOL展示蛋白三维结构,NAMD 3.0进行分子动力学模拟。
2. 结果与分析
2.1 目的基因的筛选
通过文献检索,发现来源于古菌火球菌属Pyrococcus furiosus的α-葡聚糖磷酸化酶在80 ℃下具有较高的酶活性[30],其被用于合成葡萄糖-1-磷酸[4]。以其氨基酸序列为模板,从Genbank数据库中筛选了7条不同菌种来源的葡聚糖磷酸化酶(见表2)。
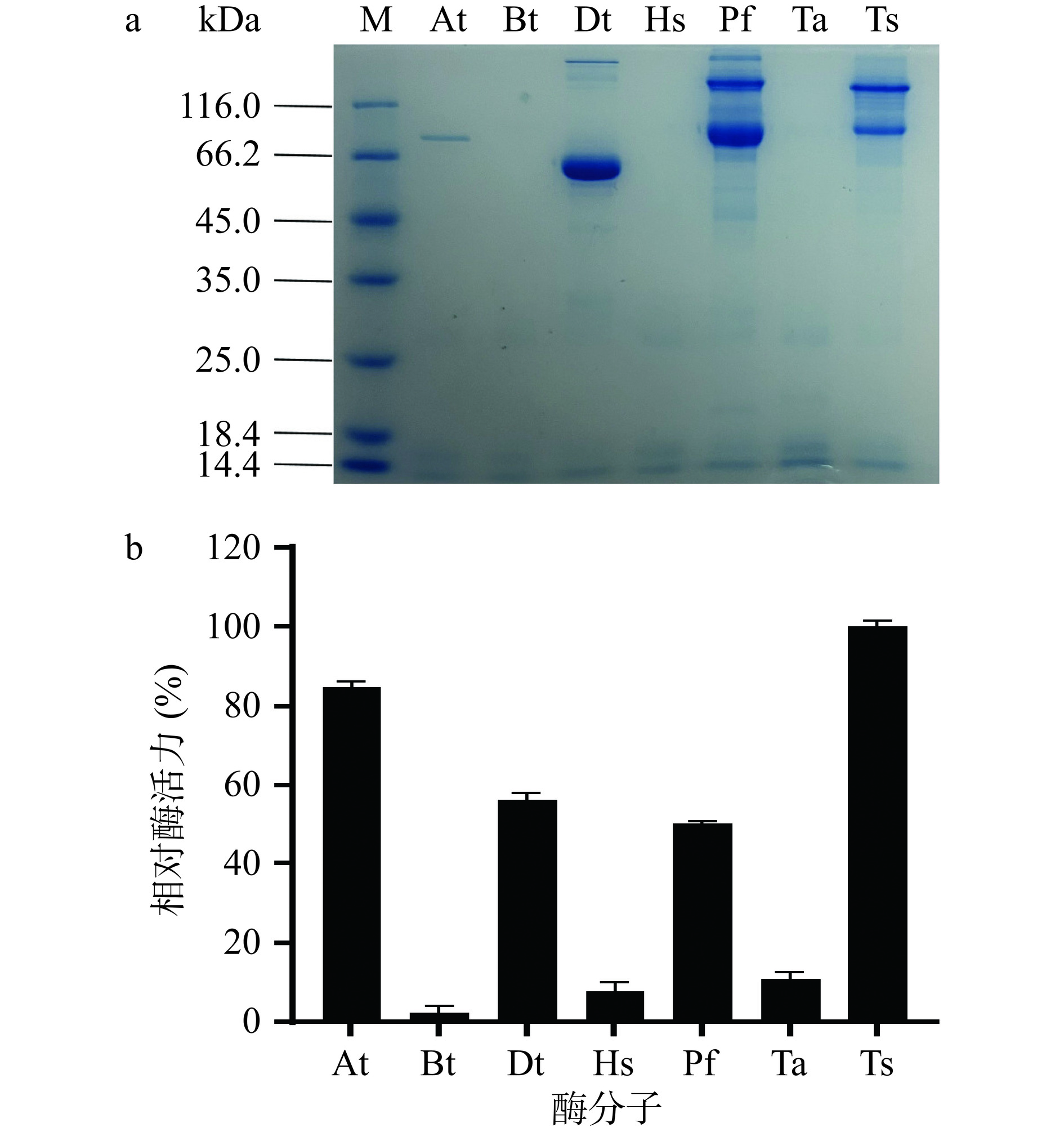
表 2 7种不同来源的α-葡聚糖磷酸化酶Table 2. Seven different sources of α-glucan phosphorylase基因名称 基因来源 分子量(kDa) Uniprot ID AtGP Anaerolinea thermolimosa 84.0 A0A3D1JG97 BtGP Bailinhaonella thermotolerans 95.4 A0A3A4B0I3 DtGP Dictyoglomus turgidum 65.4 B8E0Q6 HsGP Halodesulfovibriosp. MK-HDV 86.7 A0A7C8HW27 PfGP Pyrococcus furiosus 97.7 Q8U0Q0 TaGP Thermoplasmata archaeon M9B1D 82.1 A0A2K3J6S9 TsGP Thermococcus sp. EP1 96.5 A0A0P8ZX42 将筛选的7种不同来源的αGP在大肠杆菌E. coli BL21(DE3)中进行表达纯化。如图1a所示,SDS-PAGE的结果表明,来源于Anaerolinea thermolimosa、Dictyoglomus turgidum、Pyrococcus furiosus、Thermococcus sp. EP1的αGP成功实现异源表达,而来源于Bailinhaonella thermotolerans、Halodesulfovibrio sp. MK-HDV、Thermoplasmata archaeon M9B1D的αGP未发现有明显的蛋白条带。对7种不同αGP的酶活性进行了测定,结果表明来源于Bailinhaonella thermotolerans、Halodesulfovibrio sp. MK-HDV、Thermoplasmata archaeon M9B1D的αGP几乎无活性,与SDS-PAGE结果一致。来源于Thermococcus sp. EP1的αGP酶具有最高的比酶活,其相对活性比来源于Pyrococcus furiosus[28]的αGP比酶活提高约50%,结果如图1b所示,因此选择来源于Thermococcus sp. EP1的TsGP进行下一步研究。
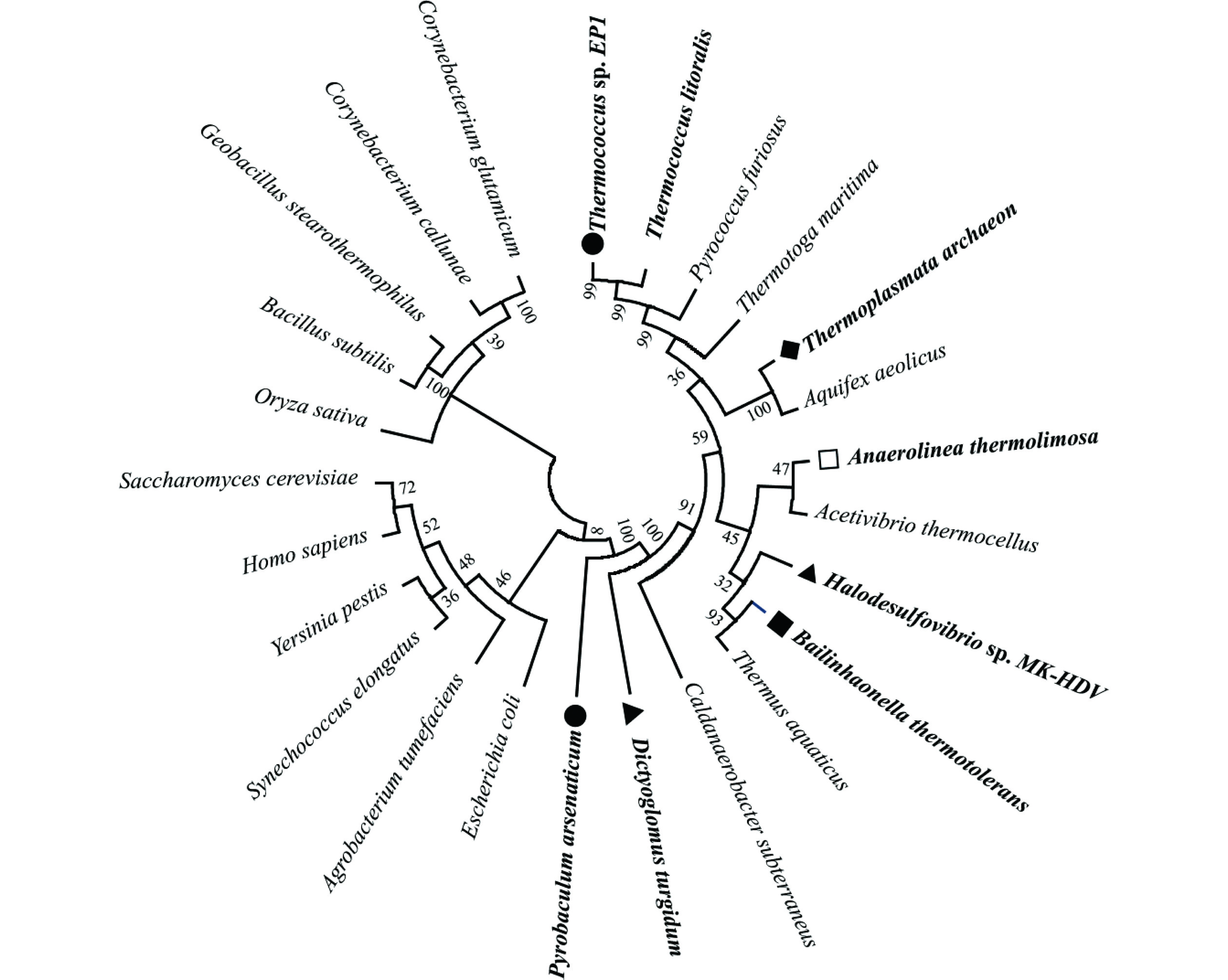
多数已报道的αGP为同源二聚体,分子量在80~100 kDa[30],TsGP共含有831个氨基酸,其预测分子量为96.5 kDa,SDS-PAGE结果显示其实际分子量与预测值相符(图1a),与多数已报道的αGP相似。由于TsGP在CAZy数据库中被归类于糖基转移酶GT35家族,选取GT35家族部分已表征的αGP蛋白序列及本研究挖掘的蛋白序列,使用软件MEGA 11构建系统发育树。如图2所示,TsGP与Thermococcus litoralis来源的αGP具有较近的亲缘关系,其同为古菌热球菌属Thermococcus。TsGP与嗜热古菌Thermococcus litoralis[25]、Pyrococcusfuriosus[28]来源的αGP蛋白序列相似性分别为86.76%、67.5%。
2.2 TsGP结构模拟
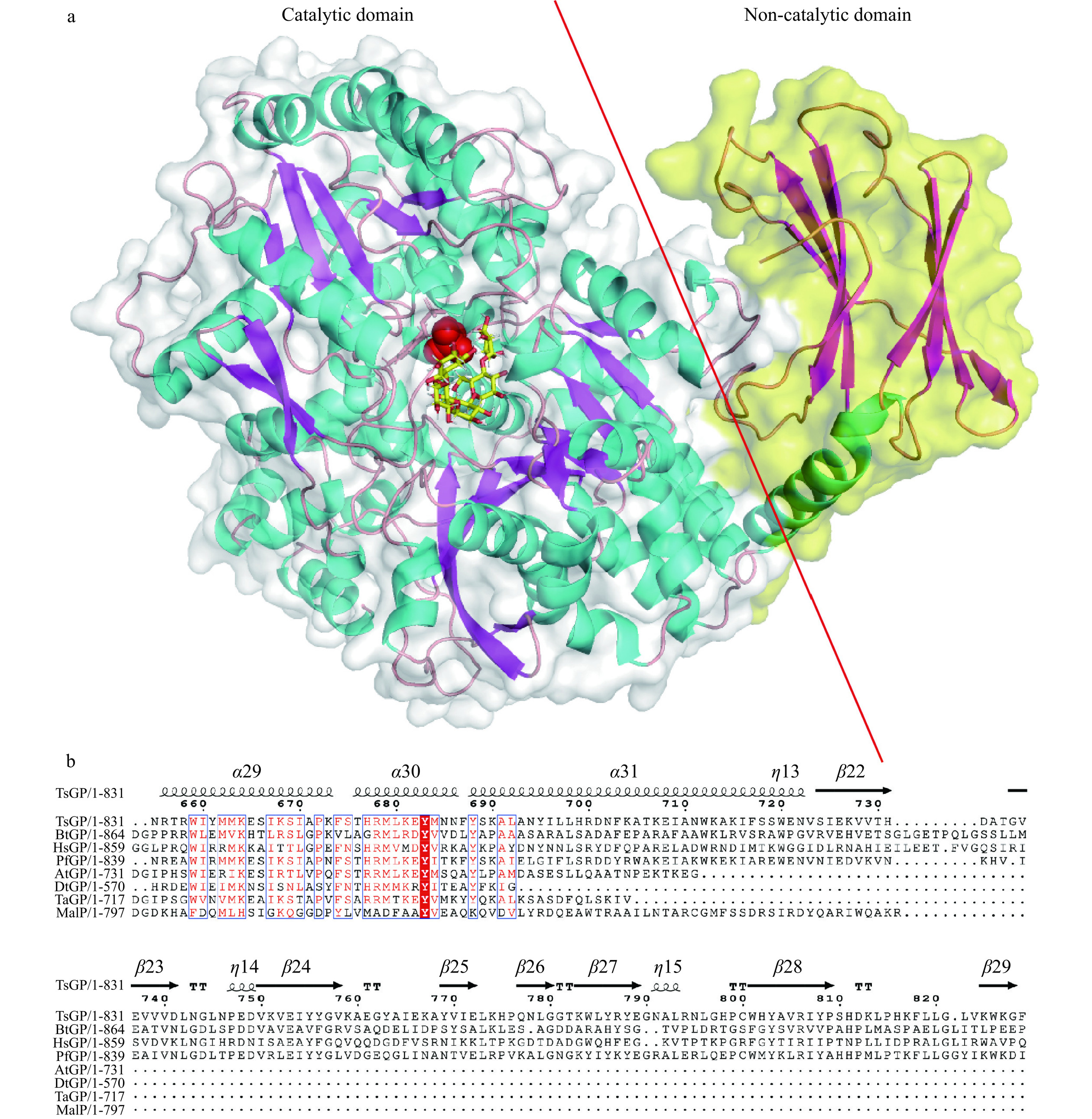
α-葡聚糖磷酸化酶的三维结构高度保守[33],来源于大肠杆菌的麦芽糊精磷酸化酶MalP的晶体结构与催化机制已被解析[34]。MalP是由两个结构域(N端结构域:19~482;C端结构域:483~829)构成的α/β蛋白,其催化位点位于两个结构域之间。根据TsGP的Uniprot ID号,从EMBL-EBI AlphaFold Protein Structure Database数据库中获得其三维结构,PyMOL软件进行分析显示其催化结构域与MalP的晶体结构高度一致,但在TsGP的C端形成一个独立结构域(721~831),其由6个β折叠构成,并由一段α螺旋(700~720)结构将其与催化结构域连接(图3a)。使用Clustal W程序对TsGP、MalP及筛选获得的蛋白质序列进行多重比对,使用ESPript 3.0展示氨基酸序列的多重序列比对结果,展示了序列的二级结构(图3b)。通过分析AlphaFold获得的三维结构,发现TsGP、BtGP、HsGP、PfGP具有同样的C端独立结构域,而AtGP、DtGP、TaGP及MalP缺乏此结构域。
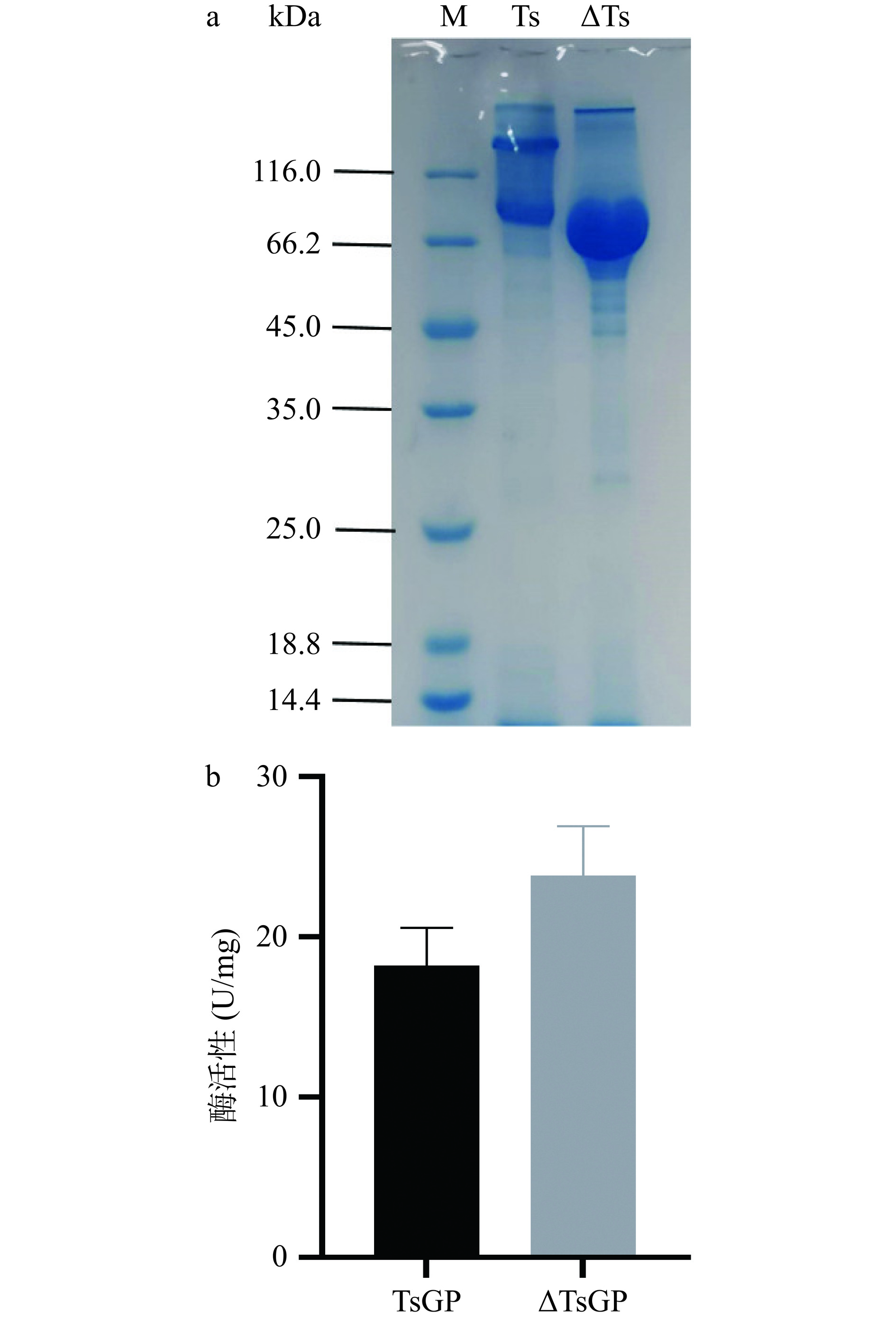
为研究TsGP的C端非催化结构域对其表达及酶学性质的影响,将其C端非催化结构域(700~831)进行了截短。对全长蛋白TsGP及截短蛋白ΔTsGP进行表达,SDS-PAGE结果显示(图4a),截短蛋白条带的分子量小于野生型,与理论分子质量一致(约81 kDa)。BCA蛋白浓度测定试剂盒测定ΔTsGP的蛋白表达量为5.33 mg/mL,较TsGP蛋白表达量提高3.76倍。以20 mg/mL麦芽七糖为底物进行酶活分析,结果如图4b所示,ΔTsGP比酶活较TsGP提高约30%。
2.3 TsGP及ΔTsGP酶学性质研究
2.3.1 最适反应温度及温度稳定性
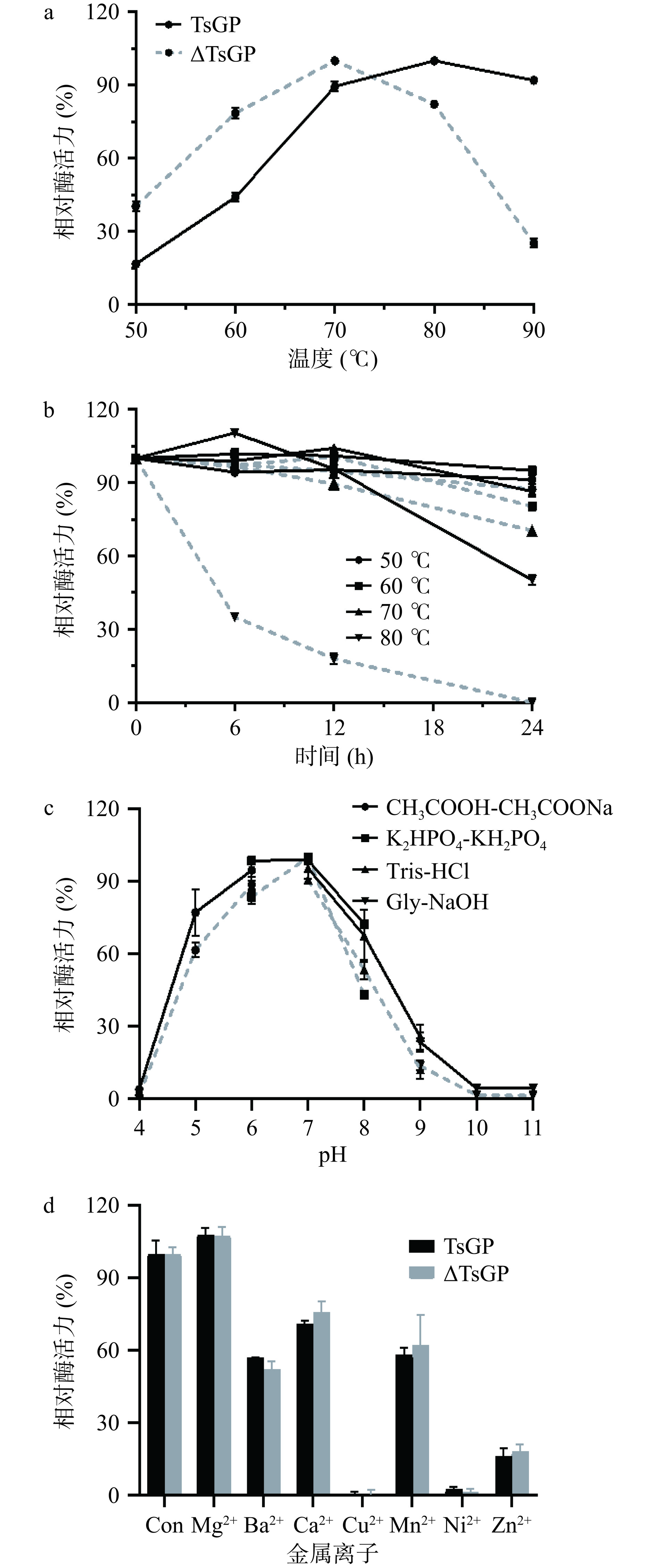
在50~90 ℃范围内,测定了TsGP及ΔTsGP的最适温度。由图5a可知,野生型酶TsGP的最适反应温度为80 ℃,在50 ℃时,酶活性约为最高酶活性的20%。在70~90 ℃时,酶活性能保持最高酶活性的90%以上。截短酶ΔTsGP的最适反应温度为70 ℃,在50 ℃时,酶活性约为最高酶活性的40%,在60~80 ℃时,酶活性仍然保留最高酶活性的80%,但当反应温度为90 ℃时,ΔTsGP仅保留最高酶活性的20%。结果表明TsGP为嗜热酶,C端非催化区域截短,降低了酶的最适反应温度,但是其在70 ℃以下时具有较高的活性。
测定TsGP与ΔTsGP的热稳定性,结果如图5b所示,野生型酶TsGP和截短酶ΔTsGP分别在50、60、70 ℃ 放置24 h,其活性均能够保持初始酶活的70%以上。TsGP在80 ℃放置24 h,其酶活性仍能保持初始酶活的50%左右,具有良好的热稳定性。然而,截短酶ΔTsGP在80 ℃放置6 h,其酶活性降至最高酶活性的40%以下,放置24 h则完全失去活性。此结果表明,C端非催化结构域能够提升TsGP在高温的热稳定性,截短酶ΔTsGP的最适反应温度由80 ℃变为70 ℃,可能是由于C端非催化结构域删除后酶的热稳定性变差引起的。
TsGP与多数已报道的嗜热αGP类似,热稳定性好,在70 ℃以上时具有最佳的酶活性,但在工业反应条件下(50~65 ℃)酶活性为最适温度条件下酶活性的50%以下[30],C端非催化结构域的截短,提升了ΔTsGP在工业反应温度条件下的酶活性,同时在70 ℃条件下保持了良好的热稳定性。
2.3.2 最适pH
在不同的缓冲体系中,对TsGP及ΔTsGP的最适pH进行了测定。由图5c可知,野生型酶TsGP和截短酶ΔTsGP的最适pH均为7.0,与Thermococcus litoralis[25]等来源的嗜热αGP最适pH相似。在pH为4.0时,TsGP和ΔTsGP均无活性。TsGP在pH5.0~8.0时,酶活性仍然保留最高酶活性的80%以上,当pH增加至9.0时酶活性迅速下降。ΔTsGP在pH5.0时,酶活性为最高酶活性的60%,在pH为8.0时,酶活性为最高酶活性的50%。此结果表明,野生型酶TsGP比截短酶ΔTsGP具有更宽泛的pH,C端非催化区域能够提升TsGP的pH耐受性。
2.3.3 金属离子对酶活性的影响
研究了不同二价金属离子对TsGP及ΔTsGP活性的影响。由图5d可知,金属离子对野生型酶TsGP和截短酶ΔTsGP的影响趋势保持一致,Mg2+能够促进酶活性,其他二价金属离子则引起酶活性不同程度的下降,特别是Cu2+和Ni2+的加入,导致酶活性完全丧失。
2.3.4 底物特异性及动力学参数
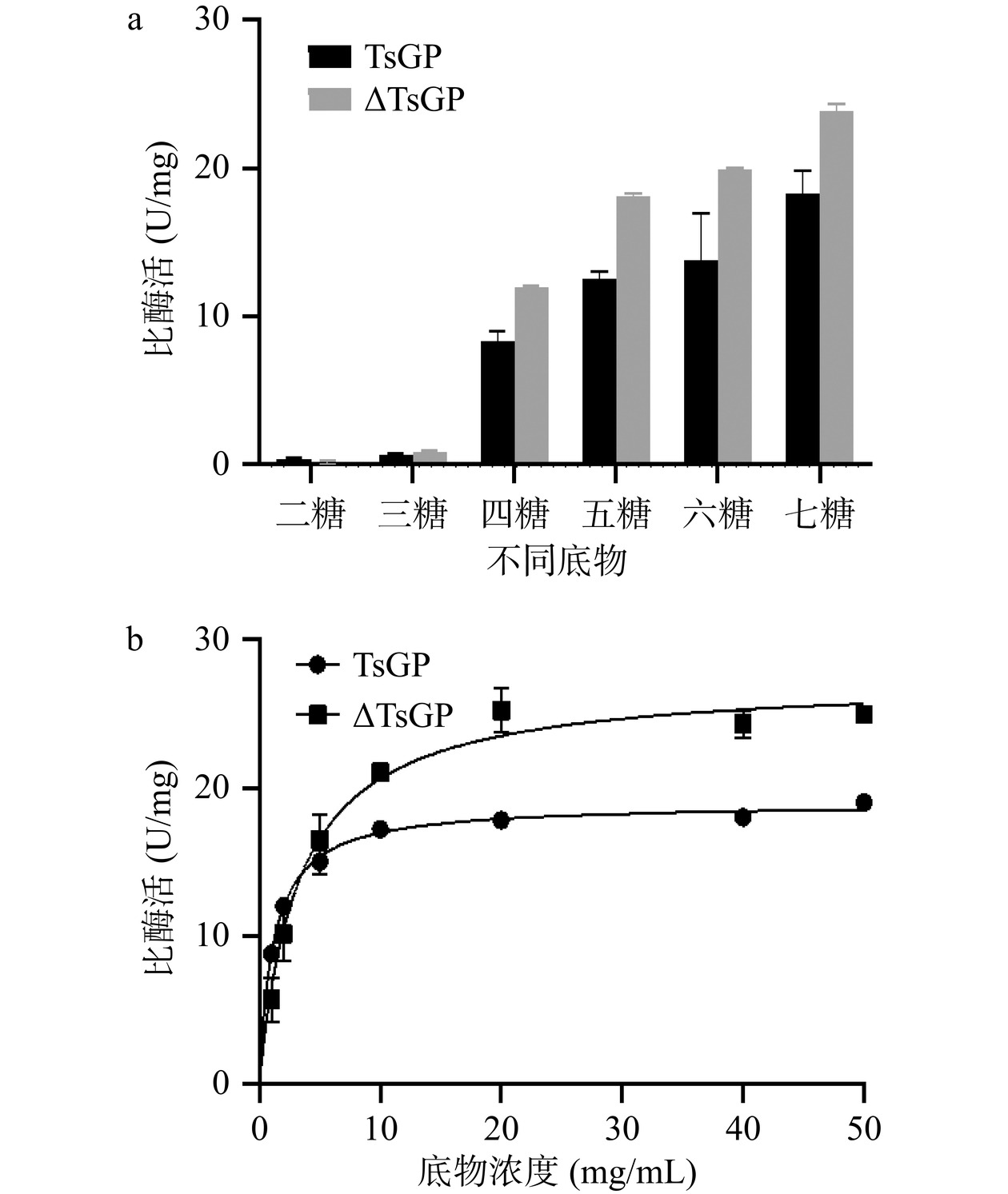
葡聚糖磷酸化酶催化α-1,4-葡聚糖的可逆磷酸解,生成α-D-葡萄糖-1-磷酸。在70 ℃下,测试了TsGP及ΔTsGP对不同链长麦芽寡糖的催化活性。结果如图6a所示,两者对不同链长的麦芽寡糖的比酶活趋势一致,对麦芽糖几乎无活性,麦芽三糖为其反应的最短底物,但比酶活力小于1 U/mg。当麦芽寡糖链长在4以上时,TsGP及ΔTsGP表现出较高的酶活性,随着链长的增加,两者的比酶活逐渐提高,其中以麦芽七糖为底物时的比酶活力最高。不同物种来源的αGP具有不同的底物特异性,来源于Dictyoglomus turgidum[14]的αGP可催化麦芽三糖生成G-1-P,来源于Pyrococcus furiosus[28]的αGP催化生成G-1-P的最短底物为麦芽四糖。
测定了TsGP及ΔTsGP在不同浓度麦芽七糖条件下的酶活力,通过Graphpad Prism 9.3.0软件酶动力学非线性拟合酶动力学曲线(图6b),TsGP的最大反应速率Vmax为18.95 U/mg,ΔTsGP的最大反应速率为27.34 U/mg。由表3可知,TsGP的kcat值为30.66 s−1、Km值为1.19 mmol/L,ΔTsGP的kcat值为37.09 s−1,Km值为3.30 mmol/L。ΔTsGP的kcat值较TsGP的kcat值略有提高,但是酶与底物的亲和力降低了2.77倍,说明C端非催化区域对底物亲和力影响显著,野生型酶TsGP的催化效率(kcat/Km)是截短酶的2.29倍。
表 3 TsGP和ΔTsGP的动力学参数Table 3. Kinetic parameters of TsGP and ΔTsGP with maltoheptaose酶 Km(mmol/L) kcat
(s−1)kcat/Km(s−1mmol/L−1) TsGP 1.19±0.13 30.66±0.35 25.76±3.61 ΔTsGP 3.30±0.17 37.09±0.47 11.24±0.62 2.3.5 C端非催化结构域影响TsGP热稳定性的机制
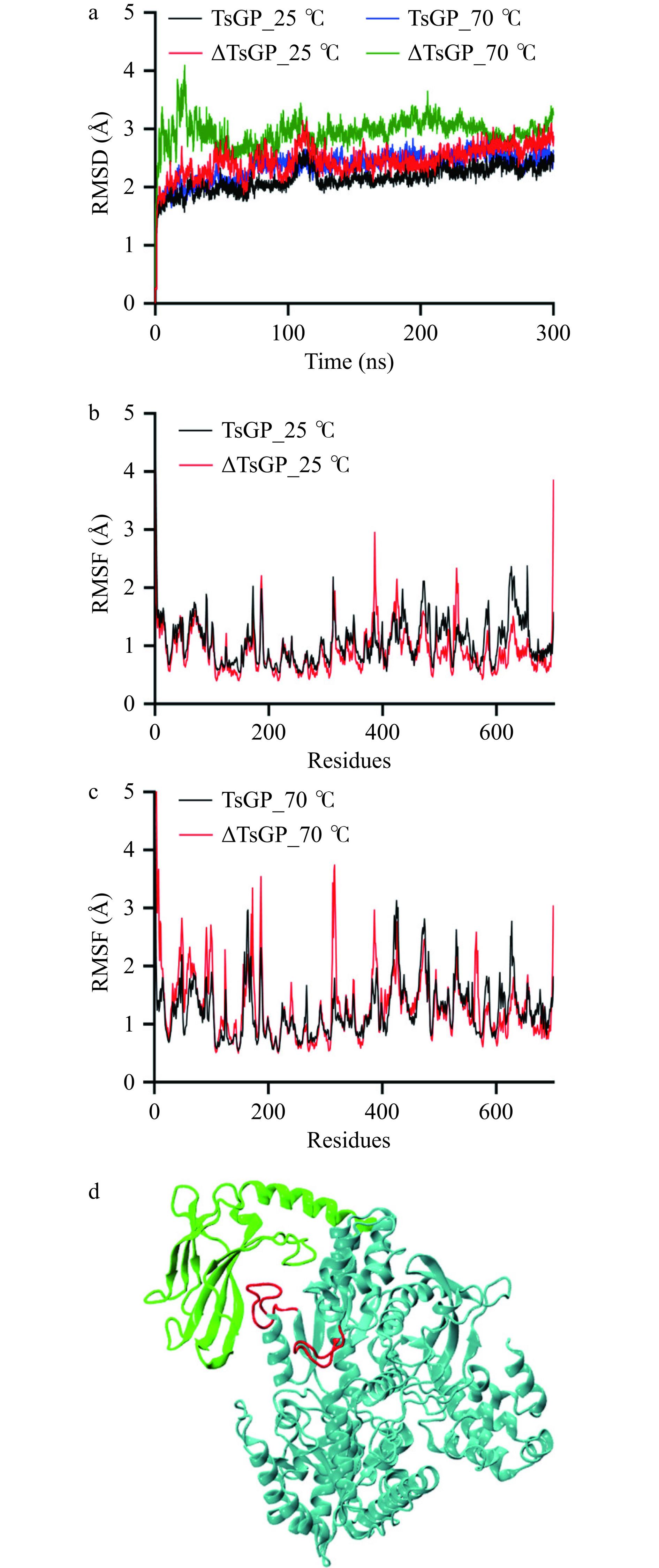
在分子动力学模拟中,均方根误差(Root Mean Square Deviation,RMSD)表示分子结构变化的程度,均方根波动(Root Mean Square Fluctuation,RMSF)表示分子中各个原子运动的自由程度,两者可以表征蛋白质的热稳定性。因此,通过分子动力学模拟,初步解析了C端非催化结构域对TsGP热稳定性的影响。如图7a所示,在模拟温度为25 ℃时,野生型和截短酶的均方根误差(RMSD)变化程度都比较小,在模拟温度为70 ℃时,野生型酶TsGP的RMSD与25 ℃的模拟结果相比变化较小,而截短酶ΔTsGP的RMSD值升高,结构偏移较大,推测C端的非催化结构域有助于稳定酶的结构。进一步统计了不同温度下TsGP与ΔTsGP氨基酸残基的均方根波动值(RMSF),如图7b与图7c所示,随着温度升高,TsGP与ΔTsGP的氨基酸残基RMSF的波动均增大,尤其是截短酶ΔTsGP变化更为明显,表明TsGP在截短后蛋白的结构稳定性降低。进一步分析发现氨基酸残基310~320和382~393的RMSF值表现出较大的波动(图7c)。从TsGP的三维结构(图7d)可以看出,这两段区域均为TsGP的loop区域,其中氨基酸残基382~393可直接与截短的C端非催化结构域相互作用导致其柔性被约束,截短后382~393残基构成的loop区域的柔性增强,进而导致截短酶ΔTsGP在70 ℃以上时酶的热稳定性下降。
3. 结论
本研究通过数据库挖掘和序列分析,成功筛选并鉴定了一种来源于嗜热古菌Thermococcus sp.EP1的新型α-葡聚糖磷酸化酶TsGP。结构分析揭示TsGP的C端为一个独立的非催化结构域。为了深入理解这一结构域对酶表达及酶学性质的影响,进一步构建了C端截短的突变体ΔTsGP。实验结果显示,ΔTsGP的表达量和酶活力分别比野生型酶TsGP提高了3.76倍和30%。ΔTsGP在金属离子影响、最适pH及底物特异性方面与TsGP保持一致。底物亲和力有所降低,而kcat值略有提高。C端非催化结构域的截短影响了TsGP的热稳定性。分子动力学模拟揭示,ΔTsGP中382~393残基构成的loop区域柔性增强,这可能是导致其热稳定性下降的原因。尽管如此,ΔTsGP在70 ℃以下仍保持良好的热稳定性,并在工业酶催化温度(50~65℃)下展现出更高的比酶活,显示出其在工业化应用中的潜力。
综上所述,本研究不仅鉴定了一种新型的嗜热α-葡聚糖磷酸化酶TsGP,还通过C端非催化结构域截短策略显著提高了其表达量和工业酶催化条件下的酶活力。这些发现为αGP的高效表达和酶活力提升提供了理论基础,未来可通过理性设计、半理性设计等酶工程手段进一步提高αGP的工业应用性能。
-
表 1 本研究中使用的菌株、质粒
Table 1 Strains and plasmids used in this study
菌株/质粒 表型 来源 菌株 E. coli DH5α F- φ80 lacZΔM15 Δ(lacZYA-argF)U169 recA1 endA1 hsdR17(rk-, mk+) phoA,
supE44 λ- thi-1 gyrA96 relA1本实验室保存 E. coli BL21(DE3) F-ompT hsdS (rB-mB-)gal dcm 本实验室保存 E. coli BL21(DE3)/pET-21a(+)-AtGP E. coli BL21(DE3) with vector pET-21a(+)-AtGP 本研究 E. coli BL21(DE3)/pET-21a(+)-BtGP E. coli BL21(DE3) with vector pET-21a(+)-BtGP 本研究 E. coli BL21(DE3)/pET-21a(+)-DtGP E. coli BL21(DE3) with vector pET-21a(+)-DtGP 本研究 E. coli BL21(DE3)/pET-21a(+)-HsGP E. coli BL21(DE3) with vector pET-21a(+)-HsGP 本研究 E. coli BL21(DE3)/pET-21a(+)-PfGP E. coli BL21(DE3) with vector pET-21a(+)-PfGP 本研究 E. coli BL21(DE3)/pET-21a(+)-TaGP E. coli BL21(DE3) with vector pET-21a(+)-TaGP 本研究 E. coli BL21(DE3)/pET-21a(+)-TsGP E. coli BL21(DE3) with vector pET-21a(+)-TsGP 本研究 E. coli BL21(DE3)/pET-21a(+)-ΔTsGP E. coli BL21(DE3) with vector pET-21a(+)-ΔTsGP 本研究 质粒 pET-21a(+)-AtGP pET-21a(+) harboring AtGP 本研究 pET-21a(+)-BtGP pET-21a(+) harboing BtGP 本研究 pET-21a(+)-DtGP pET-21a(+) harboring DtGP 本研究 pET-21a(+)-HsGP pET-21a(+) harboring HsGP 本研究 pET-21a(+)-PfGP pET-21a(+) harboring PfGP 本研究 pET-21a(+)-TaGP pET-21a(+) harboring TaGP 本研究 pET-21a(+)-TsGP pET-21a(+) harboring TsGP 本研究 pET-21a(+)-ΔTsGP pET-21a(+) harboring ΔTsGP 本研究 表 2 7种不同来源的α-葡聚糖磷酸化酶
Table 2 Seven different sources of α-glucan phosphorylase
基因名称 基因来源 分子量(kDa) Uniprot ID AtGP Anaerolinea thermolimosa 84.0 A0A3D1JG97 BtGP Bailinhaonella thermotolerans 95.4 A0A3A4B0I3 DtGP Dictyoglomus turgidum 65.4 B8E0Q6 HsGP Halodesulfovibriosp. MK-HDV 86.7 A0A7C8HW27 PfGP Pyrococcus furiosus 97.7 Q8U0Q0 TaGP Thermoplasmata archaeon M9B1D 82.1 A0A2K3J6S9 TsGP Thermococcus sp. EP1 96.5 A0A0P8ZX42 表 3 TsGP和ΔTsGP的动力学参数
Table 3 Kinetic parameters of TsGP and ΔTsGP with maltoheptaose
酶 Km(mmol/L) kcat
(s−1)kcat/Km(s−1mmol/L−1) TsGP 1.19±0.13 30.66±0.35 25.76±3.61 ΔTsGP 3.30±0.17 37.09±0.47 11.24±0.62 -
[1] KADOKAWA J I, LEE L H, YAMAMOTO K. Thermostable α-glucan phosphorylase-catalyzed enzymatic chain-elongation to produce 6-deoxygenated α(1→4)-oligoglucans[J]. Current Organic Chemistry,2021,25(11):1345−1352. doi: 10.2174/1385272825666210607005208
[2] DRULA E, GARRON M L, DOGAN S, et al. The carbohydrate-active enzyme database:Functions and literature[J]. Nucleic Acids Research,2022,50(D1):571−577. doi: 10.1093/nar/gkab1045
[3] ZHOU W, YOU C, MA H W, et al. One-pot biosynthesis of high-concentration α-glucose 1-phosphate from starch by sequential addition of three hyperthermophilic enzymes[J]. Journal of Agricultural and Food Chemistry,2016,64(8):1777−1783. doi: 10.1021/acs.jafc.5b05648
[4] 张尧, 吴绵斌, 杨立荣, 等. 重组Pyrococcus furiosus葡聚糖磷酸化酶催化淀粉合成葡萄糖-1-磷酸[J]. 高校化学工程学报,2016,30(2):417−422. [ZHANG X, WU J B, YANG L R, et al. Recombinant Pyrococcus furiosus dextran phosphorylase catalyzes the synthesis of glucose-1-phosphate from starch[J]. Journal of Chemical Engineering of Chinese Universities,2016,30(2):417−422.] doi: 10.3969/j.issn.1003-9015.2016.02.024 ZHANG X, WU J B, YANG L R, et al. Recombinant Pyrococcus furiosus dextran phosphorylase catalyzes the synthesis of glucose-1-phosphate from starch[J]. Journal of Chemical Engineering of Chinese Universities, 2016, 30(2): 417−422. doi: 10.3969/j.issn.1003-9015.2016.02.024
[5] FENG Y, YAO M, WANG Y, et al. Advances in engineering UDP-sugar supply for recombinant biosynthesis of glycosides in microbes[J]. Biotechnology Advances,2020,41:107538. doi: 10.1016/j.biotechadv.2020.107538
[6] ZHANG S, NI D, ZHU Y, et al. A comprehensive review on the properties, production, and applications of functional glucobioses[J]. Critical Reviews in Food Science and Nutrition,2023,11:1−14. doi: 10.1002/fsn3.2924
[7] WU C H, RISMONDO J, MORGAN R M L, et al. Bacillus subtilis YngB contributes to wall teichoic acid glucosylation and glycolipid formation during anaerobic growth[J]. Journal of Biological Chemistry,2021,296:100384. doi: 10.1016/j.jbc.2021.100384
[8] FUJINAKA H, NAKAMURA J, KOBAYASHI H, et al. Glucose-1-phosphate increases active transport of calcium in intestine[J]. Archives of Biochemistry and Biophysics,2007,460(2):152−160. doi: 10.1016/j.abb.2006.09.006
[9] RONCHERA-OMS C L, JIMENEZ N V, PEIDRO J. Stability of parenteral nutrition admixtures containing organic phosphates[J]. Clinical Nutrition,1995,14(6):373−380. doi: 10.1016/S0261-5614(95)80055-7
[10] ZHANG T, LIU P, WEI H, et al. Protein engineering of glucosylglycerol phosphorylase facilitating efficient and highly regio-and stereoselective glycosylation of polyols in a synthetic system[J]. ACS Catalysis,2022,12(24):15715−15727. doi: 10.1021/acscatal.2c05232
[11] XU K, CHEN X, CHEN Q, et al. Graphene oxide enabled efficient multi-enzyme immobilization for thermodynamic-driven biomanufacturing of fructose[J]. Process Biochemistry,2023,136:245−253.
[12] TIAN C Y, YANG J G, LIU C, et al. Engineering substrate specificity of HAD phosphatases and multienzyme systems development for the thermodynamic-driven manufacturing sugars[J]. Nature Communications,2022,13(1):3582. doi: 10.1038/s41467-022-31371-8
[13] LI Y J, SHI T, HAN P P, et al. Thermodynamics-driven production of value-added d-allulose from inexpensive starch by an in vitro enzymatic synthetic biosystem[J]. ACS Catalysis,2021,11(9):5088−5099. doi: 10.1021/acscatal.0c05718
[14] DAI Y W, ZHANG T, JIANG B, et al. Dictyoglomus turgidum DSM 6724 α-glucan phosphorylase:Characterization and its application in multi-enzyme cascade reaction for D-tagatose production[J]. Applied Biochemistry and Biotechnology,2021,193(11):3719−3731. doi: 10.1007/s12010-021-03624-7
[15] TIAN C Y, YANG J G, LI Y J, et al. Artificially designed routes for the conversion of starch to value-added mannosyl compounds through coupling in vitro and in vivo metabolic engineering strategies[J]. Metabolic Engineering,2020,61:215−224. doi: 10.1016/j.ymben.2020.06.008
[16] CHEN L, QIN Y, MA L, et al. Biosynthesis of Lacto-N-biose I from starch and N-acetylglucosamine via an in vitro synthetic enzymatic biosystem[J]. Synthetic and Systems Biotechnology,2023,8(3):555−562. doi: 10.1016/j.synbio.2023.08.004
[17] HAN P P, YOU C, LI Y J, et al. High-titer production of myo-inositol by a co-immobilized four-enzyme cocktail in biomi-metic mineralized microcapsules[J]. Chemical Engineering Journal,2023,461:141946. doi: 10.1016/j.cej.2023.141946
[18] WEI X, LI Q, HU C, et al. An ATP-free in vitro synthetic enzymatic biosystem facilitating one-pot stoichiometric conversion of starch to mannitol[J]. Applied Microbiology and Biotechnology,2021,105:1913−1924. doi: 10.1007/s00253-021-11154-9
[19] XU X, ZHANG W, YOU C, et al. Biosynthesis of artificial starch and microbial protein from agricultural residue[J]. Science Bulletin,2023,68(2):214−223. doi: 10.1016/j.scib.2023.01.006
[20] MENG D D, WEI X L, BAI X, et al. Artificial in vitro synthetic enzymatic biosystem for the one-pot sustainable biomanufacturing of glucosamine from starch and inorganic ammol/Lonia[J]. ACS Catalysis,2020,10(23):13809−13819. doi: 10.1021/acscatal.0c03767
[21] AMIN K, TRANCHIMAND S, BENVEGNU T, et al. Glycoside hydrolases and glycosyltransferases from hyperthermophilic archaea:Insights on their characteristics and applications in biotechnology[J]. Biomolecules,2021,11(11):1557. doi: 10.3390/biom11111557
[22] EBAID R, WANG H, SHA C, et al. Recent trends in hyperthermophilic enzymes production and future perspectives for biofuel industry:A critical review[J]. Journal of Cleaner Production,2019,238:117925. doi: 10.1016/j.jclepro.2019.117925
[23] CHEN S Q, LIU J F, PEI H D, et al. Molecular investigation of a novel thermostable glucan phosphorylase from Thermoanaerobacter tengcongensis[J]. Enzyme and Microbial Technology,2007,41(3):390−396. doi: 10.1016/j.enzmictec.2007.03.008
[24] YE X H, ROLLIN J, ZHANG Y-H P. Thermophilic α-glucan phosphorylase from Clostridium thermocellum:cloning, characterization and enhanced thermostability[J]. Journal of Molecular Catalysis B:Enzymatic,2010,65(1−4):110−116. doi: 10.1016/j.molcatb.2010.01.015
[25] XAVIER K B, PEIST R, KOSSMANN M, et al. Maltose metabolism in the hyperthermophilic archaeon Thermococcus lito-ralis:Purification and characterization of key enzymes[J]. Journal of Bacteriology,1999,181(11):3358−3367. doi: 10.1128/JB.181.11.3358-3367.1999
[26] HONG Y, WU L, LIU B, et al. Characterization of a glucan phosphorylase from the thermophilic archaeon Sulfolobus tokodaii strain 7[J]. Journal of Molecular Catalysis B:Enzymatic,2008,54(1−2):27−34. doi: 10.1016/j.molcatb.2007.11.003
[27] MUELLER M, TAHAMASA R, SCHWARZ A, et al. ''Short-chain'' α-1,4-glucan phosphorylase having a truncated N-terminal domain:Functional expression and characterization of the enzyme from Sulfolobus solfataricus[J]. Biochimica Biophysica Acta,2009,1794(11):1709−1714. doi: 10.1016/j.bbapap.2009.08.006
[28] MIZANUR R M, GRIFFIN A K, POHL N L. Recombinant production and biochemical characterization of a hyperthermostable α-glucan/maltodextrin phosphorylase from Pyrococcus furiosus[J]. Archaea,2008,2(3):169−176. doi: 10.1155/2008/549759
[29] 段绪果, 张玉华, 黄婷婷, 等. 化学分子伴侣及诱导条件协同强化 Thermotoga maritima α-葡聚糖磷酸化酶可溶性表达[J]. 生物技术通报, 2021, 37(8):233. [DUAN X G, ZHANG Y H HUANG T T, et al. Synergetic enhancing the soluble expression of thermotoga maritima α-glucan phosphorylase by chemical chaperones and induction condition optimization[J]. Biotechnology Bulletin, 2021, 37(8):233-242.] DUAN X G, ZHANG Y H HUANG T T, et al. Synergetic enhancing the soluble expression of thermotoga maritima α-glucan phosphorylase by chemical chaperones and induction condition optimization[J]. Biotechnology Bulletin, 2021, 37(8): 233-242.
[30] UBIPARIP Z, BEERENS K, FRANCEUS J, et al. Thermostable alpha-glucan phosphorylases:Characteristics and industrial applications[J]. Microbiology Biotechnology,2018,102(19):8187−8202. doi: 10.1007/s00253-018-9233-9
[31] JUMPER J, EVANS R, PRITZEL A, et al. Highly accurate protein structure prediction with alpha fold[J]. Nature,2021,596(7873):583−589. doi: 10.1038/s41586-021-03819-2
[32] PHILLIPS J C, HARDY D J, MAIA J D C, et al. Scalable molecular dynamics on CPU and GPU architectures with NAMD[J]. Journal of Chemical Physics,2020,153(4):44130. doi: 10.1063/5.0014475
[33] LEONIDAS D D, ZOGRAPHOS S E, TSITSANOU K E, et al. Glycogen phosphorylase revisited:Extending the resolution of the R-and T-state structures of the free enzyme and in complex with allosteric activators[J]. Acta Crystallographica Section F Structural Biology Communications,2021,77(9):303−311. doi: 10.1107/S2053230X21008542
[34] WATSON K A, MCCLEVERTY C, GEREMIA S, et al. Phosphorylase recognition and phosphorolysis of its oligosaccharide substrate:Answers to a long outstanding question[J]. The EMBO Journal,1999,18(17):4619−4632. doi: 10.1093/emboj/18.17.4619





 下载:
下载:
 下载:
下载:



