Diversity and in Vitro Probiotic Characteristics of Culturable Lactic Acid Bacteria from Goat's Raw Milk
-
摘要: 本研究旨在了解新疆地区山羊原奶中乳酸菌的益生特性、加工特性和安全性,以期为开发功能性乳品奠定一定的基础。实验采用16S rRNA基因测序结合Rep-PCR指纹分型技术对分离出的乳酸菌菌株进行遗传差异分析,检测它们对酸和胆盐的耐受性并对耐受性较好的菌株进行模拟胃肠液耐受性、细胞表面特性(疏水性和自凝聚)、抗菌活性、工艺逆境耐受性(溶菌酶、热)及药敏试验,来评价菌株的体外益生特性和安全性,通过主成分分析筛选最优的益生菌株。研究从15份山羊乳样品中共分离得到23株乳酸菌,包括Leuconostoc mesenteroides(9株)、Lactiplantibacillus plantarum(6株)、Latilactobacillus curvatus(4株)、Enterococcus faecium(2株)、Enterococcus durans(2株)。基于对酸和胆盐耐受性进行初筛的结果表明,有7株菌具有较好的耐受性。其中,2株植物乳植物杆菌(L. plantarum SY43-10和L. plantarum SY43-4)的模拟胃液和肠液处理后活菌数均高于5 lg CFU/mL;24 h自凝集率大于27%、并具有良好疏水性;热和溶菌酶处理后的活菌数仍大于7 lg CFU/mL;药敏试验显示2株菌对氨苄西林和氯霉素表现敏感,对头孢西丁、红霉素和青霉素表现中度敏感或敏感。因此,菌株SY43-10和SY43-4可作为优良的益生菌株,通过体内实验进一步应用于发酵乳品和功能性产品。Abstract: This study was to assess the probiotic attributes, technological properties and safety profiles of lactic acid bacteria isolated from local raw goat milk in Xinjiang, and built a cornerstone of exploitation of functional dairy products. Isolates were analyzed the genetic differences by 16S rRNA gene sequence combined Rep-PCR fingerprint typing technology. Preliminary screening under acid and bile salt base conditions were conducted and before the following experiments, including simulated gastrointestinal fluid tolerance, cell surface properties (hydrophobicity and autoaggregation), antimicrobial activity, processing stress tolerance (lysozyme and heat), and antibiotic sensitivity. Principal component analysis (PCA) was used to identify and screen the strains with optimal probiotic properties. This study found that the 23 lactic acid bacteria strains were isolated from 15 goat milk samples, including Leuconostoc mesenteroides (9 isolates), Lactiplantibacillus plantarum (6 isolates), Latilactobacillus curvatus (4 isolates), Enterococcus faecium (2 isolates) and Enterococcus durans (2 isolates). Seven strains were able to maintain considerable viable bacterial counts under pH2.0 and 0.3% bile salt conditions, indicating their high tolerance to acid and bile salt. Among them, strain SY43-10 and strain SY43-4 possessed a high tolerance towards simulated gastrointestinal fluid with over 5 lg CFU/mL after treatment, high self-agglutination rates of over 27% after 24 h and high hydrophobicit. Meanwhile, the viable counts after heat and lysozyme treatment of these two strains were more than 7 lg CFU/mL. Both of the two strains were susceptible to ampicillin and chloramphenicol, and were intermediate or susceptible to cefoxitin, erythromycin, and penicillin. In conclusion, strains Lactiplantibacillus plantarum SY43-10 and Lactiplantibacillus plantarum SY43-4 can be used as potential beneficial strains for further development of fermented dairy and functional products.
-
自从人类驯化动物、依赖畜牧业养殖业促进人类社会的发展以来,动物原料乳因为含有蛋白质、脂肪、碳水化合物、维生素、矿物质等丰富的营养物质以及微量的生物活性物质,千百年来成为人类首选的营养、功能食品[1]。山羊奶及其乳制品具有低致敏性、营养丰富、易消化等特有的生理生化性质[2−3]。不同动物原料乳中具有复杂多样的微生物,丰富的营养物质为不同的外源微生物提供了理想的栖息生境,通过各种途径进入原料乳,形成了复杂多样的原料乳微生物(microbiota)[4−5]。资料显示,门水平上,原料乳中的微生物以Firmicutes、Proteobacteria、Actinobacteria和Bacteroide为主,但是不同的动物原料乳在属水平上微生物组的差异很大[6−7]。研究发现,动物饲养场地的地理位置、植被、水源、空气,饲养动物乳头接触到的卧具、土壤、青草、饲料以及挤奶设备和人员的卫生状况,都会影响原料乳微生物组成[4]。栖息于原料乳中的微生物对原料乳本身和后续加工乳品的品质起着重要作用,如Lactococcus、Lactobacillus、Streptococcus等乳酸菌和酵母菌能够促进乳品的功能发酵,决定原料乳加工乳品的风味、质地、香味和感官品质[8−10]。
从生态学角度来看,高等哺乳动物原料乳微生物主要来自动物的栖息环境,包括植被、饲草以及接触的人和其它动物,因此动物原料乳是开发野生型优良乳酸菌及潜在益生菌的天然宝库[11−12]。近年来随着人们健康意识不断提高,从动物原料乳中开发具有益生特性的乳酸菌资源成为研究热点[11,13−14]。同时,从乳品消费群体明显的地域性以及肠道微生态理论出发,从本地特色乳品中更有可能筛选到适应于当地饮食习惯人群的益生菌,开发这些乳酸菌将有助于本地企业研发符合当地居民肠道微生态的功能性食品[15]。
我国新疆地区的畜牧业发达,乳源丰富,各民族自古就有动物原料乳自然发酵手工制作、食用的发酵乳制品,被认为是不可或缺的具有益生功能的营养食品,譬如奶疙瘩、酸马奶等。目前报道乳酸菌资源大多来自于发酵制品,而从动物原料乳中筛选益生菌的报道相对较少[16−18]。本研究以新疆哈密采集的山羊乳为研究对象,通过培养法分离乳酸菌,对分离的菌株进行体外的安全性和功能性评价,筛选出具有高效益生特性的益生菌株,以期为提高区域性山羊乳及制品的加工和发展提供支持。
1. 材料与方法
1.1 材料与仪器
山羊乳样品 采自新疆哈密地区巴里坤牧区;改良的MRS培养基、PYG培养基、TSA培养基 北京博奥拓达科技有限公司;溶菌酶(20000 U/mg)、胰蛋白酶(250 U/mg)、胃蛋白酶(30000 U/g)、盐酸、猪胆盐、氯仿、二甲苯、乙酸乙酯 天津市巴斯夫化工有限公司;PCR引物 苏州金唯智生物科技有限公司;致泻大肠埃希氏菌(CICC 10411)、鼠伤寒沙门氏菌(CICC 10420)、产肠毒素大肠埃希氏菌(CICC 10421)、出血性大肠埃希氏菌(CICC 21530) 中国工业微生物菌种保存管理中心(CICC);单核细胞增生李斯特氏菌(CGMCC 1.9136)、血清型肠炎沙门氏菌(CGMCC 1.10754)、金黄色葡萄球菌(CICC 21600) 中国普通微生物菌种保藏管理中心(CGMCC)。
Mini Beadbeater-16型珠磨式组织研磨器 美国Biospec公司;TC-512型PCR仪 英国Techne公司;Quantum-ST5型凝胶成像系统 法国VILBER LOURMAT公司。
1.2 实验方法
1.2.1 样品采集
采样时,采样人员佩戴一次性手套,用无菌生理盐水清洗山羊乳头及周围,弃去头三把奶,收集10 mL山羊乳于无菌采样管中,贴好标签,置于-20 ℃车载冰箱,72 h内运回实验室,立即进行菌种的分离鉴定。
1.2.2 乳酸菌分离与初步鉴定
取1 mL山羊乳于9 mL无菌生理盐水(浓度为0.85%)中,充分振荡混匀。采用十倍梯度稀释法稀释样品,取稀释度为10−2、10−3和10−4的样品悬浮液100 μL分别涂布到MRS固体培养皿上,每个梯度做2次平行,37 ℃培养48 h。每个培养皿挑选8个不同形态、大小和颜色的单菌落重复划线,直到出现菌落完全一致的纯菌株为止。纯菌株经革兰氏染色镜检为阳性、过氧化氢酶为阴性的视为疑似乳酸菌。疑似乳酸菌保存于−80 ℃ 25%甘油中备用。按照Randazzo等[19]描述的方法提取菌株DNA,采用16S rRNA基因通用引物27F(5’-AGAGTTTGATCCTGGCTCAG-3’)和1492R(5’-CTACGGCTACCTTGTTACGA-3’)进行PCR扩增。扩增体系及程序按照Yu等[20]的方法进行。PCR产物纯化后,送至苏州金唯智生物科技有限公司测序,测序结果提交至GenBank数据库中进行序列同源性比对(BLAST)。
1.2.3 基于rep-PCR指纹对乳酸菌的遗传差异分析
rep-PCR指纹分析采用引物ERIC1(5’-GTAAGCTCCTGGGGATTCAC-3’)和ERIC2(5’-AGTAAGTGACTGGGGTGAGCG-3’)对提取的菌株DNA进行PCR扩增。扩增体系和条件按照Matsumiya等[21]的方法进行,扩增产物用1.2%(w/v)的琼脂糖凝胶电泳检验,电泳结束后,在紫外凝胶成像仪中观察电泳结果并拍照,并用Gel Compar II(v6.5)软件对DNA指纹图谱进行聚类分析。
1.2.4 耐酸和耐胆盐特性
将纯化、保藏的乳酸菌菌株在MRS肉汤中37 ℃培养24 h,传代3次后,10000 r/min离心5 min后收集菌体并用40 mL pH7.2磷酸盐缓冲液(phosphate buffer saline,PBS)洗涤2次并重悬制备待测菌悬液。吸取5 mL待测菌液离心后加入等体积pH2.5、pH2.0的MRS肉汤,分别在0和2 h取样进行平板涂布菌落计数。用同样方法加入等体积0.3%、0.5%和1%牛胆盐处理的MRS肉汤,37 ℃保温培养3 h后取样进行平板涂布菌落计数。
1.2.5 模拟胃肠液的耐受性分析
按照Yin等[22]的方法制备模拟胃液和肠液。吸取1 mL待测液与9 mL人工胃液混合均匀后37 ℃培养,分别在0、90和180 min进行活菌计数,用同样方法将待测液与人工肠液混合,分别于0、120和240 min后进行活菌计数。
1.2.6 自凝聚能力分析
根据Li等[23]描述的方法测定自凝聚能力,乳酸菌活化三代后,离心收集菌体,磷酸盐缓冲液(PBS,pH7.2)洗涤两次。菌体用PBS(pH7.2)重悬至浓度为108 CFU/mL,将菌液在37 ℃下放置0、3、24 h,检测在600 nm处吸光度。自凝聚集能力计算公式:
(1) 式中:At表示t时刻的吸光度;A0表示t=0时的吸光度。
1.2.7 疏水性分析
根据Katarzyna等[24]描述的方法测定菌株的疏水性,将细胞悬浮液分别与等体积氯仿,二甲苯和乙酸乙酯混合,静置分层,在600 nm处测定水相的吸光度。疏水性计算公式:
(2) 式中:A0、At分别是试剂混匀前后菌液在600 nm处的吸光度。
1.2.8 常见病原菌抑菌活性测定
按照Mishra等[25]描述的方法测菌株的抗菌活性,采用牛津杯法测定菌株对7种病原菌:致泻大肠埃希氏菌(CICC 10411)、鼠伤寒沙门氏菌(CICC 10420)、产肠毒素大肠埃希氏菌(CICC 10421)、出血性大肠埃希氏菌(CICC 21530)、单核细胞增生李斯特氏菌(CGMCC 1.9136)、血清型肠炎沙门氏菌(CGMCC 1.10754)和金黄色葡萄球菌(CICC 21600)的抑制作用。将生长至对数期的致病菌用生理盐水(0.85%)梯度稀释至10−6~10−7 CFU/mL,均匀涂布在每种致病菌相对应的固体培养基表面,随后在固体培养基上放置牛津杯,并在牛津杯中加入200 μL已培养24 h的乳酸菌上清液,以不加乳酸菌的MRS培养基为空白对照。在4 ℃预扩散6 h后,转入37 ℃培养24 h,测定抑菌圈直径。
1.2.9 耐热及溶菌酶耐受性分析
将菌株在60 ℃水浴中处理5 min,取合适的稀释度进行平板菌落计数。将待测菌株细胞重悬在含有100 mg/L溶菌酶的MRS液体培养基中,分别静止0、30、90 min后,采用MRS进行平板活菌菌落计数。
1.2.10 药敏实验
根据Das等[26]方法对菌株进行12种抗生素药敏实验,抗生素药敏实验采用纸片琼脂扩散法(K-B法)进行,将培养至对数期的菌株离心,弃去上清,菌体用PBS(pH=7.2)重悬至浓度为108 CFU/mL。吸取100 μL菌悬液涂布至MRS固体培养基表面,用镊子将药敏纸片贴在涂有菌悬液的固体培养基表面,37 ℃培养24 h,测量抑菌圈直径。使用的抗生素包括:头孢西丁(FOX,30 μg)、苯唑西林(OXA,1 μg)、红霉素(ERY,15 μg)、氨苄西林(AMP,10 μg)、复方新诺明(SXT,25 μg)、左氧氟沙星(LEV,5 μg)、青霉素(PEN,10 μg)、环丙沙星(CIP,5 μg)、替考拉宁(TEC,30 μg)、氯霉素(CHL,30 μg)、庆大霉素(GEN,120 μg)、多粘菌素(PB,300 μg)。
1.3 数据处理
每个实验均进行3次重复,实验数据运用SPSS 26.0分析,结果以平均值±标准偏差表示。组间比较采用单因素方差分析,并使用Tukey事后检验矫正P值,以P<0.05为差异具有统计学意义。采用Minitab 17.0对菌株进行主成分分析(PCA),根据菌株益生特征综合得到的权重,按照Sharma[27]所使用的方法确定最佳者。采用Origin 2018绘制图表。
2. 结果与分析
2.1 乳酸菌的分离与菌株系统发育分析
从15份山羊原乳样品中共分离得到85株菌株,根据ERIC-rep-PCR指纹分析图谱,确定共有23个不同的带谱,每个rep-PCR带谱选择一个菌株,采用16S rRNA基因测序,经NCBI的BLAST同源性比对,确定为5个种,包括Leuconostoc mesenteroides(9株)、Lactiplantibacillus plantarum(6株)、Latilactobacillus curvatus(4株)、Enterococcus faecium(2株)、Enterococcus durans(2株)(表1,图1A)。
表 1 山羊原奶中筛选出的23株乳酸菌Table 1. 23 strains of LAB were screened from goat raw milk代表菌株 相同带型菌株编号 BLAST最匹配种 相似度(%) SY43-4 SY43(10、16、8、13) Lactiplantibacillus plantarum(MK045823.1) 100 SY50-1 SY50(22、21、15) Latilactobacillus curvatus(MN372055.1) 99 SY43-3 SY43(14) Enterococcus durans(JF896445.1) 99 SY42-16 SY42(15、4);SY-50(24) Leuconostoc mesenteroides(MG754625.1) 100 SY42-17 SY42(10、12);SY-50(4) Leuconostoc mesenteroides(MK182840.1) 100 SY42-18 SY42(13) Enterococcus faecium(KJ156974.1) 99 SY43-7 Lactiplantibacillus plantarum(MN720603.1) 100 SY50-11 Leuconostoc mesenteroides(KT924430.1) 100 根据ERIC引物的rep-PCR指纹分型技术对23株乳酸菌进行聚类分析,如图1B所示,23株乳酸菌获得清晰的电泳图谱,片段大小主要在100~5000 bp之间,每个菌株扩增条带在6~11之间,指纹图谱带型较丰富。根据指纹相似性,在40%的相似性水平上,23株乳酸菌可被聚为5个组。A组包含2株Enterococcus durans;B组包含2株Enterococcus faecium和1株Leuconostoc mesenteoides;C组共包含两个亚群8株菌,CⅠ和CⅡ各包含4株,全部为Leuconostoc mesenteroide;D组只有3株Latilactobacillus curvatus;6株Lactiplantibacillus plantarum则被划分到E组。
2.2 耐酸、耐胆盐特性
微生物能够作为益生菌的一个基本条件就是能够耐受人体胃肠道的恶劣环境,益生菌能够耐受酸和胆盐的暴露才能在胃和小肠中存活[28]。表2显示了23株乳酸菌在不同酸和胆盐浓度的耐受性结果。在pH2.0的酸性条件下培养2 h后23株乳酸菌均可以存活,活菌数下降约1.54~6.31 lg CFU/mL。总体上,有10株菌株(SY50-4、SY50-15、SY43-3、SY43-4、SY43-8、SY43-10、SY43-13、SY42-16、SY42-15和SY42-4)具有较好的耐酸性,活菌数下降约1.54~3.35 lg CFU/mL。随着胆盐浓度的升高,23株乳酸菌的活菌数均降低。0.3%胆盐处理3 h后,23株乳酸菌活菌数降低了2.28~6.45 lg CFU/mL。0.5%胆盐处理3 h后,1株菌(SY42-12)完全不能存活,其余菌的活菌数降低了3.08~7.97 lg CFU/mL。1%胆盐3 h处理后,8株菌完全不能存活,8株菌的活菌数降低了7.56~8.43 lg CFU/mL;而有7株菌(SY50-4、SY43-3、SY43-4、SY43-10、SY42-4、SY42-15和SY42-16)表现出较好的胆盐耐受性,其活菌数降低了5.27~6.80 lg CFU/mL。基于对23株乳酸菌的酸和胆盐的耐受性筛选,本研究最终筛选获得7株乳酸菌(SY50-4、SY43-3、SY43-4、SY43-10、SY42-4、SY42-15和SY42-16)用于进一步的分析其益生特性。
表 2 山羊原奶中筛选的23株乳酸菌的酸和胆盐耐受性特征Table 2. Acid and bile salt tolerance characteristics of 23 strains of LAB in goat raw milk种属 菌株编号 耐酸(lg CFU/mL) 耐胆盐(lg CFU/mL) 原液 pH2.0 0.3% 0.5% 1.0% 屎肠球菌 SY42-13 8.89±0.23de 5.15±0.42ef 4.26±0.09h 2.24±0.25ijkl 0.92±0.18fg SY42-18 9.91±0.38a 5.48±0.27e 5.58±0.35bcd 3.54±0.46def 1.92±0.53cd 弯曲广布乳杆菌 SY50-22 9.68±0.36abc 3.45±0.40i 5.09±0.70def 3.10±0.39fgh 0.00±0.00h SY50-21 9.70±0.66ab 5.60±0.12de 5.44±0.15bcde 3.57±0.19def 1.27±0.16efg SY50-15 8.48±0.26e 6.48±0.30bc 4.54±0.42fgh 2.06±0.14jkl 0.00±0.00h SY50-1 9.44±0.38abcd 5.45±0.33e 5.60±0.49bcd 2.55±0.33hij 0.79±0.70g 植物乳植杆菌 SY43-4 8.84±0.30de 7.30±0.32a 6.56±0.64a 5.76±0.08a 3.32±0.18ab SY43-10 8.94±0.45cde 6.51±0.56bc 6.07±0.06ab 5.38±0.21a 3.67±0.21ab SY43-16 9.94±0.34a 5.32±0.21e 3.57±0.27j 1.97±0.57jkl 0.00±0.00h SY43-13 9.24±0.19abcde 7.51±0.38a 5.25±0.08cde 3.27±0.32efg 1.68±0.48de SY43-8 9.00±0.43bcde 6.30±0.06bcd 4.55±0.29fgh 2.26±0.13ijkl 1.02±0.19fg SY43-7 9.13±0.19bcde 5.72±0.15cde 4.92±0.39efg 2.76±0.74ghi 1.39±0.51ef 耐久肠球菌 SY43-3 9.54±0.49abcd 6.29±0.41bcd 5.15±0.06cdef 4.11±0.34bcd 3.21±0.26ab SY43-14 9.42±0.39abcd 4.01±0.97hi 4.13±0.16i 2.23±0.35ijkl 0.00±0.00h 肠膜明串珠菌 SY42-16 9.16±0.08bcde 6.06±0.34cd 6.05±0.03ab 4.49±0.19b 3.65±0.44a SY42-12 9.05±0.37bcde 4.24±0.22h 4.35±0.31gh 1.62±0.48l 0.00±0.00h SY42-17 8.90±0.23de 4.44±0.37fgh 4.91±0.24efg 2.43±0.23ijk 0.00±0.00h SY42-15 9.02±0.55bcde 6.84±0.57ab 5.67±0.17bcd 3.27±0.16efg 2.22±0.10c SY42-4 8.90±0.35de 6.14±0.44cd 3.45±0.38ef 2.23±0.08c SY42-12 8.86±0.42de 3.90±0.79hi 0.00±0.00m 0.00±0.00h SY50-11 9.73±0.51ab 4.37±0.42gh 3.82±0.56cde 1.36±0.16ef SY50-24 9.71±0.53ab 3.40±0.22i 1.83±0.30kl 0.00±0.00h SY50-4 9.45±0.19abcd 6.10±0.38cd 4.26±0.30bc 2.84±0.38b 注:数值为三次平均值±标准误差;同列数据标有a~n小写字母表示差异显著(P<0.05)。 2.3 模拟胃肠液耐受性
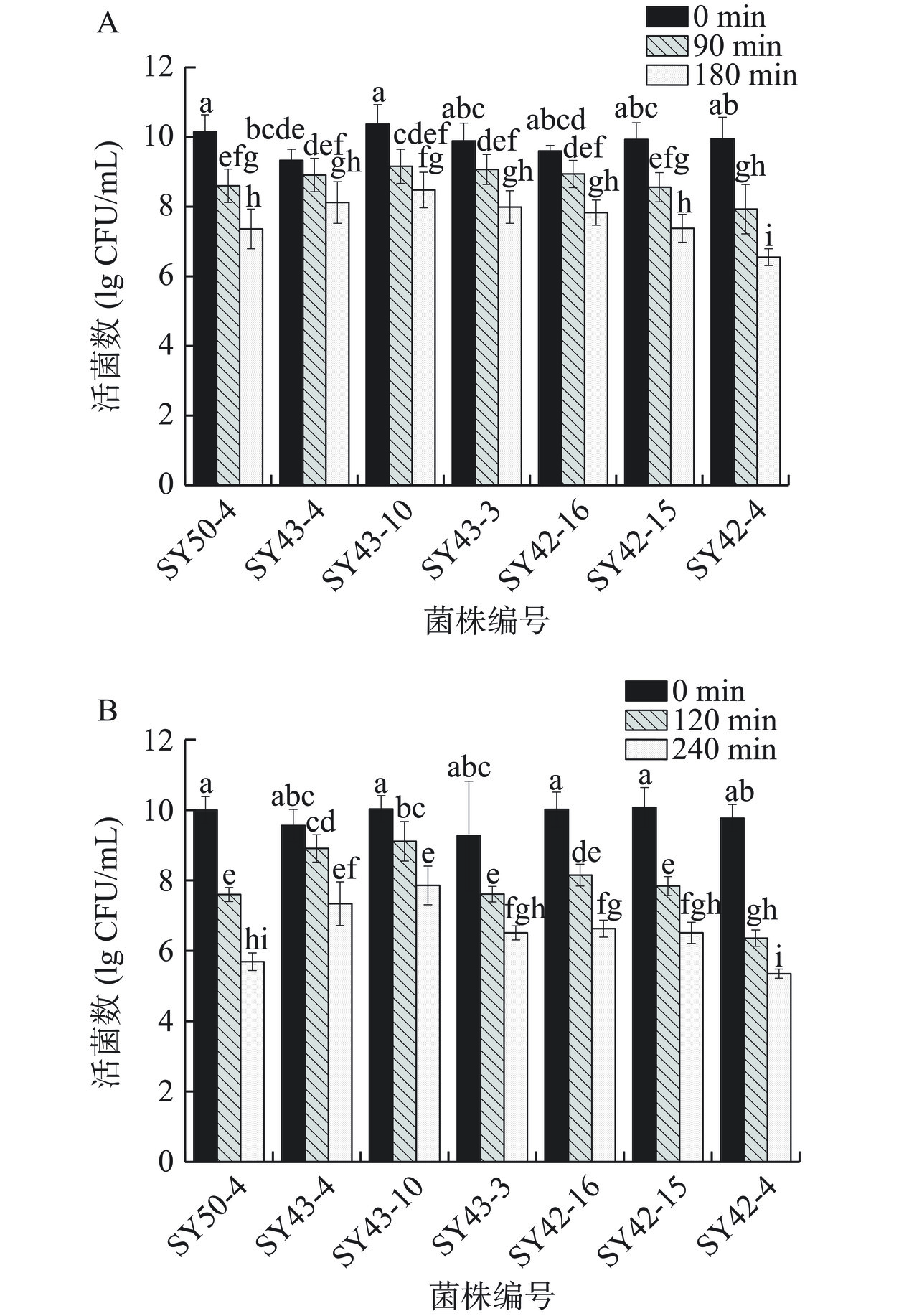
益生菌发挥益生作用的前提是以活菌状态通过人体胃肠道并且经过胃肠道屏障后在体内还有较高的存活率。人胃的环境是高酸性的,且由于饮食影响,pH会在1.5~3.0范围内变化。人体肠液主要为胆汁和胰液,益生菌经过肠液时,会受到胆汁盐和胰蛋白酶的抑制作用[29]。选择酸和胆盐耐受性较好的7株菌用于模拟胃肠液耐受性分析。由图2所示,SY43-4、SY43-10、SY43-3、SY42-16在模拟的胃液处理180 min后活菌数在7.83~8.48 lg CFU/mL,其活菌数高于其他3株菌。SY43-4和SY43-10在模拟的肠液处理240 min后活菌数高于其他菌,分别为7.34 lg CFU/mL和7.86 lg CFU/mL。结果表明,7株菌中有2株菌(SY43-4和SY43-10)表现出了较好的胃肠道耐受性。
2.4 自凝聚和疏水性
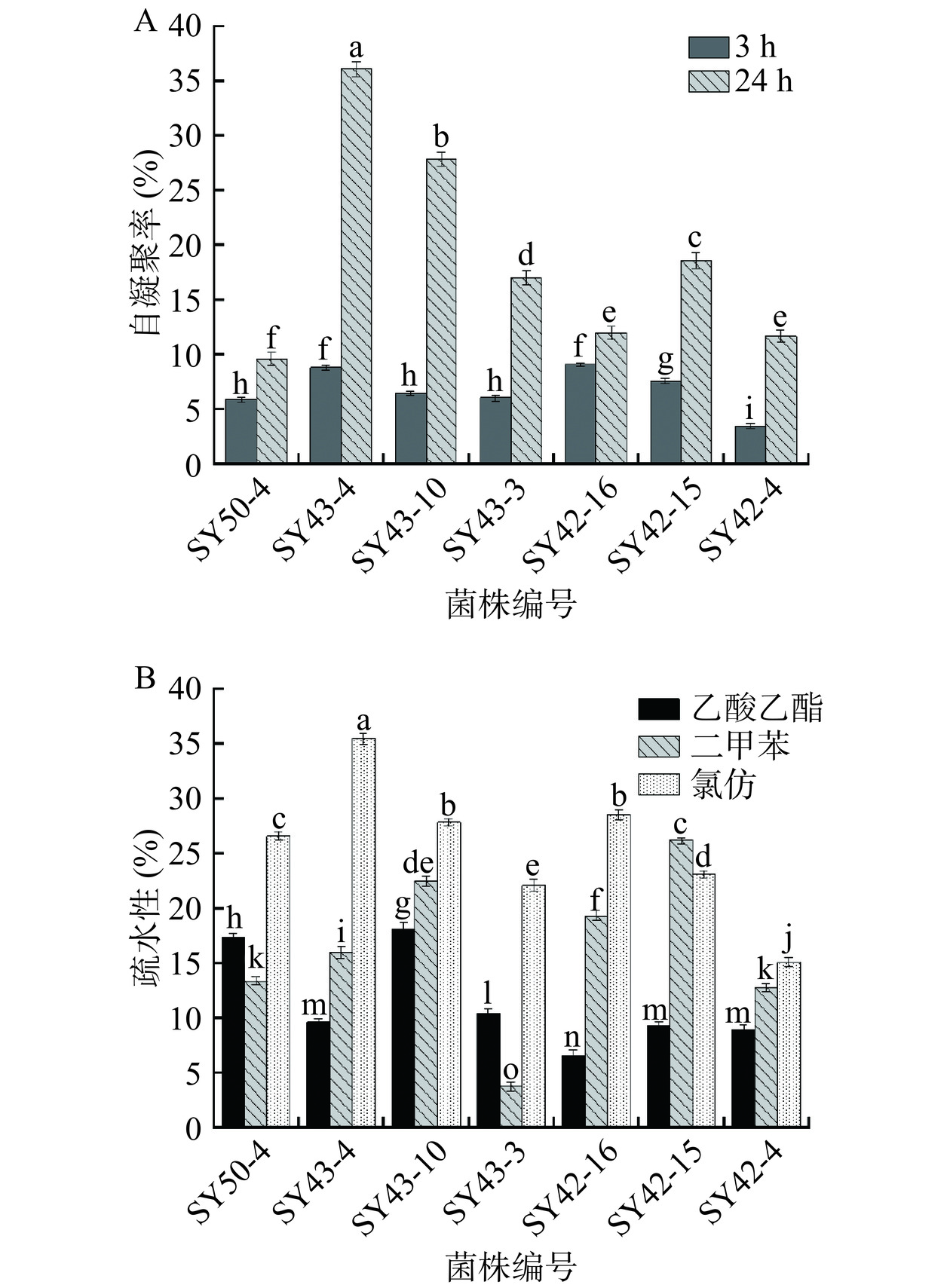
细菌粘附在粘膜表明是肠道定植和提供健康益处所必需的,因此,粘附能力是益生菌的重要筛选标准。研究表明乳酸菌的自凝聚能力和表面疏水性与其对宿主肠道细胞的黏附能力是呈正相关的,因此将自凝聚和疏水性用于筛选优良益生菌的重要指标[30]。7株菌的自凝聚和疏水性测定结果见图3。7株试验菌株在37 ℃培养3 h和24 h期间,所有菌株具有自凝聚能力,其自凝聚范围分别为3.43%~9.05%和9.55%~36.09%。培养24 h后,菌株SY43-4(36.09%)和SY43-10(27.84%)表现出相对较高的自凝聚率(图3A),其自凝聚能力显著高于其他菌(P<0.05)。选择二甲苯作为疏水性有机溶剂,检测乳酸菌的表面疏水性;选择氯仿作为路易斯酸,乙酸乙酯作为路易斯碱,检测乳酸菌的表面所带电荷[31]。由图3B可知所有菌株均具有一定的疏水性,不同试验菌株的疏水性存在显著差异(P<0.05)。除菌株SY42-15外,其余试验菌株对氯仿的亲和力高于对乙酸乙酯和二甲苯。SY43-4、SY43-10、SY42-16和SY50-4对氯仿的疏水性显著高于其他菌(P<0.05),表明其对肠道的黏附性可能较强。
2.5 菌株对热和溶菌酶的耐受性
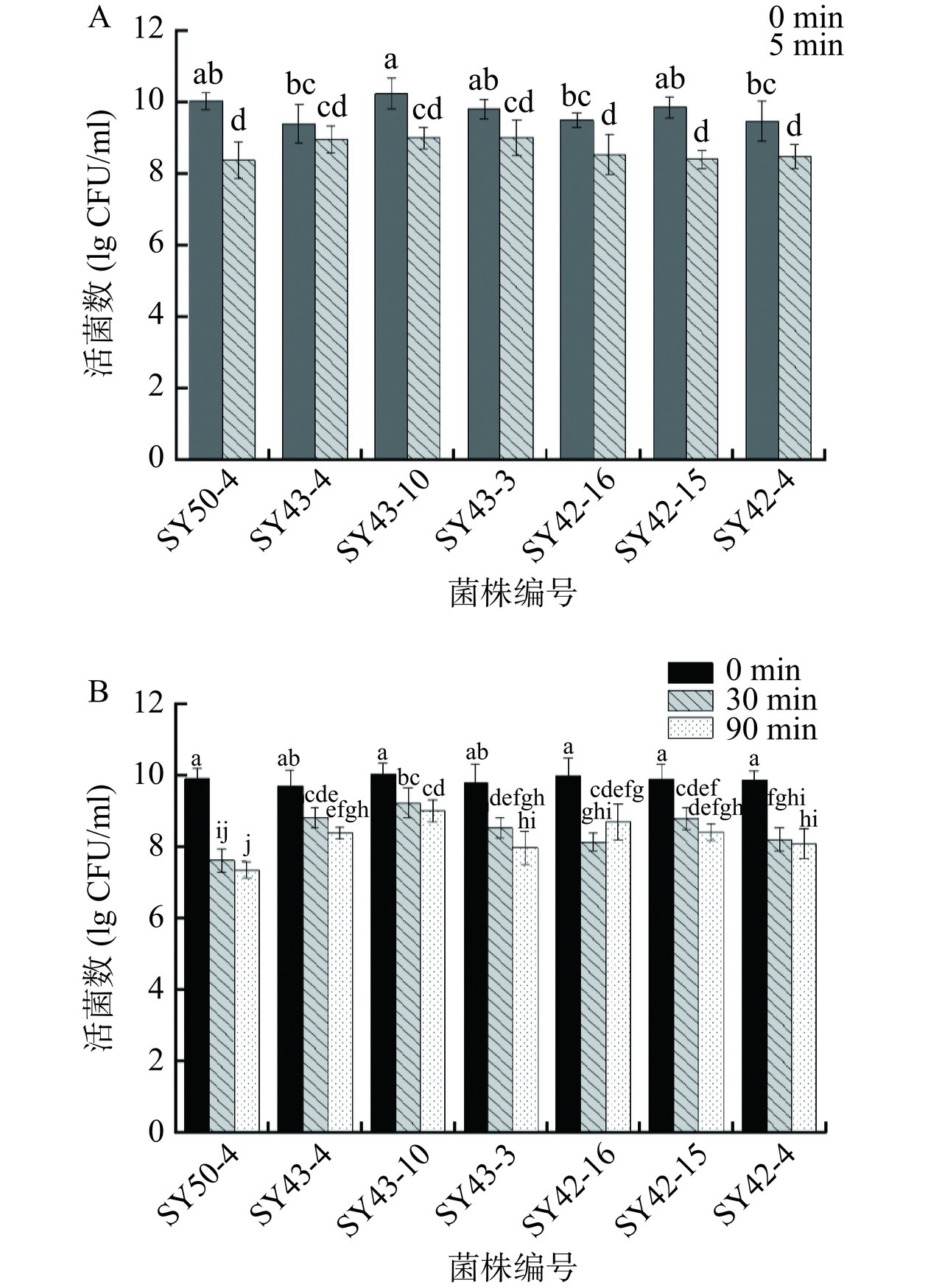
益生菌最大的缺点就是在高温环境下容易失活,限制了其的生产应用。此外,益生菌经过口腔到达人体胃肠道的过程中会受到口腔中天然杀菌物质溶菌酶的抑制,研究报道其质量浓度为0~57 μg/mL,因此益生菌能否耐受溶菌酶是重要筛选指标之一[30]。图4列出了7株试验菌株对热和溶菌酶(100 mg/mL)的耐受性结果,7株试验菌株经过60 ℃处理5 min后活菌数降低(P<0.05),下降的范围为0.65~1.22 lg CFU/mL,其中菌株SY43-10、SY43-4和SY43-3具有较好的热耐受性。在溶菌酶耐受试验中,在100 mg/mL浓度的溶菌酶培养90 min期间活菌数减少量均小于2.5 lg CFU/mL,说明7株菌均对100 mg/mL浓度的溶菌酶具有很好的耐受性。
2.6 抗菌活性
抑菌能力是评价乳酸菌益生作用的重要指标。乳酸菌通过阻断病原菌对肠道上皮细胞的侵染实现其抑菌作用[31]。本研究采用牛津杯法检测7株乳酸菌对常见致病菌的抑菌活性见表3,试验菌株中有3株乳酸菌(SY43-4、SY43-10、SY43-3)抑菌谱较广,可以抑制致泻大肠埃希氏菌,鼠伤寒沙门氏菌,出血性大肠埃希氏菌,血清型肠炎沙门氏菌和金黄色葡萄球菌。而SY50-4、SY42-16、SY42-15、SY42-4抑菌谱较窄,仅表现出对单核细胞增生李斯特氏菌的抑菌效果。
表 3 七株代表乳酸菌对指示菌的抑菌活性Table 3. Antibacterial activity of 7 representative strains of LAB against indicator pathogens菌株 21530 SM 10411 10421 10420 21600 LS SY50-4 − − − − − − +++ SY43-4 +++ +++ ++ − +++ ++ − SY43-10 + +++ + +++ +++ ++ + SY43-3 +++ +++ + + +++ + − SY42-16 − − − + − − +++ SY42-15 − − − − − − ++ SY42-4 − − − − − − +++ 注:−:阴性(=8 mm);+:阳性(8~11 mm);++:11~14 mm;+++:>14 mm;21530:出血性大肠埃希氏菌(CICC 21530);SM:血清型肠炎沙门氏菌(CGMCC 1.10754);10411:致泻大肠埃希氏菌(CICC 10411);10421:产肠毒素大肠埃希氏菌(CICC 10421);10420:鼠伤寒沙门氏菌(CICC 10420);21600:金黄色葡萄球菌(CICC 21600);LS:单核细胞增生李斯特氏菌(CGMCC 1.9136)。 2.7 药敏试验
抗生素耐药性是评估菌株是否安全的重要依据[32]。本研究选用12种常见的抗生素对7株乳酸菌进行抗生素耐药实验。结果如表4所示,7株菌对氨苄西林和氯霉素表现敏感,对头孢西丁、红霉素和青霉素表现中度敏感或敏感,而对苯唑西林、复方新诺明、左氧氟沙星、替考拉林、庆大霉素和多粘菌素则表现耐药性。
表 4 七株代表乳酸菌的抗生素耐药性分析Table 4. Antibiotic resistance of 7 representative strains of LAB菌株 FOX OXA AMP SXT LEV PEN CIP TEC CHL GEN PB ERY SY50-4 S R S R R I R R S R R I SY43-4 I R S R R I R R S R R S SY43-10 I R S R R I R R S R R S SY43-3 I R S R R I R R S R R S SY42-16 I R S R R I R R S R R I SY42-15 I R S R R I R R S R R I SY42-4 I R S R R I I R S R R I 注:FOX:头孢西丁;OXA:苯唑西林;AMP:氨苄西林;SXT:复方新诺明;LEV:左氧氟沙星;PEN:青霉素;CIP:环丙沙星;TEC:替考拉宁;CHL:氯霉素;GEN:庆大霉素;PB:多粘菌素;ERY:红霉素;S:敏感,I:中度敏感,R:耐药。 2.8 主成分分析
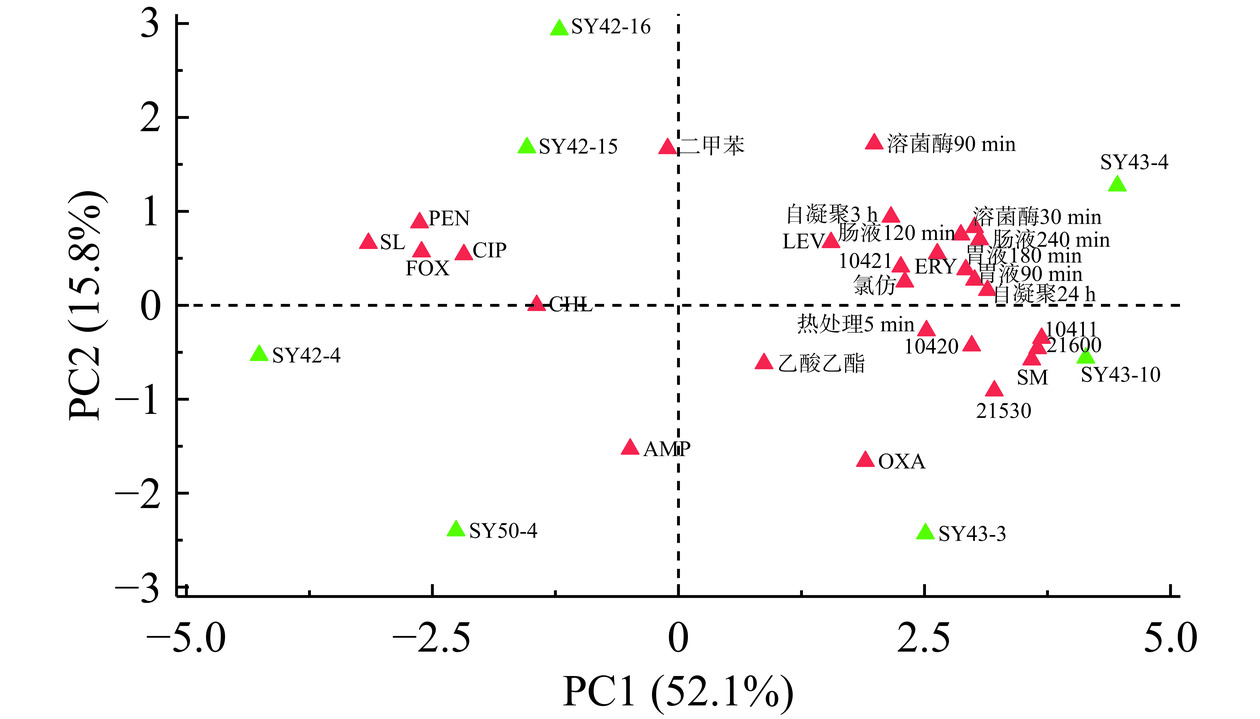
主成分分析能够区分并筛选具有潜在的益生特性的分离菌株。PCA图分析结果(图5)展示了分离菌株在不同益生特征实验中的贡献率和相关性。各分离菌株在耐酸耐胆盐、模拟胃肠液耐受性、自凝聚率和疏水性、对热和溶菌酶耐受性、抑菌活性和耐药性各个变量下总贡献率为67.9%,不同分离菌株逐渐沿着PC1(贡献率52.1%)和PC2(贡献率15.8%)拉开距离,说明在不同试验条件下,分离菌株对各变量的响应存在明显的差异;SY42-16、SY42-15、SY42-4、SY50-4与抗生素耐性的相关性最显著,SY42-4与SL、PEN、CIP、CHL、FOX表现出最高的相关性;SY43-10在模拟胃肠液试验结果中,与不同处理组的变量间表现出最高的相关性;SY43-4与SY43-3在抗菌试验中,与10420、10411、21600、21530、SM五种指示菌抑菌活性表现出最高的相关性,并且对LEV的耐受性较强。综合分析结果表明,分离乳酸菌株SY43-10和SY43-4具有较好的益生特性。
3. 讨论与结论
我国西部地区地域广袤、气候差异显著、生态环境复杂,区域特色规模化动物养殖为当地各民族提供了多样的天然乳品,孕育了极其丰富的乳酸菌资源和遗传多样性。因此,目前大部分乳酸菌资源开发来源于发酵制品,而从动物原乳中筛选益生菌鲜有报道。研究发现,不同生态来源的乳酸菌菌株,人工加工产品的发酵过程相当于驯养过程,连续生长在固定的乳品发酵环境中,菌株基因组由于长期的环境适应性,会导致基因组有关基因丢失和突变,致使重要的表型性状减弱、甚至消失[33]。相反,原奶中的微生物很可能来自动物饲养的饲草以及接触的环境中的植被,很可能孕育了丰富的野生乳酸菌资源[4],为人们筛选到优良的发酵剂、益生菌提供了来源。本研究从新疆山羊原乳中分离筛选乳酸菌并对其体外益生特性和安全性进行评价,以期筛选到优良的本土化菌株,为开发符合当地居民肠道微生态化平衡的功能性和新型食品提供理论参考依据。
本研究从新疆哈密地区15份山羊乳样品中分离筛选出23株乳酸菌,包括4个属5个种,其中Leuconostoc mesenteroides和Lactiplantibacillus plantarum是分离率最高的种,与其它地域来源有关的报道存在差异。马青雯等[34]从6份云南山羊乳饼中鉴定出46株乳酸菌,划分为3个属,6个种,其中优势菌为Enterococcus faecium。Veettil等[11]发现印度山羊原奶中优势菌群为Enterococcus durans和L. plantarum。有人对巴西山羊原奶研究显示,优势种为Weissella cibaria、Lactococcus lactis subsp. lactis和L. plantarum[14]。孟加拉国山羊原乳中乳酸菌多样性较高,分离到Lactobacillus、Leuconostoc、Enterococcus、Streptococcus、Lactococcus和Pediococcus多个属的菌株,其中Lactobacillus是最优势的属,占比50%以上[12]。因此,不同地区畜牧业环境(地理、经纬度、气候、植被等)的差异很可能是造成这种差异的原因。但是,总体上L. plantarum、干酪乳杆菌(Lacticaseibacillus casei)、副干酪乳杆菌(Lacticaseibacillus paracasei)是最广泛报道的乳酸菌。在实践上,L. plantarum是目前应用开发最广泛的乳杆菌种类,不同植物基材料和人体肠道来源的植物乳植物杆菌被广泛报道作为益生菌,应用于发酵饮料以及益生菌制剂缓解人体肥胖、结肠炎、糖尿病、癌症等疾病,显示更广泛的应用潜力[35]。未来动物原料乳仍然是开发野生型优良乳酸菌及潜在益生菌的天然宝库,尤其从消费群体的地域性以及肠道微生态角度,从本地食品更容易筛选到适应当地饮食习惯人群的益生菌菌株的认识,开发区域性的乳酸菌资源仍然很有必要。
益生菌的胃肠道耐受性是很重要的筛选特征,口服益生菌制剂,在乳酸菌通过胃、小肠到达大肠之前,因不耐受酸度和胆汁很容易被淘汰,因此是益生菌菌株能否在肠道中定殖的先决条件。本研究对分离得到的23株乳酸菌进行酸和胆盐耐受性初筛基础上,选择酸和胆盐耐受性较好的7株菌(SY50-4、SY43-3、SY43-4、SY43-10、SY42-16、SY42-15和SY42-4)进一步进行自凝聚、疏水性、耐热和溶菌酶抗性、抑菌性及药敏实验。综合这些特征,基于主成分分析来量化不同体外益生属性对最优益生菌菌株筛选的贡献,通过排名来探究筛选潜在益生菌候选者的可行性[36]。分析显示,两株植物乳植物杆菌L. plantarum SY43-10和L. plantarum SY43-4,在pH2.5人工胃液的活菌数可以保持在8 lg CFU/mL以上,在pH7.5人工肠液的活菌数达到7 lg CFU/mL以上;经过耐热和溶菌酶处理后,活菌数仍然可达到8 lg CFU/mL以上,尤其对人体常见腹泻病原菌致泻大肠埃希氏菌、鼠伤寒沙门氏菌、出血性大肠埃希氏菌、血清型肠炎沙门氏菌和金黄色葡萄球菌均有较强的抑菌功能,具有广谱抑菌性能。耐药性是微生物本身的生存策略之一,与抗生素的存在没有直接关系。相对来说,乳杆菌由质粒携带可移动耐药基因的几率较小,检测到耐药性也较低[37],一般被认为是安全的。本研究显示,除对苯唑西林、复方新诺明、左氧氟沙星、替考拉林、庆大霉素和多粘菌表现一定耐药外,对其它抗生素均表现敏感或中度敏感。
本研究从山羊原奶筛选野生型乳酸菌23株,基于对酸和胆盐耐受性选择较好的7株菌进一步进行主成分分析筛选。结果表明,依据自凝聚、疏水性、耐热、溶菌酶抗性、抑菌及药敏多个表型特征,两株植物乳植物杆菌L. plantarum SY43-10和L. plantarum SY43-4显示了优良的益生菌潜力,有望进一步通过动物造模的体内实验来验证专一的益生菌特征,为进一步应用于功能性乳品加工、后生元饮料的开发奠定基础。
-
表 1 山羊原奶中筛选出的23株乳酸菌
Table 1 23 strains of LAB were screened from goat raw milk
代表菌株 相同带型菌株编号 BLAST最匹配种 相似度(%) SY43-4 SY43(10、16、8、13) Lactiplantibacillus plantarum(MK045823.1) 100 SY50-1 SY50(22、21、15) Latilactobacillus curvatus(MN372055.1) 99 SY43-3 SY43(14) Enterococcus durans(JF896445.1) 99 SY42-16 SY42(15、4);SY-50(24) Leuconostoc mesenteroides(MG754625.1) 100 SY42-17 SY42(10、12);SY-50(4) Leuconostoc mesenteroides(MK182840.1) 100 SY42-18 SY42(13) Enterococcus faecium(KJ156974.1) 99 SY43-7 Lactiplantibacillus plantarum(MN720603.1) 100 SY50-11 Leuconostoc mesenteroides(KT924430.1) 100 表 2 山羊原奶中筛选的23株乳酸菌的酸和胆盐耐受性特征
Table 2 Acid and bile salt tolerance characteristics of 23 strains of LAB in goat raw milk
种属 菌株编号 耐酸(lg CFU/mL) 耐胆盐(lg CFU/mL) 原液 pH2.0 0.3% 0.5% 1.0% 屎肠球菌 SY42-13 8.89±0.23de 5.15±0.42ef 4.26±0.09h 2.24±0.25ijkl 0.92±0.18fg SY42-18 9.91±0.38a 5.48±0.27e 5.58±0.35bcd 3.54±0.46def 1.92±0.53cd 弯曲广布乳杆菌 SY50-22 9.68±0.36abc 3.45±0.40i 5.09±0.70def 3.10±0.39fgh 0.00±0.00h SY50-21 9.70±0.66ab 5.60±0.12de 5.44±0.15bcde 3.57±0.19def 1.27±0.16efg SY50-15 8.48±0.26e 6.48±0.30bc 4.54±0.42fgh 2.06±0.14jkl 0.00±0.00h SY50-1 9.44±0.38abcd 5.45±0.33e 5.60±0.49bcd 2.55±0.33hij 0.79±0.70g 植物乳植杆菌 SY43-4 8.84±0.30de 7.30±0.32a 6.56±0.64a 5.76±0.08a 3.32±0.18ab SY43-10 8.94±0.45cde 6.51±0.56bc 6.07±0.06ab 5.38±0.21a 3.67±0.21ab SY43-16 9.94±0.34a 5.32±0.21e 3.57±0.27j 1.97±0.57jkl 0.00±0.00h SY43-13 9.24±0.19abcde 7.51±0.38a 5.25±0.08cde 3.27±0.32efg 1.68±0.48de SY43-8 9.00±0.43bcde 6.30±0.06bcd 4.55±0.29fgh 2.26±0.13ijkl 1.02±0.19fg SY43-7 9.13±0.19bcde 5.72±0.15cde 4.92±0.39efg 2.76±0.74ghi 1.39±0.51ef 耐久肠球菌 SY43-3 9.54±0.49abcd 6.29±0.41bcd 5.15±0.06cdef 4.11±0.34bcd 3.21±0.26ab SY43-14 9.42±0.39abcd 4.01±0.97hi 4.13±0.16i 2.23±0.35ijkl 0.00±0.00h 肠膜明串珠菌 SY42-16 9.16±0.08bcde 6.06±0.34cd 6.05±0.03ab 4.49±0.19b 3.65±0.44a SY42-12 9.05±0.37bcde 4.24±0.22h 4.35±0.31gh 1.62±0.48l 0.00±0.00h SY42-17 8.90±0.23de 4.44±0.37fgh 4.91±0.24efg 2.43±0.23ijk 0.00±0.00h SY42-15 9.02±0.55bcde 6.84±0.57ab 5.67±0.17bcd 3.27±0.16efg 2.22±0.10c SY42-4 8.90±0.35de 6.14±0.44cd 3.45±0.38ef 2.23±0.08c SY42-12 8.86±0.42de 3.90±0.79hi 0.00±0.00m 0.00±0.00h SY50-11 9.73±0.51ab 4.37±0.42gh 3.82±0.56cde 1.36±0.16ef SY50-24 9.71±0.53ab 3.40±0.22i 1.83±0.30kl 0.00±0.00h SY50-4 9.45±0.19abcd 6.10±0.38cd 4.26±0.30bc 2.84±0.38b 注:数值为三次平均值±标准误差;同列数据标有a~n小写字母表示差异显著(P<0.05)。 表 3 七株代表乳酸菌对指示菌的抑菌活性
Table 3 Antibacterial activity of 7 representative strains of LAB against indicator pathogens
菌株 21530 SM 10411 10421 10420 21600 LS SY50-4 − − − − − − +++ SY43-4 +++ +++ ++ − +++ ++ − SY43-10 + +++ + +++ +++ ++ + SY43-3 +++ +++ + + +++ + − SY42-16 − − − + − − +++ SY42-15 − − − − − − ++ SY42-4 − − − − − − +++ 注:−:阴性(=8 mm);+:阳性(8~11 mm);++:11~14 mm;+++:>14 mm;21530:出血性大肠埃希氏菌(CICC 21530);SM:血清型肠炎沙门氏菌(CGMCC 1.10754);10411:致泻大肠埃希氏菌(CICC 10411);10421:产肠毒素大肠埃希氏菌(CICC 10421);10420:鼠伤寒沙门氏菌(CICC 10420);21600:金黄色葡萄球菌(CICC 21600);LS:单核细胞增生李斯特氏菌(CGMCC 1.9136)。 表 4 七株代表乳酸菌的抗生素耐药性分析
Table 4 Antibiotic resistance of 7 representative strains of LAB
菌株 FOX OXA AMP SXT LEV PEN CIP TEC CHL GEN PB ERY SY50-4 S R S R R I R R S R R I SY43-4 I R S R R I R R S R R S SY43-10 I R S R R I R R S R R S SY43-3 I R S R R I R R S R R S SY42-16 I R S R R I R R S R R I SY42-15 I R S R R I R R S R R I SY42-4 I R S R R I I R S R R I 注:FOX:头孢西丁;OXA:苯唑西林;AMP:氨苄西林;SXT:复方新诺明;LEV:左氧氟沙星;PEN:青霉素;CIP:环丙沙星;TEC:替考拉宁;CHL:氯霉素;GEN:庆大霉素;PB:多粘菌素;ERY:红霉素;S:敏感,I:中度敏感,R:耐药。 -
[1] HAJARA A, WOLFGANG M, TETYANA R, et al. The effects of dairy and dairy derivatives on the gut microbiota:A systematic literature review[J]. Gut Microbes,2020,12(1):1799533. doi: 10.1080/19490976.2020.1799533
[2] SLACANAC V, BOZANIC R, HARDI J, et al. Nutritional and therapeutic value of fermented caprine milk[J]. International Journal of Dairy Technology,2010,63(2):171−89. doi: 10.1111/j.1471-0307.2010.00575.x
[3] RANADHEERA C S, NAUMOVSKI N, AJLOUNI S. Non-bovine milk products as emerging probiotic carriers:Recent developments and innovations[J]. Current Opinion in Food Science, 2018, 22:109−114.
[4] LISA Q, ORLA O S, CATHERINE S, et al. The complex microbiota of raw milk[J]. FEMS Microbiology Reviews,2013,37(5):664−698. doi: 10.1111/1574-6976.12030
[5] PARENTE E, RICCIARDI A, ZOTTA T. The microbiota of dairy milk:A review[J]. International Dairy Journal,2020,107:104714. doi: 10.1016/j.idairyj.2020.104714
[6] NAN L, YUEZHU W, CHUNPING Y, et al. Variation in raw milk microbiota throughout 12 months and the impact of weather conditions[J]. Scientific Reports,2018,8(1):2371. doi: 10.1038/s41598-018-20862-8
[7] S S R, C S E S L, R D S M, et al. Changes in bovine milk bacterial microbiome from healthy and subclinical mastitis affected animals of the Girolando, Gyr, Guzera, and Holstein breeds[J]. International Microbiology, 2022, 25(4):803−815.
[8] VAN KRANENBURG R, KLEEREBEZEM M, VLIEG J V, et al. Flavour formation from amino acids by lactic acid bacteria:predictions from genome sequence analysis[J]. International Dairy Journal,2002,12(2−3):111−121. doi: 10.1016/S0958-6946(01)00132-7
[9] LISA Q, ORLA O S, P B T, et al. High-throughput sequencing for detection of subpopulations of bacteria not previously associated with artisanal cheeses[J]. Applied and Environmental Microbiology,2012,78(16):5717−5723. doi: 10.1128/AEM.00918-12
[10] PICON A, GARDE S, ÁVILA M, et al. Microbiota dynamics and lactic acid bacteria biodiversity in raw goat milk cheeses[J]. International Dairy Journal,2016,58:14−22. doi: 10.1016/j.idairyj.2015.09.010
[11] VEETTIL V, CHITRA V. Probiotic lactic acid bacteria from goat’s milk potential producer of bacteriocin:Evidence from liquid chromatography-mass spectrometry[J]. Journal of Pure and Applied Microbiology,2022,16(1):305−317. doi: 10.22207/JPAM.16.1.19
[12] ZAKIRUL I M, ELIAS U M, TANVIR R M, et al. Isolation and characterization of dominant lactic acid bacteria from raw goat milk:Assessment of probiotic potential and technological properties[J]. Small Ruminant Research,2021,205:106532. doi: 10.1016/j.smallrumres.2021.106532
[13] 娜日苏, 如意, 刘文俊, 等. 中东亚不同地区酸马奶中乳酸菌分离鉴定及优良菌株筛选[J]. 食品与发酵工业,2023,49(17):120−126. [NA R S, RU Y, LIU W J, et al. Isolation and identification of lactic acid bacteria in kumiss in different regions of central and East Asia and screening of excellent strains[J]. Food and Fermentation Industries,2023,49(17):120−126.] NA R S, RU Y, LIU W J, et al. Isolation and identification of lactic acid bacteria in kumiss in different regions of central and East Asia and screening of excellent strains[J]. Food and Fermentation Industries, 2023, 49(17): 120−126.
[14] SILVA L A D, NETO J H P L, CARDARELLI H R. Safety and probiotic functionality of isolated goat milk lactic acid bacteria[J]. Annals of Microbiology,2019,69(2):1497−1505.
[15] SYBESMA W, KORT R, LEE Y K. Locally sourced probiotics, the next opportunity for developing countries?[J]. Trends in Biotechnology,2015,33(4):197−200. doi: 10.1016/j.tibtech.2015.01.002
[16] NURAIDA L. A review:Health promoting lactic acid bacteria in traditional Indonesian fermented foods[J]. Food Science and Human Wellness,2015,4(2):47−55. doi: 10.1016/j.fshw.2015.06.001
[17] MAO B, YAN S. Lactic acid bacteria and fermented fruits and vegetables[J]. Lactic Acid Bacteria:Bioengineering and Industrial Applications, 2019:181−209.
[18] 袁晓霞, 韩慧玲, 李华, 等. 内蒙古地区传统制作奶豆腐和乌日莫中乳酸菌多样性及分布特征[J]. 微生物学通报,2023,50(5):2029−2043. [YUAN X X, HAN H L, LI H, et al. Diversity and distribution characteristics of lactic acid bacteria isolated from traditional milk curd and Urimo in Inner Mongolia[J]. Microbiology China,2023,50(5):2029−2043.] YUAN X X, HAN H L, LI H, et al. Diversity and distribution characteristics of lactic acid bacteria isolated from traditional milk curd and Urimo in Inner Mongolia[J]. Microbiology China, 2023, 50(5): 2029−2043.
[19] RANDAZZO C, TORRIANI S, AKKERMANS A, et al. Diversity, dynamics, and activity of bacterial communities during production of an artisanal sicilian cheese as evaluated by 16S rRNA analysis[J]. Applied and Environmental Microbiology,2002,68:1882−1892. doi: 10.1128/AEM.68.4.1882-1892.2002
[20] YU J, WANG W H, MENGHE B L G, et al. Diversity of lactic acid bacteria associated with traditional fermented dairy products in Mongolia[J]. Journal of Dairy Science,2011,94(7):3224−3229.
[21] MATSUMIYA Y, KATO N, WATANABE K, et al. Molecular epidemiological study of vertical transmission of vaginal Lactobacillus species from mothers to newborn infants in Japanese, by arbitrarily primed polymerase chain reaction[J]. J Infect Chemother,2002,8(1):43−49. doi: 10.1007/s101560200005
[22] YIN K W, UTHUMPORN U, ROSMA A, et al. Evaluation of probiotic potential and anti-hyperglycemic properties of a novel Lactobacillus strain isolated from water kefir grains[J]. Food Science and Biotechnology,2018,27(5):1369−1376. doi: 10.1007/s10068-018-0360-y
[23] LI H, A. G H, MAHMOUD A H, et al. Partial characterization of probiotic lactic acid bacteria isolated from Chinese dairy products[J]. International Journal of Food Properties,2021,24(1):446−456. doi: 10.1080/10942912.2021.1900233
[24] KATARZYNA G, ANNA S, JOANNA F F, et al. Adhesive and hydrophobic properties of the selected LAB isolated from gastrointestinal tract of farming animals[J]. Acta Biochimica Polonica,2016,63(2):311−314.
[25] MISHRA V, PRASAD D N. Application of in vitro methods for selection of Lactobacillus casei strains as potential probiotics[J]. International Journal of Food Microbiology,2005,103(1):109−115. doi: 10.1016/j.ijfoodmicro.2004.10.047
[26] DAS P, KHOWALA S, BISWAS S. In vitro probiotic characterization of Lactobacillus casei isolated from marine samples[J]. LWT,2016,73:383−390. doi: 10.1016/j.lwt.2016.06.029
[27] SHARMA S. Principal component analysis (PCA) to rank countries on their readiness for e-tail[J]. Journal of Retail & Leisure Property,2008,7:87−94.
[28] AYYASH M M, ABDALLA A K, ALKALBANI N S, et al. Invited review:Characterization of new probiotics from dairy and nondairy products-insights into acid tolerance, bile metabolism and tolerance, and adhesion capability[J]. Journal of Dairy Science,2021,104(8):8363−8379. doi: 10.3168/jds.2021-20398
[29] MADJIREBAYE P, XIAO M, MAHAMAT B, et al. In vitro characteristics of lactic acid bacteria probiotics performance and antioxidant effect of fermented soymilk[J]. Food Bioscience,2022,49:101952. doi: 10.1016/j.fbio.2022.101952
[30] RODRÍGUEZ-SÁNCHEZ S, FERNÁNDEZ-PACHECO P, SESEÑA S, et al. Selection of probiotic Lactobacillus strains with antimicrobial activity to be used as biocontrol agents in food industry[J]. LWT,2021,143:111142. doi: 10.1016/j.lwt.2021.111142
[31] BAUTISTA-GALLEGO J, ARROYO-LÓPEZ F N, RANTSIOU K, et al. Screening of lactic acid bacteria isolated from fermented table olives with probiotic potential[J]. Food Research International,2013,50(1):135−142. doi: 10.1016/j.foodres.2012.10.004
[32] ANISIMOVA E, YARULLINA D. Antibiotic resistance of Lactobacillus strains[J]. Current Microbiology,2019,76(12):1407−1416. doi: 10.1007/s00284-019-01769-7
[33] PAPIZADEH M, ROHANI M, NAHREVANIAN H, et al. Probiotic characters of Bifidobacterium and Lactobacillus are a result of the ongoing gene acquisition and genome minimization evolutionary trends[J]. Microbial Pathogenesis,2017,111:118−131. doi: 10.1016/j.micpath.2017.08.021
[34] 马青雯, 王馨聆, 王昱敬, 等. 山羊乳饼中乳酸菌的分离鉴定及优良乳酸菌的初步筛选[J]. 中国乳品工业,2018,46(3):8−12. [MA Q W, WANG X L, WANG Y J. Preliminary screening and isolation and identification of lactic acid bacteria in goat dairy cake[J]. China Dairy Industry,2018,46(3):8−12.] doi: 10.3969/j.issn.1001-2230.2018.03.002 MA Q W, WANG X L, WANG Y J. Preliminary screening and isolation and identification of lactic acid bacteria in goat dairy cake[J]. China Dairy Industry, 2018, 46(3): 8−12. doi: 10.3969/j.issn.1001-2230.2018.03.002
[35] ANDREA C, MARTINA A, GIUSEPPE C, et al. Antibiotic resistance and virulence factors in lactobacilli:Something to carefully consider[J]. Food Microbiology,2022,103:103934. doi: 10.1016/j.fm.2021.103934
[36] NATALIA G, NATALIA B, ROBERTA P, et al. Health-promoting role of Lactiplantibacillus plantarum isolated from fermented foods[J]. Microorganisms,2021,9(2):349. doi: 10.3390/microorganisms9020349
[37] MALLAPPA R H, SINGH D K, ROKANA N, et al. Screening and selection of probiotic Lactobacillus strains of Indian gut origin based on assessment of desired probiotic attributes combined with principal component and heatmap analysis[J]. LWT,2019,105:272−281. doi: 10.1016/j.lwt.2019.02.002
-
期刊类型引用(1)
1. 赵梦瑶,王法云,张立攀,王俊朋,李冰. 谷物低GI食品的研究进展. 现代食品. 2024(22): 165-169 .  百度学术
百度学术
其他类型引用(0)





 下载:
下载:

 下载:
下载:



