Production of Oligopeptide from Soybean Protein by Lactococcus lactis Fermentation
-
摘要: 筛选可以发酵分解大豆蛋白的食源性微生物,对分解产生的多肽进行分子量分析,通过分离纯化获得低聚肽并研究其抗氧化活性。结果表明:从自制泡菜中分离到一株食源性乳酸菌PZ1,经形态和16S rDNA鉴定为乳酸乳球菌;全基因组分析PZ1菌株具有多种肽酶和蛋白酶基因,具备分解蛋白的潜在能力;利用PZ1发酵大豆分离蛋白,采用凝胶渗透色谱分析发酵产生的多肽,分子量1000 Da以下占比达85%;通过超滤纯化获得300~1000 Da的低聚肽;研究大豆低聚肽的抗氧化活性,发现低聚肽对DPPH自由基、羟基自由基(·OH)和超氧阴离子自由基(O2−·)均有较好的清除作用,浓度为2 mg/mL时,清除率分别为79.31%、78.27%和84.62%。因此乳酸乳球菌PZ1能够高效降解大豆蛋白,可作为益生菌应用于大豆功能产品的开发。Abstract: In this study, foodborne microorganisms capable of fermenting and decomposing soy proteins were screened, and molecular weight analysis was performed for the peptides produced during decomposition. Subsequently, oligopeptides were obtained via isolation and purification, and their antioxidant activities were studied. The experiment results showed that a PZ1 strain was isolated from homemade kimchi and identified as Lactococcus lactis based on morphology and 16S rDNA sequence analysis. Whole genome analysis showed that the PZ1 strain contained a variety of peptidases and protease genes that had the potential to decompose proteins. Soybean proteins were then fermented by PZ1, and the polypeptides produced during fermentation were analyzed via gel permeation chromatography, revealing that 85% of polypeptides had a molecular weight below 1000 Da. The oligopeptides with molecular weight 300~1000 Da were obtained via ultrafiltration purification, and their antioxidant activity was studied. The oligopeptides demonstrated a good scavenging effect on DPPH, hydroxyl (·OH), and superoxide anion (O2−·) radicals, at an oligopeptide concentration of 2 mg/mL, the clearance rates were 79.31%, 78.27%, and 84.62%, respectively. Therefore, L. lactis PZ1 could degrade soybean protein efficiently and could be used as a probiotic for developing functional soybean products.
-
Keywords:
- Lactococcus lactis /
- soybean protein /
- oligopeptides /
- antioxidant ability
-
大豆是我国主要经济作物,含有丰富的油脂和植物蛋白等,蛋白含量达到35%~40%,具有很好的营养价值和生物活性[1]。大豆蛋白肽是大豆蛋白经酸解或酶解作用后,通过分离、精制等特殊处理而得到的蛋白质水解产物,而低聚肽则通常是由3~6个氨基酸组成,且相对分子质量低于1000 Da的低肽混合物[2]。大豆蛋白肽进入人体肠胃后不需要经过消化酶作用,可以直接被吸收,利用效果优于游离氨基酸,因此对于病弱人群是理想的蛋白营养源。同时大豆蛋白肽还具有较低的抗原性、调节免疫力、抗衰老、降低胆固醇、降血压、抗氧化等生物活性和生理功能[3−4]。目前,常采用酶解法和微生物发酵法生产大豆蛋白肽,但酶制剂价格昂贵导致酶解法生产成本高,同时酶解后容易产生苦味肽,影响产品口感,微生物发酵法生产蛋白肽则可以把酶的生产和应用合二为一,成本更低,而且发酵生产过程中可以去除大豆肽苦味,具有更好的口感[5−8]。目前报道可用于大豆蛋白水解的微生物有枯草芽孢杆菌[9]、植物乳杆菌[10]、蛹拟青霉[11]、酵母[12]、米曲霉[13]、短小芽孢杆菌[14]等。
乳酸乳球菌是常用的食品微生物,应用领域有乳品发酵、奶酪加工、泡菜和酸面团等[15]。乳酸乳球菌应用于乳品发酵研究较成熟,它可以将乳品中的乳糖转化为乳酸,同时自身的肽酶和分泌的蛋白酶可以促进乳品的蛋白质水解[16]。随着植物蛋白应用越来越广泛,乳酸乳球菌也开始逐渐用于植物蛋白的发酵分解,相对于其他发酵菌,利用乳酸乳球菌生产植物蛋白制品更安全,具有低免疫原性、降低大豆蛋白肽系酸力的优点,乳酸乳球菌在发酵过程中产生的乳酸菌素也可以起到天然防腐的作用[17]。同时由于乳酸乳球菌良好的遗传稳定性,是产业化理想的生产菌株[18−19]。
为深入开发大豆蛋白资源,从自制泡菜中筛选了一株蛋白转化能力好的菌株PZ1,经鉴定为乳酸乳球菌,对该菌进行了全基因组测定,并分析了该菌株的蛋白分解能力,利用该菌发酵大豆分离蛋白产低聚肽,对低聚肽进行了抗氧化活性研究。
1. 材料与方法
1.1 材料与仪器
乳酸乳球菌(Lactococcus lactis)PZ1 从自制泡菜中分离获得,已保藏于中国典型培养物保藏中心,保藏编号为CCTCC NO. M 2022398;米曲霉(Aspergillus oryzae)、植物乳杆菌(Lactobacillus plantarum)、枯草芽孢杆菌(Bacillus subtilis) 本实验室保存菌种;MRS培养基 蛋白胨10 g、牛肉膏5 g、酵母粉4 g、葡萄糖2 g、吐温80 1 mL、磷酸氢二钾2 g、乙酸钠5 g、柠檬酸三铵2 g、硫酸镁0.2 g、硫酸锰0.05 g、琼脂15~20 g、水1000 mL,pH6;大豆分离蛋白培养基 大豆分离蛋白2%,葡萄糖0.5%;98%大豆分离蛋白、Gly-Gly-Tyr-Arg、维生素C(VC) 上海源叶生物科技有限公司;DNA提取试剂盒 天根生化科技(北京)有限公司;DPPH Sigma公司;硫酸亚铁、水杨酸、邻苯三酚、乙醇 天津市天力化学试剂有限公司;过氧化氢 成都市科龙化工试剂厂;盐酸 成都市科隆化学品有限公司;聚乙二醇标准品 上海子起生物科技有限公司。
FA2104N电子天平 上海精其仪器有限公司;722分光光度计 天津市普瑞斯仪器有限公司;DHP-9162恒温培养箱 太仓科教仪器厂;TH2-C恒温摇床 太仓市实验设备厂;GeneAmp PCR Syetem 9700基因扩增仪 美国ABI公司;HC-2518R高速冷冻离心机 安徽中科中佳仪器有限公司;BONA-GM-011超滤仪 山东博纳生物科技集团有限公司;PL-GPC 220凝胶渗透色谱仪 美国Agilent公司。
1.2 实验方法
1.2.1 菌株筛选
从自制泡菜中收集液体,对液体进行梯度稀释,分别将102、103、104倍稀释液100 µL,涂布于MRS培养基中,32 ℃恒温培养2 d,挑取单菌落继续在MRS培养基中划线纯化,获得纯菌株,在大豆分离蛋白培养基中培养,以大豆多肽含量为指标考察菌株对大豆分离蛋白的发酵分解效果,选取效果最好的一株菌命名为PZ1。
1.2.2 菌株鉴定
形态鉴定:PZ1菌株在MRS平板上培养,观察菌落形态,具体方法参照《常见细菌系统鉴定手册》[20]。
16S rDNA鉴定:利用细菌基因组DNA快速抽提试剂盒提取菌株PZ1的总DNA,采用16S rDNA通用引物7F:5'-CAGAGTTTGATCCTGGCT-3'和1540R:5'-AGGAGGTGATCCAGCCGCA-3'进行PCR扩增,扩增程序:94 ℃ 4 min,94 ℃ 45 s,55 ℃ 45 s,72℃ 1 min,72 ℃ 10 min,4 ℃保存。PCR扩增产物送上海生工公司测序,获得的16S rDNA基因序列在NCBI网站上进行比对分析。
1.2.3 菌株PZ1全基因组测定及蛋白水解相关酶分析
将菌株PZ1送北京百迈克公司Nanopore测序技术平台进行全基因组测序。全基因组序列提交GenBank,登录号为PRJNA912843。利用基因和蛋白注释数据库对PZ1的蛋白酶和肽酶等蛋白水解相关基因进行分析。
1.2.4 不同微生物发酵大豆分离蛋白产多肽的比较
将米曲霉、植物乳杆菌、枯草芽孢杆菌(纳豆菌)和乳酸乳球菌PZ1分别接种至大豆分离蛋白培养基,32 ℃发酵2 d,发酵液测大豆多肽含量和蛋白总量,计算多肽转化率,多肽转化率(%)=肽含量/蛋白总量×100。蛋白总量采用凯氏定氮法测定[21]。
1.2.5 大豆多肽含量测定
1.2.5.1 标准曲线的制作
大豆多肽含量测定参照双缩脲法测定[22]。称取Gly-Gly-Tyr-Arg四肽20 mg,加入10 mL 5%的TCA溶解,即为四肽标准溶液。用5% TCA分别配制浓度为0.0、0.2、0.4、0.6、0.8、1.0、1.2、1.4、1.6和1.8 mg/mL的Gly-Gly-Tyr-Arg标准溶液。分别取标准溶液0.75 mL,加入0.5 mL双缩脲试剂,在漩涡混合器振荡混合均匀,静置10 min,2000 r/min,离心10 min,取上清液在540 nm下测定OD值,以不加四肽标准溶液管为空白对照测吸光度值,每个浓度做2个平行。以四肽的浓度作横坐标x(mg/mL),吸光度(OD值)作纵坐标y,制作标准曲线。得到标准曲线方程为y=0.1026x−0.0086,R2=0.9933。
1.2.5.2 多肽的测定
大豆分离蛋白经微生物发酵后,测定发酵液中多肽含量,样品用蒸馏水配成一定浓度后,取1.0 mL,加入1.0 mL 10%三氯乙酸(TCA)溶液,混合均匀后静置10 min,离心机4000 r/min离心15 min,取0.75 mL上清液转移至2 mL离心管中,加入0.5 mL双缩脲试剂,后续操作同1.2.5.1,吸光值与标准曲线对照,即可得到样品的多肽含量(mg/mL)。
1.2.6 大豆多肽的分子量分析
采用安捷伦PL-GPC 220凝胶渗透色谱仪进行大豆多肽的分子量分析,检测器为PL-GPC 50(RI),色谱柱为PL aquqgel-OH MIXED 8 μm两根串联,洗脱液为超纯水,流速1 mL/min,温度30℃,样品用蒸馏水溶解后配成浓度1 mg/mL,用0.22 μm膜过滤,取10 μL上样测试。以聚乙二醇分子量标准品进行分子量分析。
1.2.7 大豆低聚肽的制备
乳酸乳球菌PZ1转接至MRS斜面活化24 h,用接种环取2环接种至灭菌的大豆分离蛋白培养基中,32 ℃培养48 h,得大豆蛋白发酵液PZ1,将大豆蛋白发酵液放置在−20 ℃冰箱冷冻12~24 h,取出室温解冻,超声破碎15 min,超声功率为800 W,用离心管装超声后的溶液在离心机中以10000 r/min,离心10 min。取上清液在超滤装置中进行超滤,分别过5000、1000和300 Da的有机膜,收集300~1000 Da之间的滤液,得低聚肽粗品,冷冻干燥后保存(−20 ℃)备用。采用1.2.5方法测定粗品中低聚肽含量,并配成合适浓度进行活性测定。
1.2.8 低聚肽抗氧化活性测定
低聚肽、大豆分离蛋白和阳性对照维生素C(VC)分别用蒸馏水稀释,配成浓度梯度为2、1.6、1.2、0.8、0.4、0.1 mg/mL。参考卢美欢等[23]测定低聚肽、大豆分离蛋白和VC的抗氧化活性,分别检测对DPPH自由基、羟基自由基(·OH)、超氧阴离子自由基(O2−·)的清除效果。
1.3 数据处理
实验数据采用Excel 2021和DPSv7.05数据系统进行统计分析,显著性水平分析采用 Duncan’s 新复极差法。
2. 结果与分析
2.1 菌株PZ1的鉴定
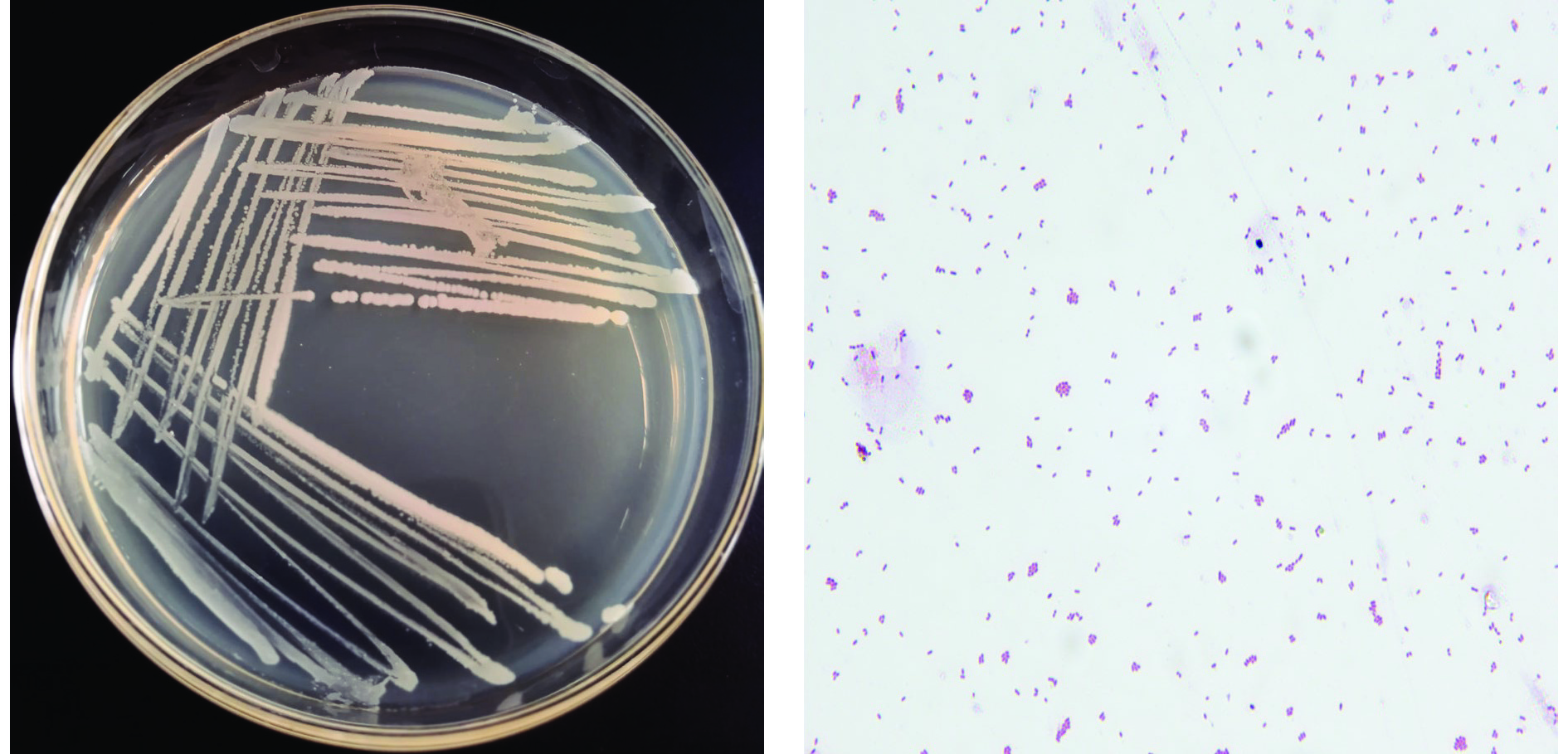
PZ1菌株在MRS平板上培养,观察菌落形态,并进行革兰氏染色。菌落形态如图1所示,菌落凸起,乳黄色,表面光滑,湿润,革兰氏染色显紫色,为阳性菌。显微观察菌株形态为圆球状,大小约0.5 μm。
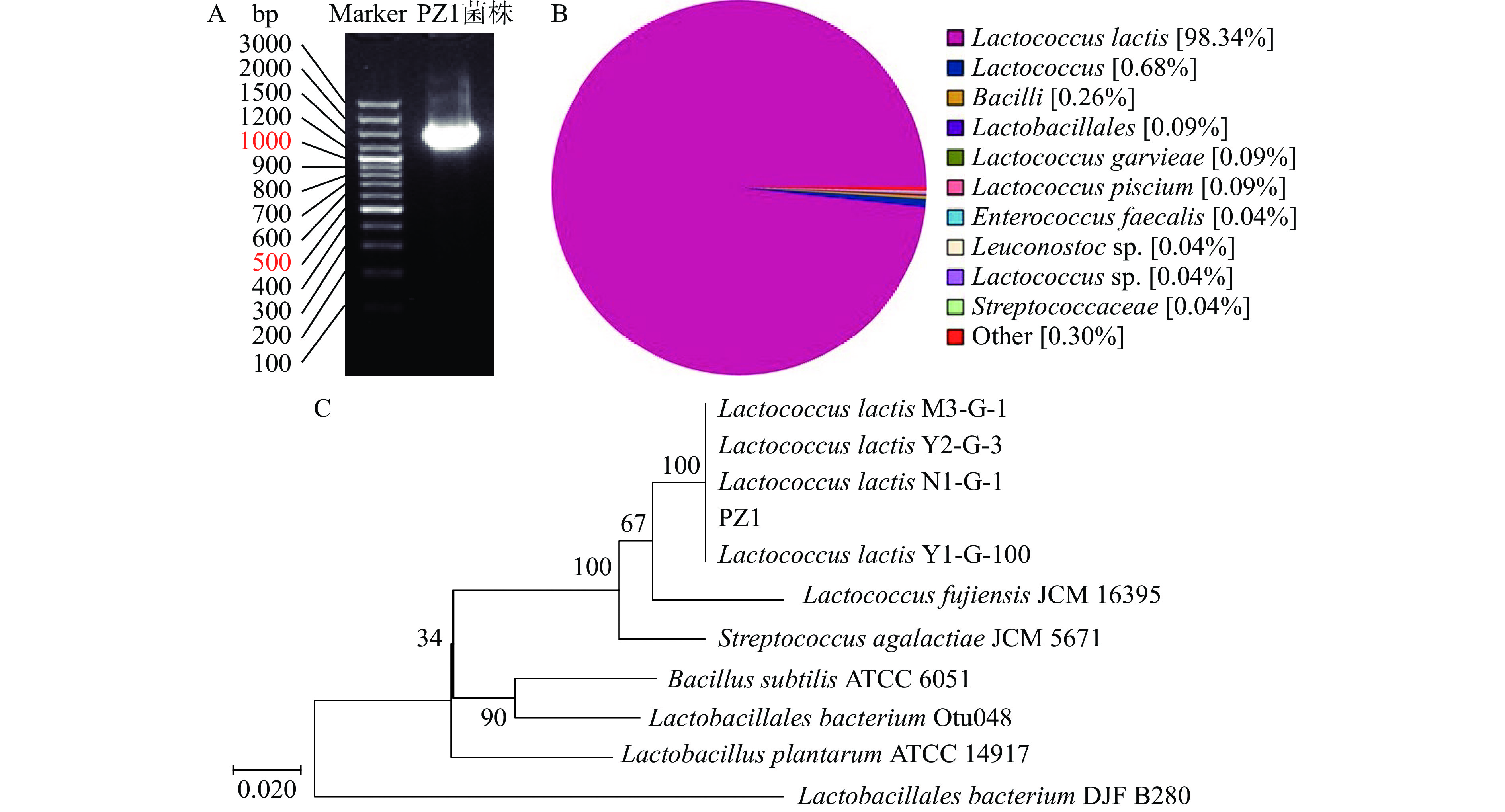
提取PZ1菌株DNA,以该DNA为模板,采用16S rDNA通用引物进行PCR扩增,电泳检测获得约1400 bp的单一特异性条带(图2A)。PCR产物进行测序,序列长度为1441 bp,通过NCBI在线比对,显示菌株PZ1与乳酸乳球菌基因序列同源性达100%,初步判定菌株PZ1为乳酸乳球菌(Lactococcus lactis)。提取乳酸乳球菌PZ1的总DNA,利用Nanopore测序技术平台进行全基因组测序。对乳酸乳球菌PZ1的全基因组序列进行Nr同源物种分布分析,结果显示与乳酸乳球菌的同源性为98.34%(图2B),系统进化树分析显示菌株PZ1和乳酸乳球菌的亲缘最近(图2C)。将乳酸乳球菌PZ1保藏于中国典型培养物保藏中心,保藏号为CCTCC NO.M 2022398。
2.2 乳酸乳球菌PZ1的全基因组分析
乳酸乳球菌PZ1的全基因组测序共得到4083885878 bp Clean Data,组装后完整基因组总长2136240 bp,GC含量(鸟嘌呤G和胞嘧啶C所占的比率)为34.86%。通过Nr数据库注释(表1),搜索和肽酶、蛋白酶相关基因,发现PZ1含有肽酶Peptidase、二肽酶Dipeptidase、内肽酶Endopeptidase、羧肽酶Carboxypeptidase、氨肽酶Aminopeptidase等肽酶基因。通过Swissprot数据库注释,乳酸乳球菌PZ1除了存在多种肽酶基因,还发现了多种蛋白酶protease基因。已有研究表明,蛋白酶的作用是将外源蛋白进行降解,生成大分子肽,肽酶则将大分子肽水解为小分子肽和氨基酸[24]。内肽酶可以切除肽链内部的氨基酸,氨肽酶和羧肽酶都属于外切蛋白酶,氨肽酶在肽链N-末端切除氨基酸,羧肽酶则专一性从肽链的C-末端切除氨基酸,同时羧肽酶可以将短肽末端的疏水性氨基酸切除,达到脱苦的目的[25]。结合以上分析可见乳酸乳球菌PZ1既能产生蛋白酶和多种肽酶将大分子蛋白质分解成短肽,也能产生羧肽酶达到脱苦的目的。
表 1 菌株PZ1在Nr和Swissprot数据库注释的蛋白酶相关基因Table 1. Protease-related genes in strain PZ1 annotated in Nr and Swissprot databases类型 基因ID Nr数据库注释 Swissprot数据库注释 基因 肽酶 GE000428 M24 family metallopeptidase − − GE000132 lipoprotein signal peptidase Lipoprotein signal peptidase lspA GE000716 M50 family peptidase − − GE000983 peptidase M13 Neutral endopeptidase pepO GE000991 peptidase T peptidase T pepT GE001163 peptidase, M16 family − − GE001181 U32 family peptidase Uncharacterized protease HI_0419 HI_0419 GE001183 U32 family peptidase − − GE001251 prepilin peptidase − − GE001274 Xaa-Pro dipeptidyl-peptidase Xaa-Pro dipeptidyl-peptidase pepX GE001510 signal peptidase I Signal peptidase I lepB GE001792 peptidase Aminopeptidase N pepN GE001887 Signal peptidase-like protein − − GE001991 Multimodular transpeptidase-transglycosylase PBP 1A − − GE002123 peptidase Nisin leader peptide-processing serine protease NisP nisP 二肽酶 GE000649 C69 family dipeptidase Probable dipeptidase B pepDB GE001737 C69 family dipeptidase Probable dipeptidase A pepDA GE002330 dipeptidase PepV Beta-Ala-Xaa dipeptidase pepV 内肽酶 GE000078 peptidoglycan endopeptidase − − GE000339 ImmA/IrrE family metallo-endopeptidase − − GE000906 oligoendopeptidase F Oligoendopeptidase F homolog pepF GE001986 gamma-glutamyl-diamino acid-endopeptidase Peptidoglycan DL-endopeptidase CwlO cwlO 羧肽酶 GE000093 D-alanyl-D-alanine carboxypeptidase family protein D-alanyl-D-alanine carboxypeptidase vanY GE000892 LD-carboxypeptidase Putative carboxypeptidase TP_0688 TP_0688 GE001515 D-alanyl-D-alanine carboxypeptidase D-alanyl-D-alanine carboxypeptidase DacA dacA 氨肽酶 GE000828 aminopeptidase P family protein Xaa-Pro dipeptidase pepQ GE001071 Aminopeptidase C Aminopeptidase C pepC GE001874 glutamyl aminopeptidase Glutamyl aminopeptidase pepA GE002061 methionyl aminopeptidase Methionine aminopeptidase map GE002155 aminopeptidase P family protein Uncharacterized peptidase SERP1271 SERP1271 蛋白酶 GE000234 − ATP-dependent Clp protease ATP-binding subunit ClpX clpX GE001348 − Putative zinc metalloprotease LL2128 LL2128 GE001354 − Serine protease Do-like HtrA htrA GE001540 − ATP-dependent zinc metalloprotease FtsH ftsH GE002005 − ATP-dependent Clp protease ATP-binding subunit ClpE clpE GE002104 − ATP-dependent Clp protease proteolytic subunit clpP GE002123 − Nisin leader peptide-processing serine protease NisP nisP 注:“−”表示未在该数据库注释到。 2.3 不同微生物发酵大豆分离蛋白产多肽的比较
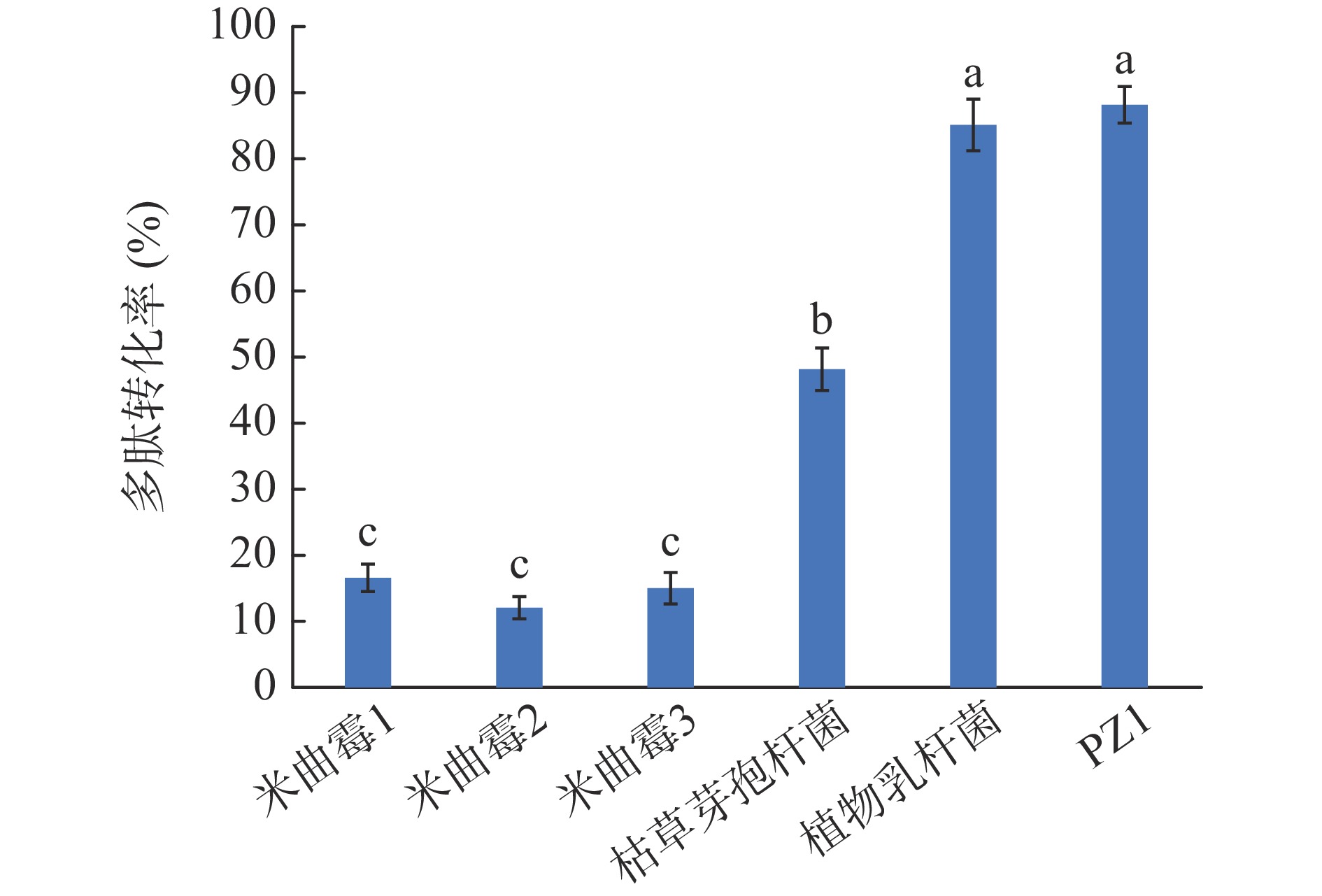
分别采用3株米曲霉,植物乳杆菌、枯草芽孢杆菌(纳豆菌)和乳酸乳球菌PZ1发酵大豆分离蛋白,测发酵后的多肽含量,计算多肽转化率见图3。3株米曲霉对大豆分离蛋白的多肽转化率均低于20%,枯草芽孢杆菌的转化率为47.84%,植物乳杆菌的多肽转化率为85.08%,乳酸乳球菌PZ1的转化率最高,为88.27%,说明PZ1可以较好地分解大豆蛋白。该结果进一步证实了PZ1存在蛋白酶和肽酶基因,可以将蛋白分解为多肽。其他研究也表明乳酸菌可以有效分解大豆蛋白,如刘海燕[26]利用乳酸菌发酵豆粕,可以将豆粕中大分子蛋白质降解为小分子肽,而且增加了豆粕中游离氨基酸含量。何勇锦等[27]利用乳酸短杆菌KLDS-1降解豆粕粉,经生料厌氧发酵后小肽含量达26.12%,比发酵前提高了22.81%。聂庆杰等[28]采用乳酸乳球菌17发酵豆粕取得良好的发酵效果,大豆球蛋白降解率为37.29%。和现有研究相比,本研究乳酸乳球菌PZ1分解大豆蛋白的效率更高,值得进一步开发利用。
2.4 乳酸乳球菌PZ1发酵大豆分离蛋白的分子量分析
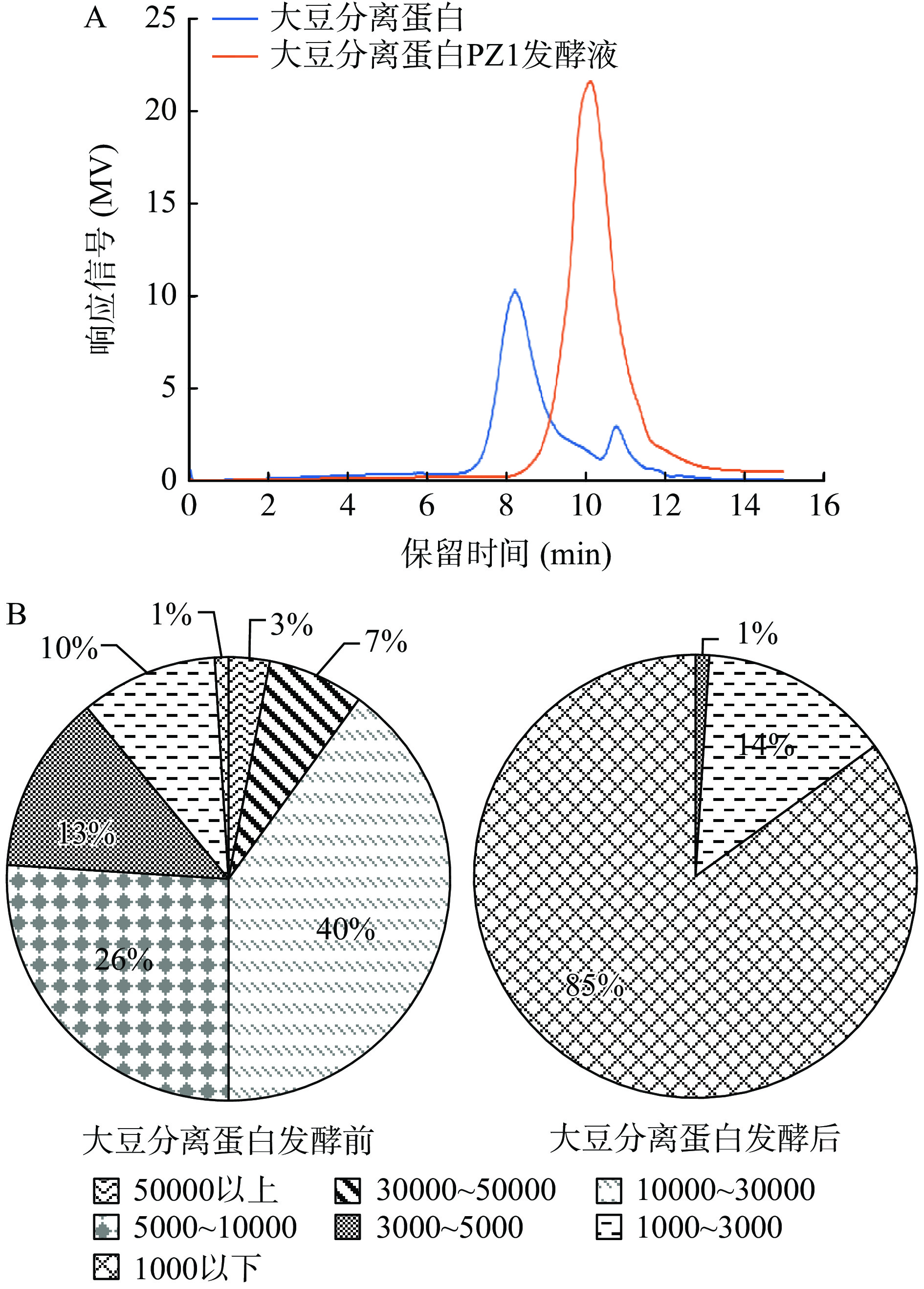
乳酸乳球菌PZ1在大豆分离蛋白培养基中发酵培养后,分别将发酵前和发酵后的蛋白溶液进行凝胶渗透色谱分析,色谱图见图4(A)。经PZ1发酵后的大豆分离蛋白出峰时间更滞后,说明大豆分离蛋白被分解成小分子的多肽。将发酵前和发酵后的蛋白分子量分布进行统计分析,结果如图4(B)所示。结果表明,发酵前的大豆蛋白分子量主要集中在5000 Da以上,占比为76%,分子量1000 Da以下仅占1%;经PZ1分解后大豆多肽分子量≤1000 Da的比例达85%,而分子量在3000~5000 Da的多肽则只有1%,此外,分子量1000~3000 Da比例为14%。WEN等[29]采用中性蛋白酶和碱性蛋白酶对大豆蛋白进行混合酶解,获得87.92%分子量≤1000 Da的大豆肽,1000~3000 Da的比例为11.03%,和本研究的分解效果接近,说明采用乳酸乳球菌PZ1发酵大豆蛋白可以获得较好的降解效果。有资料表明,乳酸菌发酵过程中可以将外源蛋白水解为能直接利用的氨基酸,从而维持自身的正常生长[30]。本研究使用的大豆蛋白培养基只含少量的碳源,其余都是大豆蛋白,营养要素匮乏促使乳酸乳球菌PZ1分泌大量的蛋白酶类对大豆蛋白进行水解,在维持自身生长的同时也产生了大量小分子低聚肽。
2.5 低聚肽抗氧化活性测定
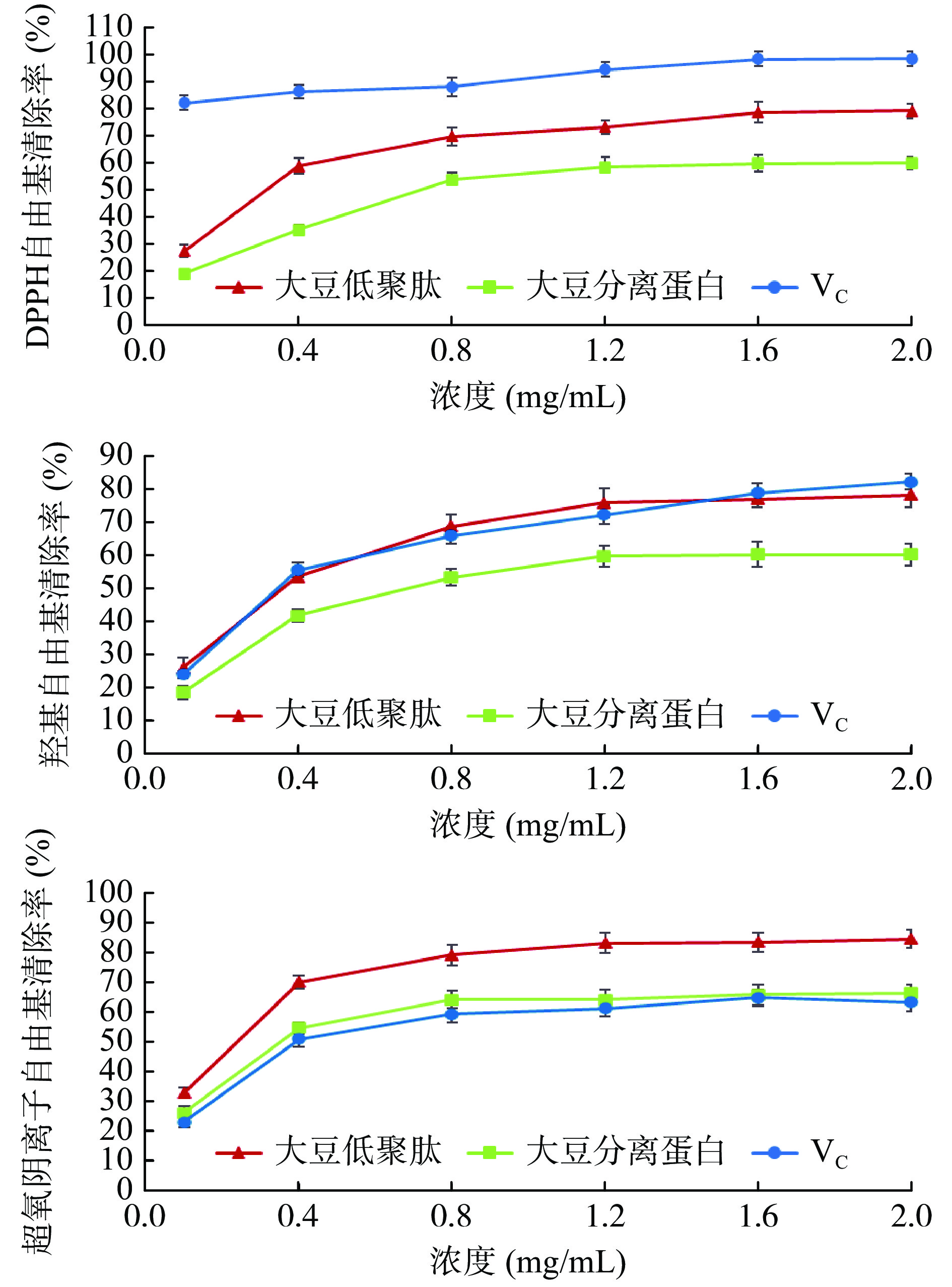
根据资料报道,分子量在多肽的生物活性中起着关键作用,分子量低于1000 Da的低聚肽具有更高的生物活性[31],在增强免疫力、抗氧化作用、吸收性方面均优于分子量较大的多肽[32]。本文通过微生物降解大豆分离蛋白为低聚肽,研究其抗氧化活性。乳酸乳球菌PZ1发酵大豆分离蛋白后,按照1.2.7方法进行低聚肽的制备。同时配制相同浓度的大豆分离蛋白和VC测定抗氧化活性。结果显示,在0.1~2 mg/mL范围内,大豆低聚肽清除DPPH自由基、羟基自由基和超氧阴离子自由基能力均大于大豆分离蛋白(见图5)。而且随着蛋白溶液浓度的增大,清除率也逐渐增大。浓度为2 mg/mL时,大豆低聚肽和大豆分离蛋白清除DPPH自由基能力分别为79.31%和59.92%,清除羟基自由基能力分别为78.27%和60.18%,清除超氧阴离子自由基能力分别为84.62%和66.49%,可见低聚肽的抗氧化活性优于大豆分离蛋白,说明低聚肽具有更好的保健功效。大豆低聚肽清除DPPH自由基、羟基自由基和超氧阴离子自由基的IC50分别为0.32、0.36、0.26 mg/mL。与阳性对照维生素C(VC)相比,在0.1~2 mg/mL范围内,VC对DPPH自由基清除能力优于大豆低聚肽,对羟基自由基清除能力两者没有较大差别,对超氧阴离子自由基清除能力则大豆低聚肽优于VC。
3. 结论
本研究从泡菜中分离出一株乳酸乳球菌PZ1,具有分解大豆蛋白的能力,全基因组分析PZ1菌株具有多种肽酶和蛋白酶基因,包括肽酶、二肽酶、内肽酶、羧肽酶、氨肽酶等基因。利用PZ1发酵大豆分离蛋白,产生的多肽分子量集中在1000 Da以下,达到85%,分子量1000~3000 Da比例为14%。通过对大豆分离蛋白发酵液的超滤纯化,获得了300~1000 Da的低聚肽。研究了大豆低聚肽的抗氧化活性,发现大豆低聚肽清除自由基能力均大于大豆分离蛋白,浓度为2 mg/mL时,大豆低聚肽对DPPH自由基、羟基自由基(·OH)和超氧阴离子自由基(O2−·)的清除率分别为79.31%、78.27%和84.62%。综上所述,乳酸乳球菌PZ1可作为大豆蛋白的降解菌株,在生产功能性低聚肽产品具有潜在的应用价值。
-
表 1 菌株PZ1在Nr和Swissprot数据库注释的蛋白酶相关基因
Table 1 Protease-related genes in strain PZ1 annotated in Nr and Swissprot databases
类型 基因ID Nr数据库注释 Swissprot数据库注释 基因 肽酶 GE000428 M24 family metallopeptidase − − GE000132 lipoprotein signal peptidase Lipoprotein signal peptidase lspA GE000716 M50 family peptidase − − GE000983 peptidase M13 Neutral endopeptidase pepO GE000991 peptidase T peptidase T pepT GE001163 peptidase, M16 family − − GE001181 U32 family peptidase Uncharacterized protease HI_0419 HI_0419 GE001183 U32 family peptidase − − GE001251 prepilin peptidase − − GE001274 Xaa-Pro dipeptidyl-peptidase Xaa-Pro dipeptidyl-peptidase pepX GE001510 signal peptidase I Signal peptidase I lepB GE001792 peptidase Aminopeptidase N pepN GE001887 Signal peptidase-like protein − − GE001991 Multimodular transpeptidase-transglycosylase PBP 1A − − GE002123 peptidase Nisin leader peptide-processing serine protease NisP nisP 二肽酶 GE000649 C69 family dipeptidase Probable dipeptidase B pepDB GE001737 C69 family dipeptidase Probable dipeptidase A pepDA GE002330 dipeptidase PepV Beta-Ala-Xaa dipeptidase pepV 内肽酶 GE000078 peptidoglycan endopeptidase − − GE000339 ImmA/IrrE family metallo-endopeptidase − − GE000906 oligoendopeptidase F Oligoendopeptidase F homolog pepF GE001986 gamma-glutamyl-diamino acid-endopeptidase Peptidoglycan DL-endopeptidase CwlO cwlO 羧肽酶 GE000093 D-alanyl-D-alanine carboxypeptidase family protein D-alanyl-D-alanine carboxypeptidase vanY GE000892 LD-carboxypeptidase Putative carboxypeptidase TP_0688 TP_0688 GE001515 D-alanyl-D-alanine carboxypeptidase D-alanyl-D-alanine carboxypeptidase DacA dacA 氨肽酶 GE000828 aminopeptidase P family protein Xaa-Pro dipeptidase pepQ GE001071 Aminopeptidase C Aminopeptidase C pepC GE001874 glutamyl aminopeptidase Glutamyl aminopeptidase pepA GE002061 methionyl aminopeptidase Methionine aminopeptidase map GE002155 aminopeptidase P family protein Uncharacterized peptidase SERP1271 SERP1271 蛋白酶 GE000234 − ATP-dependent Clp protease ATP-binding subunit ClpX clpX GE001348 − Putative zinc metalloprotease LL2128 LL2128 GE001354 − Serine protease Do-like HtrA htrA GE001540 − ATP-dependent zinc metalloprotease FtsH ftsH GE002005 − ATP-dependent Clp protease ATP-binding subunit ClpE clpE GE002104 − ATP-dependent Clp protease proteolytic subunit clpP GE002123 − Nisin leader peptide-processing serine protease NisP nisP 注:“−”表示未在该数据库注释到。 -
[1] 陈雨生, 江一帆, 张瑛. 中国大豆生产格局变化及其影响因素[J]. 经济地理,2022,42(3):87−94. [CHEN Y S, JIANG Y F, ZHANG Y. Changes of soybean production pattern and its influencing factors in China[J]. Economic Geography,2022,42(3):87−94.] CHEN Y S, JIANG Y F, ZHANG Y . Changes of soybean production pattern and its influencing factors in China[J]. Economic Geography,2022 ,42 (3 ):87 −94 .[2] WU J, DING X. Characterization of inhibition and stability of soy-protein-derived angiotensin I-converting enzyme inhibitory peptides[J]. Food Research International,2002,35(4):367−375. doi: 10.1016/S0963-9969(01)00131-4
[3] DALIRI B M, OFOSU F K, CHELLIAH R, et al. Development of a soy protein hydrolysate with an antihypertensive effect[J]. International Journal of Molecular Sciences,2019,20(6):1496. doi: 10.3390/ijms20061496
[4] DONG Yifei, SUN Na, GE Qi, et al. Antioxidant soy peptide can inhibit xanthine oxidase activity and improve LO2 cell damage[J]. Food Bioscience,2023,52:102455. doi: 10.1016/j.fbio.2023.102455
[5] 王阳, 张景, 陈聪, 等. 纳豆菌液态发酵产低分子大豆蛋白肽的工艺优化[J]. 大连工业大学学报,2012(3):161−164. [WANG Y, ZHANG J, CHEN C, et al. Optimization of Bacillus natto liquid fermentation to produce micromolecule soybean protein peptide[J]. Journal of Dalian Polytechnic University,2012(3):161−164.] doi: 10.3969/j.issn.1674-1404.2012.03.002 WANG Y, ZHANG J, CHEN C, et al . Optimization of Bacillus natto liquid fermentation to produce micromolecule soybean protein peptide[J]. Journal of Dalian Polytechnic University,2012 (3 ):161 −164 . doi: 10.3969/j.issn.1674-1404.2012.03.002[6] 李双, 魏思雯, 吴凤凤. 植物活性肽的研究进展[J]. 食品科技,2022,47(11):85−92. [LI S, WEI S W, WU F F. Research progress of plant active peptides[J]. Food Science and Technology,2022,47(11):85−92.] LI S, WEI S W, WU F F . Research progress of plant active peptides[J]. Food Science and Technology,2022 ,47 (11 ):85 −92 .[7] 黄磊, 周其洋. 大豆水解蛋白中苦味肽的研究进展[J]. 现代食品,2022,28(21):22−24. [HUANG L, ZHOU Q Y. Research progress of bitter peptides in hydrolysed soybean protein[J]. Modern Food,2022,28(21):22−24.] HUANG L, ZHOU Q Y . Research progress of bitter peptides in hydrolysed soybean protein[J]. Modern Food,2022 ,28 (21 ):22 −24 .[8] 王露露, 史茜茜, 王雨桐, 等. 大豆肽的功能活性及其在食品加工中的应用[J]. 农产品加工,2021,520(2):59−63. [WANG L L, SHI Q Q, WANG Y T. Functional activities of soybean peptides and their applications in food processing[J]. Farm Products Processing,2021,520(2):59−63.] WANG L L, SHI Q Q, WANG Y T . Functional activities of soybean peptides and their applications in food processing[J]. Farm Products Processing,2021 ,520 (2 ):59 −63 .[9] 李锡阁, 周成翀, 吴志新, 等. 微生物复合发酵对豆粕营养品质的影响[J]. 华中农业大学学报,2019,38(6):123−131. [LI X G, ZHOU C C, WU Z X, et al. Effect of microbial compound fermentation on nutritional quality of soy bean meal[J]. Journal of Huazhong Agricultural University,2019,38(6):123−131.] LI X G, ZHOU C C, WU Z X, et al . Effect of microbial compound fermentation on nutritional quality of soy bean meal[J]. Journal of Huazhong Agricultural University,2019 ,38 (6 ):123 −131 .[10] 杨紫璇, 张宾乐, 蒋慧, 等. 茅台酒曲植物乳杆菌大豆酸面团发酵面包的营养与抗氧化特性[J]. 食品与发酵工业,2018,44(6):37−42. [YANG Z X, ZHANG B L, JIANG H, et al. The nutrition and antioxidant properties of soybean sourdough and bread fermented by LAB screened from Maotai Qu starter[J]. Food and Fermentation Industries,2018,44(6):37−42.] YANG Z X, ZHANG B L, JIANG H, et al . The nutrition and antioxidant properties of soybean sourdough and bread fermented by LAB screened from Maotai Qu starter[J]. Food and Fermentation Industries,2018 ,44 (6 ):37 −42 .[11] 朱蕴兰, 陈宏伟, 陈安徽, 等. 蛹拟青霉固态发酵法制备大豆渣小肽及其理化特性[J]. 中国油脂,2019,44(12):86−91. [ZHU Y L, CHEN H W, CHEN A H, et al. Preparation and characteristics of small peptides from soybean residue by solid-state fermentation with Paecilomyces militaris[J]. China Oils and Fat,2019,44(12):86−91.] ZHU Y L, CHEN H W, CHEN A H, et al . Preparation and characteristics of small peptides from soybean residue by solid-state fermentation with Paecilomyces militaris[J]. China Oils and Fat,2019 ,44 (12 ):86 −91 .[12] JEONG D M, YOO S J, JEON M S, et al. Genomic features, aroma profiles, and probiotic potential of the Debaryomyces hansenii species complex strains isolated from Korean soybean fermented food[J]. Food Microbiology,2022,105:104011. doi: 10.1016/j.fm.2022.104011
[13] WARA P S S, ADI R, NOVI A, et al. Changes in nutritional and antinutritional aspects of soybean meals by mechanical and solid-state fermentation treatments with Bacillus subtilis and Aspergillus oryzae[J]. Bioresource Technology Reports,2022(17):100925.
[14] 李杰, 张姣锦, 杨红玲, 等. 3种益生菌发酵豆粕的营养品质研究[J]. 饲料研究,2021,44(22):56−59. [LI J, ZHANG J J, YANG H L, et al. Study on nutritional quality of three probiotics fermented soybean meal[J]. Feed Research,2021,44(22):56−59.] LI J, ZHANG J J, YANG H L, et al . Study on nutritional quality of three probiotics fermented soybean meal[J]. Feed Research,2021 ,44 (22 ):56 −59 .[15] 倪珊珊, 黄丽英. 乳酸链球菌素和乳酸乳球菌在食品工业中的应用[J]. 食品工业,2015,6(11):244−247. [NI S S, HUANG L Y. Application of Nisin and Lactococcus lactis in the food industry[J]. The Food Industry,2015,6(11):244−247.] NI S S, HUANG L Y . Application of Nisin and Lactococcus lactis in the food industry[J]. The Food Industry,2015 ,6 (11 ):244 −247 .[16] MIERAU I, KUNJI E, LEENHOUTS K J, et al. Multiple-peptidase mutants of Lactococcus lactis are severely impaired in their ability to grow in milk[J]. Journal of Bacteriology,1996,178(10):2794−2803. doi: 10.1128/jb.178.10.2794-2803.1996
[17] 劳泰财, 余忠丽, 恽辉, 等. 一种富含活性乳酸菌的大豆肽的制备方法:中国, CN111671006A[P]. 2020-9-18. [LAO T C, YU Z L, YUN H, et al. The invention relates to a preparation method of soybean peptide rich in active lactic acid bacteria:China, CN111671006A[P]. 2020-9-18.] LAO T C, YU Z L, YUN H, et al. The invention relates to a preparation method of soybean peptide rich in active lactic acid bacteria: China, CN111671006A[P]. 2020-9-18.
[18] LIU Wenjun, LI Weicheng, ZHENG huijuan, et al. Genomics divergence of Lactococcus lactis subsp. lactis isolated from naturally fermented dairy products[J]. Food Research International,2022,155:111108. doi: 10.1016/j.foodres.2022.111108
[19] 张彦位, 杨玲, 路江浩, 等. 乳酸菌全基因组测序的应用进展[J]. 食品工业科技,2022,43(15):444−450. [ZHANG Y W, YANG L, LU J H, et al. Application progress of lactic acid bacteria whole genome sequencing[J]. Science and Technology of Food Industry,2022,43(15):444−450.] ZHANG Y W, YANG L, LU J H, et al . Application progress of lactic acid bacteria whole genome sequencing[J]. Science and Technology of Food Industry,2022 ,43 (15 ):444 −450 .[20] 东秀珠, 蔡妙英. 常见细菌系统鉴定手册[M]. 北京:科学出版, 2001. [DONG X Z, CAI M Y. Handbook for the identification of common bacterial systems[M]. Beijing:Science Press, 2001.] DONG X Z, CAI M Y. Handbook for the identification of common bacterial systems[M]. Beijing: Science Press, 2001.
[21] 马晓倩, 方玲, 袁月兰, 等. 凯氏定氮法测大豆粉中蛋白质含量的不确定度评定[J]. 现代食品,2021(22):187−189. [MA X Q, FANG L, YUAN Y L, et al. Uncertainty evaluation of protein content in soybean flour by Kjeldahl method[J]. Modern Food,2021(22):187−189.] MA X Q, FANG L, YUAN Y L, et al . Uncertainty evaluation of protein content in soybean flour by Kjeldahl method[J]. Modern Food,2021 (22 ):187 −189 .[22] 鲁伟, 任国谱, 宋俊梅. 蛋白水解液中多肽含量的测定方法[J]. 食品科学,2005,26(7):169−171. [LU W, REN G P, SONG J M. Determination of content of peptides in protein hydrolysates[J]. Food Science,2005,26(7):169−171.] LU W, REN G P, SONG J M . Determination of content of peptides in protein hydrolysates[J]. Food Science,2005 ,26 (7 ):169 −171 .[23] 卢美欢, 马英辉, 王银存, 等. 荞麦醋的抑菌性能及其提取物的抗氧化性研究[J]. 食品工业科技,2012,33(9):82−84. [LU M H, MA Y H, WANG Y C, et al. Study on bacteriostasis property of Buckwheat vinegar and antioxidant activity of its extract[J]. Science and Technology of Food Industry,2012,33(9):82−84.] LU M H, MA Y H, WANG Y C, et al . Study on bacteriostasis property of Buckwheat vinegar and antioxidant activity of its extract[J]. Science and Technology of Food Industry,2012 ,33 (9 ):82 −84 .[24] 王镜岩, 沈同, 朱圣庚, 等. 生物化学[M]. 北京:高等教育出版社, 2017. [WANG J Y. SHEN T, ZHU S G, et al. Biochemistry[M]. Beijing:Higher Education Press, 2017.] WANG J Y. SHEN T, ZHU S G, et al. Biochemistry[M]. Beijing: Higher Education Press, 2017.
[25] 管风波, 宋俊梅. 大豆肽的生产及其苦味脱除方法的研究[J]. 山东轻工业学院学报(自然科学版),2008,22(3):18−20. [GUAN F B, SONG J M. Research progress on production and debitterness technology of soybean peptides[J]. Journal of Shandong Institute of Light Industry,2008,22(3):18−20.] GUAN F B, SONG J M . Research progress on production and debitterness technology of soybean peptides[J]. Journal of Shandong Institute of Light Industry,2008 ,22 (3 ):18 −20 .[26] 刘海燕. 乳酸菌发酵豆粕及其功效研究[D]. 长春:吉林农业大学, 2012. [LIU H Y. Rearch on the functional characterization of soybean meal fermented by lactic acid bacteria[D]. Changchun:Jilin Agricultural University, 2012.] LIU H Y. Rearch on the functional characterization of soybean meal fermented by lactic acid bacteria[D]. Changchun: Jilin Agricultural University, 2012.
[27] 何勇锦, 罗振达, 戴紫燕, 等. 乳酸菌生料固态发酵豆粕工艺的优化[J]. 贵州农业科学,2012(10):151−153. [[HE Y J, LUO Z D, DAI Z Y, et al. Process optimization of solid state fermentation of raw soybean meal by Lactobacillus[J]. Guizhou Agricultural Sciences,2012(10):151−153.] [HE Y J, LUO Z D, DAI Z Y, et al . Process optimization of solid state fermentation of raw soybean meal by Lactobacillus[J]. Guizhou Agricultural Sciences,2012 (10 ):151 −153 .[28] 聂庆杰, 杨红玲, 张姣锦, 等. 水产功能性发酵豆粕用乳酸菌菌株的筛选研究[J]. 饲料研究,2022,45(21):74−77. [NIE Q J, YANG H L, ZHANG J J, et al. Screening of lactic acid bacteria for aquatic functional soybean meal fermentation[J]. Feed Research,2022,45(21):74−77.] NIE Q J, YANG H L, ZHANG J J, et al . Screening of lactic acid bacteria for aquatic functional soybean meal fermentation[J]. Feed Research,2022 ,45 (21 ):74 −77 .[29] WEN Lingrong, BI Huimin, ZHOU Xuesong, et al. Structure characterization of soybean peptides and their protective activity against intestinal inflammation[J]. Food Chemistry,2022,387:132868. doi: 10.1016/j.foodchem.2022.132868
[30] 杜越欧, 侯俊财. 乳酸菌蛋白水解体系及相关基因表达的研究进展[J]. 食品工业科技,2013,34(3):383−386. [DU Y O, HOU J C. Research progress in proteolysis system of lactic acid bacteria and related gene expression[J]. Science and Technology of Food Industry,2013,34(3):383−386.] DU Y O, HOU J C . Research progress in proteolysis system of lactic acid bacteria and related gene expression[J]. Science and Technology of Food Industry,2013 ,34 (3 ):383 −386 .[31] 张连慧, 贺寅, 刘新旗. 大豆肽的研究进展及其发展前景[J]. 食品工业科技,2012,33(22):406−413. [ZHANG L H, HE Y, LIU X Q. Research advance of soy peptide and its application in food industry[J]. Science and Technology of Food Industry,2012,33(22):406−413.] ZHANG L H, HE Y, LIU X Q . Research advance of soy peptide and its application in food industry[J]. Science and Technology of Food Industry,2012 ,33 (22 ):406 −413 .[32] 易维学. 大豆低聚肽的功能性质及其应用[J]. 中国食品工业,2001(2):36−37. [YI W X. Functional properties and applications of soybean oligopeptide[J]. China Food Industry,2001(2):36−37.] YI W X . Functional properties and applications of soybean oligopeptide[J]. China Food Industry,2001 (2 ):36 −37 .





 下载:
下载:
 下载:
下载:



