Expression, Purification and Biological Characteristics Prediction of Protein HmpA of Salmonella paratyphi A
-
摘要: 目的:原核表达甲型副伤寒沙门氏菌(Salmonella paratyphi A)HmpA蛋白,并对其进行生物信息学分析,为研究副甲菌HmpA蛋白对于宿主体内一氧化氮(NO)信号通路的影响提供理论参考。方法:PCR扩增hmpA基因,亚克隆至T载体后,再构建pNdeI-hmpA表达载体,转化BL21(DE3),经IPTG诱导后,SDS-PAGE检测蛋白表达形式,利用Histrap预装柱亲和纯化HmpA蛋白,并用Western blot鉴定,生物信息学多方法分析HmpA蛋白特征。结果:成功构建了原核表达载体pNdeI-hmpA,低温诱导时,HmpA蛋白以包涵体和可溶表达形式并存;纯化后的HmpA蛋白可被His标签抗体检测。生物信息学分析提示HmpA蛋白为亲水性蛋白;无跨膜结构域、无信号肽;蛋白二级结构主要由α螺旋与不规则卷曲构成,三级结构模型类似环状;蛋白结构域分析表明含有1个功能结构域,属于PRK13289超级家族;共有32个磷酸化位点;与沙门氏菌多个降解硝酸盐和杀伤性NO的蛋白酶或转录因子存在关联。结论:本研究成功利用基因工程技术获得了甲型副伤寒沙门氏菌HmpA蛋白,并通过生物信息学方法预测了其部分生物学特征,为后续揭示甲型副伤寒沙门氏HmpA蛋白对于宿主体内的一氧化氮信号通路的影响提供理论支持。Abstract: Objective: To express the protein HmpA of Salmonella paratyphi A in prokaryotes, and perform bioinformatics analysis on it to provide a theoretical reference for studying the effect of this protein on the nitric oxide (NO) signaling pathway in the host. Methods: The hmpA gene was amplified by PCR and subcloned into the T-vector. Then the expression vector pNdeI-hmpA was constructed and transformed into BL21 (DE3). After induced by IPTG, the recombinant protein expression form was assessed by SDS-PAGE. HmpA was purified using Histrap preload column and identified by Western blot. Bioinformatics technology was used to analyze the characteristics of HmpA. Results: The prokaryotic expression vector pNdeI-hmpA was successfully constructed. HmpA coexisted in the inclusion body and soluble forms under low temperatures after being induced with IPTG. The purified HmpA protein could be detected by the anti-His antibody. Bioinformatics analysis suggested that HmpA was a hydrophilic protein with no transmembrane structural domain or signal peptide. The secondary structure of this protein was mainly composed of α-helix with irregular convolutions, and the tertiary structure model was similar to a ring. The protein structural domain analysis showed that HmpA contains one functional structural domain belonging to the PRK13289 superfamily. There were 32 phosphorylation sites in HmpA. Associated with multiple Salmonella proteases or transcription factors that degrade nitrates and killer NO. Conclusion: This study successfully obtained Salmonella paratyphi A HmpA protein by genetic engineering technology and predicted part of its biological characteristics by bioinformatics methods, which would provide theoretical support for the subsequent uncovering of the influence of HmpA of Salmonella paratyphi A on the nitric oxide signaling pathway in the host.
-
肠热症由伤寒沙门氏菌、副伤寒沙门氏菌(甲、乙、丙)所引起,主要通过污染的水源和食物而爆发流行,不良的卫生条件和生活习惯是疾病发生的主要原因,但是随着人员交际的增加,全球肠热症病发率逐渐成为大部分国家突出的卫生问题[1−3]。近年来,伤寒沙门氏菌感染占比呈下降趋势,而甲型副伤寒沙门氏菌(后简称副甲菌)在某些地区感染占比则呈现上升趋势[4−5]。据调查,近年来云南、广西、湖南、贵州、福建和广东6个省份伤寒、副伤寒的发病率均显著高于全国平均水平[6]。虽然云南省的伤寒、副伤寒的发病率总体呈逐年下降趋势,但患病率在全国仍处于较高的水平且多年位居第一[7−8]。副甲菌感染引起的疾病在临床上以持续高热,相对缓脉,肌肉酸痛,特征性中毒症,脾肿大,玫瑰疹与白细胞减少等为特征,主要并发症则包括肠出血、肠穿孔等[9−12]。因此探究副甲菌逃脱宿主免疫杀伤的机制十分必要。
多种生物体利用NO来抵抗微生物感染,NO通过促进亚硝化应激[13−15]和抑制有氧呼吸链中的血红蛋白来对微生物产生细胞毒性[16−17]。HmpA蛋白是细菌体内hmpA基因产生的NO双加氧酶[18],其在宿主一氧化氮合酶产生ROS和NO时在细菌体内表现出较高活性[19−21],功能主要是通过将宿主产生的杀伤性NO以一定方式降解为对菌体无害的硝酸盐,如在大肠杆菌中催化的主要反应是通过双加氧酶机制转化O2和NO形成硝酸根离子。但有研究显示该蛋白降解NO的效率在低氧或缺氧环境中可能受到限制[22],即在缺氧或低氧条件下,HmpA蛋白的活性会显著降低[23]。但总的来说,在细菌于宿主体内的免疫逃避过程该蛋白发挥至关重要的作用。目前关于甲型副伤寒沙门氏菌体内HmpA蛋白的功能、结构及基本理化性质尚无清晰报道。为了进一步研究副甲菌中HmpA蛋白的基本生物学信息,本研究以甲型副伤寒沙门氏菌CMCC 50973菌株为模板,克隆hmpA基因并构建了原核表达载体,诱导表达并纯化了HmpA蛋白[24−25];并通过生物信息方法对该蛋白相关的多个重要理化性质进行分析[26−27]。这为后续探究甲型副伤寒沙门氏菌体内HmpA蛋白的免疫学特性与降解宿主细胞产生的杀伤性NO机理方面的影响提供理论参考。
1. 材料与方法
1.1 材料与仪器
甲型副伤寒沙门氏菌CMCC 50973、pNdeI质粒(本实验室从pET-28a(+)质粒改造而来)、大肠杆菌BL21(DE3) 本实验室保存;pMD19T Vector、T4 DNA连接酶、DNA聚合酶、dNTPs、核酸分子质量Marker 宝生物工程有限公司;质粒小量提取试剂盒、微量琼脂糖DNA回收试剂盒 天根生化科技(北京)有限公司;PAGE凝胶快速制备试剂盒(10%)、双色预染蛋白Marker 上海雅酶生物医药科技有限公司;ULtraSignal超敏ECL化学发光底物、限制性内切酶NdeⅠ及XhoⅠ 赛默飞世尔科技公司;小鼠抗His标签单克隆抗体 德悦(北京)生物科技有限责任公司;HRP-羊抗鼠IgG、异丙基硫代半乳糖苷(IPTG) 生工生物工程(上海)股份有限公司;PAGE蛋白上样缓冲液、BeyoBlue™考马斯亮蓝超快染色液 上海碧云天生物科技有限公司;酵母提取物、胰化蛋白胨 英国Oxoid公司;氨苄青霉素、卡那霉素 BBI公司;琼脂糖 基因生物技术国际贸易(上海)有限公司;Tris/甘氨酸/SDS电泳缓冲液(10×)、10×电泳转移缓冲液(转膜液)(粉剂)、10×TBST缓冲液(粉剂)、10×Tris-甘氨酸电泳缓冲液(粉剂)、1×PBS缓冲液 北京索莱宝科技有限公司;PCR扩增引物 昆明擎科生物科技有限公司;0.22 μm无菌滤膜、0.45 μm无菌滤膜 Minipore公司;Histrap 1 mL预装柱 美国GE公司。
Nano-200核酸蛋白测定仪 杭州奥盛仪器有限公司;GZX-DH电热恒温干燥箱 上海精宏实验设备有限公司; Bio-base BSC-1500 II A2-X生物安全柜 济南鑫贝西生物安全柜;GI-1发光成像系统 上海天能有限公司;Neo 13R高速冷冻离心机 上海力申科学仪器有限公司;D3024台式高速微量离心机、D1008掌上离心机 美国赛洛捷克科技有限公司;GI-1紫外凝胶成像分析系统 通宝达成有限公司;HQ45Z恒温摇床仪 武汉中科科仪技术发展有限责任公司;EPS 200电泳仪双板垂直蛋白电泳系统 上海天能科技有限公司;JY92-IIN超声波细胞破碎仪 宁波新芝生物科技股份有限公司。
1.2 实验方法
1.2.1 目的基因的克隆
以CMCC 50973基因组为模板设计引物hmpA-F、hmpA-R(表1),为了将片段定向克隆至pNdeI载体上的NdeI和XhoI之间并与XhoI位点后的6 His标签融合,分别在上下游引物的5’端引入NdeI或XhoI酶切位点,PCR扩增hmpA基因片段,PCR程序为:94 ℃(5 min);94 ℃(30 s),57 ℃(30 s),72 ℃(70 s)共30个循环;72 ℃(5 min)。PCR产物进行1%琼脂糖凝胶电泳并扫描。
表 1 PCR引物Table 1. PCR primers引物名称 引物序列(5’→3’) 酶切位点 hmpA-F catatgcttgacgcacaaaccctc NdeI hmpA-R ctcgagcagcaccttatgcgggcc XhoI 注:下划线为引入的酶切位点。 1.2.2 亚克隆pMD19T-hmpA的构建及鉴定
将1.2.1获得的PCR产物进行1%琼脂糖凝胶电泳,将hmpA基因片段利用微量琼脂糖DNA回收试剂盒进行回收,后与pMD19T Vector载体16 ℃连接18 h。−80 ℃取出DH5α感受态细胞在冰水浴中完全融化后加入连接产物,冰水浴静置5 min;42 ℃热激90 s;迅速冰水浴静置2 min;而后加入液体LB 500 μL,37 ℃,200 r/min,培养30 min;将培养液10000 r/min离心1 min,吸弃培养基剩余约60 μL重悬菌体后,均匀涂于氨苄抗性平板上37 ℃过夜培养18 h。挑取单克隆菌落至带有氨苄抗性(30 μg/mL)的3 mL LB培养基中37 ℃,200 r/min培养18 h后利用质粒小量提取试剂盒提取质粒,进行37 ℃,30 min的NdeⅠ、XhoⅠ酶切鉴定,用1%琼脂糖凝胶进行电泳,并观察酶切结果。鉴定为正确的质粒,送昆明擎科生物科技有限公司进行测序,测序正确的载体命名为pMD19T-hmpA。
1.2.3 表达载体pNdeI-hmpA的构建及鉴定
将DH5α转化提取的pNdeI质粒及测序正确的pMD19T-hmpA质粒分别进行37 ℃,40 min的NdeⅠ、XhoⅠ双酶切,微量琼脂糖DNA回收试剂盒回收双酶切产物,将回收产物用T4 DNA连接酶16 ℃连接18 h后转化至DH5α感受态细胞,转化步骤同1.2.2。挑取转化后的单菌落至带有卡那抗性(30 μg/mL)的3 mL LB培养基中37 ℃,200 r/min培养18 h,提取质粒,进行37 ℃,30 min的NdeⅠ、XhoⅠ双酶切鉴定,用1%琼脂糖凝胶进行电泳,并观察酶切结果。构建正确的载体命名为pNdeI-hmpA。将此质粒转化入大肠杆菌BL21(DE3)感受态细胞获得重组表达菌pNdeI-hmpA/BL21(DE3)同1.2.2。
1.2.4 HmpA蛋白的诱导表达与纯化
将pNdeI-hmpA/BL21(DE3)原核表达菌株及空载体菌划线获得单克隆,挑取单克隆接种于5 mL含有50 μg/mL卡那霉素的 LB培养液中,37 ℃摇床培养过夜。第2 d将过夜培养的两种表达菌株按1%(100 μL)的比例转种于10 mL(50 μg/mL)K+ LB培养液中;37 ℃摇床培养待OD600≈0.7左右时,加入IPTG至终浓度为1 mmol/L,16 ℃诱导过夜。取1 mL经IPTG诱导菌,12000 r/min离心60 s弃上清,加入600 μL PBS重悬菌体再次以12000 r/min的速度离心60 s弃上清。按1:1体积比加PBS,冰水浴条件超声破碎(312 W,工作5 s,间隔10 s,共工作60 min)。破碎完成后,低温高速离心机4 ℃、2000 r/min,离心10 min;转移上清至新的EP管后,4 ℃、10000 r/min离心15 min,沉淀则用100 μL PBS重悬。分别取上清与沉淀重悬液与上样缓冲液混合,煮样15 min后进行SDS-PAGE电泳。
在纯化目的蛋白前先用5倍柱体积的无菌水冲洗Ni柱,然后用平衡缓冲液5 mL平衡柱子(上清平衡缓冲液:20 mmol/L PB、150 mmol/L NaCl、pH=7.40,沉淀平衡缓冲液:20 mmol/L PB、150 mmol/L NaCl、8 mol/L尿素、pH7.40),并用0.45 μm无菌滤膜过滤上清与沉淀重悬液(两者分别单独纯化)。平衡后将过滤后的上清与沉淀重悬液分批缓慢注入 Ni柱中,随后用含有咪唑的缓冲液洗柱子,上清洗柱子与洗脱缓冲液为20 mmol/L PB、150 mmol/L NaCl、50 mmol/L及1 mol/L咪唑、pH7.40,沉淀重悬液的洗柱子与洗脱缓冲液为20 mmol/L PB、150 mmol/L NaCl、50 mmol/L及1 mol/L咪唑、8 mol/L尿素、pH7.40,收集纯化洗脱的样品进行SDS-PAGE电泳。
1.2.5 纯化后蛋白的Western blot分析
重组蛋白经SDS-PAGE[28]后电转移至聚偏二氟乙烯膜(polyvinylidene fluoride,PVDF)上,用10%脱脂奶粉轻摇室温封闭2 h,TBST漂洗4次,每次10 min,加入按1:2000比例用TPBS稀释好的小鼠抗His标签单克隆抗体,于4 ℃条件下轻摇过夜后,TBST漂洗PVDF膜后并浸洗三次,每次漂洗10 min,再加入按1:10000比例用TPBS稀释好的HRP标记的羊抗鼠IgG室温轻摇1 h后,用TBST漂洗PVDF膜后并浸洗三次,每次漂洗5 min,取ULtraSignal超敏ECL化学发光底物(2种试剂)各0.5 mL至无菌EP管中(按1:1)混匀,滴加适量稀释后的发光底物于膜上,成像仪拍照保存。
1.2.6 HmpA蛋白的生物信息学功能分析
用ExPASy的ProtParam软件分析HmpA蛋白的基本理化性质;Protscale在线分析HmpA蛋白的亲水性,TMHMM Server v.2.0软件对HmpA蛋白的跨膜结构进行了氨基酸序列分析;利用SignaIP 5.0 Server软件对HmpA蛋白的信号肽预测分析;通过NPS@SOPMA在线分析软件对目的蛋白的二级结构进行氨基酸序列分析;采用ExPASy的SWISS-MODEL蛋白质库,建立了HmpA蛋白的三维结构同源模型;采用NCBI中的CDD(Conserved Domain Database)数据库通过目的蛋白的氨基酸序列分析其结构域;在NetPhos 3.1 Sever在线工具中利用目的蛋白的氨基酸序列来分析其磷酸化位点;采用ATRING 1.0蛋白互作数据库分析与HmpA相关蛋白。
2. 结果与分析
2.1 亚克隆载体pMD19T-hmpA的重组构建及鉴定
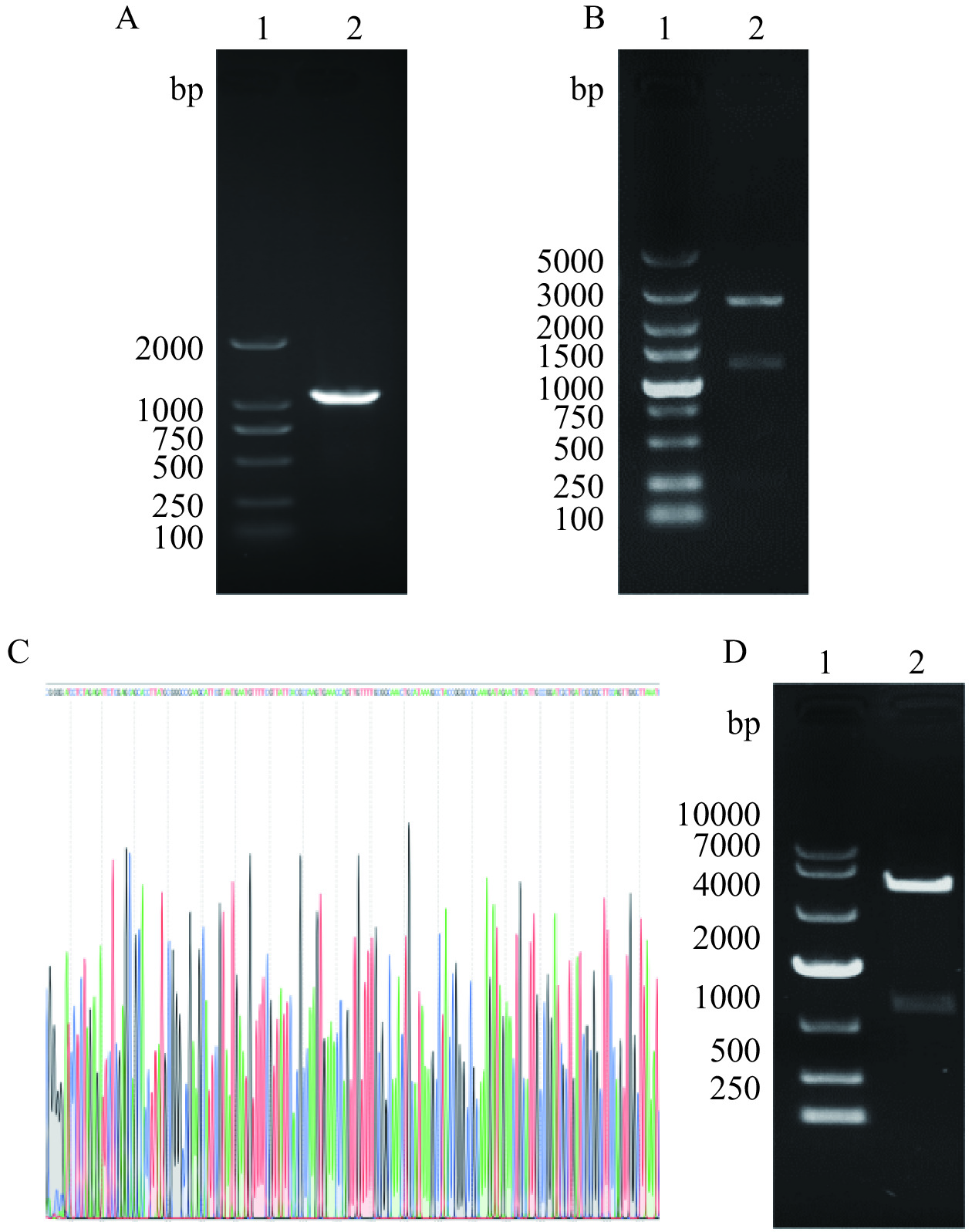
以副甲菌CMCC 50973基因组为模板,PCR扩增产物经1%琼脂糖凝胶电泳可见约1197 bp的hmpA基因,见图1A。亚克隆载体pMD19T-hmpA经NdeI和XhoI限制性内切酶双酶切后,行1%琼脂糖凝胶电泳,可见切出的目的片段(图1B)。测序结果部分峰图见图1C,碱基序列与预期相符。表达载体pNdeI-hmpA经NdeⅠ和XhoⅠ的双酶切鉴定后,电泳结果如图1D所示,可切出目的片段,表明该载体构建成功。
![]() 图 1
图 1pMD19T-hmpA载体及pNdeI-hmpA表达载体构建及鉴定 注:A:hmpA基因PCR扩增结果(l:DNA Marker DL2000;2:hmpA基因的扩增片段);B:pMD19T-hmpA重组质粒的双酶切鉴定(l:DNA Marker DL5000;2:pMD19T-hmpA质粒经NdeI和XhoI双酶切);C:pMD19T-hmpA部分测序峰图;D:pNdeI-hmpA表达载体的双酶切鉴定(l:DNA Marker DL10000;2:pNdeI-hmpA质粒经NdeI和XhoI双酶切)。Figure 1. Construction and identification of pMD19T-hmpA vector and pNdeI-hmpA expression vector2.2 HmpA蛋白的诱导表达
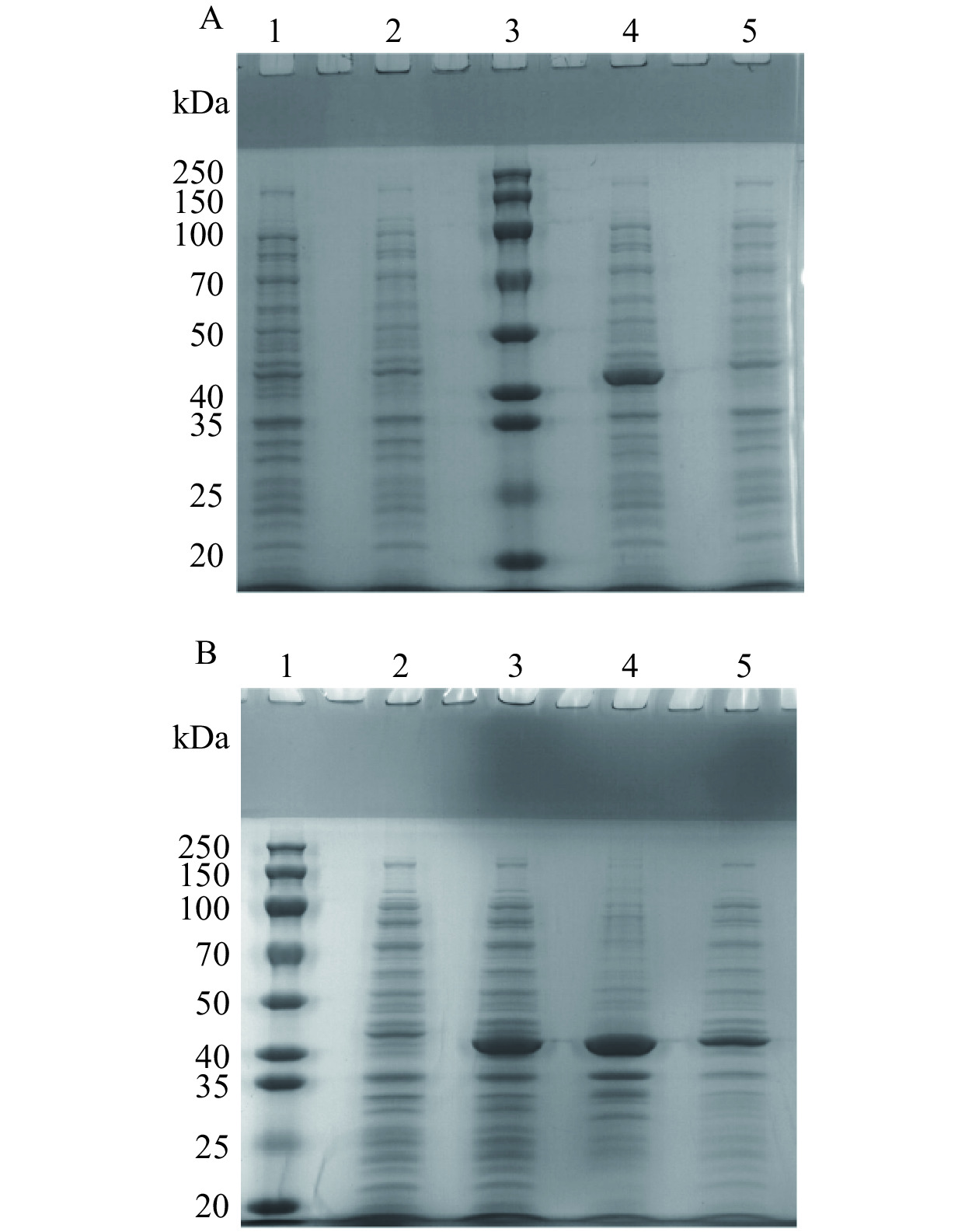
将重组pNdeI-hmpA质粒转入大肠杆菌BL21(DE3)后IPTG诱导表达,蛋白电泳条带结果如图2A所示。诱导前泳道和诱导后泳道的对比发现,诱导后在40~50 kDa间可见明显蛋白条带,说明目的蛋白诱导成功。在16 ℃条件下诱导过夜后,诱导后的pNdeI-hmpA/BL21(DE3)菌株超声破碎后,分别将离心上清及沉淀进行电泳,结果如图2B所示,表明表达载体可同时表达可溶性与包涵体两种形式的目的蛋白,泳道4是全菌超声破碎后的离心沉淀,泳道5是超声破碎后的离心上清,两者比较发现沉淀中的目的蛋白条带比上清中的目的蛋白条带粗,以此推测低温诱导表达时,目的蛋白仍然主要以包涵体形式存在。
![]() 图 2 重组菌的IPTG诱导表达及HmpA蛋白表达形式鉴定注:A:重组菌的IPTG诱导表达(l:pNdeI/BL21(DE3)菌株低温诱导前;2:pNdeI/BL21(DE3)菌株低温诱导后;3:蛋白质分子质量标准;4:pNdeI-hmpA/BL21(DE3)菌株低温诱导后;5:pNdeI-hmpA/BL21(DE3)菌株低温诱导前);B:HmpA蛋白表达形式鉴定(l:蛋白质分子质量标准;2:pNdeI-hmpA/BL21(DE3)菌株未诱导;3:pNdeI-hmpA/BL21(DE3)菌株低温诱导未破碎离心;4:pNdeI-hmpA/BL21(DE3)菌株低温诱导破碎离心沉淀(包涵体形式);5:pNdeI-hmpA/BL21(DE3)菌株低温诱导破碎离心上清(可溶性形式))。Figure 2. Protein expression of recombinated bacteria after IPTG induced and indentification of expression patternof protein HmpA
图 2 重组菌的IPTG诱导表达及HmpA蛋白表达形式鉴定注:A:重组菌的IPTG诱导表达(l:pNdeI/BL21(DE3)菌株低温诱导前;2:pNdeI/BL21(DE3)菌株低温诱导后;3:蛋白质分子质量标准;4:pNdeI-hmpA/BL21(DE3)菌株低温诱导后;5:pNdeI-hmpA/BL21(DE3)菌株低温诱导前);B:HmpA蛋白表达形式鉴定(l:蛋白质分子质量标准;2:pNdeI-hmpA/BL21(DE3)菌株未诱导;3:pNdeI-hmpA/BL21(DE3)菌株低温诱导未破碎离心;4:pNdeI-hmpA/BL21(DE3)菌株低温诱导破碎离心沉淀(包涵体形式);5:pNdeI-hmpA/BL21(DE3)菌株低温诱导破碎离心上清(可溶性形式))。Figure 2. Protein expression of recombinated bacteria after IPTG induced and indentification of expression patternof protein HmpA2.3 HmpA蛋白的纯化与Western blotting鉴定
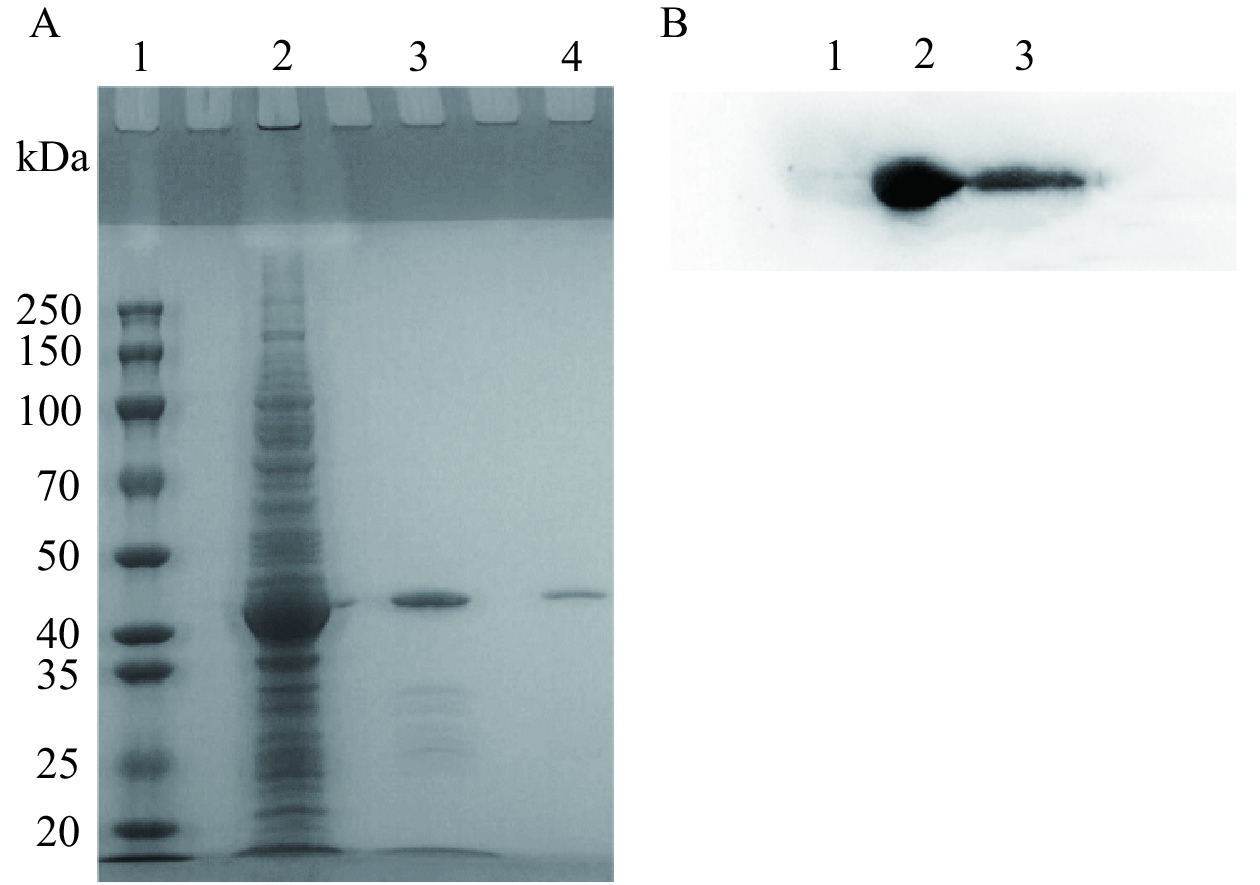
纯化后的蛋白进行SDS-PAGE检测,结果如图3A所示,纯化出的蛋白大小约为44 kDa。经Gel-Pro Analzer软件分析,可溶性表达形式纯化后的目的蛋白纯度约97.3%,包涵体表达形式纯化后的目的蛋白纯度约71.7%。为了识别HmpA重组蛋白条带,克隆设计时在hmpA基因下游添加了6个组氨酸密码子,因此表达的HmpA蛋白C端带有6个组氨酸标签(Histidine-tag,His-tag),如图3B所示,纯化后的目的蛋白均可被抗His抗体检测到。
![]() 图 3 SDS-PAGE检测纯化后的HmpA蛋白及Western blot鉴定注:A:SDS-PAGE检测纯化后的HmpA蛋白(1:蛋白质分子质量标准;2:pNdeI-hmpA/BL21(DE3)菌株低温诱导未纯化;3:pNdeI-hmpA/BL21(DE3)菌株低温诱导破碎离心沉淀(包涵体形式)纯化后;4:pNdeI-hmpA/BL21(DE3)菌株低温诱导破碎离心上清(可溶性形式)纯化后);B:Western Blot鉴定(l:阴性对照;2:包涵体形式纯化后;3:可溶性形式纯化后)。Figure 3. SDS-PAGE detection of purified HmpA protein and identification with Western blot
图 3 SDS-PAGE检测纯化后的HmpA蛋白及Western blot鉴定注:A:SDS-PAGE检测纯化后的HmpA蛋白(1:蛋白质分子质量标准;2:pNdeI-hmpA/BL21(DE3)菌株低温诱导未纯化;3:pNdeI-hmpA/BL21(DE3)菌株低温诱导破碎离心沉淀(包涵体形式)纯化后;4:pNdeI-hmpA/BL21(DE3)菌株低温诱导破碎离心上清(可溶性形式)纯化后);B:Western Blot鉴定(l:阴性对照;2:包涵体形式纯化后;3:可溶性形式纯化后)。Figure 3. SDS-PAGE detection of purified HmpA protein and identification with Western blot2.4 生物信息学功能分析结果
通过ExPASy的ProtParam软件对HmpA蛋白进行了分子式预测,其分子式为C1976H3018N540O580S12,相对分子质量为44003.69,理论等电点(pI)为5.56,原子总量为6126;在哺乳动物细胞中的半衰期约为30 h,在酵母中>20 h,大肠埃希菌中>10 h。不稳定因子约为28.32,说明其稳定性较高(不稳定因子在40以下提示蛋白质结构稳定性较好而大于40提示蛋白质可能不稳定)[29];总亲水指数为−0.268,推测是亲水性蛋白;脂肪族指数为80.38(热稳定性较高)。结果还显示该蛋白含有396个氨基酸,其中有4.3%的Ile(I);8.3%的Leu(L);1.5%的Trp(W),以及带正电荷的氨基酸残基(Arg+Lys,含碱性R基的氨基酸)的数量为33个,带负电荷的氨基酸残基(Asp+Glu,含酸性R基的氨基酸)的数量为48个。详细情况见表2。
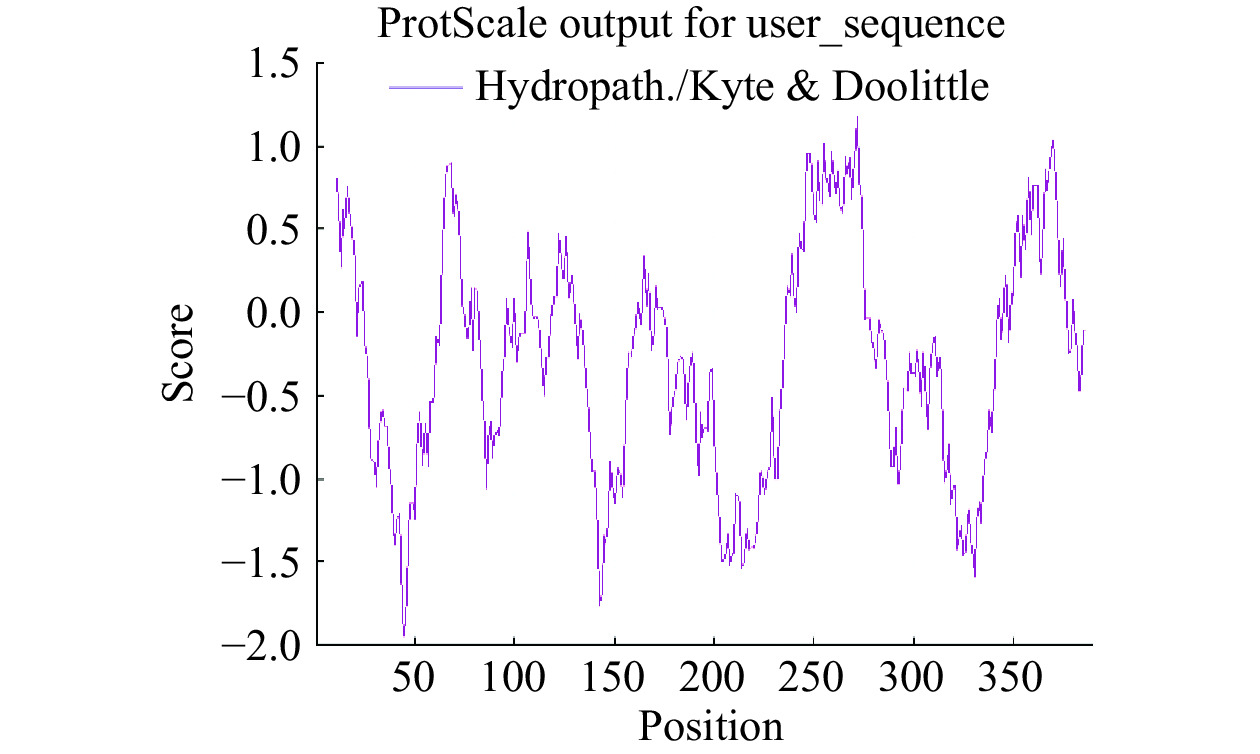
表 2 HmpA蛋白氨基酸占比Table 2. Percentage of protein amino acid of HmpA氨基酸 占比(%) 氨基酸 占比(%) Ala(A) 11.4 Ile(I) 4.3 Arg(R) 4.3 Leu(L) 8.3 Asn(N) 4.5 Lys(K) 4.0 Asp(D) 4.8 Met(M) 2.5 Cys(C) 0.5 Phe(F) 5.3 Gln(Q) 4.8 Pro(P) 5.3 Glu(E) 7.3 Ser(S) 4.0 Gly(G) 7.3 Thr(T) 5.6 His(H) 4.3 Trp(W) 1.5 Tyr(Y) 3.0 Val(V) 6.8 Pyl(O) 0 Sec(U) 0 利用Protscale分析HmpA蛋白亲水性,选用Kyte & Doolittle模式计算方法,将亲水性与疏水性峰值分析图谱间隔设置为21,所得结果见图4。图中纵坐标分值大于0表示疏水性强弱,纵坐标分值小于0表示亲水性强弱。结果显示目标蛋白的第45位氨基酸亲水性最强(分值为−1.948),第272位氨基酸疏水性最强(分值为1.186),同时肽链的亲水区占比多于疏水区占比,提示目标蛋白为亲水性蛋白的可能性较大,这和ProtParam预测的结果相同。
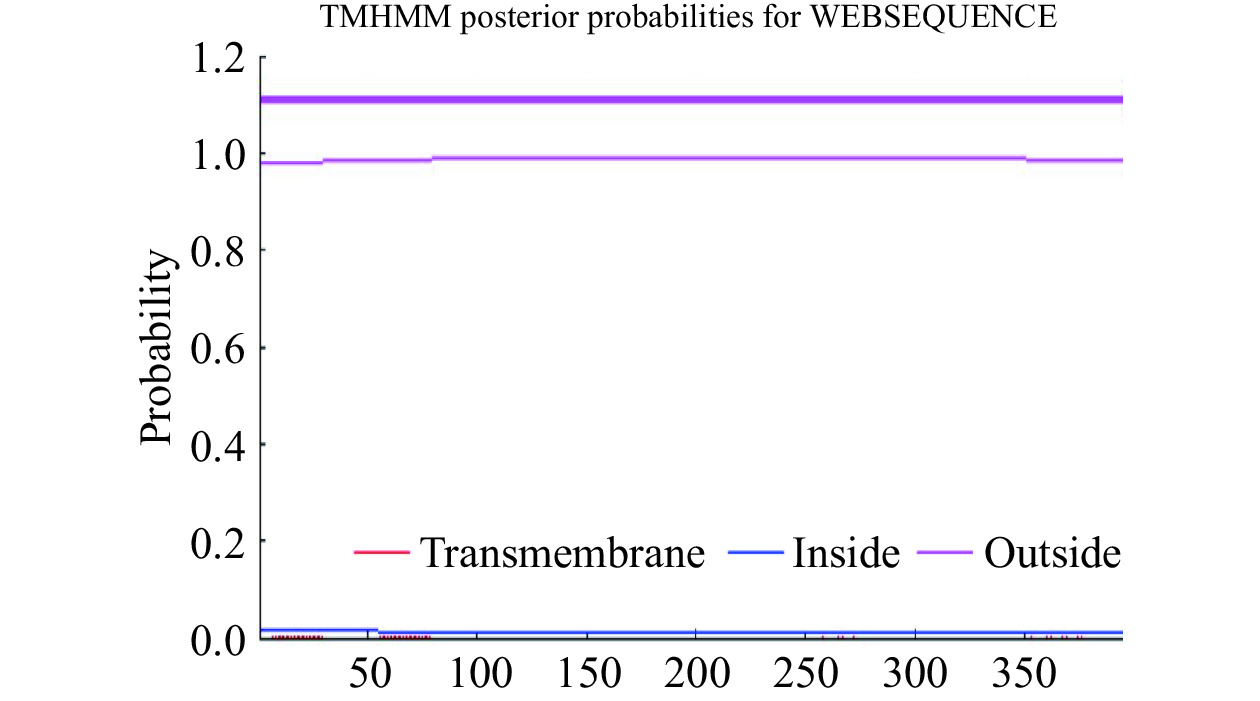
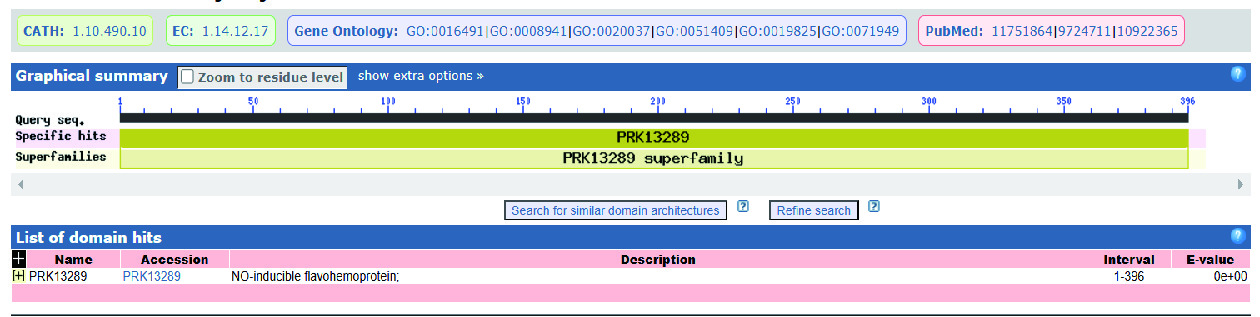
通过TMHMM Server v.2.0软件对HmpA蛋白的跨膜结构进行了氨基酸序列分析,其中跨膜螺旋值趋于0,所以推测该蛋白没有跨膜区的结构(图5)。利用SignaIP 5.0 Server软件对HmpA蛋白是否存在信号肽进行预测分析(信号肽是引导蛋白质进行膜转移的小分子氨基酸,根据信号肽的存在与否,可预测蛋白是否为分泌型),结果显示HmpA蛋白为Sec/SPI信号肽的概率为1.22%,TAT/SPI信号肽的概率为0.16%,Sec/SPII信号肽的概率为0.1%,其他类型的概率为98.52%,综合上述结果,表明目标蛋白是分泌型蛋白的可能性很低(表3)。采用NCBI中的CDD(Conserved Domain Database)数据库通过目的蛋白的氨基酸序列分析其结构域,结果显示HmpA蛋白含一个结构域,属于PRK13289超级家族(图6)。
表 3 HmpA蛋白的信号肽类型预测Table 3. Signaling peptide type prediction for HmpA proteins信号肽类型 由 Sec 转座子转运且由信号肽
酶I切割的分泌信号肽(Sec/SPI)由 Tat 转座子转运并由信号肽
酶I切割的 Tat 信号肽(TAT/SPI)由 Sec 转座子转运并由信号肽
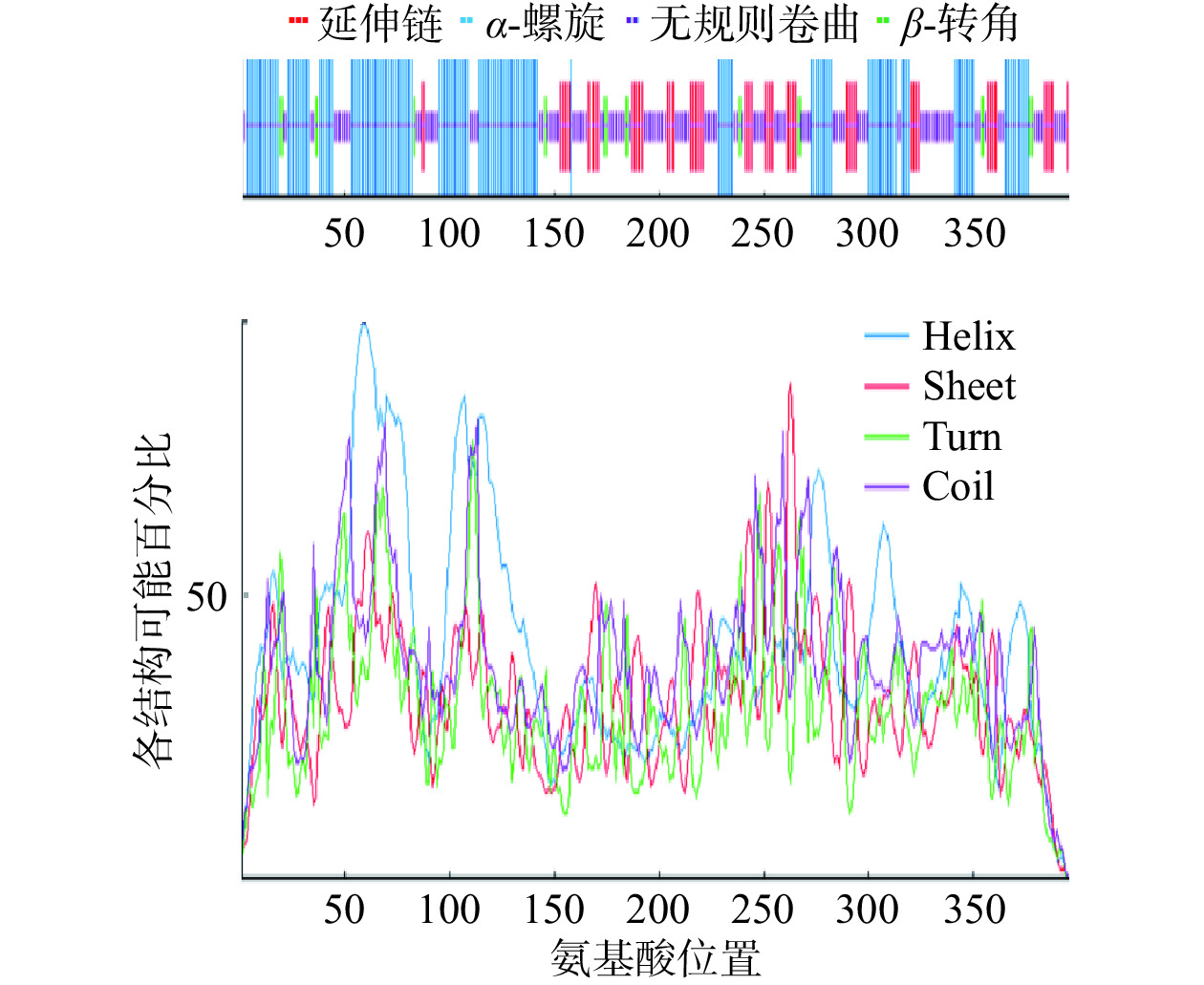
酶II切割的脂蛋白信号肽(Sec/SPII)其他 可能性 0.0122 0.0016 0.001 0.9852 通过在线软件NPS@SOPMA对HmpA蛋白的二级结构进行预测(图7),其中α-螺旋占42.17%,延伸链占16.67%,β-转角占4.8%,无规则卷曲占36.36%。HmpA蛋白的三级结构通过ExPASy的SWISS-MODEL蛋白质库预测,模板序列为1gvh.1.A,序列相似度为91.16%,结果如图8所示。
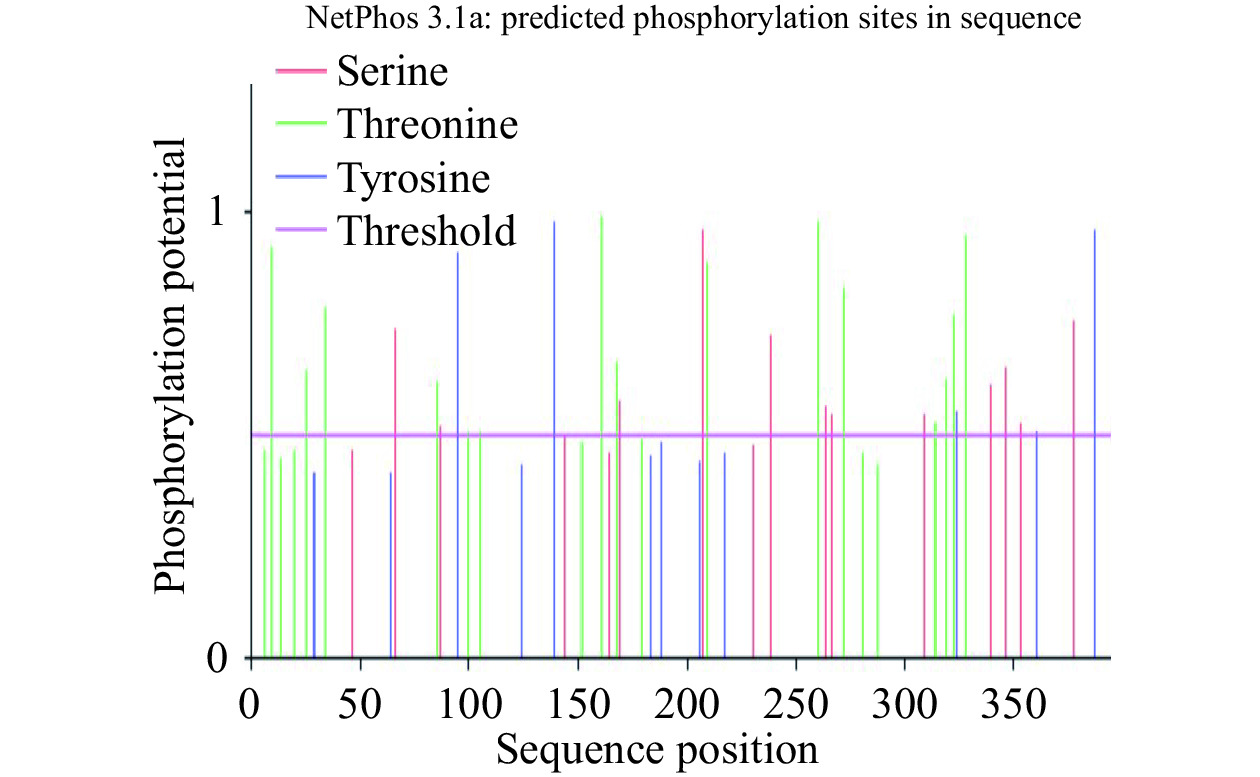
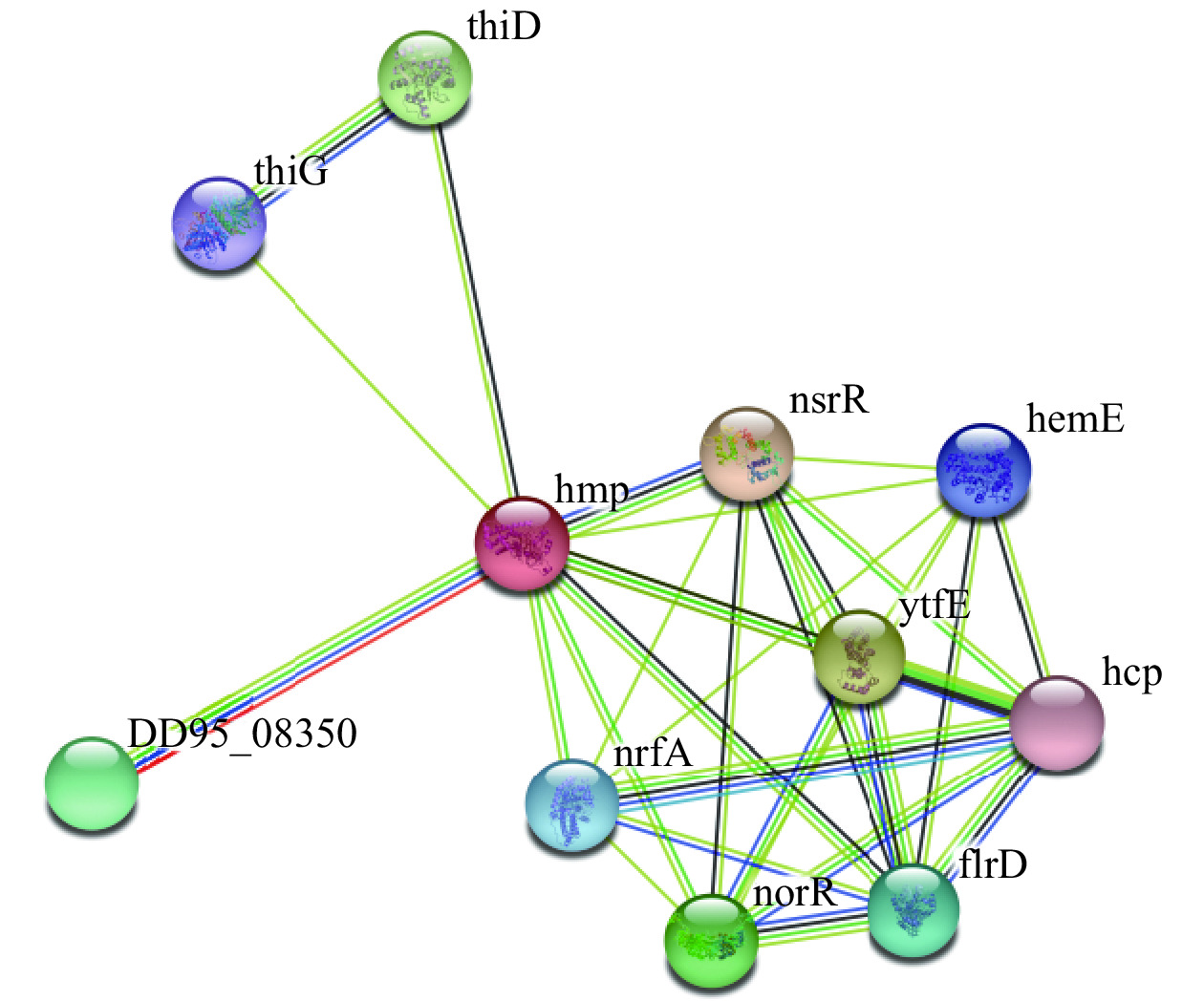
在NetPhos 3.1 Sever在线工具中,利用目的蛋白的氨基酸序列来分析其磷酸化位点。结果显示HmpA蛋白具有多个磷酸化位点,其中磷酸化能力超阈值有12个丝氨酸(Serine),15个苏氨酸(Threonine),5个酪氨酸(Tyrosine),所以HmpA蛋白共有32个磷酸化位点(图9)。采用ATRING11.0蛋白互作数据库预测与HmpA蛋白相关蛋白,图中每个节点代表一个互作蛋白,若两个蛋白质之间存在相互作用,则以连线连接,连线的颜色反映了互作类型,包括试验验证或预测得到的,也包含直接物理作用、共表达、基因融合等关系[30]。结果显示,HmpA在鼠伤寒沙门氏菌中的同源蛋白为hmp(hmp蛋白在鼠伤寒沙门氏菌体内有氧过程中参与NO解毒,从而被称为一氧化氮双加氧酶),与hmp蛋白有较紧密联系的蛋白质有HTH型转录抑制因子NsrR(参与细胞亚硝化应激基因激活的一氧化氮敏感抑制因子)、铁硫簇修复蛋白YtfE(参与修复被氧化和亚硝酸盐应激条件破坏的铁硫簇的含二铁蛋白)、细胞色素C亚硝酸盐还原酶nrfA(催化亚硝酸盐还原为氨),其他还有同源蛋白DD95_08350及在其他文章中一起被提及的thiD、thiG、hemE、hcp等与细菌NO解毒途径有关的转录因子和酶[31−33],据此推测HmpA蛋白的功能与副甲菌降解宿主产生的杀伤性NO机制通路密切相关(图10)。
3. 结论
本研究成功构建了pNdeI-hmpA原核表达载体,转入BL21(DE3)后经IPTG诱导、亲和层析法纯化可得到重组蛋白。生物信息学分析结果显示HmpA蛋白为亲水性蛋白,无信号肽、跨膜区域;属PRK13289超级家族蛋白;二级结构较松散,主要由α螺旋与不规则卷曲构成;含有多个磷酸化位点,与副甲菌体内降解NO的多种酶、多肽及转录因子等关联,初步验证了HmpA蛋白与副甲菌NO解毒途径有关,但关于该重组蛋白的体外NO降解活性、在副甲菌体内产生的机理途径,以及同宿主细胞的抗原抗体反应分子机制在研究中均未涉及。后续研究将对HmpA蛋白的体外活性及其对宿主免疫细胞功能的影响做进一步探究,以分析该蛋白在副甲菌感染过程中所起到的作用,为副伤寒疾病的治疗及副甲菌的防治提供帮助。
生物信息学分析结果显示HmpA蛋白为亲水性蛋白,但实验结果显示诱导的目的蛋白既有包涵体形式又有可溶性形式,这提示我们在典型的大肠杆菌原核表达系统中,蛋白的两亲性跟目的蛋白的表达形式可能不是互相对应的。本实验所用到的载体是基于T7 RNA聚合酶及其强启动子之间的特异性和转录高效性建立,该系列载体诱导目的蛋白表达,同时将大量菌体内资源用于高效率的蛋白表达,且蛋白产量极高。因此在诱导时由于细胞内肽链合成过快却来不及正确折叠进而不能形成天然构象蛋白。同时影响外源基因在大肠杆菌系统中表达的因素还包括目的蛋白的分子质量、培养条件的控制(温度的选择和诱导及培养的时间)、密码子的选用等。后续实验将从表达载体的选取、诱导表达条件的优化等方面改善以提高HmpA蛋白的表达效率。
-
图 1
pMD19T-hmpA载体及pNdeI-hmpA表达载体构建及鉴定 注:A:hmpA基因PCR扩增结果(l:DNA Marker DL2000;2:hmpA基因的扩增片段);B:pMD19T-hmpA重组质粒的双酶切鉴定(l:DNA Marker DL5000;2:pMD19T-hmpA质粒经NdeI和XhoI双酶切);C:pMD19T-hmpA部分测序峰图;D:pNdeI-hmpA表达载体的双酶切鉴定(l:DNA Marker DL10000;2:pNdeI-hmpA质粒经NdeI和XhoI双酶切)。
Figure 1. Construction and identification of pMD19T-hmpA vector and pNdeI-hmpA expression vector
图 2 重组菌的IPTG诱导表达及HmpA蛋白表达形式鉴定
注:A:重组菌的IPTG诱导表达(l:pNdeI/BL21(DE3)菌株低温诱导前;2:pNdeI/BL21(DE3)菌株低温诱导后;3:蛋白质分子质量标准;4:pNdeI-hmpA/BL21(DE3)菌株低温诱导后;5:pNdeI-hmpA/BL21(DE3)菌株低温诱导前);B:HmpA蛋白表达形式鉴定(l:蛋白质分子质量标准;2:pNdeI-hmpA/BL21(DE3)菌株未诱导;3:pNdeI-hmpA/BL21(DE3)菌株低温诱导未破碎离心;4:pNdeI-hmpA/BL21(DE3)菌株低温诱导破碎离心沉淀(包涵体形式);5:pNdeI-hmpA/BL21(DE3)菌株低温诱导破碎离心上清(可溶性形式))。
Figure 2. Protein expression of recombinated bacteria after IPTG induced and indentification of expression patternof protein HmpA
图 3 SDS-PAGE检测纯化后的HmpA蛋白及Western blot鉴定
注:A:SDS-PAGE检测纯化后的HmpA蛋白(1:蛋白质分子质量标准;2:pNdeI-hmpA/BL21(DE3)菌株低温诱导未纯化;3:pNdeI-hmpA/BL21(DE3)菌株低温诱导破碎离心沉淀(包涵体形式)纯化后;4:pNdeI-hmpA/BL21(DE3)菌株低温诱导破碎离心上清(可溶性形式)纯化后);B:Western Blot鉴定(l:阴性对照;2:包涵体形式纯化后;3:可溶性形式纯化后)。
Figure 3. SDS-PAGE detection of purified HmpA protein and identification with Western blot
表 1 PCR引物
Table 1 PCR primers
引物名称 引物序列(5’→3’) 酶切位点 hmpA-F catatgcttgacgcacaaaccctc NdeI hmpA-R ctcgagcagcaccttatgcgggcc XhoI 注:下划线为引入的酶切位点。 表 2 HmpA蛋白氨基酸占比
Table 2 Percentage of protein amino acid of HmpA
氨基酸 占比(%) 氨基酸 占比(%) Ala(A) 11.4 Ile(I) 4.3 Arg(R) 4.3 Leu(L) 8.3 Asn(N) 4.5 Lys(K) 4.0 Asp(D) 4.8 Met(M) 2.5 Cys(C) 0.5 Phe(F) 5.3 Gln(Q) 4.8 Pro(P) 5.3 Glu(E) 7.3 Ser(S) 4.0 Gly(G) 7.3 Thr(T) 5.6 His(H) 4.3 Trp(W) 1.5 Tyr(Y) 3.0 Val(V) 6.8 Pyl(O) 0 Sec(U) 0 表 3 HmpA蛋白的信号肽类型预测
Table 3 Signaling peptide type prediction for HmpA proteins
信号肽类型 由 Sec 转座子转运且由信号肽
酶I切割的分泌信号肽(Sec/SPI)由 Tat 转座子转运并由信号肽
酶I切割的 Tat 信号肽(TAT/SPI)由 Sec 转座子转运并由信号肽
酶II切割的脂蛋白信号肽(Sec/SPII)其他 可能性 0.0122 0.0016 0.001 0.9852 -
[1] MOGASALE V V, RAMANI E, MOGASALE V, et al. Estimating typhoid fever risk associated with lack of access to safe water:A systematic literature review[J]. Journal of Environmental and Public Health,2018,2018:9589208.
[2] FEASEY N A, DOUGAN G, KINGSLEY R A, et al. Invasive non-typhoidal salmonella disease:An emerging and neglected tropical disease in africa[J]. Lancet,2012,379(9835):2489−2499. doi: 10.1016/S0140-6736(11)61752-2
[3] BROCKETT S, WOLFE M K, HAMOT A, et al. Associations among water, sanitation, and hygiene, and food exposures and typhoid fever in case-control studies:A systematic review and meta-analysis[J]. The American Journal of Tropical Medicine and Hygiene,2020,103(3):1020−1031. doi: 10.4269/ajtmh.19-0479
[4] AMANO F. Interaction of Salmonella with macrophages-critical roles of Salmonella sep22, a pathogenicity-related protein, and macrophage reactive-oxygen intermediate species (rois) on the infection and survival of Salmonella[J]. Yakugaku Zasshi:Journal of the Pharmaceutical Society of Japan,2019,139(4):617−627.
[5] PETERSEN E, MILLER S I. The cellular microbiology of Salmonellae interactions with macrophages[J]. Cellular Microbiology,2019,21(11):e13116.
[6] 阳波, 张静, 刘凤凤, 等. 2015-2016年全国和高发省份伤寒、副伤寒流行病学分析[J]. 疾病监测,2018,33(5):407−412. [YANG Bo, ZHANG Jing, LIU Fengfeng, et al. Epidemiological analysis of typhoid and paratyphoid in national and high-prene provinces from 2015-2016[J]. Disease Surveillance,2018,33(5):407−412.] YANG Bo, ZHANG Jing, LIU Fengfeng, et al. Epidemiological analysis of typhoid and paratyphoid in national and high-prene provinces from 2015-2016[J]. Disease Surveillance, 2018, 33(5): 407−412.
[7] 伏晓庆, 古文鹏, 尹建雯, 等. 云南省2003-2012年伤寒副伤寒流行特征分析[J]. 现代预防医学,2014,41(14):2497−2499,2502. [FU Xiaoqing, GU Wenpeng, YIN Jianwen, et al. Analysis of the epidemic characteristics of typhoid and paratyphoid fever in Yunnan Province from 2003-2012[J]. Modern Preventive Medicine,2014,41(14):2497−2499,2502.] FU Xiaoqing, GU Wenpeng, YIN Jianwen, et al. Analysis of the epidemic characteristics of typhoid and paratyphoid fever in Yunnan Province from 2003-2012[J]. Modern Preventive Medicine, 2014, 41(14): 2497−2499,2502.
[8] 孟银平, 姜黎黎, 周永明, 等. 云南省2010-2020年伤寒副伤寒流行特征分析[J]. 现代预防医学,2021,48(15):2689−2691,2715. [MENG Yinping, JIANG Lili, ZHOU Yongming, et al. Analysis of the epidemic characteristics of typhoid and paratyphoid fever in Yunnan Province from 2010-2020[J]. Modern Preventive Medicine,2021,48(15):2689−2691,2715.] MENG Yinping, JIANG Lili, ZHOU Yongming, et al. Analysis of the epidemic characteristics of typhoid and paratyphoid fever in Yunnan Province from 2010-2020[J]. Modern Preventive Medicine, 2021, 48(15): 2689−2691,2715.
[9] GBD 2017 Typhoid and Paratyphoid Collaborators. The global burden of typhoid and paratyphoid fevers:A systematic analysis for the global burden of disease study 2017[J]. The Lancet Infectious Diseases, 2019, 19(4):369-381.
[10] JOHNSON R, MYLONA E, FRANKEL G. Typhoidal Sal monella:Distinctive virulence factors and pathogenesis[J]. Cellular Microbiology,2018,20(9):e12939. doi: 10.1111/cmi.12939
[11] RAFFATELLU M, WILSON R P, WINTER S E, et al. Clinical pathogenesis of typhoid fever[J]. Journal of Infection in Developing Countries,2008,2(4):260−266.
[12] CHONG A, LEE S, YANG Y A, et al. The role of typhoid toxin in Salmonella typhi virulence[J]. The Yale Journal of Biology and Medicine,2017,90(2):283−290.
[13] LANCASTER J R. Nitroxidative, nitrosative, and nitrative stress:Kinetic predictions of reactive nitrogen species chemistry under biological conditions[J]. Chemical Research in Toxicology,2006,19(9):1160−1174. doi: 10.1021/tx060061w
[14] FORRESTER M T, FOSTER M W. Protection from nitrosative stress:A central role for microbial flavohemoglobin[J]. Free Radical Biology & Medicine,2012,52(9):1620−1633.
[15] FITZSIMMONS L F, LIU L, KIM J S, et al. Salmonella reprograms nucleotide metabolism in its adaptation to nitrosative stress[J]. mBio, 2018, 9(1):e00211-18.
[16] 李德东, 王彦, 李莹, 等. 一氧化氮对微生物生长影响作用的两面性[J]. 第二军医大学学报,2010,31(12):1356−1358. [LI Dedong, WANG Yan, LI Ying, et al. Two-sided effects of nitric oxide on microbial growth[J]. Academic Journal of Naval Medical University,2010,31(12):1356−1358.] LI Dedong, WANG Yan, LI Ying, et al. Two-sided effects of nitric oxide on microbial growth[J]. Academic Journal of Naval Medical University, 2010, 31(12): 1356−1358.
[17] CHAN J, TANAKA K, CARROLL D, et al. Effects of nitric oxide synthase inhibitors on murine infection with Mycobacterium tuberculosis[J]. Infection and Immunity,1995,63(2):736−740. doi: 10.1128/iai.63.2.736-740.1995
[18] PARK Y M, LEE H J, JEONG J H, et al. Branched-chain amino acid supplementation promotes aerobic growth of Salmonella Typhimurium under nitrosative stress conditions[J]. Archives of Microbiology,2015,197(10):1117−1127. doi: 10.1007/s00203-015-1151-y
[19] 孙聪, 陈鑫, 上官春雨, 等. 钝齿棒杆菌黄素血红蛋白Hmp在L-精氨酸合成中的作用[J]. 应用与环境生物学报,2022,28(3):561−568. [SUN Cong, CHEN Xin, SHANGGUAN Chunyu, et al. The role of bacillus lutein hemoglobin Hmp in L-arginine synthesis[J]. Chinese Journal of Applied and Environmental Biology,2022,28(3):561−568.] SUN Cong, CHEN Xin, SHANGGUAN Chunyu, et al. The role of bacillus lutein hemoglobin Hmp in L-arginine synthesis[J]. Chinese Journal of Applied and Environmental Biology, 2022, 28(3): 561−568.
[20] STEVANIN T M, READ R C, POOLE R K. The hmp gene encoding the NO-inducible flavohaemoglobin in Escherichia coli confers a protective advantage in resisting killing within macrophages, but not in vitro:Links with swarming motility[J]. Gene,2007,398(1-2):62−68. doi: 10.1016/j.gene.2007.03.021
[21] CHO Y, PARK Y M, BARATE A K, et al. The role of rpoS, hmp, and ssrAB in Salmonella enterica Gallinarum and evaluation of a triple-deletion mutant as a live vaccine candidate in Lohmann layer chickens[J]. Journal of Veterinary Science,2015,16(2):187−194. doi: 10.4142/jvs.2015.16.2.187
[22] POOLE R K. Flavohaemoglobin:The pre-eminent nitric oxide-detoxifying machine of microorganisms[J]. F1000 Research, 2020, 9:7.
[23] MILLS P C, ROWLEY G, SPIRO S, et al. A combination of cytochrome c nitrite reductase (NrfA) and flavorubredoxin (NorV) protects Salmonella enterica serovar Typhimurium against killing by NO in anoxic environments[J]. Microbiology (Reading, England), 2008, 154:1218-1228.
[24] 吴嫚, 李森, 陈国薇, 等. 单增李斯特菌nox基因的克隆、表达以及功能[J]. 食品科学,2017,38(2):46−51. [WU Man, LI Sen, CHEN Guowui, et al. Cloning, expression and function of the Listeria nox gene[J]. Food Science,2017,38(2):46−51.] WU Man, LI Sen, CHEN Guowui, et al. Cloning, expression and function of the Listeria nox gene[J]. Food Science, 2017, 38(2): 46−51.
[25] 杨亚军, 何金科, 宋胜男, 等. 奶牛妊娠相关糖蛋白BoPAG7和BoPAG8的表达及其结构与功能预测[J]. 畜牧与兽医,2020,52(8):56−63. [YANG Yajun, HE Jinke, SONG Shengnan, et al. Expression, structure and function prediction of pregnancy-related glycoprotein BoPAG7 and BoPAG8 in dairy cows[J]. Animal Husbandry & Veterinary Medicine,2020,52(8):56−63.] YANG Yajun, HE Jinke, SONG Shengnan, et al. Expression, structure and function prediction of pregnancy-related glycoprotein BoPAG7 and BoPAG8 in dairy cows[J]. Animal Husbandry & Veterinary Medicine, 2020, 52(8): 56−63.
[26] 徐丽, 邱雪梅, 骆海龙, 等. 肺炎克雷伯菌CRP蛋白表达与纯化及生物学特征分析[J]. 食品工业科技,2021,42(4):98−102,127. [XU Li, QIU Xuemei, LUO Hailong, et al. Expression and purification of CRP protein and biological characteristics of Klebsiella pneumoniae[J]. Science and Technology of Food Industry,2021,42(4):98−102,127.] XU Li, QIU Xuemei, LUO Hailong, et al. Expression and purification of CRP protein and biological characteristics of Klebsiella pneumoniae[J]. Science and Technology of Food Industry, 2021, 42(4): 98−102,127.
[27] 毛耀芳, 魏亚琴, 杨宇泽, 等. 腾冲嗜热厌氧杆菌Cmr3的生物信息学分析及原核表达[J]. 基因组学与应用生物学,2021,40(Z2):2643−2648. [MAO Yaofang, WEI Yaqin, YANG Yuze, et al. Bioinformatics analysis and prokaryotic expression of Tengchong thermophilic anaerobic bacillus Cmr3[J]. Genomics and Applied Biology,2021,40(Z2):2643−2648.] MAO Yaofang, WEI Yaqin, YANG Yuze, et al. Bioinformatics analysis and prokaryotic expression of Tengchong thermophilic anaerobic bacillus Cmr3[J]. Genomics and Applied Biology, 2021, 40(Z2): 2643−2648.
[28] 岳婷婷, 井申荣, 曾韦锟, 等. 呼吸道合胞病毒F2蛋白与结核分枝杆菌早期分泌抗原靶6蛋白的融合表达及纯化[J]. 中国生物制品学杂志,2013,26(3):324−327. [YUE Tingting, JING Shenrong, ZENG Weikun, et al. Fusion, expression and purification of early antigen target 6 protein secreted by respiratory syncytial virus F2 protein and mycobacterium tuberculosis[J]. Chinese Journal of Biologicals,2013,26(3):324−327.] YUE Tingting, JING Shenrong, ZENG Weikun, et al. Fusion, expression and purification of early antigen target 6 protein secreted by respiratory syncytial virus F2 protein and mycobacterium tuberculosis[J]. Chinese Journal of Biologicals, 2013, 26(3): 324−327.
[29] 刘威, 付玉荣, 伊正君. 结核分枝杆菌PKnG蛋白结构与功能的生物信息学分析[J]. 中国病原生物学杂志,2018,13(6):567−571. [LIU Wei, FU Yurong, YI Zhengjun. Bioinformatics analysis of the structure and function of mycobacterium tuberculos is PKnG protein[J]. Journal of Pathogen Biology,2018,13(6):567−571.] LIU Wei, FU Yurong, YI Zhengjun. Bioinformatics analysis of the structure and function of mycobacterium tuberculos is PKnG protein[J]. Journal of Pathogen Biology, 2018, 13(6): 567−571.
[30] 王建. 蛋白质相互作用数据库[J]. 中国生物化学与分子生物学报,2017,33(8):760−767. [Wang Jian. Database of protein interactions[J]. Chinese Journal of Biochemistry and Molecular Biology,2017,33(8):760−767.] Wang Jian. Database of protein interactions[J]. Chinese Journal of Biochemistry and Molecular Biology, 2017, 33(8): 760−767.
[31] CORKER H, POOLE R K. Nitric oxide formation by Escherichia coli. Dependence on nitrite reductase, the NO-sensing regulator Fnr, and flavohemoglobin Hmp[J]. The Journal of Biological Chemistry,2003,278(34):31584−31592. doi: 10.1074/jbc.M303282200
[32] TTUCKER N P, D'AUTRÉAUX B, SPIRO S, et al. Mechanism of transcriptional regulation by the Escherichia coli nitric oxide sensor NorR[J]. Biochemical Society Transactions, 2006, 34:191−194.
[33] KARLINSEY J E, BANG I S, BECKER L A, et al. The NsrR regulon in nitrosative stress resistance of Salmonella enterica serovar Typhimurium[J]. Molecular Microbiology,2012,85(6):1179−1193. doi: 10.1111/j.1365-2958.2012.08167.x





 下载:
下载:

 下载:
下载:



