Establishment and Application of Rapid Detection Method for Polymyxin Resistance Gene mcr-1 Based on RPA-LFD Method
-
摘要: 目的:建立一种快速、高效、可视化的细菌多黏菌素耐药基因mcr-1检测方法,为其基层检测的展开提供依据和便利。方法:利用重组酶聚合酶扩增结合胶体金侧向流试纸条技术(Recombinase polymerase amplification combined with a lateral flow dipstick,RPA-LFD),辅以手持式胶体金读数仪;根据mcr-1基因保守序列设计合成一对特异性RPA引物,通过对反应条件和体系的优化,以及特异性试验、灵敏度试验、模拟食样试验和实际样品试验,成功建立了可视化定量检测细菌多黏菌素耐药基因mcr-1的RPA-LFD方法。结果:在引物浓度400 nmol/L,引物比例1:1时,该方法最佳反应条件为Mg2+浓度14.0 mmol/L,反应温度37 ℃,反应时间20 min;灵敏度好,标准曲线方程为y=0.117x+0.051,定量限为101~108 copies/μL,检出限为101 copies/μL,比PCR法低一个数量级且模拟样品检出结果与PCR法一致。利用建立的RPA-LFD法对猪肉样品、鸡肉样品、生猪养殖场环境样品、肉鸡养殖场环境样品、大肠杆菌分离株和弯曲肠杆菌分离株各15份中多黏菌素耐药基因mcr-1携带情况进行分析;RPA-LFD法与常规PCR法阳性样本检出率一致,共检出9份mcr-1基因阳性样品。RPA-LFD定量分析显示,阳性样品中mcr-1基因浓度在4.5×102~8.6×104 copies/µL之间。结论:本研究建立的细菌多黏菌素耐药基因 mcr-1的RPA-LFD检测法特异性强、灵敏性高、操作简单,可广泛应用于基层检验。
-
关键词:
- 多黏菌素耐药基因mcr-1 /
- 重组酶聚合酶扩增技术 /
- 胶体金侧向流试纸条 /
- 快速检测 /
- 可视化 /
- 定量
Abstract: Objective: To develop a rapid, efficient and visual method for the detection of bacterial colistin resistance gene mcr-1, so as to provide the basis and convenience for the development of its detection at the grassroots level. Methods: Using recombinase polymerase amplification combined with a lateral flow dipstick (RPA-LFD) assay, supplemented by a hand-held colloidal gold reader. According to the conserved sequence of the mcr-1 gene, a pair of specific RPA primers were designed and synthesized. Through the optimization of the reaction conditions and system, as well as the specificity test, sensitivity test, simulated food sample test and actual sample test, the RPA-LFD assay for visual and quantitative detection of bacterial colistin resistance gene mcr-1 was successfully established. Results: When the primer concentration was 400 nmol/L and the primer ratio was 1:1, the optimal reaction conditions of this method are Mg2+ concentration 14.0 mmol/L, reaction temperature 37 ℃ and reaction time 20 min. The sensitivity was good, the standard curve equation was y=0.117x+0.051, the quantification limit was 101~108 copies/μL, and the detection limit was 101 copies/μL, which was an order of magnitude lower than the PCR method and the detection result of the simulated sample was consistent with the PCR method. Carrying status of colistin resistance gene mcr-1 in each 15 pork samples, chicken samples, pig farm environmental samples, broiler farm environmental samples, Escherichia coli isolates and Enterobacter campylobacter isolates were analyzed by the established RPA-LFD assay. The detection rate of positive samples by RPA-LFD assay was consistent with that of conventional PCR method, and a total of 9 mcr-1 gene positive samples were detected. RPA-LFD quantitative analysis showed that the concentration of mcr-1 gene in positive samples was between 4.5×102~8.6×104 copies/µL. Conclition: The RPA-LFD detection method of the bacterial colistin resistance gene mcr-1 established in this study had strong specificity, high sensitivity, and simple operation, and could be widely used in grassroots inspections. -
多黏菌素是一种提取于多粘芽孢杆菌变种的多肽类抗生素,临床上用于治疗多重耐药革兰氏阴性菌引起的感染,被称为抵抗多重耐药细菌感染的“最后一道防线”[1-2]。此外,硫酸粘菌素也在全球养殖业中作为细菌性疾病治疗用药而广泛应用[3]。然而,自2015年质粒介导的多黏菌素耐药基因mcr-1首次被报道后[4],其在临床、食品、畜牧养殖、水产及相关环境中屡屡检出[5-6]。Borowiak等[7]分析了德国联邦风险评估研究所从动物、食物、饲料和环境中收集的耐药沙门氏菌,发现mcr-1基因在德国家畜和食品来源的沙门氏菌中广泛流行。此外,mcr-1基因也普遍存在于大肠埃希菌、产气肠杆菌、肺炎克雷伯菌等肠杆菌科细菌中[8-11]。有报道显示,动物源、动物性食品源和人源肠杆菌科细菌中,mcr-1基因检出率在5%~30%[12-13]。与其他国家相比,我国的mcr-1基因检出率较高,在5%~80%之间[14-16]。mcr-1基因的流行率高且传播速度快,严重威胁了公众安全和人类健康,因此亟需建立一种适用于基层的的检测方法对该耐药基因进行快速诊断和及时监测。
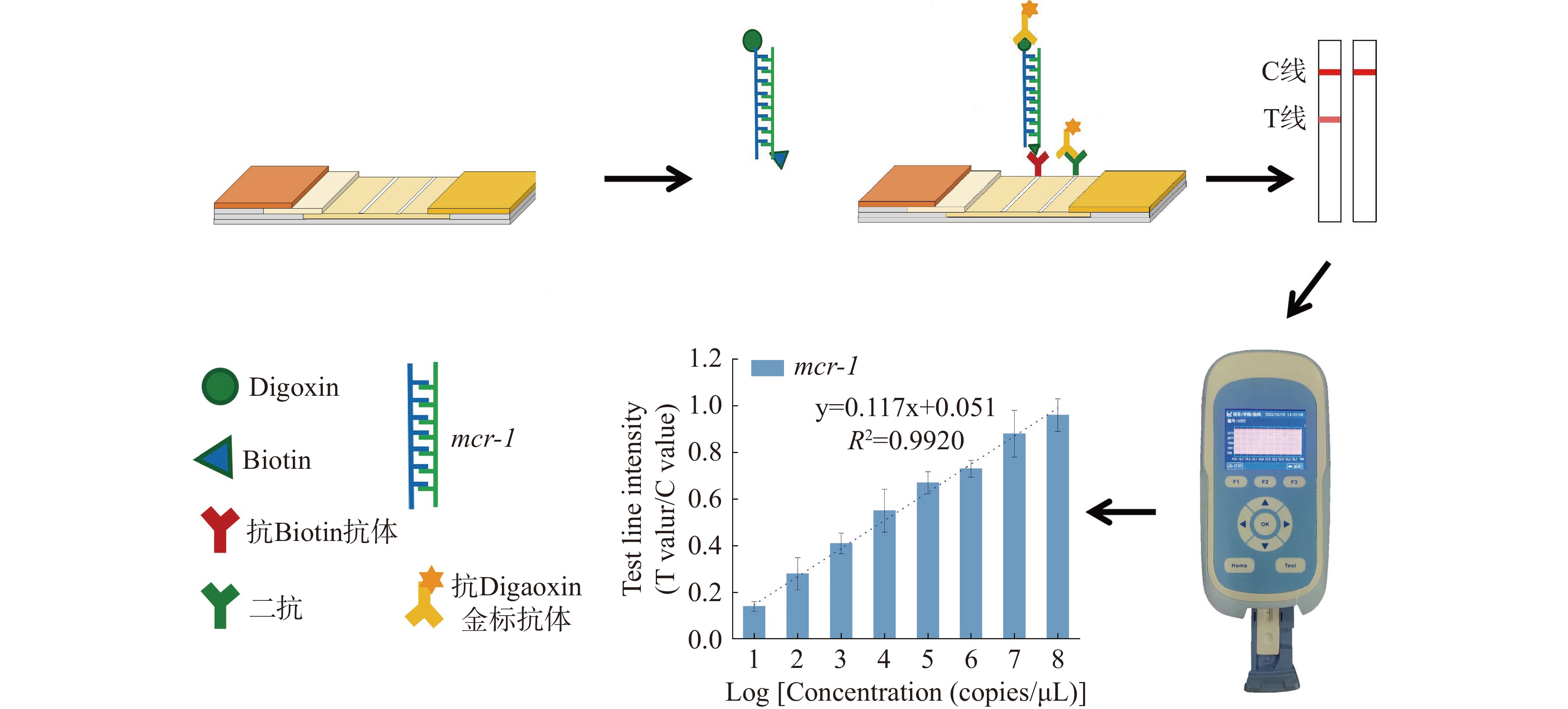
目前,对mcr-1基因的检测,主要是基于传统PCR、荧光定量PCR以及数字PCR等技术的分子检测方法[17-20]。这些检测方法重复性、特异性均较好,但大多数仍存在反应时间长、操作较为繁琐等弊端,且相关实验设备昂贵,对技术人员的熟练度要求高,不适用于基层对目的样品的快速检测。重组酶聚合酶扩增技术(recombinase polymerase amplification,RPA)被称为是可以替代PCR的核酸检测技术,其特点是能够在常温条件下实现核酸扩增[21]。RPA技术使用重组酶促进寡核苷酸引物与其双链DNA分子互补序列结合,该过程由单链DNA结合蛋白辅助,可防止引物解离,之后由DNA聚合酶进行复制[22]。RPA技术具有反应时间快速、操作简便、灵敏度高等优点,一般在20~30 min即可获得检出范围内的扩增产物,非常适用于基层检测[23-26]。通过与荧光基团标记后,获得的扩增产物可与侧流层析试纸条(lateral flow dipstick,LFD)相结合,能够实现可视化检测。RPA技术在临床检验、动物疫病诊断、畜牧养殖产业、微生物及其耐药性检测等领域均有涉及,已成功应用到日本血吸虫、非洲猪瘟病毒、猪嵴病毒、布鲁氏菌病等的检测中,结合LFD后,其定性检测结果可在2~5 min内直接进行判定[27-30]。此外,LFD检测结果可通过手持式胶体金读数仪扫描,读取T/C值,进一步实现目的基因的定量检测。
本研究利用RPA-LFD技术辅以手持式胶体金读数仪,建立了一种快速、高效、可应用于基层的细菌多黏菌素耐药基因mcr-1的检测方法,这对多黏菌素耐药基因mcr-1的快速检测、切断传播途径、保障人们身体健康具有十分重要的意义[31-33]。
1. 材料与方法
1.1 材料与仪器
携带mcr-1基因的重组质粒pGM-T-mcr-1、携带质粒pGM-T-mcr-1的重组大肠杆菌 Escherichia coli Top10-pGM-T-mcr-1 本实验室前期构建;质粒pGM-T、大肠杆菌E. coli Top10 杭州纽贝生物科技有限公司;质控菌株大肠杆菌E. coli ATCC25922(不含耐药基因) 美国典型培养物保藏中心;不携带mcr-1基因的大肠杆菌E. coli、弯曲肠杆菌Enterobacter campylobacter、霍氏肠杆菌E. hormaechei、干酪乳杆菌Lactobacillus casei 实验室前期分离鉴定;15株大肠杆菌分离株E. coli(菌株编号E1~E15)、15株弯曲肠杆菌分离株E. campylobacter(菌株编号EC1~EC15) 来源于浙江省某养殖场的猪粪样品,为实验室前期分离保存;15份生猪养殖场环境样品(样品编号SZ1~SZ15) 来源于浙江省某养殖场猪粪(取生猪新鲜粪便分别密封于无菌均质袋中,置于装有冰袋的泡沫箱中,密封后在2 h内运送回实验室处理);15份肉鸡养殖场环境样品(样品编号RJ1~RJ15) 来源于浙江省某养殖场鸡粪(处理方法同上);15份猪肉样品(样品编号ZR1~ZR15)和15份鸡肉样品(样品编号JR1~JR15) 浙江省某农贸市场(处理方法同上);营养琼脂培养基、LB液体培养基和甘油等生化试剂 青岛海博生物技术有限公司;RPA核酸扩增试剂盒(基础型) 英国TwistDX公司;其它常规生化试剂 美国Sigma公司;PCR试剂、细菌质粒提取试剂盒、细菌基因组DNA提取试剂盒等分子生物学试验用试剂 宝生物工程(大连)有限公司。
SW-CJ-2FD/2F超净工作台 上海BOXUN公司;GT14DP高压蒸汽灭菌锅 美国ZEALWAYF公司;NanoDrop 2000c超微量紫外分光光度计 美国Thermo Scientific公司;EPS-600电泳装置 上海Tanon公司;Arktik PCR仪 美国Thermo Scientific公司;Gel Doc EZ凝胶成像分析系统 美国Bio-Rad公司;TSR-200手持式胶体金读数仪 杭州奥盛仪器有限公司。
1.2 实验方法
1.2.1 携带mcr-1基因的标准质粒pGM-T-mcr-1制备
在−80 ℃冰箱中取出实验室冻存的携带mcr-1基因的重组大肠杆菌E. coli Top10-pGM-T-mcr-1划线于LB琼脂培养基,于37 ℃过夜培养,挑取单菌落于LB液体培养基中,摇床中37 ℃,200 r/min培养至菌液OD600为1.0,参照细菌质粒提取试剂盒操作说明书方法提取携带mcr-1基因标准质粒pGM-T-mcr-1。利用Nanodrop 2000c紫外分光光度计测定标准质粒浓度。采用煮沸法制备待测大肠杆菌E. coli、弯曲肠杆菌E. campylobacter、霍氏肠杆菌E. hormaechei、干酪乳杆菌L. casei等菌液模板DNA:取1 μL待测菌液加入30 μL Tris缓冲液,煮沸5 min,冰上冷却2 min,离心12000 r/min,2 min,取上清液用作DNA模板[34]。
1.2.2 PCR和RPA引物设计
登录NCBI(https://www.ncbi.nlm.nih.gov/)下载mcr-1基因序列,分析目标基因mcr-1序列的多态性并确定其保守区域。针对保守区域序列,利用Primer Premier 5.0软件设计PCR与RPA引物。PCR与RPA引物序列见表1,由上海生工生物工程有限公司合成。
表 1 RPA 扩增和PCR 扩增引物序列Table 1. Sequence of RPA amplification and PCR amplification primers引物名称 引物序列(5’-3’) 引物序列大小(bp) mcr-1-F1 5'-CGGTCAGTCCGTTTGTTC-3’ 309 mcr-1-R1 5'-CTTGGTCGGTCTGTAGGG-3’ mcr-1-rpa-F2 5'-GTCCGTTTGTTCTTGTG
GCGAGTGTTGCCGTT-3’298 mcr-1-rpa-R2 5'-TCGGTCTGTAGGGCATTT
TGGAGCATGGTCGT-3’1.2.3 PCR与RPA反应体系与反应程序
PCR反应体系:总体系25.0 μL,Premix TaqTM 12.5 μL,上下游引物(10 μmol/L)各1 μL,DNA样本2 μL,灭菌去离子水8.5 μL。PCR反应程序:94 ℃预变性1 min;98 ℃变性30 s,55 ℃退火30 s,72 ℃延伸20 s,30次循环;72℃延伸20 s,72 ℃再延伸10 min,4 ℃保存。取6 μL扩增产物经1.2%琼脂糖凝胶电泳后,分析凝胶成像结果。RPA反应体系(50.0 μL):反应干粉1管,Rehydration Buffer 29.5 μL,上下游引物(10 μmol/L)各2 μL,Mg2+ 2.5 μL,DNA样本5.0 μL,灭菌去离子水11.0 μL。37 ℃孵育30 min。RPA扩增产物纯化后进行1.2%琼脂糖凝胶电泳,分析凝胶成像结果。
1.2.4 RPA-LFD可视化检测策略
RPA-LFD可视化检测策略如图1所示。RPA引物荧光集团标记:mcr-1-rpa-F2引物5’端标记Biotin,mcr-1-rpa-R2引物5’端标记Digoxin;LFD试纸条制备:利用金标点膜仪将0.65 mg/mL抗Biotin单克隆抗体、1 mg/mL羊抗鼠多克隆二抗和10 μg/mL的胶体金标记Digoxin单抗溶液分布均匀标记在LFD试纸条NC膜的检测线T线、质控线C线和结合垫上。将标记好的NC膜和结合垫置于烘箱37 ℃干燥12 h。将干燥好的NC膜、结合垫与样品垫、吸收垫和底板组合组装成LFD试纸条;RPA-LFD可视化:将稀释后的RPA扩增产物滴加到LFD试纸条样品垫,标记有Biotin和Digoxin的扩增产物侧向层析后被包被在检测线T的抗Biotin单克隆抗体捕获,被捕获的扩增产物因标记有Digoxin,将胶体金标记Digoxin单抗捕获在T线,多余的胶体金标记Digoxin单抗在控制线C线被包被的多克隆二抗捕获,最终实现T线、C线显色;当扩增产物无目标产物时,则只有C线显色;可用TSR-200手持式胶体金读数仪读取试纸条T/C值,记录数据,分析检测定量结果。
1.2.5 RPA-LFD方法的优化
RPA反应体系见1.2.3。用LFD试纸条缓冲液将RPA扩增产物稀释50倍进行LFD侧流层析。5~10 min内读取结果。为提高目标基因的扩增效率,获得最优的反应条件,对RPA-LFD可视化体系的反应温度、反应时间以及Mg2+反应浓度进行了优化。设置反应温度梯度为:30、35、37、39、45和50 ℃;固定反应温度以及Mg2+反应浓度,设置反应时间梯度为:2.5、5、7.5、10、12.5、15、17.5、20、22.5和30 min;设置Mg2+反应浓度梯度为:2.8、5.6、8.4、11.2、14和16.8 mmol/L。在研究一个变量时,RPA反应的其它条件按照1.2.3里的条件固定。
1.2.6 RPA-LFD方法特异性分析
采用优化后的RPA-LFD体系,以重组质粒pGM-T-mcr-1和重组大肠杆菌E. coli Top10-pGM-T-mcr-1为阳性对照,以质粒pGM-T、大肠杆菌E. coli Top10、耐药敏感菌株大肠杆菌E. coli ATCC25922、以经鉴定不携带mcr-1基因的大肠杆菌 E. coli、弯曲肠杆菌E. campylobacter、霍氏肠杆菌E. hormaechei、干酪乳杆菌L. casei分离株为阴性对照,以灭菌去离子水为空白对照,分析RPA-LFD方法特异性。
1.2.7 RPA-LFD方法灵敏度分析
利用Nanodrop 2000c紫外分光光度计测定提取的pGM-T-mcr-1标准质粒浓度,并计算标准质粒拷贝数。拷贝数计算公式为:拷贝数/μL=质粒浓度(ng/μL)×6.02×1023/(660×质粒长度),其中6.02×1023为1摩尔中含有的摩尔分子(拷贝数);660为计算dsDNA平均分子量的常数。用Tris-HCl缓冲液以10为倍数梯度稀释标准质粒,制备108、107、106、105、104、103、102、101、100和10−1 copies/μL浓度梯度的标准质粒溶液。以10倍浓度梯度稀释的标准质粒溶液为模板,采用优化的最优RPA-LFD反应,分析RPA-LFD方法灵敏度。同时,利用读数仪读取LFD试纸条T/C值,以T/C值为纵坐标轴,以标准质粒拷贝数浓度的lg10对数为横坐标轴,建立mcr-1基因定量标准曲线。
1.2.8 RPA-LFD方法用于模拟食样检测
从当地农贸市场购买猪肉和鸡肉等样品,参考GB 4789.1进行样品前处理,制成样品液。用细菌基因组DNA提取试剂盒法提取样品DNA,采用常规PCR方法确证所有样品中不含mcr-1耐药基因。将标准质粒添加到猪肉和鸡肉样品,使得样品中标准质粒菌株的终浓度为104~100 copies/μL,制备样品液并提取样品DNA,用建立的RPA-LFD方法分析模拟食样中携带mcr-1基因情况。
1.2.9 RPA-LFD方法用于实际样品检测
利用RPA-LFD方法和常规PCR方法,分析15份猪肉样品(样品编号ZR1~ZR15)、15份鸡肉样品(样品编号JR1~JR15)、15份生猪养殖场环境样品(样品编号SZ1~SZ15)、15份肉鸡养殖场环境样品(样品编号RJ1~RJ15)、15株大肠杆菌分离株E. coli(菌株编号E1~E15)和15株弯曲肠杆菌分离株样品E. campylobacter(菌株编号EC1~EC15)中携带mcr-1基因情况,分析RPA-LFD方法与常规PCR方法检测结果一致性。
1.3 数据处理
利用Adobe Photoshop CC 2019将所得电泳、试纸条图片进行标注;利用OriginLab Origin 2021对数据进行处理并制作柱状图。
2. 结果与分析
2.1 RPA-LFD检测方法反应条件优化
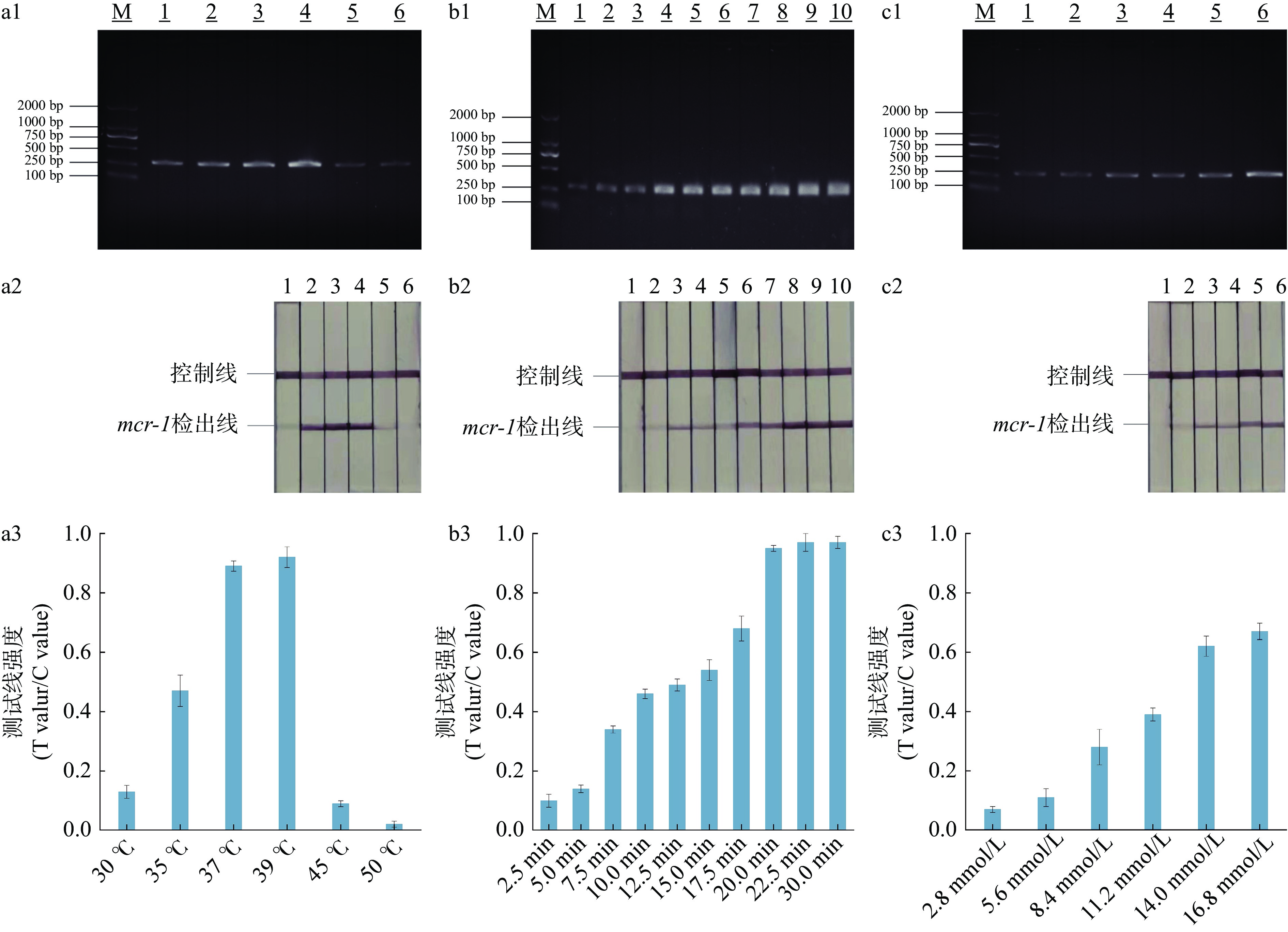
以mcr-1基因标准质粒pGM-T-mcr-1为模板,对RPA-LFD方法的反应温度、反应时间以及Mg2+浓度进行优化。设置30~50 ℃温度梯度,对反应温度进行优化,结果显示,在35、37和39 ℃的反应温度下,mcr-1基因检出效果均较好,电泳条带明亮且试纸条的目标检测线较为清晰(图2a1~图2a3)。设置2.5~30 min反应时间梯度,对反应时间进行优化,结果显示,在电泳条带和试纸条颜色在反应20、22.5和30 min处清晰明亮,mcr-1基因检出效果均较好(图2b1~图2b3)。设置2.8~16.8 mmol/L的Mg2+反应浓度梯度,优化Mg2+浓度,结果显示,Mg2+浓度达到14.0和16.8 mmol/L时mcr-1耐药基因检出效果均较好(图2c1~图2c3)。综上所述,结合高效与节约角度,37 ℃为最优反应温度,14 mmol/L为最优Mg2+浓度,22.5 min为最佳反应时间。
![]() 图 2 RPA-LFD反应条件优化注:a1为反应温度优化电泳结果;a2为反应温度优化LFD试纸条检测结果;a3为反应温度优化手持式设备检测结果;b1为反应时间优化电泳结果;b2为反应时间优化LFD试纸条检测结果;b3为反应时间优化手持式设备检测结果;c1为Mg2+浓度优化电泳结果;c2为Mg2+浓度优化LFD试纸条检测结果;c3为Mg2+浓度优化手持式设备检测结果;M表示DNA Marker (DL2000);a系列图中,1~6分别表示30、35、37、39、45、50 ℃;b系列图中,1~10分别表示2.5、5、7.5、10、12.5、15、17.5、20、22.5、30 min;c系列图中,1~6分别表示2.8、5.6、8.4、11.2、14.0、16.8 mmol/L。Figure 2. Optimization of RPA-LFD reaction conditions
图 2 RPA-LFD反应条件优化注:a1为反应温度优化电泳结果;a2为反应温度优化LFD试纸条检测结果;a3为反应温度优化手持式设备检测结果;b1为反应时间优化电泳结果;b2为反应时间优化LFD试纸条检测结果;b3为反应时间优化手持式设备检测结果;c1为Mg2+浓度优化电泳结果;c2为Mg2+浓度优化LFD试纸条检测结果;c3为Mg2+浓度优化手持式设备检测结果;M表示DNA Marker (DL2000);a系列图中,1~6分别表示30、35、37、39、45、50 ℃;b系列图中,1~10分别表示2.5、5、7.5、10、12.5、15、17.5、20、22.5、30 min;c系列图中,1~6分别表示2.8、5.6、8.4、11.2、14.0、16.8 mmol/L。Figure 2. Optimization of RPA-LFD reaction conditions2.2 特异性试验
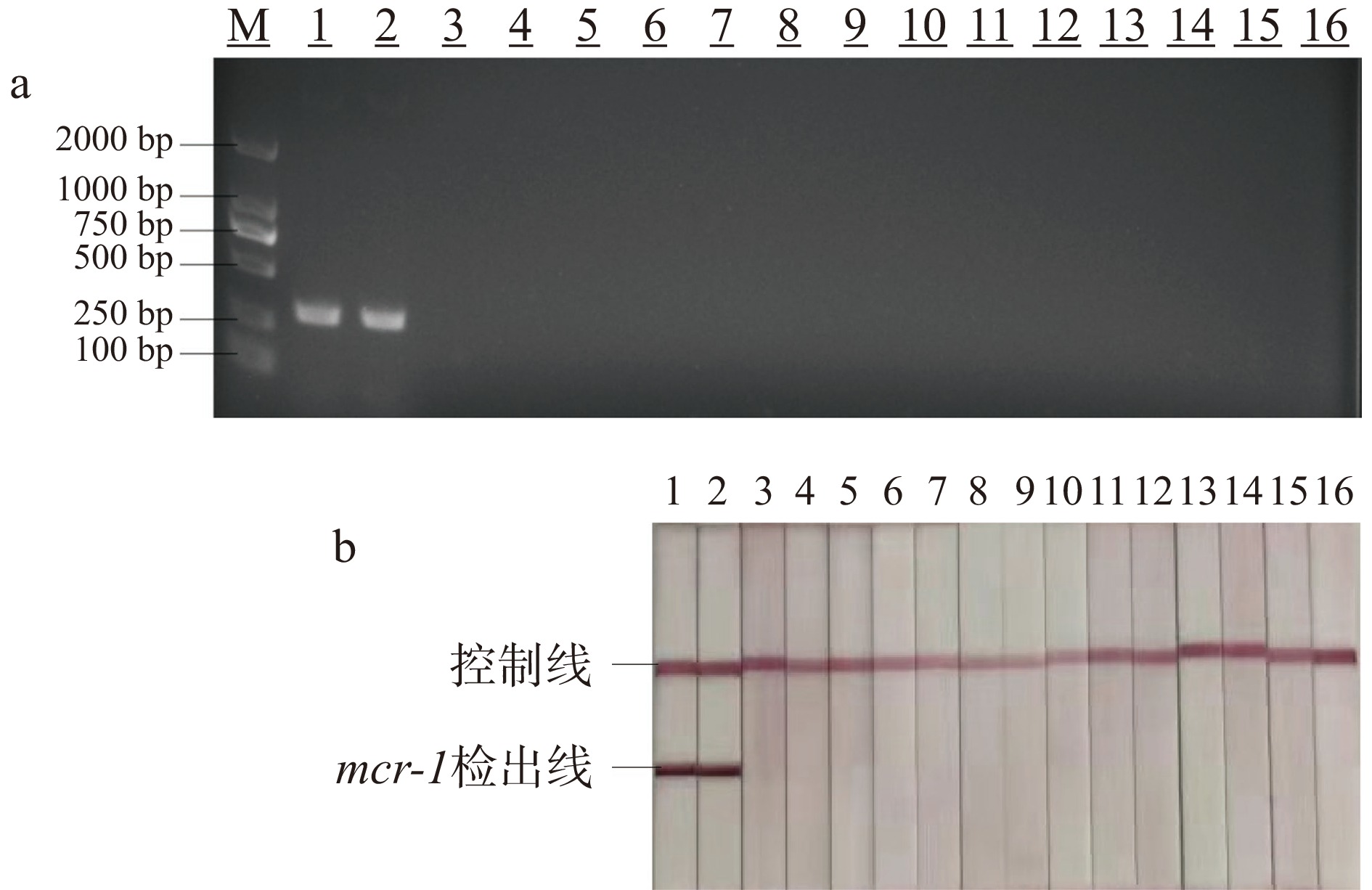
特异性试验按照1.2.6进行,检测结果如图3所示。经RPA-LFD检测,仅重组质粒pGM-T-mcr-1和重组大肠杆菌E. coli Top10-pGM-T-mcr-1为的检测结果显示为阳性。质粒pGM-T、大肠杆菌E. coli Top10、耐药敏感菌株大肠杆菌E. coli ATCC25 922、不携带mcr-1基因的大肠杆菌E. coli、弯曲肠杆菌E. campylobacter、霍氏肠杆菌E. hormaechei、干酪乳杆菌L. casei分离株等不含mcr-1耐药基因样品检测结果均显示为阴性。试验结果表明,本研究建立的RPA-LFD检测mcr-1耐药基因的方法特异性好,与其他不携带mcr-1基因的质粒和分离株无交叉反应。
![]() 图 3 RPA-LFD特异性检测注:a为RPA-LFD特异性电泳结果;b为RPA-LFD特异性LFD试纸条检测结果;M:DNA Marker(DL2000);1:质粒pGM-T-mcr-1;2:E. coli Top10-pGM-T-mcr-1;3:质粒pGM-TT;4:E. coli Top10;5:耐药敏感菌株E. coli ATCC25922;6~8:不携带mcr-1基因的大肠杆菌分离株E. coli;9~10:不携带mcr-1基因的弯曲肠杆菌分离株E. campylobacter;11~12:不携带mcr-1基因的霍氏肠杆菌分离株E. hormaechei;13~14:不携带mcr-1基因的干酪乳杆菌分离株L. casei;15:灭菌去离子水;16:空白对照。Figure 3. RPA-LFD specificity test
图 3 RPA-LFD特异性检测注:a为RPA-LFD特异性电泳结果;b为RPA-LFD特异性LFD试纸条检测结果;M:DNA Marker(DL2000);1:质粒pGM-T-mcr-1;2:E. coli Top10-pGM-T-mcr-1;3:质粒pGM-TT;4:E. coli Top10;5:耐药敏感菌株E. coli ATCC25922;6~8:不携带mcr-1基因的大肠杆菌分离株E. coli;9~10:不携带mcr-1基因的弯曲肠杆菌分离株E. campylobacter;11~12:不携带mcr-1基因的霍氏肠杆菌分离株E. hormaechei;13~14:不携带mcr-1基因的干酪乳杆菌分离株L. casei;15:灭菌去离子水;16:空白对照。Figure 3. RPA-LFD specificity test2.3 灵敏度试验
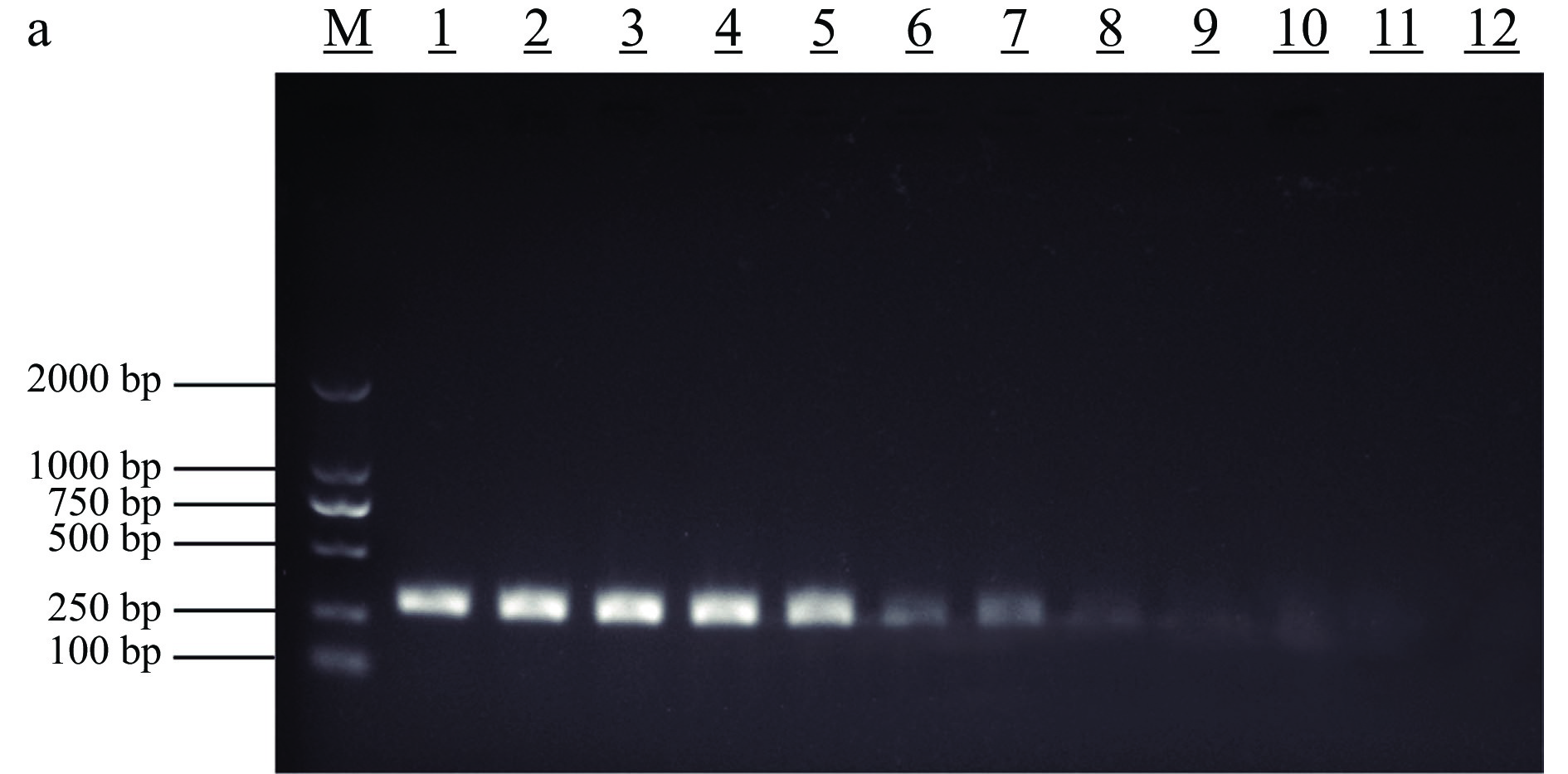
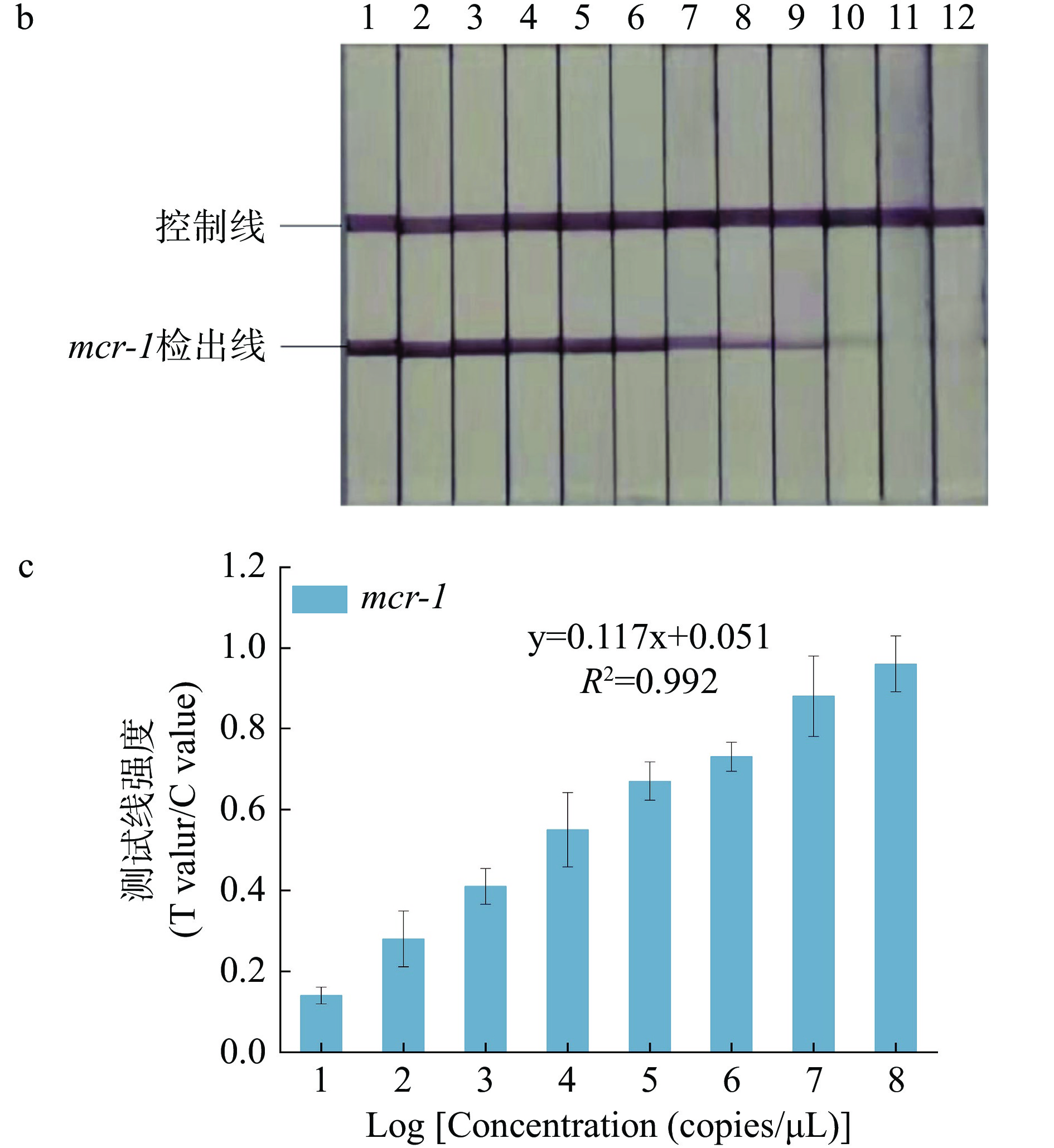
灵敏度试验按照1.2.7进行,检测结果如图4所示。以108~10−1 copies/μL浓度梯度的 mcr-1基因标准质粒为RPA扩增模板,进行RPA-LFD灵敏度试验。结果表明 RPA-LFD方法检测mcr-1耐药基因的检测灵敏度为 101 copies/μL,具有较好的检测灵敏度。根据LFD试纸条T/C值和对应mcr-1基因标准质粒拷贝数浓度的对数,建立mcr-1基因RPA-LFD定量标准曲线,结果显示,T/C值和对应mcr-1基因标准质粒拷贝数浓度的对数存在显著的正相关性,标准曲线方程为y=0.117x+0.051,R2=0.9920。
2.4 模拟食样检测
模拟食样检测按照1.2.8进行,检测结果如表2所示。RPA-LFD方法对添加mcr-1基因标准质粒的模拟食样的检出结果传统PCR方法检测结果一致,且RPA-LFD方法检测模拟样品中的携带mcr-1耐药基因的菌液的检出限可达101 copies/μL,比PCR方法的检出限102 copies/μL低1个数量级。
表 2 模拟食样检测结果Table 2. Test results of simulated food samples样品 添加浓度(copies/μL) RPA-LFD检测 PCR检测 猪肉 1×104 9.45×103 是 1×103 1.10×103 是 1×102 8.70×101 是 1×101 1.16×101 否 1×100 否 否 鸡肉 1×104 8.78×103 是 1×103 9.30×102 是 1×102 1.36×102 是 1×101 1.07×101 否 1×100 否 否 2.5 实际样品检测
实际样品检测按照1.2.9进行,检测结果如表3所示。在实际样品的检测试验中,RPA-LFD方法与常规PCR方法对15份猪肉样品(样品编号ZR1~ZR15)、15份鸡肉样品(样品编号JR1~JR15)、15份生猪养殖场环境样品(样品编号SZ1~SZ15)、15份肉鸡养殖场环境样品(样品编号RJ1~RJ15)、15株大肠杆菌分离株E. coli(菌株编号E1~E15)和15株弯曲肠杆菌分离株样品E. campylobacter(菌株编号EC1~EC15)中mcr-1基因携带情况进行分析,结果显示,RPA-LFD方法与常规PCR方法阳性样本检出率一致,共检出9份mcr-1基因阳性样品。RPA-LFD定量分析显示,阳性样品中mcr-1基因浓度在4.5×102~8.6×104 copies/µL之间。
表 3 实际样品检测结果Table 3. Test results of actual samples样品 数量
(份或株)RPA-LFD检测结果 常规PCR
检测结果阳性数 定量(copies/µL) 阳性数 定量 猪肉样品 15 2 4.5×102~
3.5×1032 − 鸡肉样品 15 0 − 0 − 生猪养殖场环境样品 15 3 2.1×104~
8.6×1043 − 肉鸡养殖场环境样品 15 0 − 0 − 猪肉源大肠杆菌 15 1 7.5×102 1 − 肉鸡养殖场环境
来源弯曲肠杆菌15 3 6.5×102~
7.6×1033 − 3. 讨论与结论
自细菌多黏菌素耐药基因mcr-1被报道以来,基于PCR方法[17-20]和重组酶介导扩增方法[35]等检测方法相继出现。如屈素洁等[17]建立了blaNDM-1和mcr-1基因双重TaqMan荧光定量PCR检测方法,该法对mcr-1基因检出下限为1.63×101 copies/μL,并且能够特异性的检出blaNDM-1和mcr-1基因质粒标准品,重复性好。沈伟伟等[18]建立了双重荧光定量PCR检测携带mcr-1基因的鼠伤寒沙门菌的检测方法,该法对mcr-1基因检测的线性范围为1.40×101~1.40×108 copies/μL,能够实现对携带mcr-1基因的鼠伤寒沙门菌的筛查与耐药性持续监测。车勇良等[35]建立了多黏菌素耐药基因mcr-1重组酶介导扩增结合荧光定量PCR的方法,可在21 min后出结果,能特异性地检测多黏菌素耐药基因mcr-1,最低检出限为102 copies/μL的mcr-1阳性质粒拷贝数。这些检测方法重复性、特异性均好,但大多数仍存在依赖大型仪器等不足,目前仍未见适用于基层进行耐药筛查和监测的mcr-1耐药基因快速检测方法。
本研究利用RPA-LFD技术,是一项将重组酶聚合酶扩增、胶体金标记(三明治夹心法)、分子杂交和侧向流层析等方法相结合,且结果可视化的核酸检测技术[36],与PCR方法相比,不需要复杂的热循环和PCR反应仪器,用时短,在此次细菌多黏菌素耐药基因mcr-1的检测中,只需要37 ℃恒温扩增20 min即可完成,且检出限为101 copies/μL,比PCR法低一个数量级且模拟样品检出结果与PCR法一致。若无恒温条件也可以手握进行核酸扩增,随后将扩增产物滴加在试纸条上便可用肉眼读数,也可用手持式胶体金读进行定量分析,十分简便快速。
综上,本研究建立的RPA-LFD方法检测特异性、灵敏度好,缩短了检测时长,降低了对仪器设备及实验人员的依赖性。这对在生猪养殖场和肉鸡养殖场开展mcr-1耐药基因监测、切断传播途径、保障人们身体健康具有重要意义[37];同时,可为其它致病微生物及其耐药基因的快速检测方法研究提供借鉴,使致病微生物及其耐药性基因的检测向高通量、高灵敏度、简便快捷、经济适用的方向发展。
-
图 2 RPA-LFD反应条件优化
注:a1为反应温度优化电泳结果;a2为反应温度优化LFD试纸条检测结果;a3为反应温度优化手持式设备检测结果;b1为反应时间优化电泳结果;b2为反应时间优化LFD试纸条检测结果;b3为反应时间优化手持式设备检测结果;c1为Mg2+浓度优化电泳结果;c2为Mg2+浓度优化LFD试纸条检测结果;c3为Mg2+浓度优化手持式设备检测结果;M表示DNA Marker (DL2000);a系列图中,1~6分别表示30、35、37、39、45、50 ℃;b系列图中,1~10分别表示2.5、5、7.5、10、12.5、15、17.5、20、22.5、30 min;c系列图中,1~6分别表示2.8、5.6、8.4、11.2、14.0、16.8 mmol/L。
Figure 2. Optimization of RPA-LFD reaction conditions
图 3 RPA-LFD特异性检测
注:a为RPA-LFD特异性电泳结果;b为RPA-LFD特异性LFD试纸条检测结果;M:DNA Marker(DL2000);1:质粒pGM-T-mcr-1;2:E. coli Top10-pGM-T-mcr-1;3:质粒pGM-TT;4:E. coli Top10;5:耐药敏感菌株E. coli ATCC25922;6~8:不携带mcr-1基因的大肠杆菌分离株E. coli;9~10:不携带mcr-1基因的弯曲肠杆菌分离株E. campylobacter;11~12:不携带mcr-1基因的霍氏肠杆菌分离株E. hormaechei;13~14:不携带mcr-1基因的干酪乳杆菌分离株L. casei;15:灭菌去离子水;16:空白对照。
Figure 3. RPA-LFD specificity test
表 1 RPA 扩增和PCR 扩增引物序列
Table 1 Sequence of RPA amplification and PCR amplification primers
引物名称 引物序列(5’-3’) 引物序列大小(bp) mcr-1-F1 5'-CGGTCAGTCCGTTTGTTC-3’ 309 mcr-1-R1 5'-CTTGGTCGGTCTGTAGGG-3’ mcr-1-rpa-F2 5'-GTCCGTTTGTTCTTGTG
GCGAGTGTTGCCGTT-3’298 mcr-1-rpa-R2 5'-TCGGTCTGTAGGGCATTT
TGGAGCATGGTCGT-3’表 2 模拟食样检测结果
Table 2 Test results of simulated food samples
样品 添加浓度(copies/μL) RPA-LFD检测 PCR检测 猪肉 1×104 9.45×103 是 1×103 1.10×103 是 1×102 8.70×101 是 1×101 1.16×101 否 1×100 否 否 鸡肉 1×104 8.78×103 是 1×103 9.30×102 是 1×102 1.36×102 是 1×101 1.07×101 否 1×100 否 否 表 3 实际样品检测结果
Table 3 Test results of actual samples
样品 数量
(份或株)RPA-LFD检测结果 常规PCR
检测结果阳性数 定量(copies/µL) 阳性数 定量 猪肉样品 15 2 4.5×102~
3.5×1032 − 鸡肉样品 15 0 − 0 − 生猪养殖场环境样品 15 3 2.1×104~
8.6×1043 − 肉鸡养殖场环境样品 15 0 − 0 − 猪肉源大肠杆菌 15 1 7.5×102 1 − 肉鸡养殖场环境
来源弯曲肠杆菌15 3 6.5×102~
7.6×1033 − -
[1] SALOME N, MARKUS H, VINCENT P, et al. Extended-spectrum cephalosporin-resistant gram-negative organisms in livestock: An emerging problem for human health?[J]. Drug Resistance Updates,2013,16(1−2).
[2] PATERSON D. Resistance in gram-negative bacteria: Enterobacteriaceae[J]. American Journal of Medicine, 2006, 119: S20-S28.
[3] 陈愿, 张永安, 周洋. 噬菌体在水产养殖业中的研究进展[J]. 水产学报,2021,45(9):1605−1615. [CHEN Y, ZHANG Y A, ZHOU Y. Research progress of bacteriophage in aquaculture[J]. Journal of Fisheries of China,2021,45(9):1605−1615. CHEN Y, ZHANG Y A, ZHOU Y. Research progress of bacteriophage in aquaculture[J]. Journal of Fisheries of China, 2021, 45(9): 1605-1615.
[4] 李一鸣, 王少林. 肠杆菌科细菌耐药基因表达的遗传和环境调控[J]. 生物工程学报,2021,37(4):1092−1106. [LI Y M, WANG S L. The impact of genetic and environmental regulation on theexpression of antibiotic resistance genes in Enterobacteriaceae[J]. Chinese Journal of Biotechnology,2021,37(4):1092−1106. doi: 10.13345/j.cjb.200392 LI Y M, WANG S L. The impact of genetic and environmental regulation on theexpression of antibiotic resistance genes in Enterobacteriaceae[J]. Chinese Journal of Biotechnology, 2021, 37(4): 1092-1106. doi: 10.13345/j.cjb.200392
[5] WANG Y, ZHANG R, LI J, et al. Comprehensive resistome analysis reveals the prevalence of NDM and MCR-1 in Chinese poultry production[J]. Nature microbiology, 2017, 2.
[6] WANG X, WANG Y, ZHOU Y, et al. Emergence of a novel mobile colistin resistance gene, mcr-8, in NDM-producing Klebsiella pneumoniae[J]. Emerging Microbes & Infections,2018,7(1).
[7] BOROWIAK M, BAUMANN B, FISHER J, et al. Development of a novel mcr-6 to mcr-9 multiplex PCR and assessment of mcr-1 to mcr-9 occurrence in colistin-resistant Salmonella enterica isolates from environment, feed, animals and food (2011-2018) in Germany[J]. Frontiers in Microbiology,2020,11:80. doi: 10.3389/fmicb.2020.00080
[8] 孙赫, 张桉潮, 郭越, 等. 吉林省鸡源携带多黏菌素耐药基因mcr-1大肠杆菌的流行病学调查[J]. 中国兽医杂志,2020,56(9):68−73. [SUN H, ZHANG A C, GUO Y, et al. Epidemiological investigation of colistin resistance gene mcr-1 in chickens from Jilin province[J]. Chinese Journal of Veterinary Medicine,2020,56(9):68−73. SUN H, ZHANG A C, GUO Y, et al. Epidemiological Investigation of Colistin Resistance Gene mcr-1 in Chickens from Jilin Province[J]. Chinese Journal of Veterinary Medicine, 2020, 56(9): 68-73.
[9] 王新兴, 翟真真, 常维山, 等. 多粘菌素耐药基因mcr-1的研究进展[J]. 中国动物传染病学报,2020,28(2):110−114. [WANG X X, QU Z Z, CHANG W S, et al. Dvances in research on polymyxin resistence mechanism mcr-1[J]. Chinese Journal of Animal Infectious Diseases,2020,28(2):110−114. WANG X X, QU Z Z, CHANG W S, et al. Dvances in research on polymyxin resistence mechanism mcr-1[J]. Chinese Journal of Animal Infectious Diseases, 2020, 28(2): 110-114.
[10] MICHAELl R M, LAURA F M, JAMES R, et al. Dissemination of the mcr-1 colistin resistance gene[J]. The Lancet Infectious Diseases,2016,16(3):144−145.
[11] 刘向君, 吕媛. 质粒介导的多黏菌素耐药基因mcr-1研究进展[J]. 中华微生物学和免疫学杂志,2018,38(4):305−312. [LIU X J, LÜ Y. Progress in plasmid-mediated colistin resistance gene mcr-1[J]. Chinese Journal of Microbiology and Immunology,2018,38(4):305−312. LIU X J, LV Y. Progress in plasmid-mediated colistin resistance gene mcr-1[J]. Chinese Journal of Microbiology and Immunology, 2018, 38(4): 305-312.
[12] 王齐晖, 刘学佳, 胡锦瑞, 等. 肠杆菌科细菌mcr-1基因筛查及其与多黏菌素耐药表型的相关性[J]. 中国医科大学学报,2018,47(8):713−716. [WANG Q H, LIU X J, HU J R, et al. Screening for the mcr-1 gene in the members of the Enterobacteriaceae and its correlation with colistin resistant phenotype[J]. Journal of China Medical University,2018,47(8):713−716. WANG Q H, LIU X J, HU J R, et al. Screening for the mcr-1 gene in the members of the Enterobacteriaceae and its correlation with colistin resistant phenotype[J]. Journal of China Medical University, 2018, 47(8): 713-716.
[13] 陆上, 毕颖敏, 杨洋, 等. 临床分离肠杆菌科细菌中 mcr-1 的筛查及其阳性菌株的药敏结果[J]. 中国感染与化疗杂志,2018,18(4):408−412. [LU S, BI Y M, YANG Y, et al. Distribution of mcr-1 gene among clinical isolates of Enterobacteriaceae and antimicrobial susceptibility profile of mcr-1 positive strains[J]. Chinese Journal of Infection and Chemotherapy,2018,18(4):408−412. LU S, BI Y M, YANG Y, et al. Distribution of mcr-1 gene among clinical isolates of Enterobacteriaceae and antimicrobial susceptibility profile of mcr-1 positive strains[J]. Chinese Journal of Infection and Chemotherapy, 2018, 18(4): 408-412.
[14] 郑效瑾, 吕东月, 俞全喜, 等. blaNDM 和 mcr-1~mcr-5 基因在中国不同标本中的分布[J]. 中国校医,2022,36(1):21−25. [ZHENG X J, LV D Y, YU Q X, et al. Distribution of blaNDM and mcr-1~mcr-5 genes in different types of samples in China[J]. Chinese Journal of School Doctor,2022,36(1):21−25. ZHENG X J, LV D Y, YU Q X, et al. Distribution of blaNDM and mcr-1~mcr-5 genes in different types of samples in China[J]. Chinese Journal of School Doctor, 2022, 36(1): 21-25.
[15] 樊融. 中国不同来源的革兰阴性细菌mcr-1及bla-(NDM)基因的筛选及分析[D]. 北京: 中国疾病预防控制中心, 2020 FAN R. Retrospective screening and analysis of mcr-1 and bla-NDM in gram-negative bacteria in China[D]. Beijing: Chinese Center for Disease Control and Prevention, 2020.
[16] 陈莉. 粘菌素耐药基因(mcr-1-mcr-8)的流行病学调查及mcr-1传播机制的研究[D]. 扬州: 扬州大学, 2019 CHEN L. Molecular epidemiology of mobile colistin resistance genes(mcr-1- mcr-8) and transmission mechanism of mcr-1 positive Escherichia coli[D]. Yangzhou: Yangzhou University, 2019.
[17] 屈素洁, 施开创, 尹彦文, 等. 超级细菌bla_(NDM-1)和mcr-1基因双重TaqMan荧光定量PCR检测方法的建立[J]. 中国动物传染病学报,2020,28(5):23−30. [QU S J, SHI K C, YIN Y W, et al. Establishment of duplex taoman real-time PCR fordetection of bla_(NDM-1) and mcr-1 genes of superbug[J]. Chinese Journal of Animal Infectious Diseases,2020,28(5):23−30. QU S J, SHI K C, YIN Y W, et al. Establishment of duplex taoman real-time PCR fordetection of bla_(NDM-1) and mcr-1 genes of superbug[J]. Chinese Journal of Animal Infectious Diseases, 2020, 28(5): 23-30.
[18] 沈伟伟, 盛莹, 管雅雅, 等. 双重荧光定量PCR检测携带mcr-1基因的鼠伤寒沙门菌的建立与应用[J]. 中国卫生检验杂志,2020,30(12):1412−1415. [SHEN W W, SHENG Y, GUAN Y Y, et al. Establishment and application of double fluorescent quantitative PCR in detection of Salmonella typhimurium carrying mcr-1 gene[J]. Chinese Journal of Health Laboratory Technology,2020,30(12):1412−1415. [SHEN W W, SHENG Y, GUAN Y Y, et al. Establishment and application of double fluorescent quantitative PCR in detection of Salmonella typhimurium carrying mcr-1 gene[J]. Chinese Journal of Health Laboratory Technology, 2020, 30(12): 1412-1415. ]
[19] 张克旭, 滚双宝, 车勇良, 等. 耐药基因mcr-1和bla_(NDM-1)的双重荧光定量PCR检测方法的建立[J]. 动物医学进展,2021,42(10):9−13. [ZHANG K X, GUN S B, CHE Y L, et al. Establishment of a duplex reatime PCR method for detecting drug resistance genes mcr-l and bla__(NDM-1)[J]. Progress in Veterinary Medicine,2021,42(10):9−13. ZHANG K X, GUN S B, CHE Y L, et al. Establishment of A Duplex Reatime PCR Method for Detecting Drug Resistance Genes mcr-l and bla NDM-1[J]. Progress In Veterinary Medicine, 2021, 42(10): 9-13.
[20] YONGBING L, HAIBEI L, CHUNYAN C, et al. Profile of antibiotic resistance genes in the terminal tap water from the center area of Tianjin[J]. Asian Journal of Ecotoxicology,2021(2):195−202.
[21] MIAO F, ZHANG J, LI N, et al. Rapid and sensitive recombinase polymerase amplification combined with lateral flow strip for detecting African swine fever virus[J]. Frontiers in Microbiology,2019,10(1004):1−7.
[22] LI J L, MA B, FANG J H, et al. Recombinase polymerase amplification (RPA) combined with lateral flow immunoassay for rapid detection of Salmonella in food[J]. Foods,2020,9(27):1−12.
[23] CHEN K, MA B, LI J, et al. A rapid and sensitive europium nanoparticle-based lateral flow immunoassay combined with recombinase polymerase amplification for simultaneous detection of three food-borne pathogens[J]. International Journal of Environmental Research and Public Health,2021,18(9):4574. doi: 10.3390/ijerph18094574
[24] SRISRATTAKARN A, PANPRU P, TIPPAYAWAT P, et al. Rapid detection of methicillin-resistant Staphylococcus aureus in positive blood-cultures by recombinase polymerase amplification combined with lateral flow strip[J]. PLoS One,2022,17(6):e0270686. doi: 10.1371/journal.pone.0270686
[25] 赵玉梅. 三种重要疫霉菌 LFD-RPA 快速检测技术研究[D]. 福州: 福建农林大学, 2020 ZHAO Y M. Rapid detection of three main Phytophthora spp. using lateral flow dipstick-recombinase polymerase amplification assay[D]. Fuzhou: Fujian Agriculture and Forestry University, 2020.
[26] DAHEER R K, STEWART G, BOISSINOT M, et al. Recombinase polymerase amplification for diagnostic applications[J]. Clinical Chemistry,2016,62(7):947−958. doi: 10.1373/clinchem.2015.245829
[27] 邓王平, 洪清华, 徐斌, 等. 基于RPA-LFD的日本血吸虫循环核酸快速可视化检测方法的建立及初步评价[J]. 中国寄生虫学与寄生虫病杂志,2020,38(3):286−292. [DENG W P, HONG H Q, XU B, et al. Development and preliminary evaluation of a rapid visualization detection method for circulating nucleic acids of Schistosoma japonicum based on RPA-LFD[J]. Chinese Journal of Parasitology and Parasitic Diseases,2020,38(3):286−292. DENG W P, HONG H Q, XU B, et al. Development and preliminary evaluation of a rapid visualization detection method for circulating nucleic acids of Schistosoma japonicum based on RPA-LFD[J]. Chinese Journal of Parasitology and Parasitic Diseases, 2020, 38(3): 286-292.
[28] 蔡应奎, 刘新生, 张丽萍, 等. 猪嵴病毒重组酶聚合酶扩增结合侧流层析试纸条(RPA-LFD)快速诊断方法的建立与应用[J]. 中国兽医科学,2020,50(7):820−824. [CAI Y K, LIU X S, ZHANG L P, et al. Development and application of a recombinase polymerase amplification combined with a lateral flow dipstick (RPA-LFD) assay for rapid diagnosis of porcine kobuvirus[J]. Veterinary Science in China,2020,50(7):820−824. CAI Y K, LIU X S, ZHANG L P, et al. Development and application of a recombinase polymerase amplification combined with a lateral flow dipstick (RPA-LFD) assay for rapid diagnosis of porcine kobuvirus[J]. Veterinary Science in China, 2020, 50(7): 820-824.
[29] 于博, 李博宇, 赵博, 等. 布鲁氏菌RPA-LFD检测方法的建立[J]. 中国预防兽医学报,2019,41(12):1233−1237. [YU B, LI B Y, ZHAO B, et al. Establishment of RPA-LFD detection method for Brucella[J]. Chinese Journal of Preventive Veterinary Medicine,2019,41(12):1233−1237. YU B, LI B Y, ZHAO B, et al. Establishment of RPA-LFD detection method for Brucella[J]. Chinese Journal of Preventive Veterinary Medicine, 2019, 41(12): 1233-1237.
[30] 林彦星, 吴江, 赵现锋, 等. 非洲猪瘟病毒 LFD-RPA 快速检测方法的建立[J]. 中国兽医科学,2020,50(8):952−956. [LIN Y X, WU J, ZHAO X F, et al. Development of LFD-RPA assay for rapid detection of African swine fever virus[J]. Veterinary Science in China,2020,50(8):952−956. LIN Y X, WU J, ZHAO X F, et al. Development of LFD-RPA assay for rapid detection of African swine fever virus[J]. Veterinary Science in China, 2020, 50(8): 952-956.
[31] JIN B, MA B, LI J, et al. Simultaneous detection of five foodborne pathogens using a mini automatic nucleic acid extractor combined with recombinase polymerase amplification and lateral flow immunoassay[J]. Microorganisms,2022,10(7):1352. doi: 10.3390/microorganisms10071352
[32] LIU H, CAO R, XU W, et al. A cost-effective method for the rapid detection of chicken adulteration in meat using recombinase polymerase amplification combined with nucleic acid hybridization lateral flow strip[J]. Journal of Food Composition and Analysis,2022,111:104602. doi: 10.1016/j.jfca.2022.104602
[33] LAI F Y, CHANG K C, CHANG C S, et al. Development of a rapid sex identification method for newborn pigeons using recombinase polymerase amplification and a lateral-flow dipstick on farm[J]. Animals,2022,12(21):2969. doi: 10.3390/ani12212969
[34] HOLMES D S, QUIGLEY M. A rapid boiling method for the preparation of bacterial plasmids[J]. Analytical Biochemistry,1981,114(1):193−197. doi: 10.1016/0003-2697(81)90473-5
[35] 车勇良, 陈秋勇, 陈如敬, 等. 多黏菌素耐药基因mcr-1重组酶介导扩增方法的建立及应用[J]. 动物医学进展,2022,43(8):46−49. [CHE Y L, CHEN Q Y, CHEN J R, et al. Development and application of recombinase-aided amplification assay (RAA) for detecting polymyxin-resistant mcr-1 gene[J]. Progress in Veterinary Medicine,2022,43(8):46−49. CHE Y L, CHEN Q Y, CHEN J R, et al. Development and application of recombinase-aided amplification assay (RAA) for detecting polymyxin-resistant mcr-1 gene[J]. Progress In Veterinary Medicine, 2022, 43(8): 46-49.
[36] 于灵芝, 陶凌云, 魏晓锋. 可视化核酸检测技术RPA-LFD的研究和应用进展[J]. 实验动物与比较医学,2021,41(6):547. [YU L Z, TAO L Y, WEI X F. Research and application progress in visualized rpa-lfd nucleic acid detection technology[J]. Laboratory Animal and Comparative Medicine,2021,41(6):547. YU L Z, TAO L Y, WEI X F. Research and application progress in visualized rpa-lfd nucleic acid detection technology[J]. Laboratory Animal and Comparative Medicine, 2021, 41(6): 547.
[37] 张闪闪, 何斌, 李书光, 等. 可视化RPA-LFD技术快速检测猪链球菌[J]. 畜牧兽医学报,2022,53(2):538−547. [ZHANG S S, HE B, LI S G, et al. Rapid detection of Streptococcus suis with visual RPA-LFD technology[J]. Acta Veterinaria et Zootechnica Sinica,2022,53(2):538−547. ZHANG S S, HE B, LI S G, et al. Rapid detection of Streptococcus suis with visual RPA-LFD technology[J]. Acta Veterinaria et Zootechnica Sinica, 2022, 53(2): 538-547.
-
期刊类型引用(2)
1. 逄梦玉,隋昕怡,刘丹,刘养山,任洪飞,张景,杜秀菊. 酿酒酵母发酵法制备桑黄多糖工艺优化及其结构表征和抗氧化活性研究. 天然产物研究与开发. 2025(01): 85-94 .  百度学术
百度学术
2. 逄梦玉,隋昕怡,刘丹,任洪飞,刘养山,张景,杜秀菊. 桑黄多糖提取工艺优化、结构表征及抗氧化活性研究. 食品安全质量检测学报. 2024(12): 48-57 .  百度学术
百度学术
其他类型引用(5)





 下载:
下载:
 下载:
下载:
