Rapid Detection of Viable Penicillium expansum in Apple by PMA-qPCR
-
摘要: 目的:为建立一种叠氮溴化丙锭(Propidium Monoazide,PMA)与实时定量聚合酶链式反应(Quantitative real-time Polymerase Chain Reaction,qPCR)联用的快速检测扩展青霉活菌方法。方法:通过优化PMA处理浓度、黑暗孵育及曝光时间,筛选扩展青霉特异性引物,结合qPCR技术,建立一种基于PMA-qPCR联用快速检测扩展青霉活菌的方法,构建定量标准曲线,应用于人工污染的苹果样品中活菌的检测,与平板菌落计数比较评估该方法的可靠性。结果:PMA处理浓度10 µg/mL、黑暗孵育5 min、曝光10 min为最佳PMA处理条件。4种引物中Pexp-patF对扩展青霉具有极强的特异性,可作为引物用于PMA-qPCR检测。构建的定量标准曲线的R2=0.9948,最低检测限为102.6 CFU/mL,方法检测结果与平板菌落计数无明显差异,并发现在苹果的未腐烂部分中可检测出扩展青霉活菌。结论:研究建立的PMA-qPCR技术可应用于苹果中扩展青霉活菌的检测,为扩展青霉精准防控提供一定技术支撑。
-
关键词:
- 扩展青霉 /
- 叠氮溴化丙锭 /
- 实时定量聚合酶链式反应 /
- 苹果 /
- 活菌
Abstract: Objective: To establish a rapid detection method for viable Penicillium expansum by propidium monoazide (PMA) combined with quantitative real-time polymerase chain reaction (qPCR). Methods: PMA-qPCR detection method for viable P. expansum was established, including the optimization of the treatment concentration, dark incubation and exposure time of PMA, the screening of specific primers of P. expansum, and the conduction of qPCR. In addition, the standard curve was constructed, which was applied to the detection of artificially contaminated apples samples. The reliability of this method was also evaluated by comparing with the plate counting method. Results: The optimal PMA treatment conditions were: 10 µg/mL for PMA concentration, 5 min for the dark incubation and 10 min for the exposure time. Among the 4 pairs of primers, Pexp-patF showed strong specificity for P. expansum, which could be used as a optimal primer for PMA-qPCR detection. The correlation coefficient of the established quantitative standard curve was 0.9948, and the detection limit of the method was 102.6 CFU/mL. There was no obvious difference between the detection result of this method and the plate counting method, and the viable P. expansum could be detected in the non-rotted part of apples. Conclusion: The established PMA-qPCR technique could be applied to the detection of P. expansum in apples, which would provide technical support for the prevention and control of P. expansum. -
以扩展青霉(Penicillium expansum)为主的植物病原菌感染导致每年都会有大量的水果被侵蚀腐烂,造成大量的经济损失,大概有25%的农产品会受到真菌毒素的污染,部分国家受污染比例可达到农产品产量的一半及以上[1]。扩展青霉产生的展青霉素具有肠、肝、肾、免疫、基因毒性等[2],对多种人体器官造成伤害,被国际癌症研究机构(International Agency for Research on Cancer,IARC)认定为“第三类可疑致癌物”[3]。早年就有研究者发现切除腐烂部位并不能完全去除展青霉素,扩展青霉和毒素会向未腐烂的组织迁移,并且未腐烂组织中的毒素含量可能会超过最低限量[4],展青霉素迁移远慢于扩展青霉菌落的迁移[5]。根据毒素含量与菌落浓度之间的对应关系可以通过菌落浓度来反映毒素含量[6]。近年来越来越多研究者开始利用qPCR技术对扩展青霉进行检测从而研究毒素含量。Tannous等[7]创建了利用qPCR检测苹果中扩展青霉菌DNA从而估计展青霉素含量的方法。Rodríguez等[8]也利用SYBR Green和TaqMan两种方法进行qPCR技术检测展青霉素含量。从控制展青霉素污染角度来说对扩展青霉菌活菌检测具有重要意义。但普通的qPCR技术并不能分辨出活菌和死菌的区别,极易出现假阳性结果。
PMA和叠氮溴化乙锭(Ethidium Monoazide Bromide,EMA)都属于对DNA有着高亲和力的光敏DNA染料[9]。PMA可以选择性穿过死菌破损细胞膜,在钨灯强光照射下,PMA的叠氮基团生成的nitrene基,在结合部位与DNA中的碳氢氧键结合生成稳共价氮碳键,从而抑制了死菌DNA扩增,qPCR检测中假阳性信号的产生[10]。PMA比EMA多一个正点荷,更加温和、更难进入活菌细胞膜[11],近年来研究者更倾向用PMA结合qPCR技术研究活菌检测。目前该技术已广泛应用于药物活性检测[12]、医源及食源性病原菌检测[13-14]、污水处理检测[15]、植物病原菌检测[16]等。对于利用PMA-qPCR这一技术对植物病原菌进行研究,已应用于青枯菌(Pseudomonas solanacearum)[11]、十字花科黑斑病菌(Pseudomonas syringae pv. maculicola)[17]、梨火疫病菌(Erwinia amylovory)[9]、丁香假单胞菌(Pseudomonas syringae)[18]的活菌检测等,但该技术对扩展青霉菌活菌检测及相关的研究较少。
因此,为了深入研究扩展青霉菌活菌在苹果上的迁移规律,本文将以产自新疆阿克苏的红富士苹果为材料,人工接种扩展青霉进行常温培养,改进Crespo-Sempere等[19]的PMA处理方法,利用PMA-qPCR技术快速、准确地对扩展青霉活菌数进行计数,为苹果中扩展青霉的防控提供了理论依据,为食品中扩展青霉活菌检测提供新方法。
1. 材料与方法
1.1 材料与仪器
扩展青霉菌ACCC 31688 中国农业微生物菌种保藏管理中心;红富士苹果(Malus domestica Borkh.CV.Red Fuji) 同一批次的成熟的大小相近、无创伤,新疆乌鲁木齐市;马铃薯葡萄糖琼脂(PDA)培养基 北京陆桥技术股份有限公司;PMA染料 北京百瑞极生物科技有限公司;真菌DNA提取试剂盒、CTAB缓冲液、蛋白酶K溶液、RNase A溶液、DNA提取液、核酸提取液 北京索莱宝公司;2×Taq PCR MasterMixⅡ、2×SuperReal PreMix Plus 天根生化科技(北京)有限公司;异丙醇 分析醇,天津永晟精细化工有限公司。
生物安全柜ESCO 生命科学集团;霉菌培养箱 上海精宏实验设备有限公司;Thermo Fisher Scientific离心机 德国贺利氏公司;PCR仪(SureCycle 8800) 安捷伦科技有限公司;实时荧光定量PCR仪(LightCycler® 96) 德国罗氏诊断有限公司。
1.2 实验方法
1.2.1 霉菌孢子悬浮液配制
无菌条件下,向已培养7 d的扩展青霉菌的PDA培养基中加入10 mL无菌水,利用无菌涂布器刮下真菌孢子,四层纱布过滤,将滤液转至50 mL锥形瓶中,旋涡30 s,使孢子分布均匀后加入无菌水进行梯度稀释。采用血球计数板法计算孢子浓度,最终将孢子悬浮液稀释至4.0×107 CFU/mL。
取10 mL稀释好的孢子悬浮液放入水浴锅,99.9 ℃水浴30 min进行热灭活处理。取1 mL经热灭活处理的孢子悬浮液涂布于PDA培养基28 ℃培养72 h,验证灭活效果。PDA培养基上无菌生长则认为已完全灭活,可用于后续实验。
1.2.2 霉菌及苹果样品DNA提取
1.2.2.1 霉菌DNA提取
取500 µL孢子悬浮液,12000 r/min条件下离心2 min,弃上清,沉淀按照真菌DNA提取试剂盒说明书提取DNA。检测前放入−20 ℃保存。
1.2.2.2 苹果样品DNA提取
参考Crespo-Sempere等[19]的方法,根据样品略有改动。取5 g样品(受扩展青霉侵染的苹果病斑)装入50 mL试管,加入5 mL的磷酸缓冲盐溶液(Phosphate Buffered Saline,PBS)。匀质3 min,4层纱布过滤。将滤液转移至离心管中,12000 r/min离心10 min,弃去上清。沉淀重悬于1 mL PBS缓冲液后进行PMA处理。PMA处理后,12000 r/min离心2 min,弃去上清。
参考Tannous等[7]的方法,根据实验条件略做修改,提取沉淀DNA。将沉淀重悬于经95 ℃预热100 µL无菌水中并在冰上放置10 min。加入500 µL CTAB缓冲液,10 µL蛋白酶K溶液,65 ℃水浴1 h,4 ℃条件下13000 r/min离心10 min。吸取上清,加等体积的DNA提取液(酚/氯仿/异戊醇25:24:1)静置5 min,4 ℃条件下1000 r/min离心20 min。吸取上清,加入10 µL RNase A溶液(10 mg/mL),37 ℃恒温水浴1 h。水浴后,加等体积的核酸提取液(氯仿/异戊醇24:1),4 ℃条件下13000 r/min离心5 min。取上清液用等体积的异丙醇−20 ℃沉淀过夜。4 ℃条件下13000 r/min离心20 min,弃去上清,沉淀中加入500 µL的75%乙醇进行洗脱,12000 r/min离心5 min,重复洗脱2次。弃上清,加入室温干燥5~10 min,加入30 µL的dd H2O,−20 ℃保存。
1.2.3 PMA处理条件优化
1.2.3.1 PMA浓度优化
称取1 mg PMA加入1 mL的无菌水,制备浓度为2 mmol/L的PMA溶液,4 ℃储存。取500 µL浓度为4.0×107 CFU/mL的热灭活孢子悬浮液,加入不同体积的PMA溶液,使最终每管的PMA浓度分别为0、5、10、15、20和 25 µg/mL。充分混匀后黑暗孵育20 min,然后将试管放置冰上在钨灯强光下照射10 min。12000 r/min离心 2 min,弃上清,取沉淀提取孢子DNA,具体步骤同1.2.2.1。所提DNA按照1.2.5中的反应体系及条件进行qPCR检测,获得循环阈值(Cycle Threshold,Ct值),根据Ct值变化来研究能够抑制死菌DNA扩增的PMA最小浓度。同样方法处理活孢子悬浮液,研究不影响活菌DNA扩增的PMA最大浓度。
1.2.3.2 黑暗孵育时间优化
分别添加PMA溶液于500 µL浓度为4.0×107 CFU/mL的热灭活孢子悬浮液,充分混匀,使PMA最终浓度为10 µg/mL,随后置于黑暗处孵育。设置孵育时间分别为5、10、15、20和25 min,黑暗孵育后放置冰上在钨灯下照射10 min,之后步骤按1.2.3.1进行。
1.2.3.3 曝光时间优化
分别添加PMA溶液于500 µL浓度为4.0×107 CFU/mL的热灭活孢子悬浮液,充分混匀,获得PMA最终浓度为10 µg/mL,随后置于黑暗处孵育5 min,设置曝光时间分别为0、5、10、15和20 min。之后步骤按1.2.3.1进行。
1.2.4 引物选择及验证
实验所用的4种引物均参考已发表文献,并由生工生物工程(上海)股份有限公司合成,详情见表1。
以所提的扩展青霉菌DNA作为模板,各引物经PCR扩增后,用1%琼脂糖凝胶电泳法验证扩增产物的大小及引物的特异性,PCR反应体系详情见表2。PCR总共35个循环,每个循环包括94 ℃预热 4 min,94 ℃变性30 s,52 ℃ 30 s,72 ℃延伸45 s,72 ℃最后延伸10 min。
表 2 PCR反应体系Table 2. PCR reaction system体系成分 体积(µL) 2×Taq PCR MasterMixⅡ 25 扩展青霉菌DNA 4 正引物 2 反引物 2 dd H2O 17 总体积 50 1.2.5 扩展青霉菌荧光定量PCR实验
选取实验1.2.4选择出特异性最好的引物按照表3中的体系用实时荧光定量PCR仪进行聚合酶链式反应。热循环条件为:95 ℃作用3 min,40个循环95 ℃作用20 s,65 ℃作用20 s(在此期间测量荧光),57~95 ℃绘制熔解曲线,升温速率为1 ℃/min。
表 3 qPCR反应体系Table 3. qPCR reaction system体系成分 体积(µL) 2×SuperReal PreMix Plus 10 样品DNA 4 正引物 1 反引物 1 ddH2O 4 总体积 20 1.2.6 标准曲线的制作
参考Crespo-Sempere等[19]的标准曲线的制作方法,略有改动。挑选无病害的苹果,用清水清洗,75%酒精消毒,晾干后打浆。取5 g样品装入50 mL试管,加入5 mL的PBS缓冲液。配制106 CFU/mL的扩展青霉孢子悬浮液,加入无菌水梯度稀释至浓度为105、104、103 CFU/mL。加入孢子悬浮液,使样品中孢子最终浓度达到105、104、103、102 CFU/mL。按照1.2.2.2提取DNA,通过qPCR测定对应的Ct值,反应体系参照1.2.5,重复3次。建立菌落浓度(log10 CFU/mL)与Ct值之间的线性关系,并通过公式E=10−1/s−1计算扩增效率,其中s为标准曲线的斜率。
1.2.7 人工染菌样品活菌检测
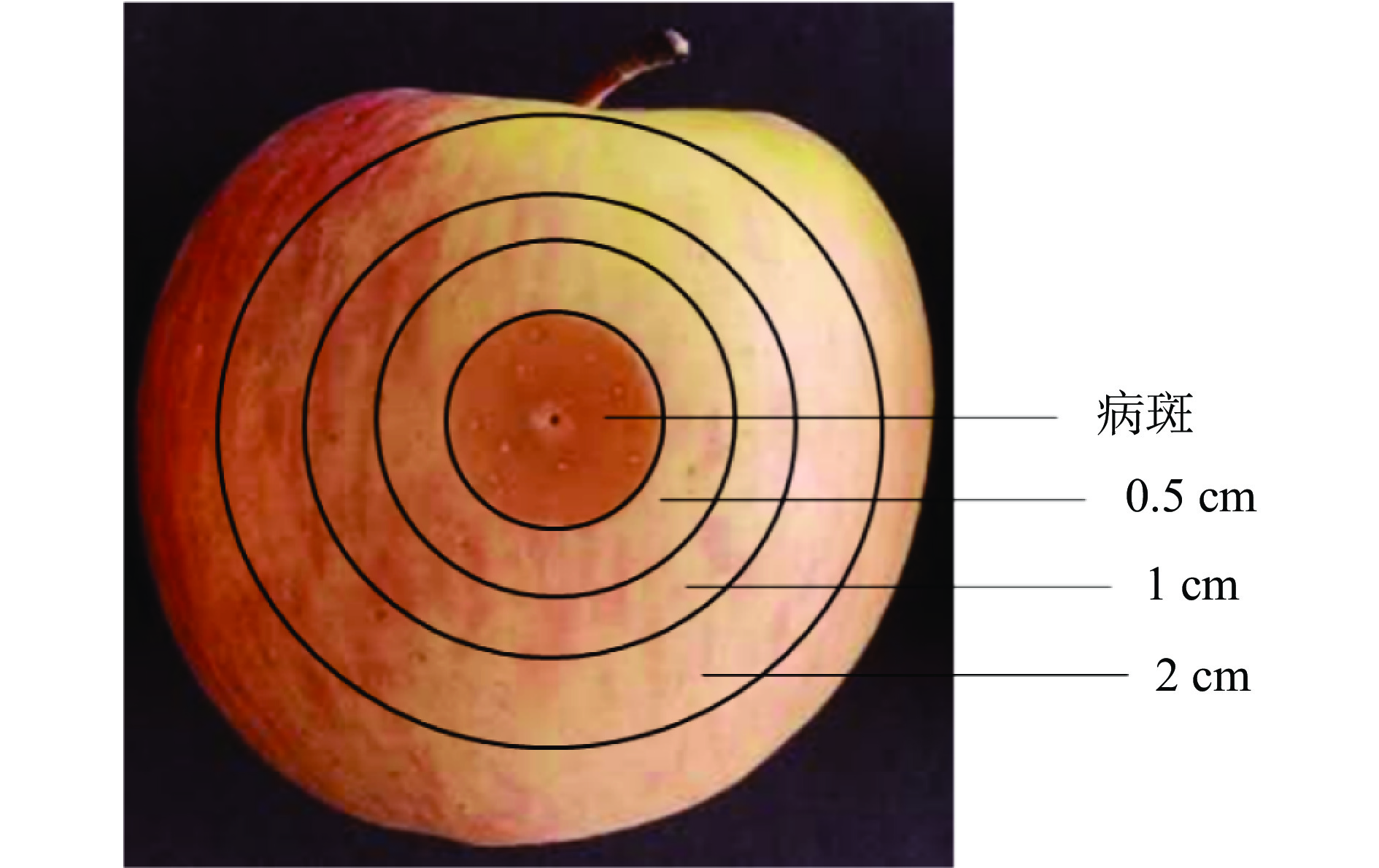
用清水清洗苹果晾干后,用75%酒精擦拭表面。用灭菌过的移液枪头在每个苹果赤道部位对称刺2个直径2~3 mm、深度5~7 mm的伤口,每个伤口接种10 µL孢子悬浮液(1.0×105 CFU/mL),待孢子悬浮液晾干后,放入经紫外灭菌的纸箱中。在室温条件下培养3 d后,以病斑为中心,分别取外扩0.5、1、2 cm处苹果组织为样品(如图1所示)。取1 g样品参照尉冬梅等[23]的方法进行平板菌落计数。
冰浴研磨后,称取5g 样品装入50 mL试管,加入5 mL PBS缓冲液。匀质3 min,4层纱布过滤。将滤液转移至离心管中,12000 r/min离心10 min,弃去上清。沉淀中加入1 mL PBS缓冲液,按照优化后的PMA处理条件进行处理。按照1.2.2.2的方法提取样品DNA后,进行qPCR反应。反应条件及体系同1.2.5。所有DNA样品在qPCR检测之前都经过常规PCR和凝胶电泳检测,以防后期因苹果DNA样品问题出现假阴性结果。
1.3 数据处理
所有实验均重复3次,数据结果以平均值±标准差表示。利用OriginPro 2019作图,Excel 2016进行数据分析,IBM SPSS Statistics 23软件进行单因素ANOVA 检验及独立样本t检验分析差异显著性。
2. 结果与分析
2.1 PMA条件优化
2.1.1 PMA浓度优化
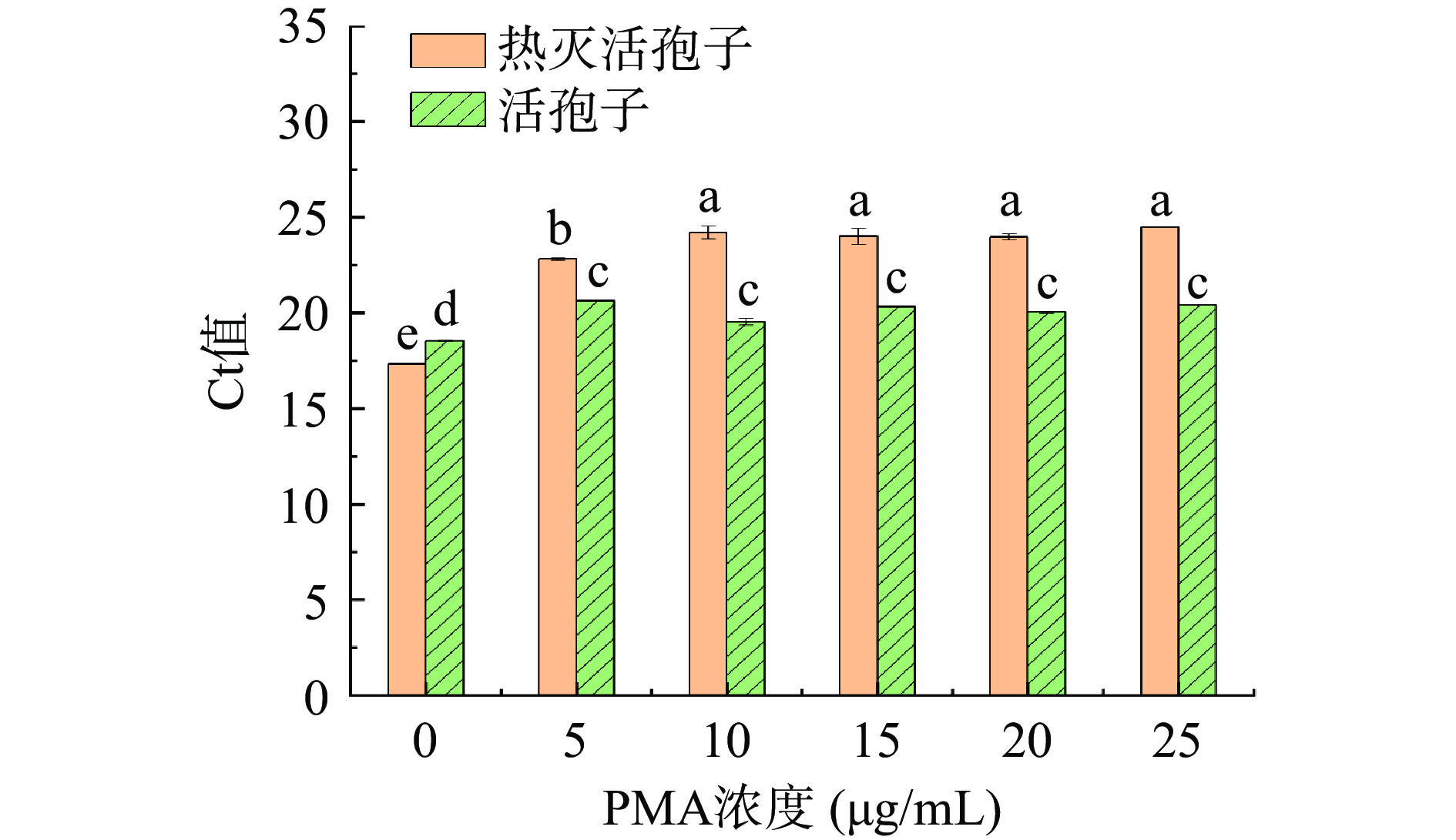
添加不同浓度的PMA对扩展青霉热灭活孢子悬浮液和活孢子悬浮液进行处理,研究不同浓度PMA对扩展青霉热灭活孢子和活孢子qPCR扩增的影响。图2结果显示使用PMA溶液能够明显抑制热灭活扩展青霉的DNA扩增,当PMA浓度=0时,Ct值与PMA处理组Ct值具有显著差异(P<0.05);当PMA浓度<10 µg/mL时,Ct值随着PMA浓度增加而增大,这说明低浓度的PMA不能完全抑制热灭活孢子DNA扩增;当PMA浓度>10 µg/mL时,Ct值不存在显著差异(P>0.05),说明10 µg/mL的PMA能完全与热灭活孢子DNA结合,并完全抑制热灭活孢子DNA扩增。不同浓度的PMA对活孢子进行处理,对活孢子qPCR扩增无显著抑制作用(P>0.05)。故选10 µg/mL为最佳PMA处理浓度。
2.1.2 黑暗孵育时间优化
如图3所示,黑暗孵育处理5、10、15、20和25 min的Ct值无显著差异(P>0.05),说明黑暗孵育5 min时PMA就能够完全与热灭活孢子DNA结合,抑制热灭活孢子DNA的扩增;同时不同黑暗孵育处理,对活孢子DNA扩增并无显著抑制作用(P>0.05),说明黑暗处理5~25 min均不会对活孢子产生毒性,且不会影响活孢子DNA的扩增,黑暗孵育5 min处理能够抑制热灭活孢子DNA的扩增,但不影响活孢子DNA的扩增,故选5 min为最佳黑暗孵育时间。
2.1.3 曝光时间优化
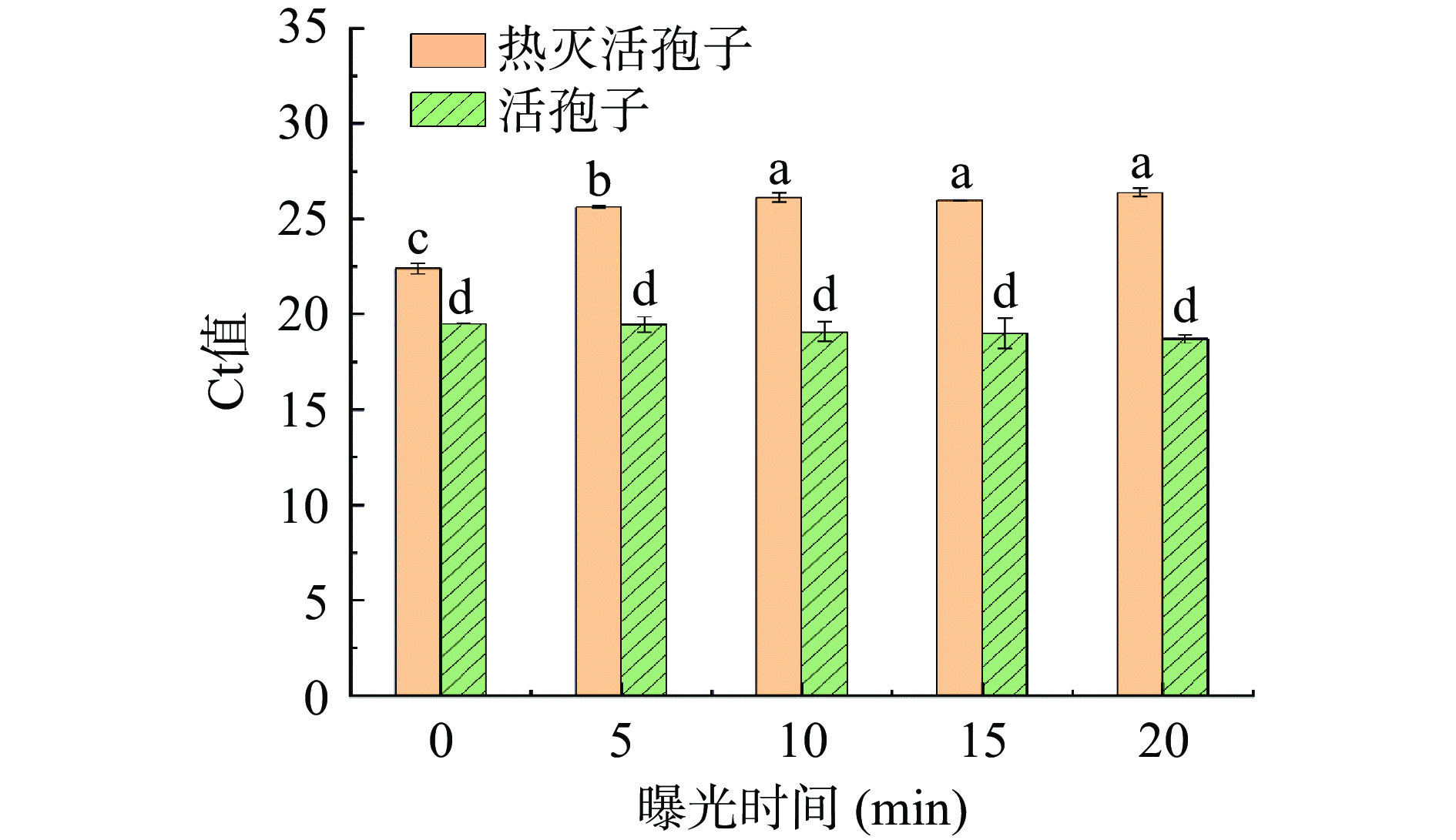
如图4所示,使用PMA对扩展青霉热灭活孢子进行处理,曝光时间<10 min时,Ct值会随着曝光时间的增加而增大,说明不足的曝光时间,不能使残留的PMA充分光解,从而不能完全抑制热灭活孢子DNA扩增;当曝光时间>10 min时,Ct值无显著差异(P>0.05),说明曝光10 min就能够使残留的PMA充分光解。使用PMA处理扩展青霉活菌处理,不同的曝光时间组的Ct值无显著差异(P>0.05),说明不同的曝光时间并不影响活孢子DNA扩增。故选10 min为最佳曝光时间。
本研究最终发现10 µg/mL为PMA最佳浓度,5 min为最佳黑暗孵育时间,10 min为最佳曝光时间,与于璇等[17]设计检测十字花科黑斑病原菌的最佳处理条件一致,与王帅等[11]建立的青枯菌PMA-qPCR检测的最佳处理条件(PMA浓度为15 ng/µL、黑暗孵育处理10 min、曝光处理5 min)不同。两种方法的处理条件不同可能是菌种品类不同、PMA产品不同、钨灯型号不同所致[9]。
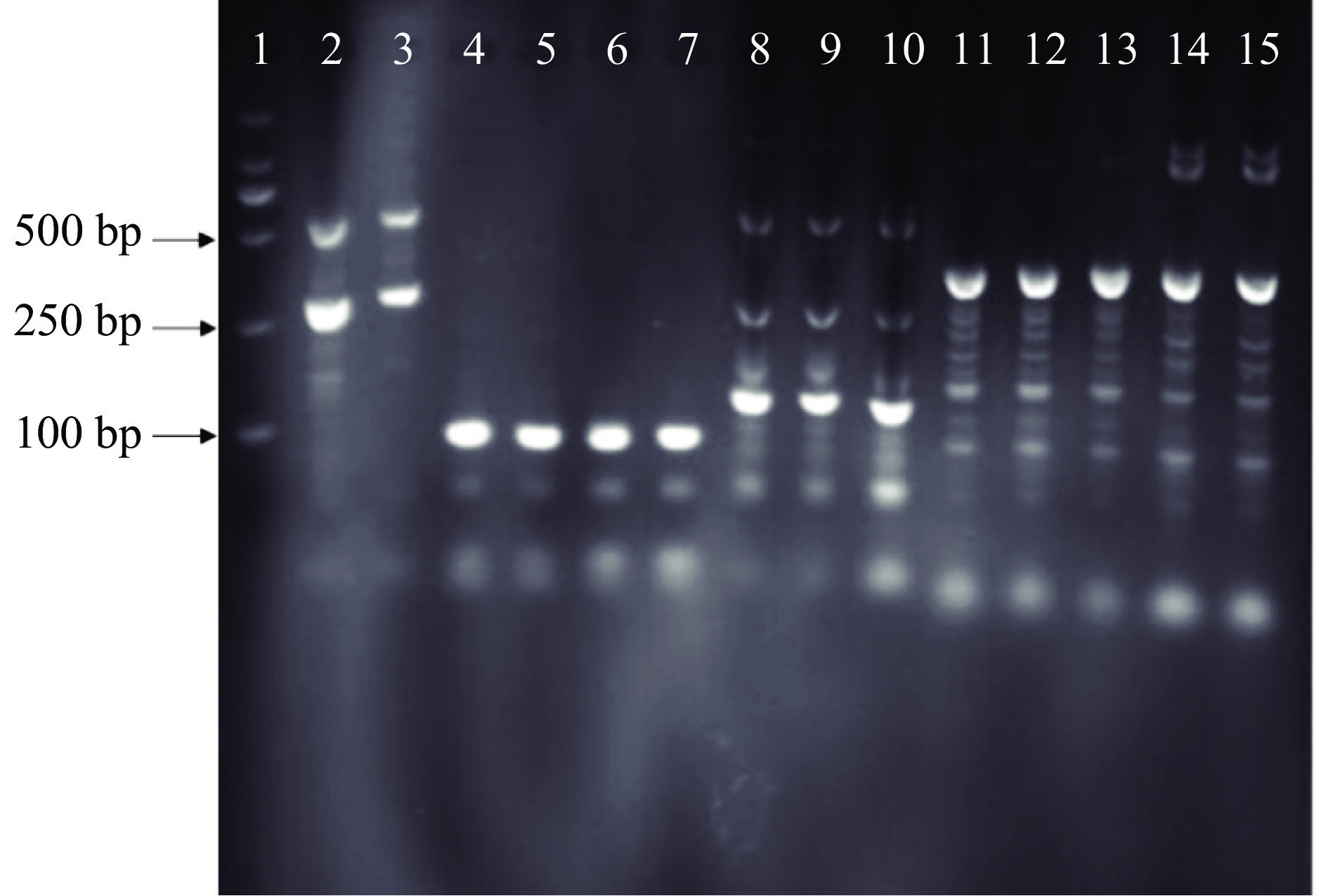
2.2 引物特异性
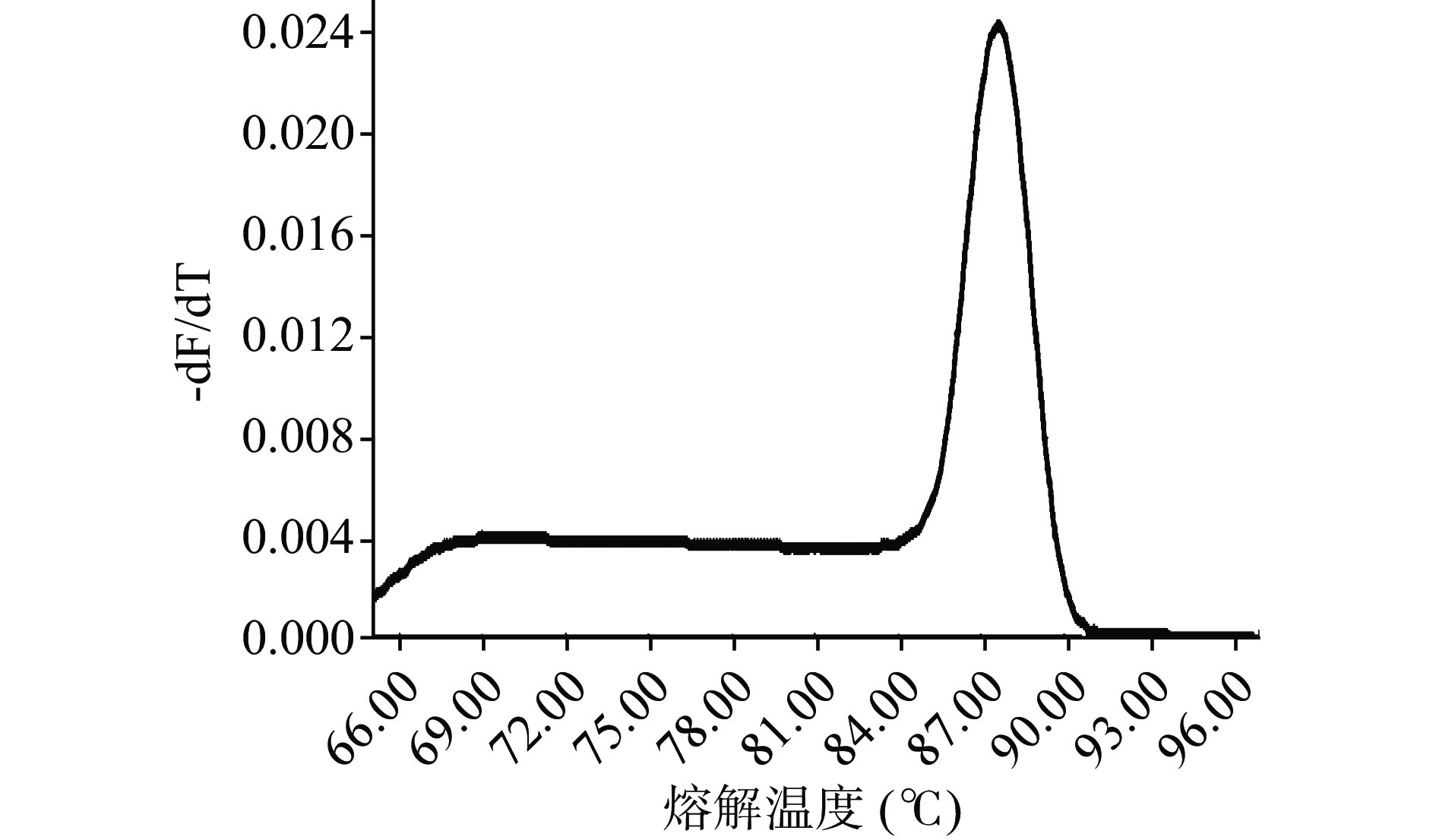
通过对各引物PCR扩增、凝胶电泳验证,结果(图5)显示Pexp-patF引物扩增出的片段大小与Tannous等[7]方法中扩增片段大小一致,为92 bp,且其只产生一条带,并无引物二聚体的存在,与本实验所用菌株基因特异性结合,特异性最强,而PG、POL、HHJ等3个引物扩增产物为多个条带,另外,对Pexp-patF进行qPCR扩增,观察反应熔解曲线在熔解温度(Melting Temperature,Tm)为87.4 ℃时有单一的熔解峰,无引物二聚体和非特异性出现(图6),因此,Pexp-patF可以用于后期PMA-qPCR实验。Pexp-patF是Tannous等[7]通过展青霉素合成调控基因PatF所设计,PatF基因是调控新棒曲霉素合成酶的基因,展青霉素合成的中间物质叶点霉素在新棒曲霉素合成酶作用下会生成新棒曲霉素,新棒曲霉素在乙醇脱氢酶、葡萄糖-甲醇-胆碱氧化还原酶的作用下最终转化为展青霉素[24],这可能是Pexp-patF能够与扩展青霉基因结合、且特异性良好的原因。PG引物实验所用菌株是从腐烂柑橘中分离,POL菌株来源于加拿大国家食品安全研究所,HHJ菌株来源于中国微生物中国普通微生物菌种保藏管理中心中分离均与该实验所用菌株不同。PG、POL、HHJ引物没有跟本实验所用扩展青霉基因特异性结合可能与引物设计所用扩展青霉的菌株与本实验不同所致。以后的引物设计中,可选择菌种的合成代谢物基因来设计引物,从而设计出具有更高的特异性的引物用于qPCR检测中,能够减少假阳性结果的产生,并提高检测的准确性。
2.3 PMA-qPCR标准曲线及检出限的评价
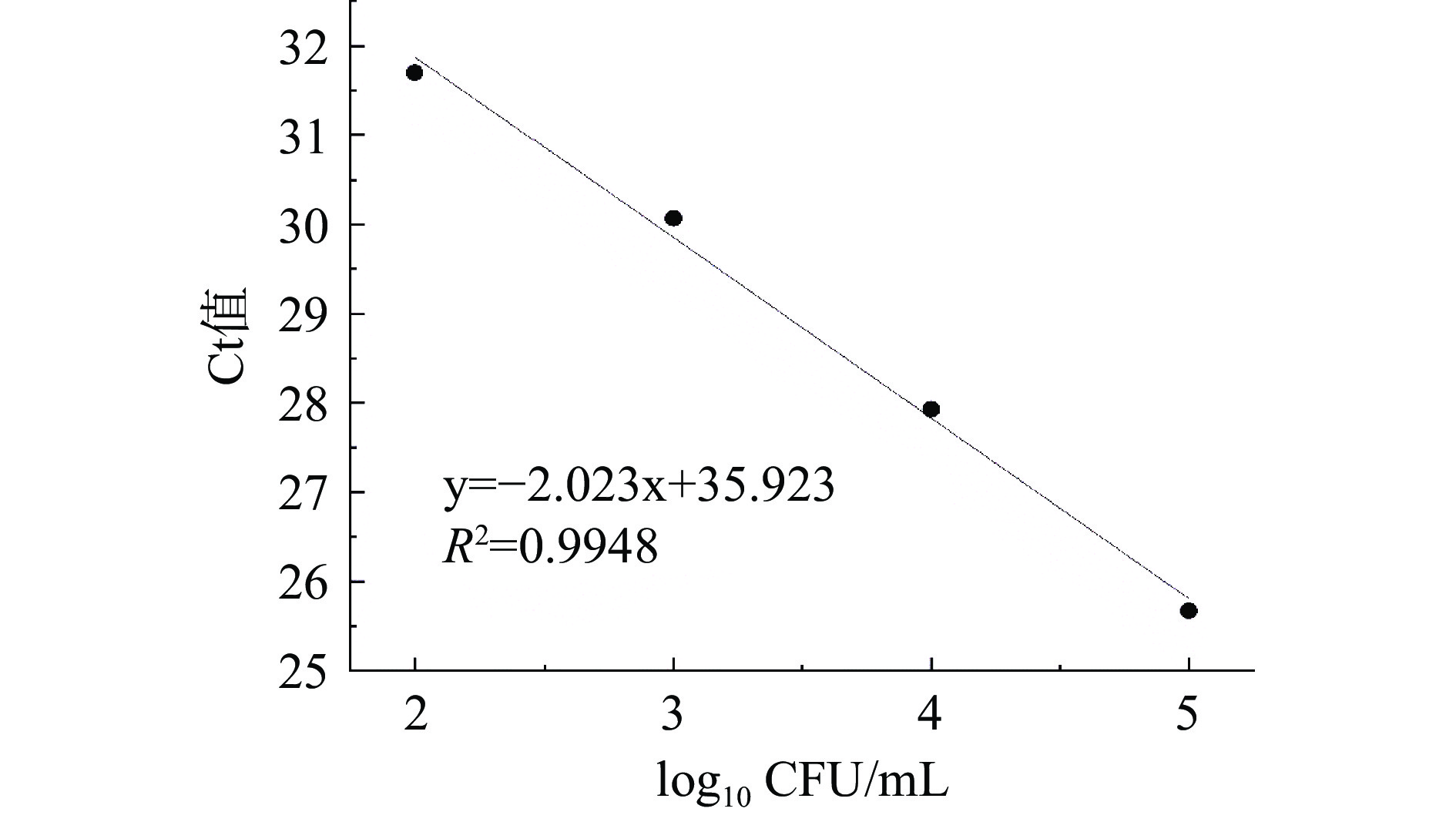
利用扩展青霉孢子悬浮液梯度稀释进行PMA-qPCR扩增,菌落浓度与Ct值的相关性如图7所示。两者之间具有良好的线性相关关系,y=−2.023x+35.925,R2=0.9948,标准曲线的重复性和稳定性良好。经计算PMA-qPCR扩增效率(E)为90%。检测限(Limit of Detection,LOD)通过公式Ct(LOD)=Ct(空白)–3计算[25],为102.6 CFU/mL。该检测限比王银环等[26]利用PMA-qPCR对地衣芽孢杆菌活菌制品中金黄色葡萄球菌最低检测限(104 CFU/mL)低,与Crespo-Sempere等[19]建立的PMA-qPCR检测人工接种番茄样品中链格孢菌检出限(102 CFU/g)类似。与Frisch等[27]优化的一种环介导的等温扩增检测扩展青霉的方法的检测下限(103 CFU/mL)相比较,本研究采用的PMA-qPCR检测扩展青霉检出限更低,具有更高的灵敏度。
2.4 人工染菌苹果样品活菌检测
对人工接种扩展青霉苹果样品进行PMA-qPCR检测,根据标准曲线计算人工染菌苹果样品中扩展青霉活菌数。结果如表4和图8所示,PMA-qPCR检测出人工污染3 d后病斑处扩展青霉活菌数已达到1.61×106 CFU/g,平板菌落计数按照国标进行计数[28]结果为1.4×106 CFU/g。距离病斑0.5、1 cm处,PMA-qPCR检测出结果及平板菌落计数法检测出活菌。这与Wei等[5]利用平板菌落计数方法发现当病斑直径为1 cm时仍可在距病斑2 cm处检测出扩展青霉活菌类似,这结果再次验证了扩展青霉菌会向未腐烂组织迁移。距离病斑2 cm处提取的DNA未检测出荧光信号,含量低于最低检测线,同时平板菌落计数法结果小于10。两者方法结果进行配对t检验,无显著差异,这一发现与Dias等[29]的结果一致,这证明该本实验方法结果可靠,可利用PMA-qPCR来检测定量扩展青霉活菌。在实际操作过程中,PMA-qPCR具有显著的优势。传统的平板菌落计数需将样品稀释液与未凝固培养基混合均匀后,凝固倒置培养。若培养基温度过高会烫死原本活的孢子,从而影响结果。除此之外,样品的破碎不均匀、环境污染都会影响平板菌落计数的结果,然而利用PMA-qPCR技术检测则会很好地避免这些影响因素。传统的平板菌落计数法需花费3~4 d进行菌落培养,而PMA-qPCR仅需花1 d时间便可得到准确结果。与平板菌落计数这一传统活菌检测手段相比PMA-qPCR检测更便捷,且具有高度敏感性和特异性[30]。
表 4 人工染菌样品 PMA-qPCR结果与平板法计数比较Table 4. Comparison of PMA-qPCR results and plate counts results of artificially infected samples距离病斑
位置(cm)Ct值 PMA-qPCR结果
(CFU/g)平板计数结果
(CFU/g)0 21.86±0.17 1.61×106±9.05×104 1.4×106±1.29×105 0.5 29.22±0.16 3.74×102±1.51×101 3.6×102±1.83×101 1 34.01±0.01 1.77±0.03 <10 2 − − <10 注:“−”为无Ct值。 3. 结论
PMA-qPCR能够弥补PCR、qPCR不能检测活菌的缺陷,同时比传统平板菌落计数方法更快捷,且具有特异性。本实验根据已发表文献筛选特异引物并优化PMA的处理条件,最终建立了以10 µg/mL PMA、黑暗孵育5 min、曝光处理10 min为最优处理条件的PMA-qPCR检测扩展青霉活菌方法,该方法具有较高的灵敏性和可靠性,能够快速检测出样品中的扩展青霉活菌数,最低检测限为102.6 CFU/mL。将其应用到人工染菌样品活菌检测,其结果与传统的平板菌落计数结果无明显差异,并且在未腐烂的苹果组织中发现扩展青霉活菌。未来将该方法应用于食品加工检测中,可进一步降低人们暴露在展青霉素的潜在危险,为扩展青霉防控提供一种新的技术支持。但本研究中样品DNA提取步骤较为繁琐,花费时间较长,后期可优化样品DNA提取方法,从而提高PMA-qPCR检测扩展青霉活菌的便捷性。
-
表 1 PCR 扩增引物序列
Table 1 PCR amplification primer sequences
表 2 PCR反应体系
Table 2 PCR reaction system
体系成分 体积(µL) 2×Taq PCR MasterMixⅡ 25 扩展青霉菌DNA 4 正引物 2 反引物 2 dd H2O 17 总体积 50 表 3 qPCR反应体系
Table 3 qPCR reaction system
体系成分 体积(µL) 2×SuperReal PreMix Plus 10 样品DNA 4 正引物 1 反引物 1 ddH2O 4 总体积 20 表 4 人工染菌样品 PMA-qPCR结果与平板法计数比较
Table 4 Comparison of PMA-qPCR results and plate counts results of artificially infected samples
距离病斑
位置(cm)Ct值 PMA-qPCR结果
(CFU/g)平板计数结果
(CFU/g)0 21.86±0.17 1.61×106±9.05×104 1.4×106±1.29×105 0.5 29.22±0.16 3.74×102±1.51×101 3.6×102±1.83×101 1 34.01±0.01 1.77±0.03 <10 2 − − <10 注:“−”为无Ct值。 -
[1] LI Y, ZHANG X, NIE J, et al. Occurrence and co-occurrence of mycotoxins in apple and apple products from China[J]. Food Control,2020,118:107354. doi: 10.1016/j.foodcont.2020.107354
[2] 李勇, 单硕, 吴丹舟, 等. 棒曲霉素的毒性及其毒性机制的研究进展[J/OL]. 食品科学: 1−12[2022-10-14]. http://kns.cnki.net/kcms/detail/11.2206.TS.20220728.1846.030.html 1−12 LI Y, SHAN S, WU D Z, et al. Research progress on toxicity and toxicity mechanism of patulin[J/OL]. Food Science: 1−12[2022-10-14]. http://kns.cnki.net/kcms/detail/11.2206.TS.20220728.1846.030.html 1−12.
[3] IMAN S I. The characteristics, occurrence, and toxicological effects of patulin[J]. Food & Chemical Toxicology An International Journal Published for the British Industrial Biological Research Association,2019,129:301−311.
[4] COTON M, BREGIER T, POIRIER E, et al. Production and migration of patulin in Penicillium expansum molded apples during cold and ambient storage[J]. International Journal of Food Microbiology,2020,313:108177.
[5] WEI D M, XU J, DONG F S, et al. Penicillium and patulin distribution in pears contaminated with Penicillium expansum. Determination of patulin in pears by UHPLCMS/MS[J]. Journal of Integrative Agriculture,2017,16(7):1645−1651. doi: 10.1016/S2095-3119(16)61543-5
[6] ATOUI A, KHOURY A E, KALLASSY M, et al. Quantification of Fusarium graminearum and Fusarium culmorum by real-time PCR system and zearalenone assessment in maize[J]. International Journal of Food Microbiology,2012,154(1-2):59−65. doi: 10.1016/j.ijfoodmicro.2011.12.022
[7] TANNOUS J, ATOUI A, KHOURY A E, et al. Development of a real-time PCR assay for Penicillium expansum quantification and patulin estimation in apples[J]. Food Microbiology,2015,50:28−37. doi: 10.1016/j.fm.2015.03.001
[8] RODRIGUEZ A, LUQUE M I, ANDRADE M J, et al. Development of real-time PCR methods to quantify patulin-producing molds in food products[J]. Food Microbiology,2011,28(6):1190−1199. doi: 10.1016/j.fm.2011.04.004
[9] 袁英哲, 韩剑, 王岩, 等. 梨火疫病菌活菌快速定量检测方法的建立[J]. 果树学报,2020,37(9):1425−1433. [YUAN Y Z, HAN J, WANG Y, et al. Establishment of rapid quantitative detection of viable Erwinia amylovora[J]. Journal of Fruit Science,2020,37(9):1425−1433. YUAN Y Z, HAN J, WANG Y, et al. Establishment of rapid quantitative detection of viable Erwinia amylovora[J]. Journal of Fruit Science, 2020, 37(9): 1425-1433.
[10] NOCKER A, CAMPER A K. Novel approaches toward preferential detection of viable cells using nucleic acid amplification techniques[J]. Fems Microbiology Letters,2009,291(2):137−142. doi: 10.1111/j.1574-6968.2008.01429.x
[11] 王帅, 徐进, 许景升, 等. PMA-qPCR定量检测青枯菌活菌方法的建立[J]. 植物保护,2018,44(6):122−128. [WANG S, XU J, XU J S, et al. Development of a PMA-qPCR method for quantitative detection of Ralstonia solanacearum[J]. Plant Protection,2018,44(6):122−128. WANG S, XU J, XU J S, et al. Development of a PMA-qPCR method for quantitative detection of Ralstonia solanacearum[J]. Plant Protection, 2018, 44(6): 122-128.
[12] 张静, 陈曦, 王彬, 等. 改良单叠氮丙啶-荧光定量PCR法的建立及其检测抗结核药物活性的价值[J]. 中国防痨杂志,2020,42(5):472−480. [ZHANG J, CHEN X, WANG B, et al. Establishment of modified propidium monoazide (PMAxx)-quantitative PCR assay and its application for identification of antituberculosis drug activity[J]. Chinese Journal of Antituberculosis,2020,42(5):472−480. ZHANG J, CHEN X, WANG B, et al. Establishment of modified propidium monoazide (PMAxx)-quantitative PCR assay and its application for identification of antituberculosis drug activity[J]. Chinese Journal of Antituberculosis, 2020, 42(5): 472-480.
[13] HU L, FU Y D, ZHANG S, et al. An assay combining droplet digital PCR with propidium monoazide treatment for the accurate detection of live cells of Vibrio vulnificus in plasma samples[J]. Frontiers in Microbiology,2022,13:927285. doi: 10.3389/fmicb.2022.927285
[14] 柯振华. 餐饮食品中6病原菌叠氮丙啶-定量聚合酶链反应检测方法开发与检验数据分析[J]. 食品安全质量检测学报,2020,11(19):6855−6861. [KE Z H. Development of detection method and test data analysis of 6 pathogenic bacteria azidopropidine-quantitative polymerase chain reaction detection method in catering food[J]. Journal of Food Safety & Quality,2020,11(19):6855−6861. KE Z H. Development of detection method and test data analysis of 6 pathogenic bacteria azidopropidine-quantitative polymerase chain reaction detection method in catering food[J]. Journal of Food Safety & Quality, 2020, 11(19): 6855-6861.
[15] TANG M L Y, LAU S C K. Strategy to evaluate changes in bacterial community profiles and bacterial pathogen load reduction after sewage disinfection[J]. Frontiers in Microbiology,2022,13:919207. doi: 10.3389/fmicb.2022.919207
[16] 曹学仁, 周益林. 基于PCR技术的植物病原菌分子定量检测技术研究进展[J]. 植物保护,2020,46(4):7−11. [CAO X R, ZHOU Y L. Research progress of quantitative detection of plant pathogens using PCR technique[J]. Plant Protection,2020,46(4):7−11. CAO X R, ZHOU Y L. Research progress of quantitative detection of plant pathogens using PCR technique[J]. Plant Protection, 2020, 46(4): 7-11.
[17] 于璇, 王卫芳, 李献锋, 等. PMA-qPCR检测十字花科黑斑病菌活菌方法的建立[J]. 植物检疫,2021,35(4):49−54. [YU X, WANG W F, LI X F, et al. PMA-qPCR assay for the detection of viable Pseudomonas syringae pv.maculicola[J]. Plant Quarantine,2021,35(4):49−54. YU X, WANG W F, LI X F, et al. PMA-qPCR assay for the detection of viable Pseudomonas syringae pv. maculicola[J]. Plant Quarantine, 2021, 35(4): 49-54.
[18] 张丹丹, 许晓丽, 李健强, 等. PMA-qPCR检测两种丁香假单胞菌活性研究[C]//成都: 中国植物病理学会2019年学术年会论文集, 2019: 379 ZHANG D D, XU X L, LI J Q, et al. PMA-qPCR detection of two species of Pseudomonas syringae activity[C]//Chengdu: Proceedings of the 2019 Annual Conference of the Chinese Society of Plant Pathology, 2019: 379.
[19] CRESPO-SEMPERE A, ESTIARTE N, MARIN S, et al. Propidium monoazide combined with real-time quantitative PCR to quantify viable Alternaria spp. contamination in tomato products[J]. International Journal of Food Microbiology,2013,165(3):214−220. doi: 10.1016/j.ijfoodmicro.2013.05.017
[20] 樊明涛, 毕静莹, 刘邻渭, 等. 浓缩苹果汁中扩展青霉菌实时PCR快速检测条件的优化[J]. 西北农林科技大学学报(自然科学版),2007(11):84−89. [FAN M T, BI J Y, LIU L W, et al. Condition optimization of real-time PCR used in rapid detection of Penicillium expansum in apple juice concentrate[J]. Journal of Northwest A&F University (Natural Science Edition),2007(11):84−89. FAN M T, BI J Y, LIU L W, et al. Condition optimization of real-time PCR used in rapid detection of Penicillium expansum in apple juice concentrate[J] Journal of Northwest A & F University (Natural Science Edition), 2007(11): 84-89.
[21] 赵利娜, 李慧芳, 余江, 等. 扩展青霉侵染柑橘机制分析(英文)[J]. 食品科学,2019,40(16):97−106. [ZHAO L N, LI H F, YU J, et al. A mechanistic study of citrus infection by Penicillium expansum[J]. Food Science,2019,40(16):97−106. ZHAO L N, LI H F, YU J, et al. A mechanistic study of citrus infection by Penicillium expansum[J]. Food Science, 2019, 40(16): 97-106.
[22] 何鸿举, 焦凌霞, 樊明涛, 等. 应用PCR技术快速检测腐烂苹果中扩展青霉菌[J]. 食品科学,2011,32(12):183−187. [HE H J, JIAO L X, FAN M T, et al. Rapid detection of Penicillium expansum in rotten apples by polymerase chain reaction[J]. Food Science,2011,32(12):183−187. HE H J, JIAO L X, FAN M T, et al. Rapid detection of Penicillium expansum in rotten apples by polymerase chain reaction[J]. Food Science, 2011, 32(12): 183-187.
[23] 尉冬梅, 徐军, 董丰收, 等. 大久保桃扩展青霉病斑外延组织中青霉菌及展青霉素的扩散范围检测[J]. 农产品质量与安全,2016(4):35−39. [WEI D M, XU J, DONG F S, et al. Detection of the spread range of Penicillium and patulin in the epitaxy tissue of Okubo peach extended Penicillium[J]. Quality and Safety of Agro-Products,2016(4):35−39. WEI D M, XU J, DONG F S, et al. Detection of the spread range of Penicillium and Patulin in the epitaxy tissue of Okubo peach extended Penicillium[J]. Quality and Safety of Agro-Products, 2016, (4): 35-39.
[24] 王艳玲, 郭小洁, 张紊玮, 等. 棒曲霉素生物合成及分子调控研究进展[J]. 食品科学,2020,41(17):267−274. [WANG Y L, GUO X J, ZHANG W W, et al. Recent advances in patulin biosynthesis and its molecular regulation[J]. Food Science,2020,41(17):267−274. WANG Y L, GUO X J, ZHANG W W, et al. Recent advances in patulin biosynthesis and its molecular regulation[J]. Food Science, 2020, 41(17): 267-274.
[25] LÜ X C, LI Y, QIU W W, et al. Development of propidium monoazide combined with real-time quantitative PCR (PMA-qPCR) assays to quantify viable dominant microorganisms responsible for the traditional brewing of Hong Qu glutinous rice wine[J]. Food Control,2016,66:69−78. doi: 10.1016/j.foodcont.2016.01.040
[26] 王银环, 郑小玲, 阮昊, 等. 叠氮溴化丙锭与实时荧光定量PCR结合检测地衣芽孢杆菌活菌制品中金黄色葡萄球菌[J]. 中国药学杂志,2022,57(3):231−236. [WANG Y H, ZHEN X L, RUAN H, et al. Detection ofStaphylococcus aureus inBacillus licheniformis live products by PMA-qPCR[J]. Chinese Pharmaceutical Journal,2022,57(3):231−236. WANG Y H, ZHEN X L, RUAN H, et al. Detection of Staphylococcus aureus in Bacillus licheniformis live products by PMA-qPCR[J]. Chinese Pharmaceutical Journal, 2022, 57(03): 231-236.
[27] FRISCH L M, MANN M A, MAREK D N, et al. Development and optimization of a loop-mediated isothermal amplification (LAMP) assay for the species-specific detection of Penicillium expansum[J]. Food Microbiology,2021,95:103681. doi: 10.1016/j.fm.2020.103681
[28] 中华人民共和国卫生部. GB 4789.15-2010食品安全国家标准 食品微生物学检验 霉菌和酵母计数[S]. 北京: 中国标准出版社, 2012: 151−158. Ministry of Health of the Ministry of Health of the People's Republic of China. GB 4789.15-2010 National Food Safety Standard for Microbiological Examination of Food-Count of mold and yeast[S]. Beijing: China Standards Publishing House, 2012: 151−158.
[29] DIAS C O, SCARIOT M C, AMBONI R, et al. Application of propidium monoazide coupled with quantitative PCR to evaluate cell viability of Bifidobacterium animalis subsp. lactis in a non-dairy probiotic beverage[J]. Annals of Microbiology,2020,70:22. doi: 10.1186/s13213-020-01566-9
[30] BARRETTA C, VERRUCK S, MARAN B M, et al. Listeria monocytogenes survival in raw Atlantic salmon (Salmo salar) fillet under in vitro simulated gastrointestinal conditions by culture, qPCR and PMA-qPCR detection methods[J]. LWT-Food Science and Technology,2019,107:132−137. doi: 10.1016/j.lwt.2019.03.015
-
期刊类型引用(22)
1. 熊波,杨丽涓,尹涛,普娅丽. QuEChERS净化-气相色谱-串联质谱法测定玫瑰花中24种农药残留. 食品安全导刊. 2025(02): 56-60 .  百度学术
百度学术
2. 邓涵玉,鲁莎莎,陈非凡,毛涛,邓祥宜,李继伟. 白萝卜精酿啤酒酿造工艺优化及品质分析. 中国酿造. 2025(02): 231-238 .  百度学术
百度学术
3. 和丽媛. 蓝莓-玫瑰复合饮料的研制. 轻工科技. 2024(01): 1-3+32 .  百度学术
百度学术
4. 丁献华,周红琴,马雪玲. 芳香植物缓解大学生焦虑情绪的比较研究. 现代园艺. 2024(06): 13-15 .  百度学术
百度学术
5. 刘晓雨,李雪松,单艳琴,张博雅,李景明. PDMS膜渗透浓缩对天然玫瑰冻干露香气的影响. 食品科学. 2024(05): 158-165 .  百度学术
百度学术
6. 欧阳雪灵,江新凤,刘书玲,王国行,彭玉辅,彭勇,彭火辉,陈紫梅,杨雪珍. 基于顶空固相微萃取-气相色谱-质谱法和相对气味活性值分析不同发酵时间玫瑰花茶香气的差异. 食品安全质量检测学报. 2024(10): 243-250 .  百度学术
百度学术
7. 和丽媛,李安佳,玉叫勐,钱星蓉. 藜麦玫瑰面包研制. 粮食科技与经济. 2024(02): 109-112 .  百度学术
百度学术
8. 高飞,尹思雨,皮汨源,朱月星. 大马士革玫瑰纯露发酵液制备工艺及功效评价. 广东化工. 2024(16): 49-51+27 .  百度学术
百度学术
9. 李芬,夏瑞萍,王智慧,王玮,李梅. 不同花源窨制红茶研究进展. 蚕桑茶叶通讯. 2024(05): 17-19 .  百度学术
百度学术
10. 丁献华,段丽君,乔晓丽,万丽,肖敏. 芳香植物玫瑰缓解高职学生焦虑情绪的研究. 现代园艺. 2023(03): 175-177+180 .  百度学术
百度学术
11. 赵珊,杨飞洋,秦琳,李曦,黄世群,郑幸果,雷欣宇,仲伶俐. 代用茶茶汤中功能性成分组成及含量分析. 食品研究与开发. 2023(02): 162-168 .  百度学术
百度学术
12. 代丽凤,蒋洁琳,官兴丽,邹小林,杨智,符宗林,罗赛. 普洱茶玫瑰花葡萄籽复合饮料的研制及其抗氧化活性评价. 茶叶学报. 2023(02): 1-13 .  百度学术
百度学术
13. 和丽媛,杨志龙,樊丹敏. 黑松露玫瑰酥性饼干的研制. 现代食品. 2022(02): 81-84 .  百度学术
百度学术
14. 黄一承,史玉,马艳丽,丁云峰,马琳,李丹,李晓磊. 玫瑰、百合煎饼风味成分比较研究. 食品工业科技. 2022(05): 302-309 .  本站查看
本站查看
15. 姜苗,黄晓琴,伊冉,曹美琪,丁凯华,代智慧,刘红,焦春梅,扈兴强,王庆卫,石磊. 玫瑰花红茶抗氧化性研究. 山东林业科技. 2022(05): 39-47 .  百度学术
百度学术
16. 姜苗,黄晓琴,伊冉,曹美琪. 不同加工工艺对玫瑰花红茶品质的影响. 中国茶叶. 2022(12): 34-40 .  百度学术
百度学术
17. 杨丽,柯南,龚秋霞,刘雯雯,魏琴. 响应面法优化玫瑰风味酸奶工艺. 宜宾学院学报. 2022(12): 56-61+84 .  百度学术
百度学术
18. 郭香香,胥鑫萌,张恺欣,宋锦博,刘军海. 几种植物提取物在抗衰老化妆品中的应用. 中国洗涤用品工业. 2021(04): 94-97 .  百度学术
百度学术
19. 孙瑶,朱雅婧,李武,赵付安. 玫瑰花冠茶总黄酮提取的工艺优化. 农产品加工. 2021(14): 31-34 .  百度学术
百度学术
20. 丁强,董纪超,荚启安,吴泽宇,张文成. 响应面法优化固定超声辅助提取玫瑰花黄酮的工艺. 安徽化工. 2021(05): 37-40 .  百度学术
百度学术
21. 青舒婷,杨丰,张海仑,李宇龙,许军,岳进. 远红外辅助热泵干燥食用玫瑰花瓣及产品品质分析. 食品工业科技. 2021(22): 246-253 .  本站查看
本站查看
22. 熊海燕,林敏,鲁萌. 蟠桃玫瑰米露酒加工工艺研究. 食品安全导刊. 2021(31): 114-116 .  百度学术
百度学术
其他类型引用(1)





 下载:
下载:


 下载:
下载:



