Optimization of Fermentation Conditions, Enzymatic Properties and Degradation Products of Agarase Produced by Marine Bacterium Sphingomonas sp. Q2
-
摘要: 本研究旨在对从江蓠中筛选获得的Sphingomonas sp. Q2菌株产琼胶酶能力条件进行优化并对其酶学性质及降解产物进行研究。通过响应面法对发酵条件进行优化,采用硫酸铵沉淀、离子交换层析和凝胶层析等方法对发酵所得酶液进行纯化并对纯化后的酶液进行酶学性质研究。发酵优化结果表明该菌株产琼胶酶的最佳培养基组成为:琼脂4.42 g/L、磷酸氢二钾1.30 g/L、氯化钠10.51 g/L。优化后的酶活力为1085.71 U/mL,较优化前提高了1.58倍。纯化后的琼胶酶比活为112048.82 U/mg,纯化倍数为7倍,回收率为48.04%。酶学性质结果表明该酶最适反应温度为40 ℃,最适pH为6.5,且在最适温度下保存8 h,酶活仍保持在90%以上。MS和13C-NMR结果表明,该琼胶酶的降解产物主要为新琼四糖。该琼胶酶具有良好的热稳定性及较高的酶活力,为琼胶寡糖的开发制备提供了基础。Abstract: This study was aimed at optimization of the fermentation conditions for agarase production by Sphingomonas sp.Q2 which was isolated from Gracilaria and characterization of its enzymatic properties and degradation products. The optimum fermentation conditions were determined by response surface analysis. The agarase was purified by (NH4)2SO4 precipitation and column separation and its enzymatic properties was investigated. The results indicated that the optimal culture medium components for agarase production were agar 4.42 g/L, K2HPO4 1.30 g/L, NaCl 10.51 g/L. The highest enzyme activity of 1085.71 U/mL was obtained ,which was 1.58 times compared with the initial activity. The crude enzyme was purified 7 times with 112048.82 U/mg of enzyme activity. The recovery rate of agarase was 48.04%. The enzyme had optimal temperature and pH of 40 ℃ and 6.5, respectively. The enzyme activity remained above 90% when stored at the optimum temperature for 8 h. The capital hydrolysates were identified as neoagarotetraose by MS and 13C-NMR. The agarase showed a good thermal stability and high stability, which laid the foundation for the development and application of functional oligosaccharides agar.
-
琼胶酶是一类可以催化琼胶降解形成琼胶寡糖的糖苷水解酶,分为α-琼胶酶和β-琼胶酶两类[1],α-琼胶酶作用于琼脂糖的α-1,3糖苷键,产生以3,6-内醚-α-L-半乳糖为还原端的琼寡糖(Agarooligosaccharides,AOS);β-琼胶酶作用于β-1,4糖苷键,产生以β-D-半乳糖为还原端的新琼寡糖(Neoagarooligosaccharides,NAOS)[2]。在食品医药化工领域琼胶酶可高效降解琼胶制备具有抑制淀粉老化[3]、防腐[4]、抗氧化[5]、保湿[6]、美白[7]、抗炎[8]、益生元[9]多种生理活性的琼胶寡糖。此外,琼胶酶还可用于分离海藻单细胞和制备海藻原生质体,以及从琼胶糖中回收DNA和RNA[10]等。
琼胶酶产生菌主要来源于海洋环境,目前报道的琼胶酶产生菌主要包括:假单胞菌属(Pseudomonas)[11]、弧菌属(Vibrio)[12]、链霉菌属(Streptomyces)[13]、交替单胞菌属(Alteromonas)[14]、不动杆菌属(Acinetobacter)[15]等。由于海洋环境的特殊性,菌株在产琼胶酶时存在酶活力不高且产酶条件不稳定等问题[10],限制琼胶酶开发应用。因此,筛选获得新型产酶菌株并通过发酵优化改良其产酶能力,对于琼胶酶研究应用具有重要意义。由于琼胶降解菌大多是海洋菌,所产琼胶酶的最适pH多偏弱碱性且耐热性较差,不利于高温下酶促反应的进行。目前对产琼胶酶菌株的研究多集中在假单胞菌、弧菌等菌株上,而对鞘氨醇杆菌的研究较少且缺乏对其酶学性质及降解产物的研究,限制了鞘氨醇杆菌在产琼胶酶研究中的进一步应用。
随着人们对健康生活需求的提升,酶法水解琼脂得到的琼胶寡糖需求量不断增长,其广泛应用于食品、药品、保健品中,因此,开发一种产酶能力强且稳定性高的新型琼胶酶尤为重要。本文从江蓠中筛选获得一株新型产琼胶酶鞘氨醇菌株Sphingomonas sp. Q2,对Sphingomonas sp. Q2菌株发酵产琼胶酶的培养基组成进行优化,提高其产酶能力。通过硫酸铵沉淀、离子交换层析和凝胶层析技术对Sphingomonas sp. Q2琼胶酶进行纯化,并对其酶学性质及降解产物进行研究,为琼胶寡糖的高效制备提供理论基础。
1. 材料与方法
1.1 材料与仪器
鞘氨醇杆菌Sphingomonas sp. Q2 本实验室分离保存;种子培养基(w/v) 琼脂2 g,氯化铵1 g,磷酸氢二钾1 g,硫酸镁0.5 g,氯化钠15 g,蒸馏水1000 mL,pH7.0;发酵基础培养基同种子培养基;琼脂、NaCl、K2HPO4、(NH4)2SO4、NH4Cl、十二烷基硫酸钠等 分析纯,国药集团化学试剂有限公司。
ZWY-2102C型全温摇床 上海智城分析仪器制造有限公司;CF1524R型台式高速冷冻离心机 美国赛洛捷克公司;HWS24型电热恒温水浴锅 上海一恒科学仪器有限公司;UV-6000PC紫外可见分光光度计 上海元析仪器有限公司。 1.2 实验方法
1.2.1 琼胶酶活力测定
采用3,5-二硝基水杨酸法测定酶活力[16]。操作步骤如下:用0.05 mol/L、pH7.0的柠檬酸缓冲液配制浓度为0.2%的琼胶溶液,向1.9 mL琼胶溶液中加入0.1 mL酶液,40 ℃反应30 min,加入2 mL DNS,沸水浴中加热5 min显色,迅速冷却,用蒸馏水定容至25 mL并摇匀,于520 nm处测定吸光值,用灭活酶液作空白对照。以D-半乳糖绘制标准曲线计算酶活力。
酶活力单位(U)定义为在实验条件下,每分钟催化底物产生1 μg还原糖所需的酶量。发酵液酶活力单位定义为每毫升发酵液含酶活单位(U/mL)。
以Bradford法[17]测定蛋白含量,采用牛血清白蛋白为标准蛋白。
1.2.2 发酵培养优化
前期Plackett-Burman试验与最陡爬坡实验结果表明琼脂、磷酸氢二钾、氯化钠对酶活力影响较大(另文发表),因此通过Box-Behnken实验设计三因素三水平实验(表1),以琼脂、磷酸氢二钾、氯化钠为影响因素,琼胶酶活力为响应值,建立响应面回归模型,确定最优培养基及各组分的交互作用。
表 1 Box-Behnken试验设计因素与水平Table 1. Experimental design factors and level of Box-Behnken编码 因素 水平 −1 0 1 A 琼脂(g/L) 3 4 5 B 磷酸氢二钾(g/L) 0.5 1 1.5 C 氯化钠(g/L) 10 15 20 1.2.3 琼胶酶的分离纯化
1.2.3.1 硫酸铵分级沉淀
发酵上清液加入硫酸铵至0%~90%饱和度,4 ℃静置过夜,8000 r/min冷冻离心20 min,收集沉淀及上清液,测定不同饱和度下的琼胶酶活力和蛋白质含量,由此确定硫酸铵沉淀的最佳饱和度。
1.2.3.2 QFF阴离子交换层析
将所得粗酶液进行透析脱盐并经0.22 μm滤膜过滤后上样于QFF阴离子交换柱,用0~1 mol/L NaCl溶液梯度洗脱,洗脱液经紫外吸收检测并进行收集(2 mL/管)。检测波长为280 nm,测定有紫外吸收峰试管中的洗脱液酶活力。
1.2.3.3 Sephdex G75凝胶柱层析
将1.2.3.2中得到的酶液上样于Sephdex G75凝胶柱,用pH7.0 Tris-HCl缓冲液进行洗脱,流速控制为0.2 mL/min,洗脱液经紫外吸收检测并进行收集(2 mL/管),测定有紫外吸收峰试管中的洗脱液酶活力。
1.2.3.4 SDS-PAGE
SDS-PAGE凝胶电泳由12%的分离胶和8%的浓缩胶组成,蛋白质的染色与脱色均采用常规考马斯亮蓝染色法和脱色技术[18]。
1.2.4 酶学性质
1.2.4.1 酶的最适温度及温度稳定性
分别在25、30、35、40、45、50、55、60 ℃温度下测定酶活力,以最高酶活力为100%,考察酶的最适反应温度。将酶液分别在不同温度下保温8 h,测定其对应的剩余酶活力,以未处理的酶活力为100%,考察酶的温度稳定性。
1.2.4.2 酶的最适pH及pH稳定性
分别用50 mmol/L pH为3.0~5.0的柠檬酸盐缓冲液,50 mmol/L pH为6.0~8.0的Na2HPO4-NaH2PO4缓冲液和50 mmol/L pH为9.0~10.0的甘氨酸-NaOH缓冲液,将酶液分别置于以上不同pH的底物缓冲液中测定琼胶酶的活力,以最高酶活为100%,研究酶的最适pH。将酶液在最适pH下40 ℃放置6 h,测定酶的剩余活力,以未处理的酶活力为100%,研究酶的pH稳定性。
1.2.4.3 不同金属离子对酶活力的影响
向酶液中分别加入终浓度为50 mmol/L的不同金属离子,室温下放置1 h,测定剩余酶活力,以未添加金属离子的酶活力为100%,考察金属离子对酶活力的影响。
1.2.4.4 动力学参数测定
将酶液与不同浓度的琼脂作用,测定酶反应的初速度,用Lineweaver-Burk双倒数作图法求出以琼脂为底物的酶的Km值。
1.2.5 降解产物分析
将纯化后的酶液添加到含0.2%琼脂糖的50 mmol/L酸盐缓冲液(pH7),在40 ℃下,反应2 h,沸水浴10 min终止反应,8000 r/min冷冻离心10 min,收集上清液在安捷伦6460三重四极质谱仪上进行负离子电喷雾电离质谱(ESI-MS)分析,进样量10 μL,流动相为乙腈:水(1:1)。用重水处理降解样品并反复冷冻干燥三次,样品最终用重水溶解,利用安捷伦DD2-500光谱仪记录13C-NMR光谱。
1.3 数据处理
每个实验设置3组重复,且实验数据为3次平行试验的平均值。使用Origin 2021及Expert 8.0.6软件对实验数据及图片进行分析处理。
2. 结果与分析
2.1 发酵培养条件优化
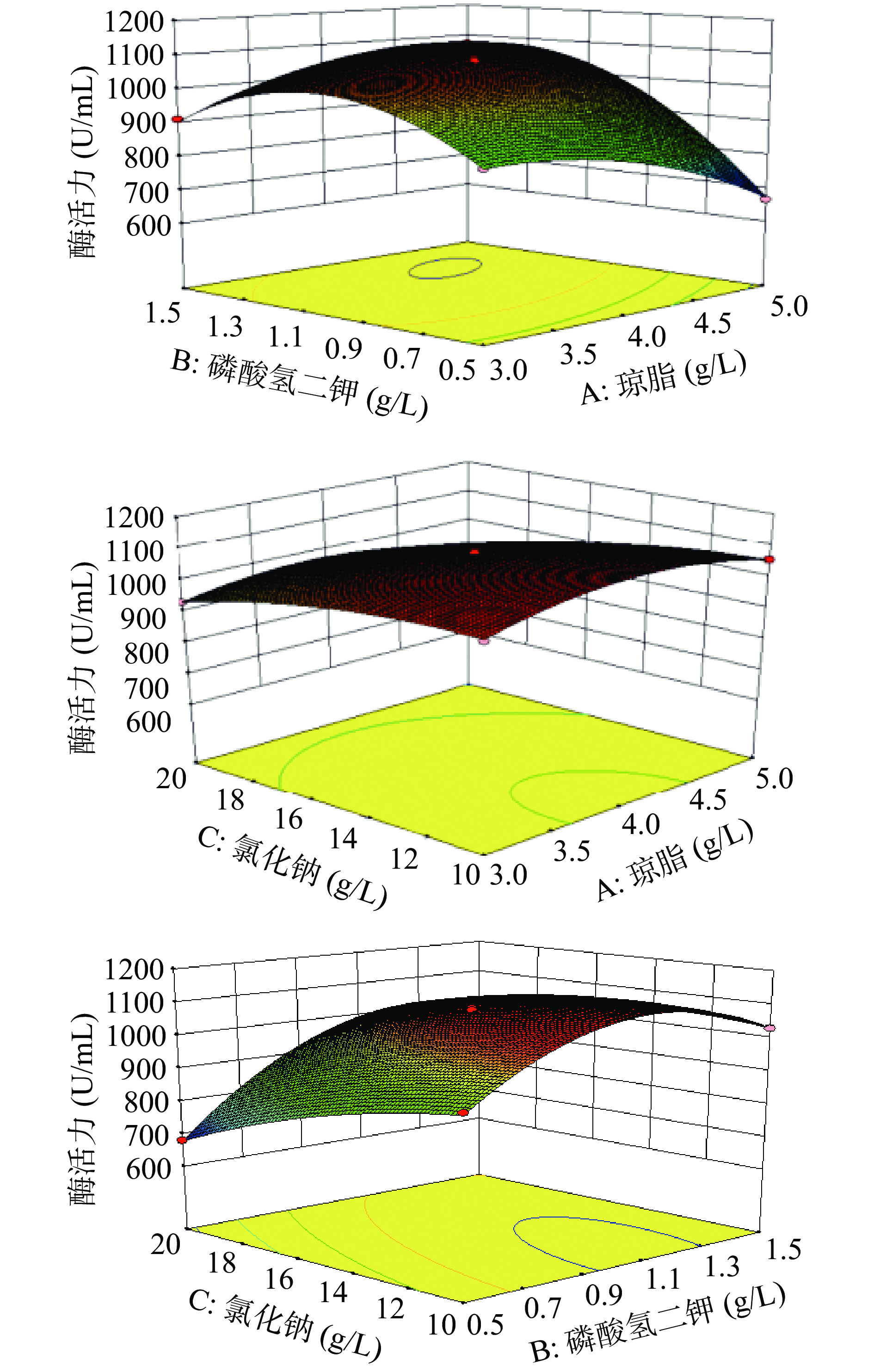
采用Box-Behnken试验设计响应面试验,以琼胶酶活力为响应值,结果见表2。用Design Expert 8.0.6软件对表2中数据进行多元回归拟合,得到的回归方程为Y=1078.94−7.91A+107.85B−68.13C+91.58AB−11.03AC+50.57BC−61.44A2−138.12B2−35.51C2,并对回归模型进行方差分析,结果见表3。由表3可知,回归模型的P<0.0001,极显著;失拟项P>0.05,不显著,说明模型可靠,决定系数R2=0.9988,校正决定系数R2adj=0.9973,CV%=0.72%。通过方程可以看出琼胶酶活性与3个因素呈线性关系,该模型拟合性较好,可以用该模型来分析和预测琼脂粉、磷酸氢二钾、氯化钠对菌株产琼胶酶活力的影响。
表 2 Box-Behnken试验设计与结果Table 2. Design and results of Box-Behnken tests实验号 A琼脂 B磷酸氢二钾 C氯化钠 酶活(U/mL) 1 −1 −1 0 870.19 2 1 −1 0 664.96 3 −1 1 0 910.64 4 1 1 0 1071.73 5 −1 0 −1 1041.57 6 1 0 −1 1054.08 7 −1 0 1 931.97 8 1 0 1 900.34 9 0 −1 −1 922.41 10 0 1 −1 1029.07 11 0 −1 1 680.41 12 0 1 1 989.35 13 0 0 0 1079.09 14 0 0 0 1077.62 15 0 0 0 1085.71 16 0 0 0 1072.47 17 0 0 0 1079.83 表 3 回归模型的方差分析Table 3. Variance analysis of regression model方差来源 平方和 自由度 均方 F值 P值 显著性 模型 25043.60 9 2782.62 658.78 <0.0001 ** A 44.04 1 44.04 10.43 0.0145 * B 8194.12 1 8194.12 1939.93 <0.0001 ** C 3269.95 1 3269.95 774.15 <0.0001 ** AB 2953.93 1 2953.93 699.33 <0.0001 ** AC 42.88 1 42.88 10.15 0.0154 * BC 900.74 1 900.74 213.25 <0.0001 ** A2 1399.49 1 1399.49 331.33 <0.0001 ** B2 7073.05 1 7073.05 1674.52 <0.0001 ** C2 467.51 1 467.51 110.68 <0.0001 ** 残差 29.57 7 4.22 失拟项 21.62 3 7.21 3.63 0.1226 纯误差 7.95 4 1.99 总和 25073.16 16 注:“*”表示对结果影响显著(P<0.05);“**”表示对结果影响极显著(P<0.01)。 利用软件Design Expert 8.0.6根据二次回归方程作出响应面及等高线如图1所示。图中响应面坡度越陡峭表示该因素对菌株产酶活力影响越大。等高线形状可以直观反映交互作用的强弱,椭圆形为交互作用显著,圆形则为不显著,由图1和表3分析可知,琼脂和磷酸氢二钾、磷酸氢二钾和氯化钠对酶活力影响的P<0.0001,说明交互作用极显著,琼脂与氯化钠的P<0.05,交互作用较显著。当琼脂浓度为4 g/L时,琼胶酶活力随着磷酸氢二钾浓度的增加而升高,当磷酸氢二钾浓度达到1.30 g/L时,琼胶酶活力升高至最优值,随着两个变量增加,酶活力呈先上升后下降趋势,而琼脂对菌株产酶活力影响较小。
通过上述二次多项回归模型,利用软件Design-Expert 8.0.6优化培养基组成,获得最优发酵培养基:琼脂4.42 g/L、氯化钠10.51 g/L、磷酸氢二钾1.30 g/L。此最优发酵培养基中的酶活力预测值为1107.40 U/mL,对上述条件进行验证实验,最终得到的琼胶酶活力为1085.71 U/mL,与预测值接近,说明优化后的回归方程对预测菌株的产酶活力具有可靠性。与目前已报道的产琼胶酶海洋细菌多食鞘氨醇杆菌(酶活力18.40 U/mL)[19]和弧菌(酶活力51.60 U/mL)[12]相比,本研究菌株Sphingomonas sp.Q2产琼胶酶活力较高。
2.2 琼胶酶的分离纯化
2.2.1 硫酸铵沉淀结果
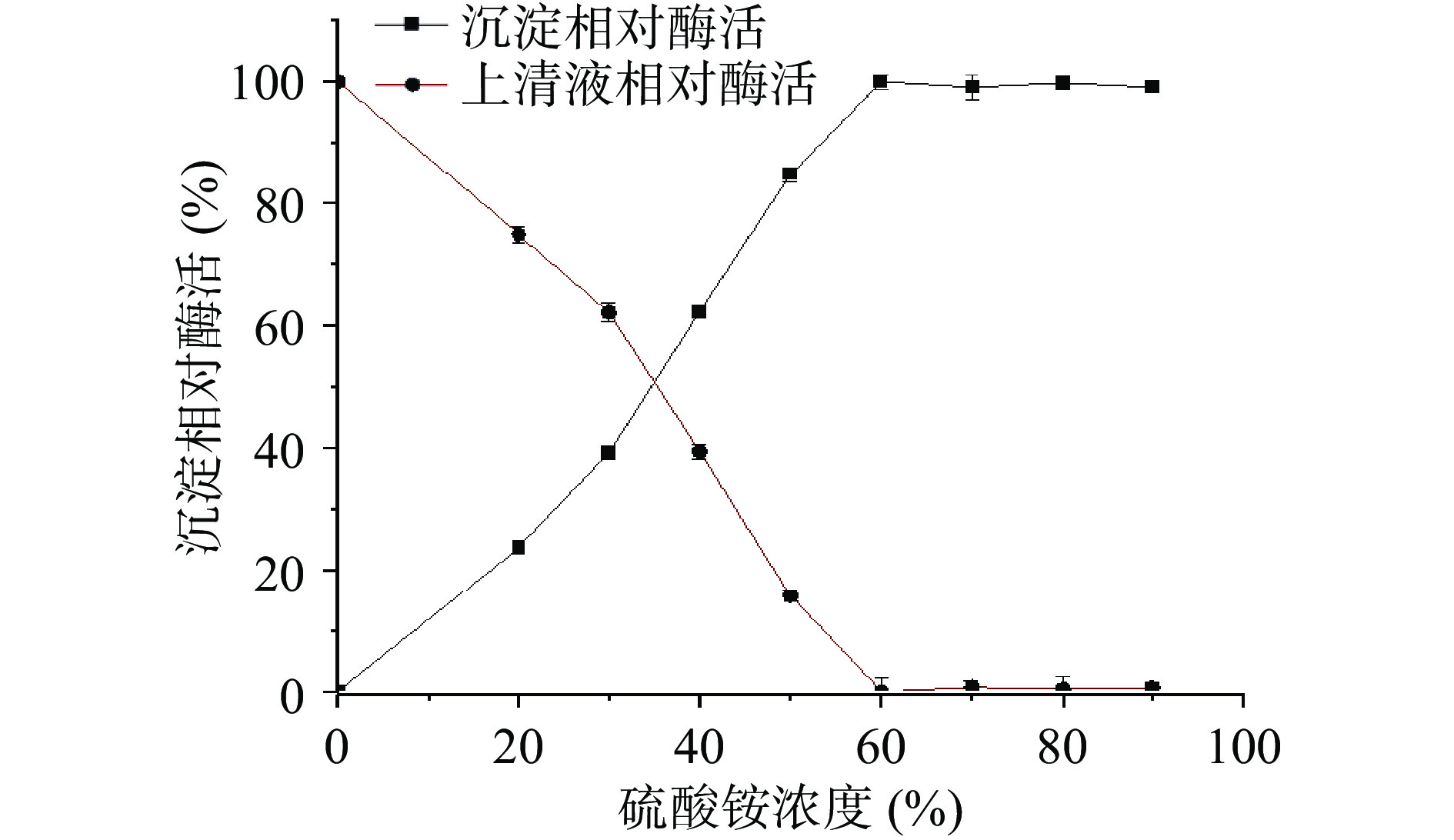
硫酸铵沉淀法可从粗酶液中浓缩和纯化蛋白质,且不易使蛋白质变性[1]。不同硫酸铵饱和度下沉淀物及上清液中的相对酶活如图2所示。结果显示,随着硫酸铵饱和度的增加,沉淀物的酶活逐渐增加。当硫酸铵饱和度为60%时沉淀中的酶活力接近最大值,且上清液中几乎检测不到酶活力,故在后续步骤中,选择饱和度为60%的硫酸铵进行琼胶酶粗分离。
2.2.2 柱层析纯化
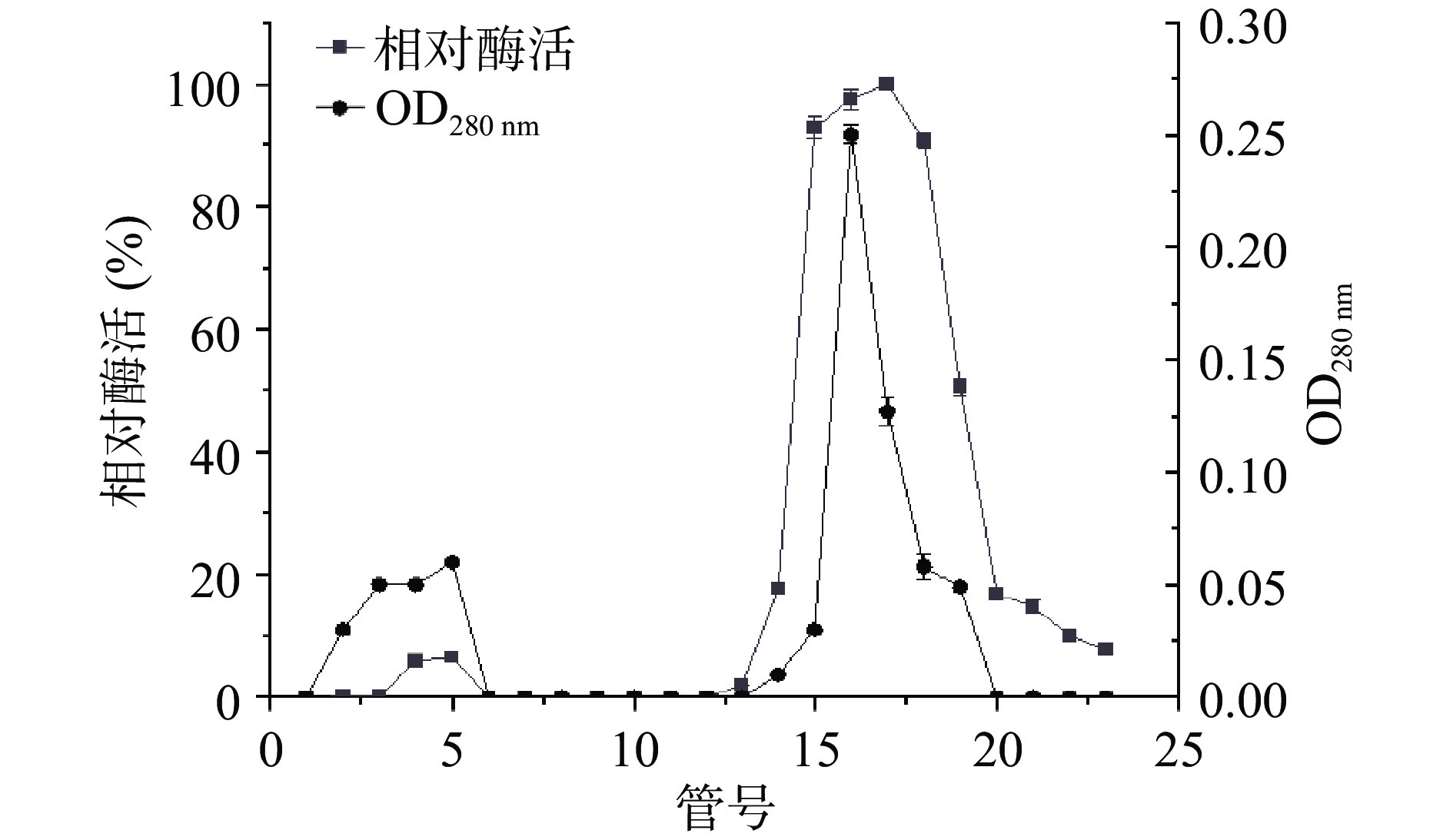
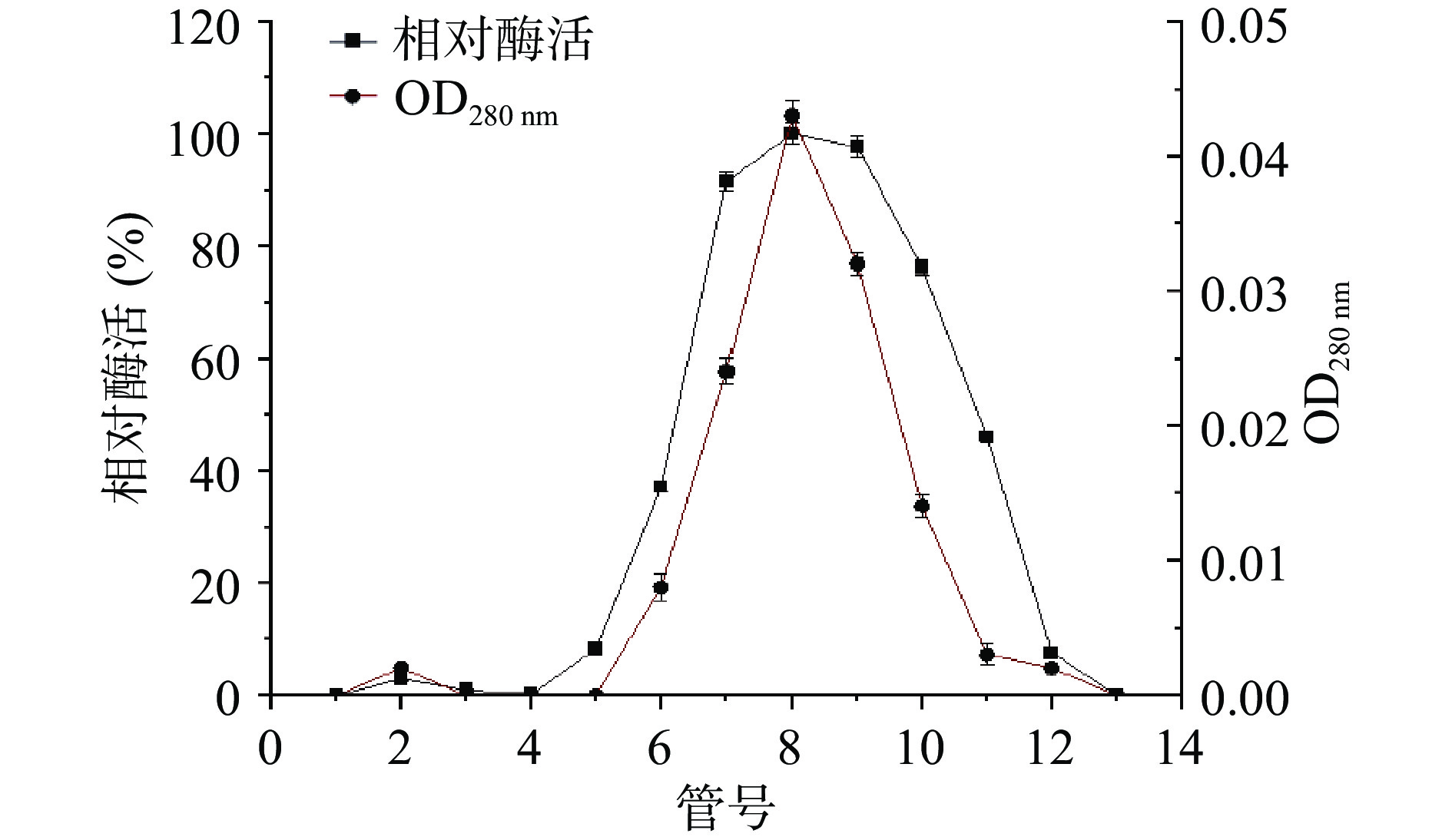
洗脱液紫外吸收及酶活力测定结果如图3所示,洗脱梯度为:0~14 min,0% B;14~34 min,0~100% B;34~45 min,100% B。对两个洗脱峰的酶活力进行测定,结果显示主要酶活力集中在第二个峰中,因此,收集第二个峰的洗脱液进行透析除盐并进一步用凝胶层析柱进行纯化,结果如图4所示,在280 nm条件下检测到一个峰,经酶活检测结果表明该峰为活性集中部分,因此,收集洗脱液进行纯度和分子量检测。
2.2.3 琼胶酶纯化结果分析
琼胶酶的分离纯化结果如表4所示,经硫酸铵沉淀、QFF阴离子交换层析、Sephdex G75凝胶柱层析后,琼胶酶的比活为112048.82 U/mg,纯化倍数为7倍,回收率为48.04%,较李驰等[20]的纯化倍数与回收率明显提高。这是由于在离子交换层析时,蛋白质的分配系数对离子强度非常敏感,本研究在纯化过程中通过改变缓冲液的离子强度进行分离纯化,损失蛋白较少,并进一步通过排阻层析提高纯化倍数。
表 4 琼胶酶分离纯化结果Table 4. Summary of isolation and purification procedures of agarase纯化步骤 总酶活(U) 总蛋白(mg) 比活(U/mg) 纯化倍数 回收率(%) 发酵液 105164 6.57 16006.70 1 100 硫酸铵沉淀 86850.29 1.67 52006.16 3.25 82.59 QFF阴离子交换层析 53181.91 0.60 88459.59 5.53 50.57 Sephdex G75凝胶柱层析 50522.81 0.45 112048.82 7.00 48.04 2.2.4 SDS-PAGE纯度鉴定及相对分子量测定
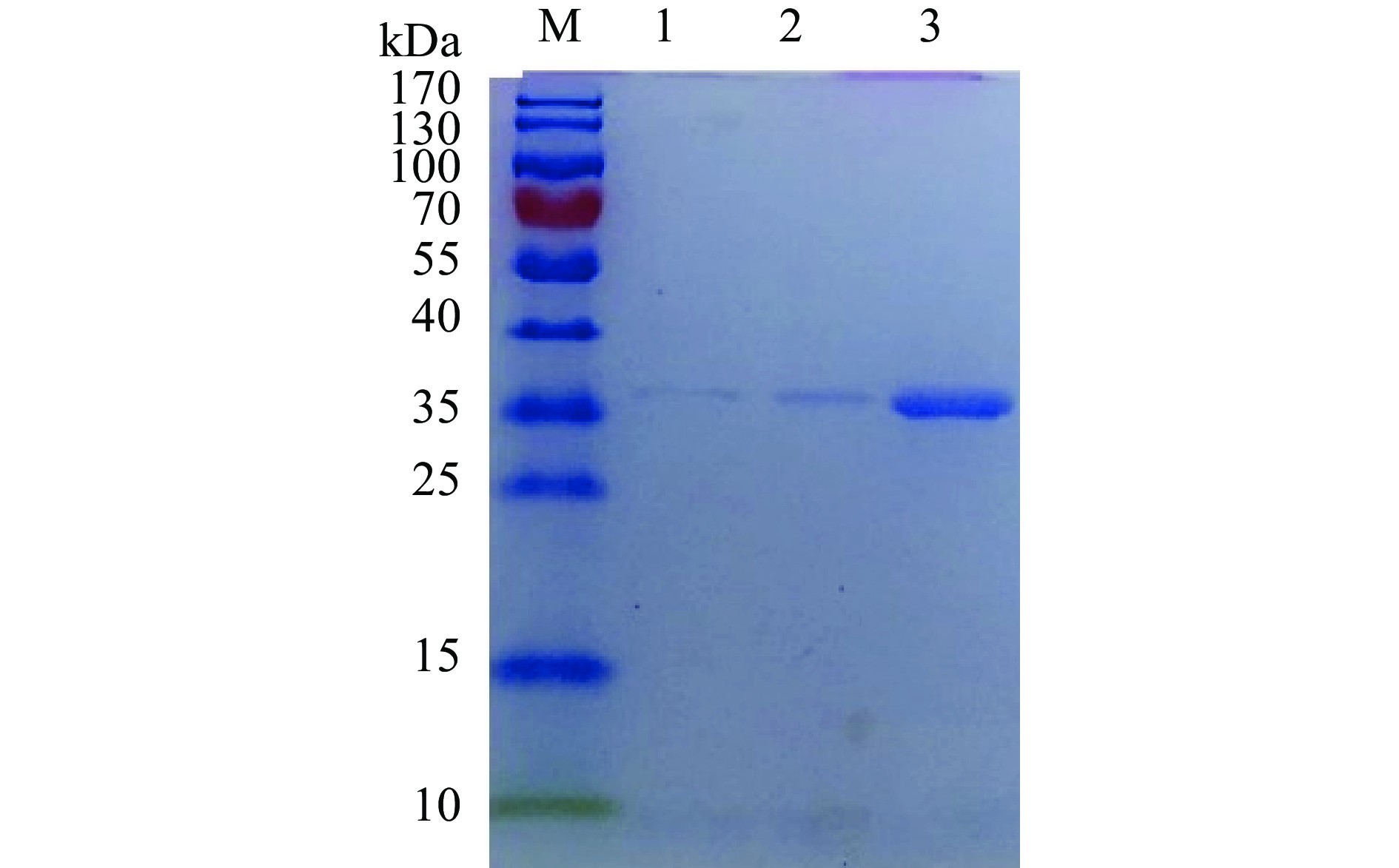
由图5所示,该琼胶酶在SDS-PAGE图谱上呈现单一条带,表明分离效果较好并且经过以上纯化步骤可以得到电泳纯酶,该酶分子量为35 kDa,与Li等[21]的结果相近。
2.3 琼胶酶酶学性质研究
2.3.1 琼胶酶最适温度及热稳定性
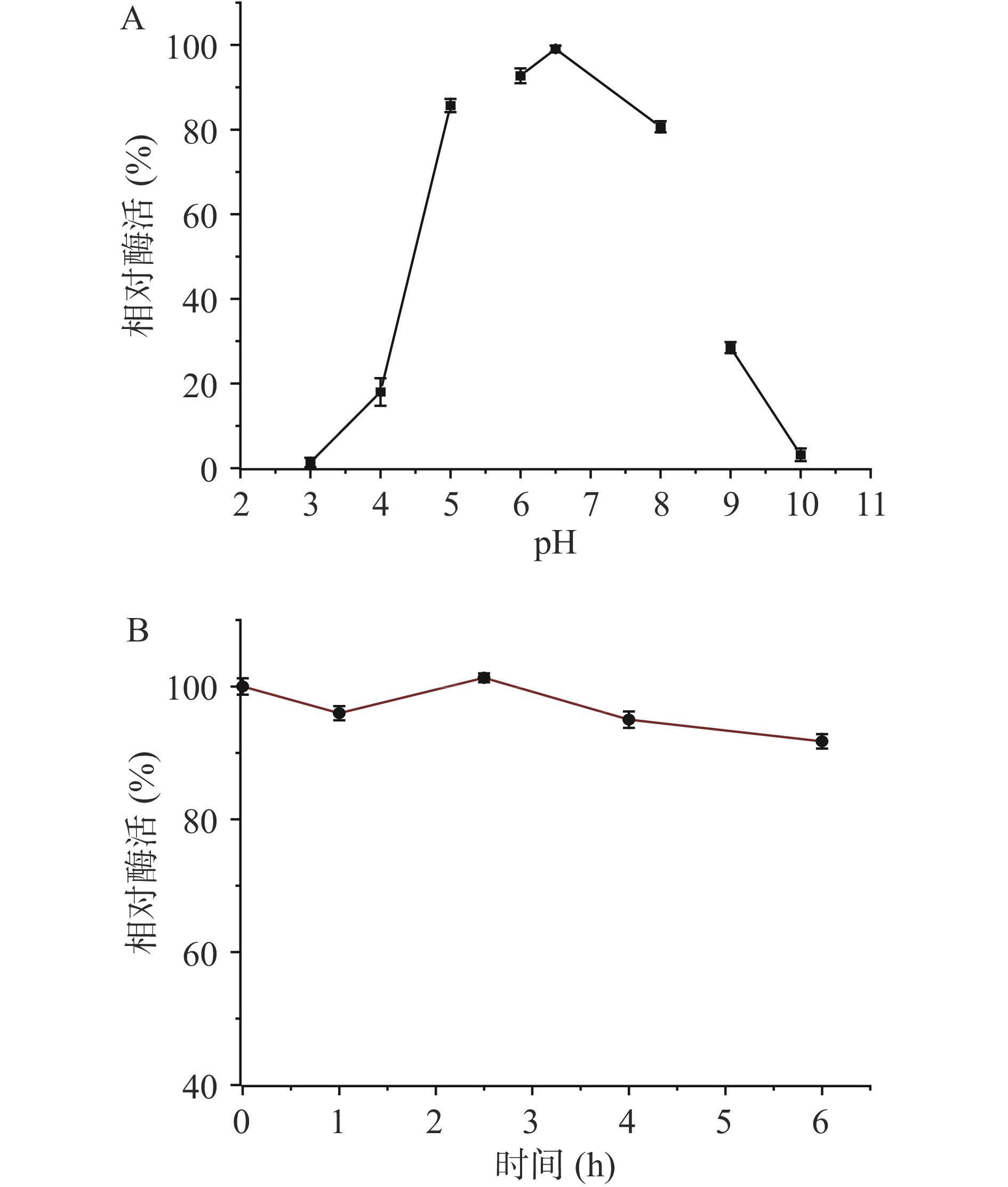
琼胶酶的最适温度及热稳定性研究结果如图6所示,图6A中的结果表明琼胶酶的酶活随温度的升高而增加,温度为40 ℃时酶活最高,当温度超过40 ℃后,高温使蛋白变性失活,酶促反应会受到温度的抑制,酶活力快速下降。因此该酶的最适反应温度为40 ℃,该酶在60 ℃下酶活下降80%左右,说明该琼胶酶具有一定耐热性。将琼胶酶在20、35 ℃保温8 h,酶活保持在95%以上;在40 ℃保温8 h内,酶活保持在90%以上;而当温度为45 ℃,酶活在1 h内降低到20%,证明该酶在20~40 ℃稳定性较好。与金佳等[22]的研究结果相比,本研究的琼胶酶温度作用范围较广且热稳定性较好。
2.3.2 琼胶酶最适pH及pH稳定性
由图7A可知,琼胶酶的最适pH为6.5,且该琼胶酶在pH5~8范围内具有较高的酶活力。天然海水的pH在7.9~8.4之间,而该酶在弱酸性环境下也具有较高的酶活,说明其具有一定的耐酸性。当pH<5或pH>8时,该琼胶酶的酶活大大降低,这是由于过低或过高的pH都会因为影响蛋白质中解离基团的解离状态而影响酶活,甚至使蛋白质失活。由图7B可知,在最适pH环境下,该琼胶酶6 h内的酶活维持在90%以上,结果表明该琼胶酶在弱酸性条件下稳定。
2.3.3 不同金属离子对酶活力影响结果
许多酶需要金属离子的存在才能发挥其催化功能,金属离子在酶的催化过程中起到的作用主要有传递电子、促进酶与底物的结合、稳定酶的分子的构象等作用。不同金属离子对琼胶酶活力影响结果如表5所示。在50 mmol/L浓度下,Cu2+、Zn2+、Fe3+、Al3+对琼胶酶活力具有强烈的抑制作用;Mg2+、Fe2+、Mn2+对琼胶酶活力也均有不同程度的抑制作用;原因可能在于反应体系中存在这些金属离子时影响了一些功能基团的亲和力,导致酶催化位点的结构发生变化,从而使得酶活降低[23]。而Na+、K+、Ca2+等在海水中含量丰富的离子对该酶的影响均不大,这可能与该菌株来源于海洋环境有关[24]。
表 5 金属离子对琼胶酶活性的影响Table 5. Effects of metal ions on agarase activity金属离子 浓度(mmol/L) 相对酶活(%) 对照 0 100.00±0.35 Na+ 50 98.48±0.46 K+ 50 98.70±2.50 Ca2+ 50 100.86±3.24 Mg2+ 50 79.91±4.63 Cu2+ 50 0.87±0.70 Fe2+ 50 84.91±2.33 Zn2+ 50 0.33±0.46 Mn2+ 50 78.39±1.85 Fe3+ 50 0 Al3+ 50 0 2.3.4 琼胶酶动力学参数结果
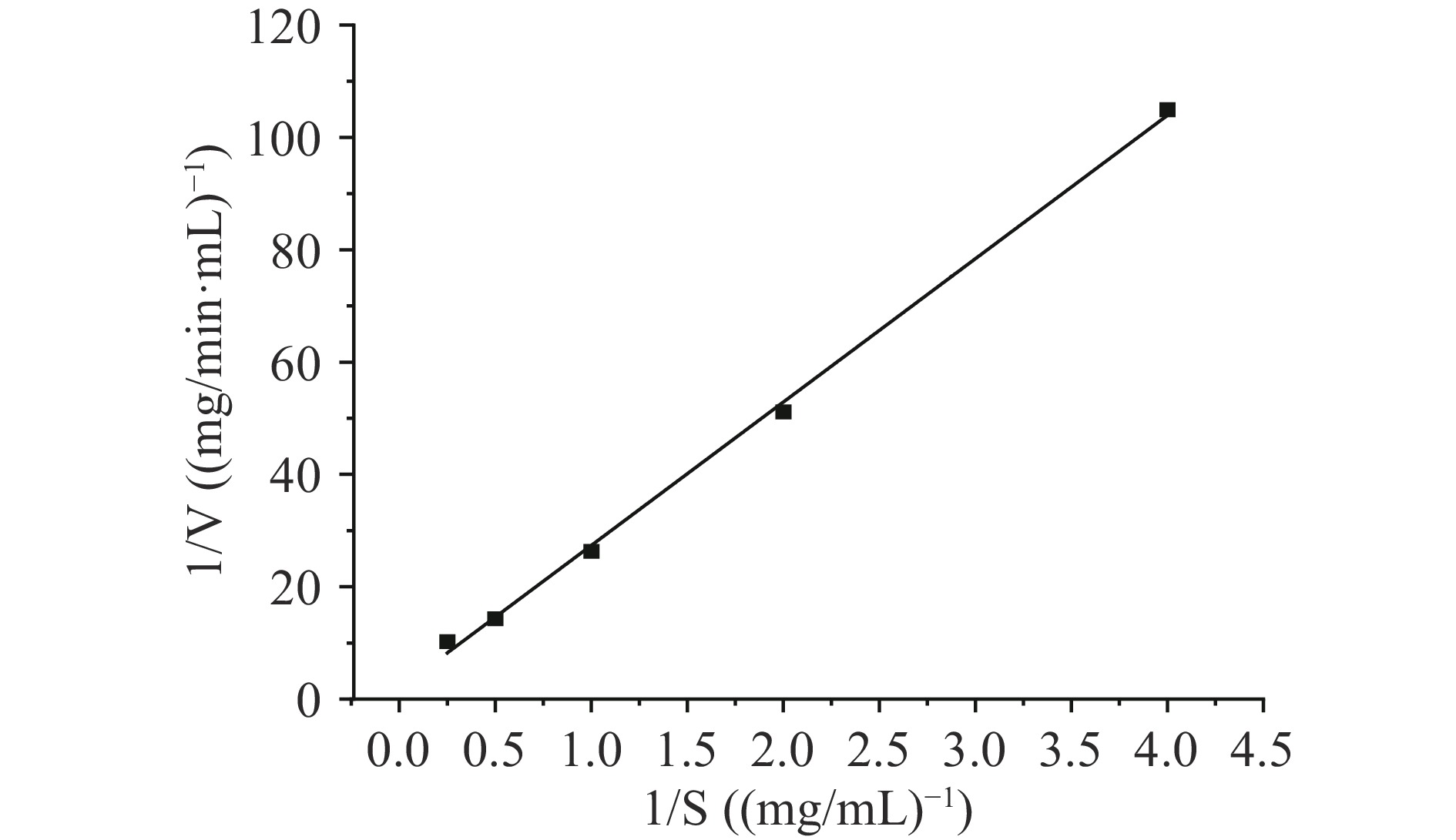
分别配制浓度为0.25、0.5、1、2、4 mg/mL的底物,测定底物反应速率,按照Lineweaver-Burk方程[25]处理,由图8中直线在横纵轴的截距分别计算出该琼胶酶的米氏常数为13.84 mg/mL,最大反应速度Vmax为0.54 mg/mL·min。1/Km可以近似地表示酶对底物亲和力的大小,1/Km越大,Km越小,则达到酶促反应最大反应速率的一半所需要的底物浓度越小,可以表明酶和底物的亲和力越大[26]。与Alkotaini 等[27]报道的Km值16.67 mg/mL相比较而言,该琼胶酶对琼脂具有相对较高的亲和力以及催化效率。
2.4 降解产物分析
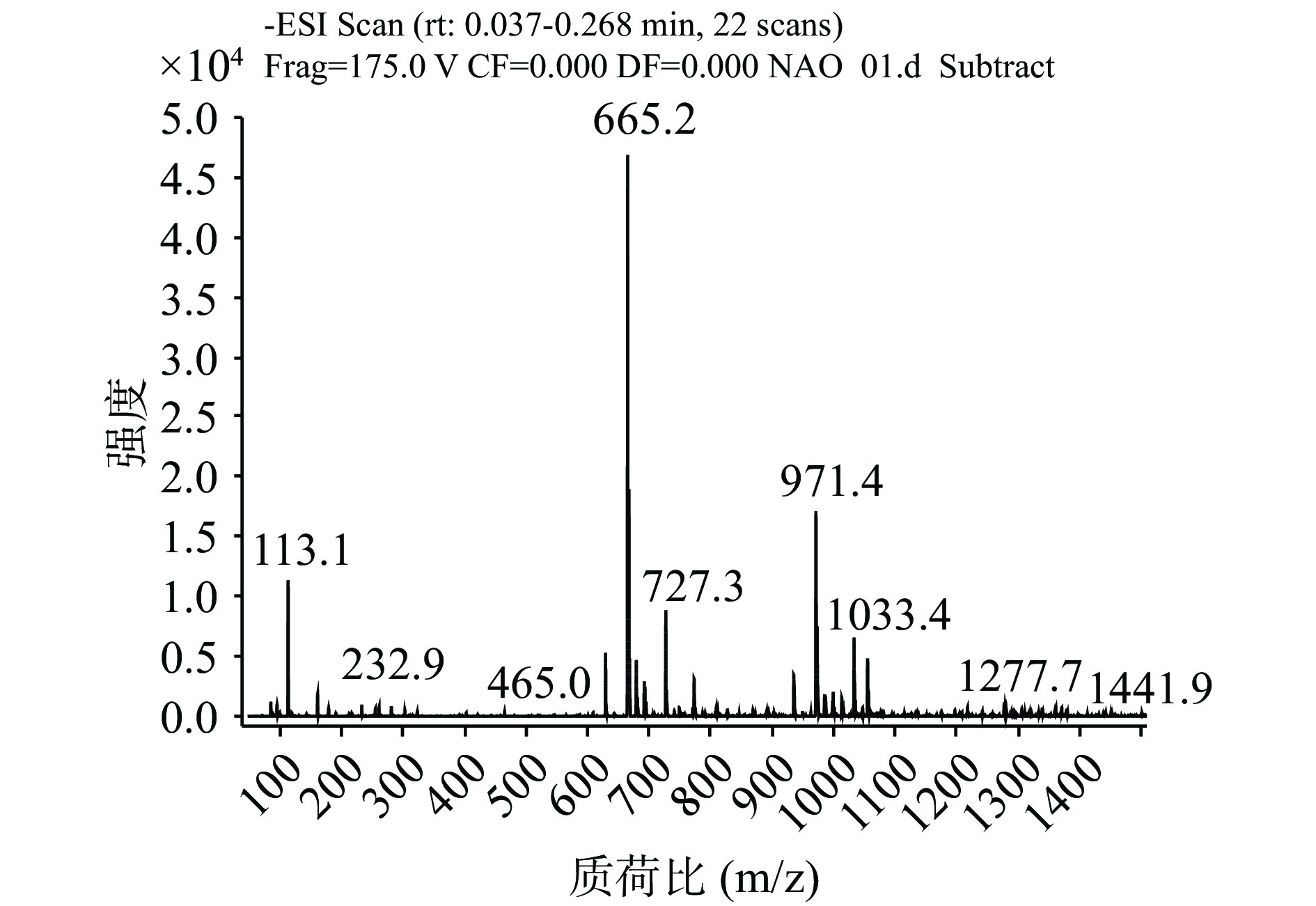
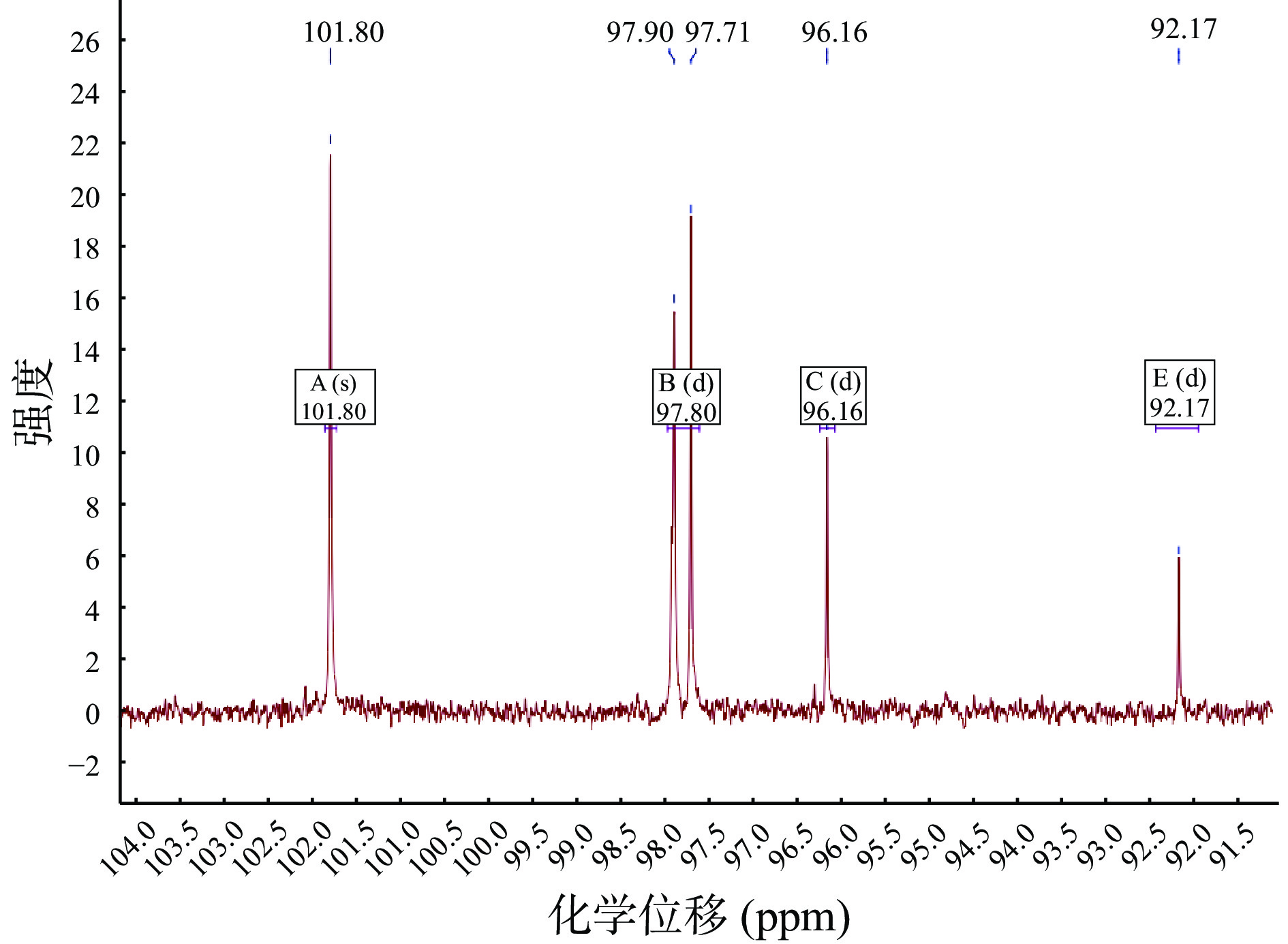
利用电喷雾质谱技术对降解产物进行验证,如图9所示,测得的离子峰质荷比(m/z)为:665.2[M-H]+、971.4[M-H]+,根据降解产物的分子量可推知,该酶的降解产物为四糖和六糖,其中主要产物为四糖。通过13C-NMR进一步鉴定琼胶酶的降解产物,如图10所示,在90.72 ppm处未检测到信号,92 ppm和96 ppm处检测到的信号是由还原端的α-和β-异头碳引起的[28],在97 ppm处的信号对应于非还原端3,6-脱水乳糖,这些特征信号表明了该琼胶酶为β-琼胶酶。经MS和13C-NMR分析,该酶主要降解产物为新琼四糖。
3. 结论
本研究针对自主筛选的Sphingomonas sp.Q2菌株,通过产酶条件优化、盐析沉淀以及不同形式的柱层析等技术手段,实现电泳纯琼胶酶的分离制备。经纯化后的琼胶酶比活为112048.82 U/mg,纯化倍数为7倍,回收率为48.04%。与现有的研究相比,得到的琼胶酶比活高且回收率高。通过对酶学性质的考察得到了最佳的反应条件:最适温度为40 ℃,最适pH为6.5,其性质符合海洋来源琼胶酶的基本特征。动力学参数结果表明,该琼胶酶的米氏常数为13.84 mg/mL,最大反应速度Vmax为0.54 mg/mL·min,具有较高的亲和力。通过MS、13C-NMR证明该酶为β-琼胶酶,主要降解产物为新琼四糖,能够对其结构进行验证。
通过以上对Sphingomonas sp.Q2菌株所产琼胶酶的酶学性质及降解产物的研究,充分了解其酶学性质,初步研究结果表明,该琼胶酶具有较高的酶活且产酶条件稳定,为新琼寡糖相关产物的制备提供技术基础,也为琼胶寡糖功能产品开发提供一种新型酶资源。但本文目前只局限于对野生菌产琼胶酶的初步探索,随着研究的深入还需进一步对该琼胶酶的结构、催化特异性及异源表达等方面进行探索并有望将其广泛应用到食品工业。
-
表 1 Box-Behnken试验设计因素与水平
Table 1 Experimental design factors and level of Box-Behnken
编码 因素 水平 −1 0 1 A 琼脂(g/L) 3 4 5 B 磷酸氢二钾(g/L) 0.5 1 1.5 C 氯化钠(g/L) 10 15 20 表 2 Box-Behnken试验设计与结果
Table 2 Design and results of Box-Behnken tests
实验号 A琼脂 B磷酸氢二钾 C氯化钠 酶活(U/mL) 1 −1 −1 0 870.19 2 1 −1 0 664.96 3 −1 1 0 910.64 4 1 1 0 1071.73 5 −1 0 −1 1041.57 6 1 0 −1 1054.08 7 −1 0 1 931.97 8 1 0 1 900.34 9 0 −1 −1 922.41 10 0 1 −1 1029.07 11 0 −1 1 680.41 12 0 1 1 989.35 13 0 0 0 1079.09 14 0 0 0 1077.62 15 0 0 0 1085.71 16 0 0 0 1072.47 17 0 0 0 1079.83 表 3 回归模型的方差分析
Table 3 Variance analysis of regression model
方差来源 平方和 自由度 均方 F值 P值 显著性 模型 25043.60 9 2782.62 658.78 <0.0001 ** A 44.04 1 44.04 10.43 0.0145 * B 8194.12 1 8194.12 1939.93 <0.0001 ** C 3269.95 1 3269.95 774.15 <0.0001 ** AB 2953.93 1 2953.93 699.33 <0.0001 ** AC 42.88 1 42.88 10.15 0.0154 * BC 900.74 1 900.74 213.25 <0.0001 ** A2 1399.49 1 1399.49 331.33 <0.0001 ** B2 7073.05 1 7073.05 1674.52 <0.0001 ** C2 467.51 1 467.51 110.68 <0.0001 ** 残差 29.57 7 4.22 失拟项 21.62 3 7.21 3.63 0.1226 纯误差 7.95 4 1.99 总和 25073.16 16 注:“*”表示对结果影响显著(P<0.05);“**”表示对结果影响极显著(P<0.01)。 表 4 琼胶酶分离纯化结果
Table 4 Summary of isolation and purification procedures of agarase
纯化步骤 总酶活(U) 总蛋白(mg) 比活(U/mg) 纯化倍数 回收率(%) 发酵液 105164 6.57 16006.70 1 100 硫酸铵沉淀 86850.29 1.67 52006.16 3.25 82.59 QFF阴离子交换层析 53181.91 0.60 88459.59 5.53 50.57 Sephdex G75凝胶柱层析 50522.81 0.45 112048.82 7.00 48.04 表 5 金属离子对琼胶酶活性的影响
Table 5 Effects of metal ions on agarase activity
金属离子 浓度(mmol/L) 相对酶活(%) 对照 0 100.00±0.35 Na+ 50 98.48±0.46 K+ 50 98.70±2.50 Ca2+ 50 100.86±3.24 Mg2+ 50 79.91±4.63 Cu2+ 50 0.87±0.70 Fe2+ 50 84.91±2.33 Zn2+ 50 0.33±0.46 Mn2+ 50 78.39±1.85 Fe3+ 50 0 Al3+ 50 0 -
[1] MENG Y, MAO X, NAN L, et al. Purification and characterization of two agarases from Agarivorans albus OAY02[J]. Process Biochemistry,2014,49(5):905−912. doi: 10.1016/j.procbio.2014.02.015
[2] WU Y R, ZHOU Z R, ZHAO M, et al. Molecular characterization of the thermostability and carbohydrate-binding module from a newly identified GH118 family agarase, AgaXa[J]. Process Biochemistry,2016,52:192−199.
[3] GIORDANO A, ANDREOTTI G, TRAMICE A, et al. Marine glycosyl hydrolases in the hydrolysis and synthesis of oligosaccharides[J]. Biotechnol,2006,1(5):511−530.
[4] 唐传核, 彭志英. 功能性食品基料低聚糖及膳食纤维类开发现状[J]. 粮食与油脂,2000,3(8):33−35. [TANG C H, PENG Z Y. Recent development of functional food ingredient-oligosocchorides and dietary fibres[J]. Cereals and Oils,2000,3(8):33−35. TANG C H, PENG Z Y. Recent development of functional food ingredient-oligosocchorides and dietary fibres[J]. Cereals and Oils, 2000, 3(8): 33-35.
[5] ROSELINE T L, SACHINDRA N M. Characterization of extracellular agarase production by Acinetobacter junii PS12B, isolated from marine sediments[J]. Biocatalysis and Agricultural Biotechnology,2016,6:219−226. doi: 10.1016/j.bcab.2016.04.007
[6] DONG Q, RUAN L, SHI H. A β-agarase with high pH stability from Flammeovirga sp. SJP92[J]. Carbohydrate Research,2016,432:1−8. doi: 10.1016/j.carres.2016.05.002
[7] LEE D G, JANG M K, LEE O H, et al. Over-production of a glycoside hydrolase family 50 β-agarase from Agarivorans sp. JA-1 in Bacillus subtilis and the whitening effect of its product[J]. Biotechnology Letters,2008,30(5):911−918. doi: 10.1007/s10529-008-9634-4
[8] ENOKI T, TANABE M, SHIMOMURA M, et al. Induction nechanism of heme oxygenase-1 and anti-inflammatory activity by agaro-oligosaccharides[J]. Nippon Shokuhin Kagaku Kogaku Kaishi,2010,57(4):157−162. doi: 10.3136/nskkk.57.157
[9] HU B, GONG Q, WANG Y, et al. Prebiotic effects of neoagaro-oligosaccharides prepared by enzymatic hydrolysis of agarose[J]. Anaerobe,2006,12(5-6):260−266. doi: 10.1016/j.anaerobe.2006.07.005
[10] CHEN X L, LIN H T, JIN M, et al. Characterization of a novel alkaline β-agarase and its hydrolysates of agar[J]. Food Chemistry,2019,295:311−319.
[11] IMOTOT, YAGISHITA K. A simple activity measurement of lysozyme[J]. Agricultural and Biological Chemistry,1971,35(7):1154−1156. doi: 10.1080/00021369.1971.10860050
[12] 姚艳艳, 常丽荣, 王晓辉, 等. 皱纹盘鲍来源弧菌Y-AG3产琼胶酶发酵培养基优化[J]. 中国酿造,2019,38(12):135−139. [YAO Y Y, CHANG L R, WANG X H, et al. Optimization of fermentation medium for agarase production by Vibrio Y-AG3 fromHaliotis discus hannai[J]. China Brewing,2019,38(12):135−139. YAO Y Y, CHANG L R, WANG X H, et al. Optimization of fermentation medium for agarase production by Vibrio Y-AG3 from Haliotis discus hannai[J]. China Brewing, 2019, 38(12): 135-139.
[13] PARK J, CHANG Y K. Production of DagA, a β-agarase by Streptomyces lividans in Mixed-sugar medium simulating microalgae hydrolysate[J]. Journal of Microbiology and Biotechnology,2014,24(12):1622−1628. doi: 10.4014/jmb.1405.05019
[14] CHANDRAN P R, CRI J. Isolation, purification and characterization of an extracellular agarase from Alteromonas sp[J]. Journal of Pure and Applied Microbiology,2012,6(1):351−356.
[15] ROSELINE T L, SACHINDRA N. Purification and characterization of agarase from marine bacteria Acinetobacter sp. PS12B and its use for preparing bioactive hydrolysate from agarophyte red seaweed Gracilaria verrucosa[J]. Applied Biochemistry & Biotechnology,2018,186(1):66−84.
[16] BELAS R . Sequence analysis of the agra gene encoding 3-agarase from Pseudomonas atlantica[J]. Journal of Bacteriology 2018, 171(1): 602-605.
[17] BRADFORD M M. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding[J]. Analytical Biochemistry,1976,72(1−2):248−254. doi: 10.1016/0003-2697(76)90527-3
[18] AKASSOU M , LEMAY J F , QUESSY P , et al. Expression of pullulanase from Thermus thermophilus HB8 in Pichia pastoris and its characterization[C]. The 3rd World Congress on New Technologies, 2017: 119.
[19] 陈惠源, 蔡俊鹏, 刘江涛. 海洋细菌产琼胶酶的条件优化[J]. 现代食品科技,2005,21(3):48−50. [CHEN H Y, CAI J P, LIU J T. Optimization growth conditions of an agarase-producing marine bacterium Sphingobacterium multivorumd[J]. Guangzhou Food Science and Technology,2005,21(3):48−50. CHEN H Y, CAI J P, [LIU J T. Optimization growth conditions of an agarase-producing marine bacterium Sphingobacterium multivorumd[J]. Guangzhou Food Science and Technology, 2005, 21(3): 48-50
[20] 李驰, 李春生, 杨贤庆, 等. 海洋细菌Vibrio fluvialis的分离鉴定, 产琼胶酶条件优化及酶的分离纯化[J]. 食品与发酵工业,2020,46(7):35−42. [LI C, LI C S, YANG X Q, et al. Isolation and identification of agarase-producing Vibrio fluvialis for agarase preparation and purification[J]. Food and Fermentation Industries,2020,46(7):35−42. LI C, LI C S, YANG X Q, et al. Isolation and identification of agarase-producing Vibrio fluvialis for agarase preparation and purification[J]. Food and Fermentation Industries, 2020, 46( 7): 35-42.
[21] LI R K, CHEN Z, YING X J, et al. A novel GH16 beta-agarase isolated from a marine bacterium, Microbulbifer sp. BN3 and its characterization and high-level expression in Pichia pastoris[J]. International Journal of Biological Macromolecules,2018,119:1164−1170. doi: 10.1016/j.ijbiomac.2018.08.053
[22] 金佳, 江承程, 毛相朝. α-琼胶酶OUC-GaJJ96的异源表达及酶学性质[J]. 食品科学技术学报,2020,38(6):47−54. [JIN J, JIANG C C, MAO X Z. Heterologous expression and enzymatic properties of α-agarase OUC-GaJJ96[J]. Journal of Food Science and Technology,2020,38(6):47−54. JIN J, JIANG C C, MAO X Z. Heterologous expression and enzymatic properties of α-agarase OUC-GaJJ96 [J]. Journal of Food Science and Technology, 2020, 38(6): 47-54.
[23] CHEN X, XIA W, YU X. Purification and characterization of two types of chitosanase from Aspergillus sp. CJ22-326[J]. Food Research International,2005,38:315−322. doi: 10.1016/j.foodres.2004.04.012
[24] 产竹华, 侯艳平, 狄文婕, 等. 深海太平洋火色杆菌琼胶降解基因分析和琼胶酶Aga0950的表达及酶学性质[J]. 微生物学报,2018,58(3):411−422. [CHAN Z H, HOU Y P, DI W J, et al. Analysis of agar degradation genes and agarase Aga0950 expression and enzymatic properties of Proteus pacifica from deep sea J]. Acta Microbiologica Sinica,2018,58(3):411−422.
[25] 景一娴, 饶菁菁, 廖飞, 等. 低于米氏常数底物浓度下酶动力学参数的测定[J]. 重庆医科大学学报,2018,43(11):1464−1468. [JING Y X, RAO J J, LIAO F, et al. An integration strategy for the estimation of kinetic parameters of an enzyme at substrate concentrations lower than Michaelis-Menten constant[J]. Journal of Chongqing Medical University,2018,43(11):1464−1468. JING Y X, RAO J J, LIAO F, et al. An integration strategy for the estimation of kinetic parameters of an enzyme at substrate concentrations lower than Michaelis-Menten constant[J]. Journal of Chongqing Medical University, 2018, 43(11): 1464-1468.
[26] 夏宁, 王晓琪, 严文冰, 等. 豆浆脂肪氧合酶测定方法的优化及酶动力学的研究[J]. 大豆科学,2018,37(5):769−775. [XIA N, WANG X Q, YAN W B, et al. Method optimization for the determination of lipoxygenase in soybean milk and the evaluation of its kinetics[J]. Soybean Science,2018,37(5):769−775. XIA N, WANG X Q, YAN W B, et al. Method optimization for the determination of lipoxygenase in soybean milk and the evaluation of its kinetics[J]. Soybean Science, 2018, 37(5): 769-775.
[27] ALKOTAINI B, HAN N S, KIM B S. et al. Enhanced catalytic efficiency of endo-beta-agarase I by fusion of carbohydrate-binding modules for agar prehydrolysis[J]. Enzyme & Microbial Technology,2016,93:142−149.
[28] TEMUUJIN U, CHI W J, CHANG Y K, et al. Identification and biochemical characterization of Sco3487 from Streptomyces coelicolor A3(2), an exo-and endo-type β-agarase-producing neoagarobiose[J]. Journal of Bacteriology,2012,194(1):142−149. doi: 10.1128/JB.05978-11
-
期刊类型引用(1)
1. 宋美玲,郭三保,廖莉,吴晴阳,黄胜和. 16S rDNA鉴定腐败椰果罐头中的微生物. 食品安全导刊. 2024(24): 71-74 .  百度学术
百度学术
其他类型引用(2)





 下载:
下载:

 下载:
下载:



