Progress on Enzymatic Properties and Gene Structure of Bile Salt Hydrolase
-
摘要: 胆盐水解酶(Bile salt hydrolase, BSH)广泛存在于哺乳动物胃肠道中,是肠道菌群在生长、繁殖过程中产生的一种胞内酶,可以调节宿主的胆汁酸平衡,影响脂质代谢,有控制胆固醇、调节肠道疾病的作用,并且BSH这种调控作用可以实现益生菌部分益生功能,因此BSH一直是研究热点。BSH结构和底物特异性的详细知识是开发BSH相关产品的坚实基础。本文介绍了BSH的来源及活性检测方法,BSH基因结构、酶学性质及底物识别机制,最后讨论了BSH在畜禽业食品工业及医药等方面的应用,期望为胆盐水解酶的深入研究及其在食品、保健品及医药方面的开发利用提供参考。Abstract: Bile salt hydrolase (BSH) is an intracellular enzyme which is produced by intestinal flora during the period of growth and reproduction. This enzyme has been suggested to be involved in the host's bile acid balance, lipid metabolism and metabolic responses. Therefore, BSH has always been a research hotspot. Detailed knowledge of BSH structure and substrate preferences is a solid foundation for the development of BSH related products. This review explains the source and distribution of BSH, test method for BSH activity. It also introduces enzymatic properties and genetic organization. Finally, the potential applications of BSH research are briefly discussed. This review would provide theoretical basis for the in-depth research of BSH, developments of related functional foods, and other industry applications.
-
Keywords:
- bile salt hydrolase /
- intestinal microflora /
- enzymatic properties /
- structural basis /
- applications
-
胆盐水解酶(Bile salt hydrolase,BSH)是肠道菌群在生长发育过程中产生的一类代谢产物,属于N-端亲核水解酶(Ntn)超家族中胆酰甘氨酸水解酶(CGH)家族,是bsh基因编码的胞内酶,具有重要的生理作用[1]。BSH作用于酰胺键,能够调节胆酸池的组成,将结合态胆汁酸催化水解为游离氨基酸和游离态胆汁酸,该反应在胆汁酸代谢及肝肠循环中具有重要作用[2]。20世纪70年代就开始了对BSH的研究,1967年首次从产气荚膜梭菌中部分纯化了BSH,关于BSH的研究主要集中在BSH基因克隆及纯化、高BSH菌株筛选、BSH在胃肠道中的作用机制及降胆固醇机理等方面[3]。
越来越多研究发现BSH对肠道微生物和宿主生理代谢具有重要调节作用,仅BSH活性就可以显著影响宿主的脂质代谢和体重增加[1]。胆盐水解酶的解结合会导致脂质吸收不良,这可能导致宿主体重减轻,通过抑制肠道BSH的活性,将促进脂肪代谢和能量收获,从而改善食用动物的生长性能和饲料效率[4]。BSH的解结合作用可以影响宿主体内胆固醇的动态平衡[5]。广泛的肠道微生物区系研究表明,BSH是开发新型非抗生素食品添加剂、提高动物饲料产量和设计控制人类肥胖的新措施的关键因素,通过BSH介导饮食干预肝肠疾病具有巨大潜力,BSH活性作为益生菌筛选的标记,在功能性食品研究与开发中具有重要意义。胆盐水解酶与益生菌降胆固醇有一定关系,但具体机制尚不明确,BSH对肠道菌的重要性也不清楚,为了对胆盐水解酶有更深入全面的认识,本文对BSH来源分布、酶学性质以及结构和应用方面进行了综述,对于进一步深入研究胆盐水解酶作用机制及其在食品工业、医药及其他方面的应用提供参考。
1. 胆盐水解酶来源及分布
研究发现BSH广泛分布在肠道细菌中,并且在人类和小鼠肠道环境中观察到的BSH蛋白存在细微差异,这反映该酶的宿主物种特异性。系统发育分析表明,以共轭胆汁酸形式存在的选择性压力促使BSH活性的表达,富含结合胆汁酸和非结合胆汁酸的自然环境,如肠道粪便通常容易分离出有BSH活性的菌株[6]。哺乳动物和人体的肠道和粪便是分离产BSH菌株的主要来源,例如,研究者从小鸡胃肠道菌群中分离了12株Lactobacillus菌株(如L. reuteri、L. brevis、L. salivarius、L. gallinarum和L. panis),人体粪便中分理出3株菌株(L. fermentum、Streptococcus bovis ATCC 43143和 Enterococcus faecalis UK873),这些菌株具有水解结合胆汁盐的能力,并且这些微生物作为益生菌制剂与宿主肠道微生物群具有良好相容性。后面研究者也从蔬菜、温泉和牛奶中分离出了具有BSH活性的微生物[7]。从奶酪中分离出来的植物乳杆菌菌株DGIA1对甘胆酸钠、甘氨酸脱氧胆酸盐、牛磺胆酸盐和牛磺酸脱氧胆酸盐有很高的水解能力[8]。表1中列出了部分产BSH菌株,BSH主要分布在革兰氏阳性的肠道细菌中,例如乳杆菌属(Lactobacillus)、双歧杆菌属(Bifidobacterium)、肠球菌属(Bifidobacterium)、梭状芽孢杆菌属(Clostridium)和拟杆菌属(Bacteroides)等。其中双歧杆菌和乳杆菌等益生菌尤为受到人们关注,并且研究发现双歧杆菌来源的胆盐水解酶的活性较乳杆菌来源的胆盐水解酶的酶活高得多[9]。
表 1 部分产胆盐水解酶菌株及来源Table 1. Microbial sources of bile salt hydrolase菌株 来源 参考文献 植物乳杆菌(L. plantarum DGIA1) 奶酪 [8] 嗜酸乳杆菌(L. acidophilus LA15) 开菲尔颗粒 [7] 植物乳杆菌(L. plantarum WCFS1) 人体唾液 [7] 副干酪乳杆菌(L. paracasei RN025880) 传统酵素 [10] 芽孢杆菌(Bacillus polyfermenticus) 朝鲜泡菜 [11] 枯草芽孢杆菌(B. subtilis Bn1) 牛奶 [7] 克里斯滕森菌(C. minuta DSM33407) 人体 [12] 粪肠球菌(E. faecalis UK873) 人体 [7] 酵母菌(Yeasts) 绿橄榄 [13] 片球菌(Pediococcus spp) 红酒 [14] 2. 胆盐水解酶活性检测方法
BSH活性很大程度上代表了一种常见的作用于胆汁的微生物生物转化能力,BSH活性与胆盐耐受性之间存在一定联系,并可以作为筛选益生菌的一项重要标准[15]。基于BSH与底物反应的原理已有多种检测其活性的方法,其中定性检测的方法包括平板筛选法、放射性同位素标记法和薄层色谱法等,定量方法包括茚三酮法、高效液相色谱法、糠醛法和三硝基苯磺酸法等[16-17]。
2.1 定性检测方法
平板法是实验过程中最常用的一种方法,操作简便,可以快速地从待测菌株中筛选出目标菌株,其检测的主要原理是结合胆汁酸和非结合胆汁酸的不同物理化学性质,BSH将结合型胆汁酸水解成游离型胆汁酸和氨基酸,游离型胆汁酸在水中溶解性差,从而在琼脂中沉淀出来从而产生光晕。平板法筛选时培养基中常用的底物有牛磺脱氧胆酸和牛磺胆酸,由于BSH表现出不同的底物特异性,在平板中添加其他结合胆盐(如甘氨鹅去氧胆酸、甘氨胆酸等)可以达到特定的筛选目的[18]。放射性同位素标记法采用放射性同位素标记的胆盐作为底物,结果准确,灵敏度高,稳定性好,但价格昂贵不易操作[19]。薄层色谱法利用各成分的极性不同从而达到分离目的,可用于胆盐的定性分离和鉴定,与光密度法相结合可实现胆盐的定量分析,该方法准确度较高,但成本高。随着数字图像技术的发展,将数字图像分析软件与平面色谱法相结合的高效薄层色谱法与传统方法相比具有更高的分离效率及准确度,操作简单,经济有效[20]。
2.2 定量检测方法
定量检测方法根据检测对象的不同分为两种类型,一种是通过检测BSH水解结合态胆汁盐生成的氨基酸的浓度来反应BSH活力,如茚三酮法和三硝基苯磺酸法;另一种则是通过测定结合胆盐或游离胆盐变化确定酶活,如高效液相色谱法和糠醛法。糠醛法和三硝基苯磺酸法因具有一定毒害作用,已较少使用。茚三酮法在酶活性定量检测方法中具有成本低、操作便捷的优势,在国内外实验中得到广泛应用,其检测过程主要包括胆盐水解反应和反应生成氨基酸的量化[21]。高效液相色谱法准确度与灵敏度都较高,并且高效液相色谱方法可以同时检测多种胆盐为底物时的反应,可研究BSH对不同底物的降解区别,通过液相色谱图可以直观地看到BSH的降解偏好,便于进行特定的筛选[22]。
3. 胆盐水解酶的基本结构和酶学性质
3.1 胆盐水解酶空间结构
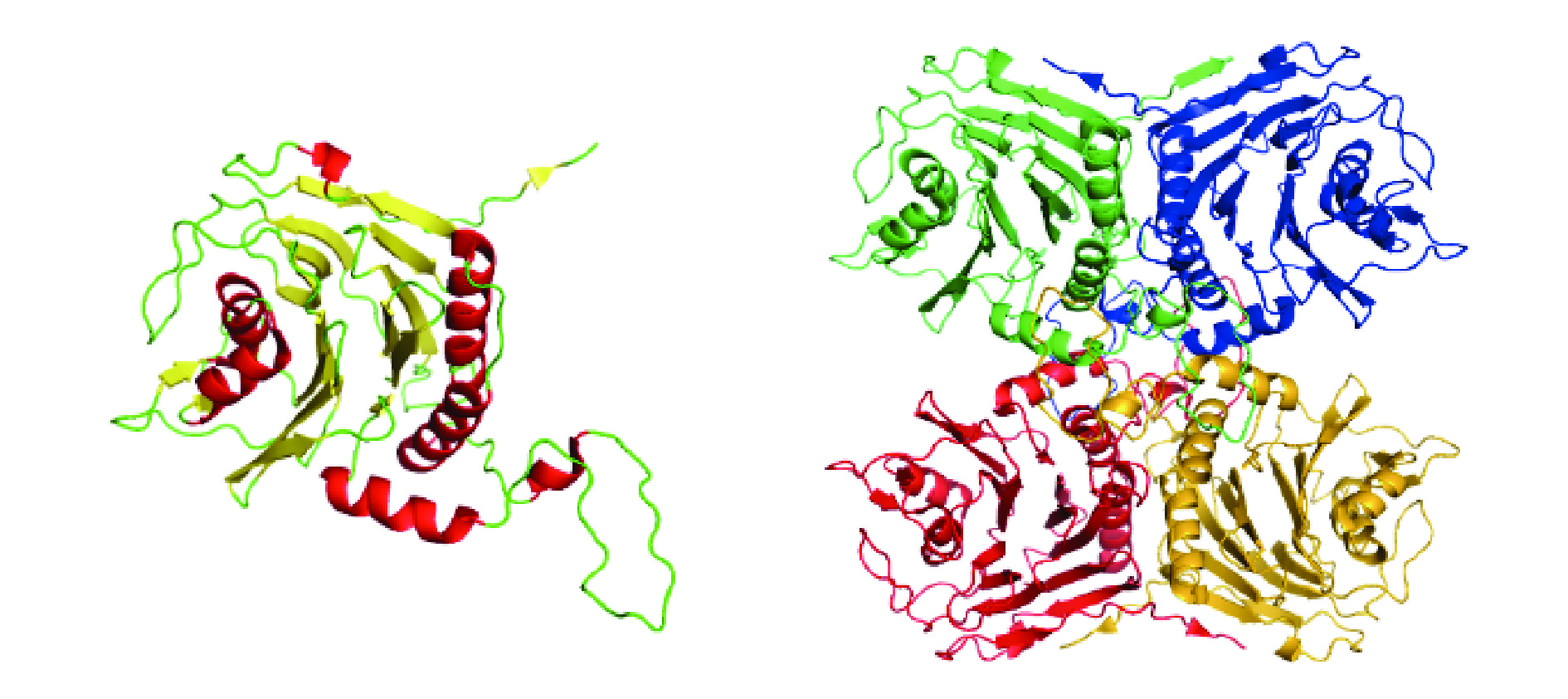
目前,已经报道了四个特定物种的BSH晶体结构,包括来自长双歧杆菌的BlBSH[23]、产气荚膜双歧杆菌的CpBSH[24]、粪肠球菌的EfBSH[25]和唾液乳杆菌的LsBSH[26],BSH主要以同源四聚体的结构存在,BSH分子单体采用NTN水解酶的αββα四层三级结构折叠,两个反向平行的β-片层夹在两个α-螺旋之间,单体和单体之间通过氢键、疏水作用及静电相互作用连接形成四聚体,如图1所示,单体的总体四聚体排列和其他NTN水解酶家族成员所描述的生理组装是一致的[27]。部分BSH以同源六聚体或八聚体形式存在,同一生物体中的BSH同系物可能表现出不同的单体结构[2],在脆弱芽孢杆菌ATCC 25285中发现了同源二聚体、同源六聚体和同源八聚体三种形式的BSH[28]。
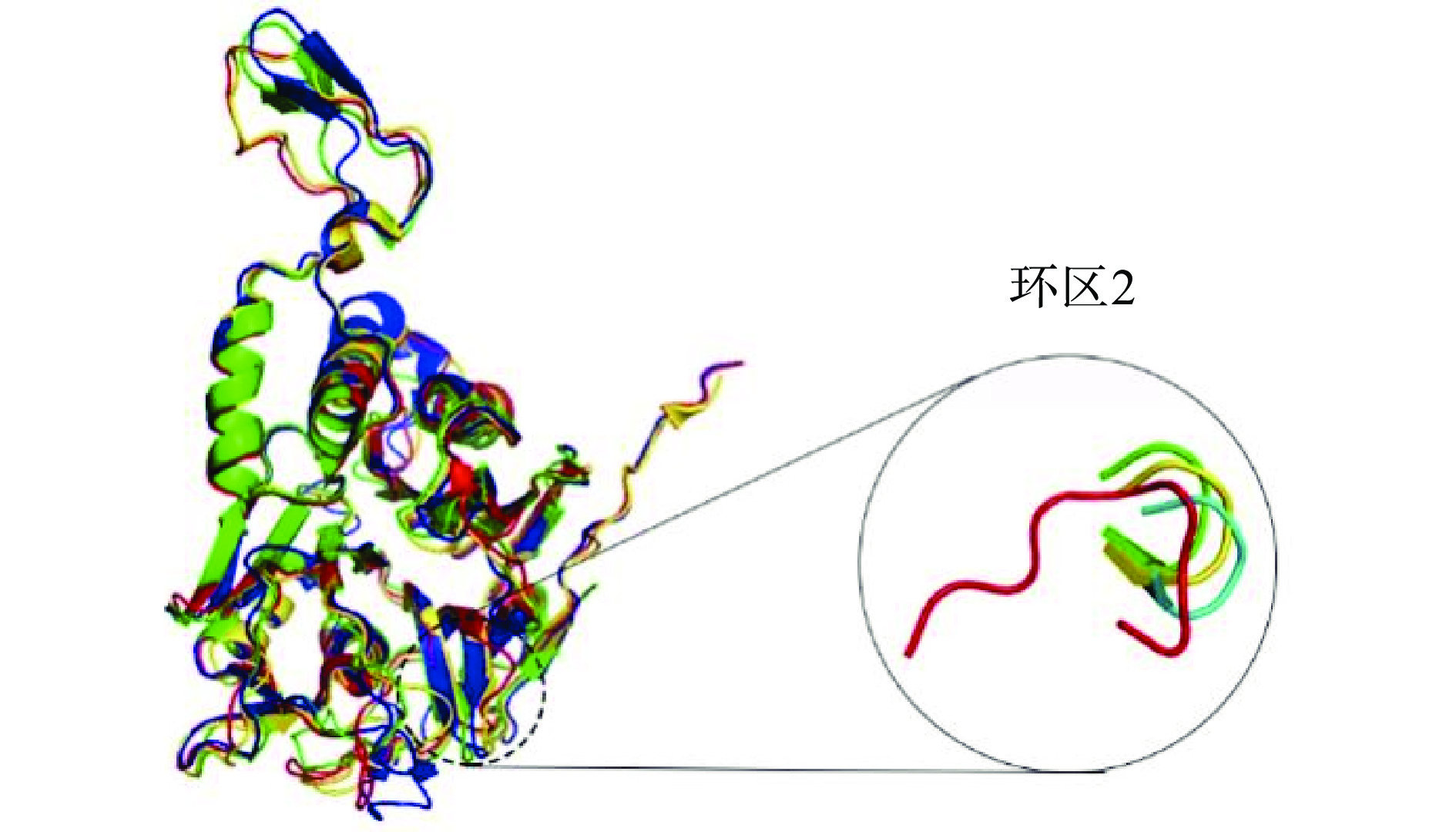
BSH的结构解析有助于理解酶分子的底物选择性及作用机理,已知胆酰甘氨酸水解酶类有四个结合口袋环,它们构成底物结合口袋,影响酶活力及底物亲和能力[20]。Ef BSH中的这四个环由残基21~25(环区1)、57~64(环区2)、125~137(环区3)和255~269(环区4)组成。与其他BSHS相比,Ef BSH的环区2延伸的更多,动态性和灵活性增加(图2),此外,与Cp BSH和Bl BSH相比,Ef BSH的结合口袋具有更强的疏水性。活性中心性质的这些变化(如较小的结合袋体积和较高的疏水/亲水比),可能会影响底物的结合[22]。
3.2 酶学性质
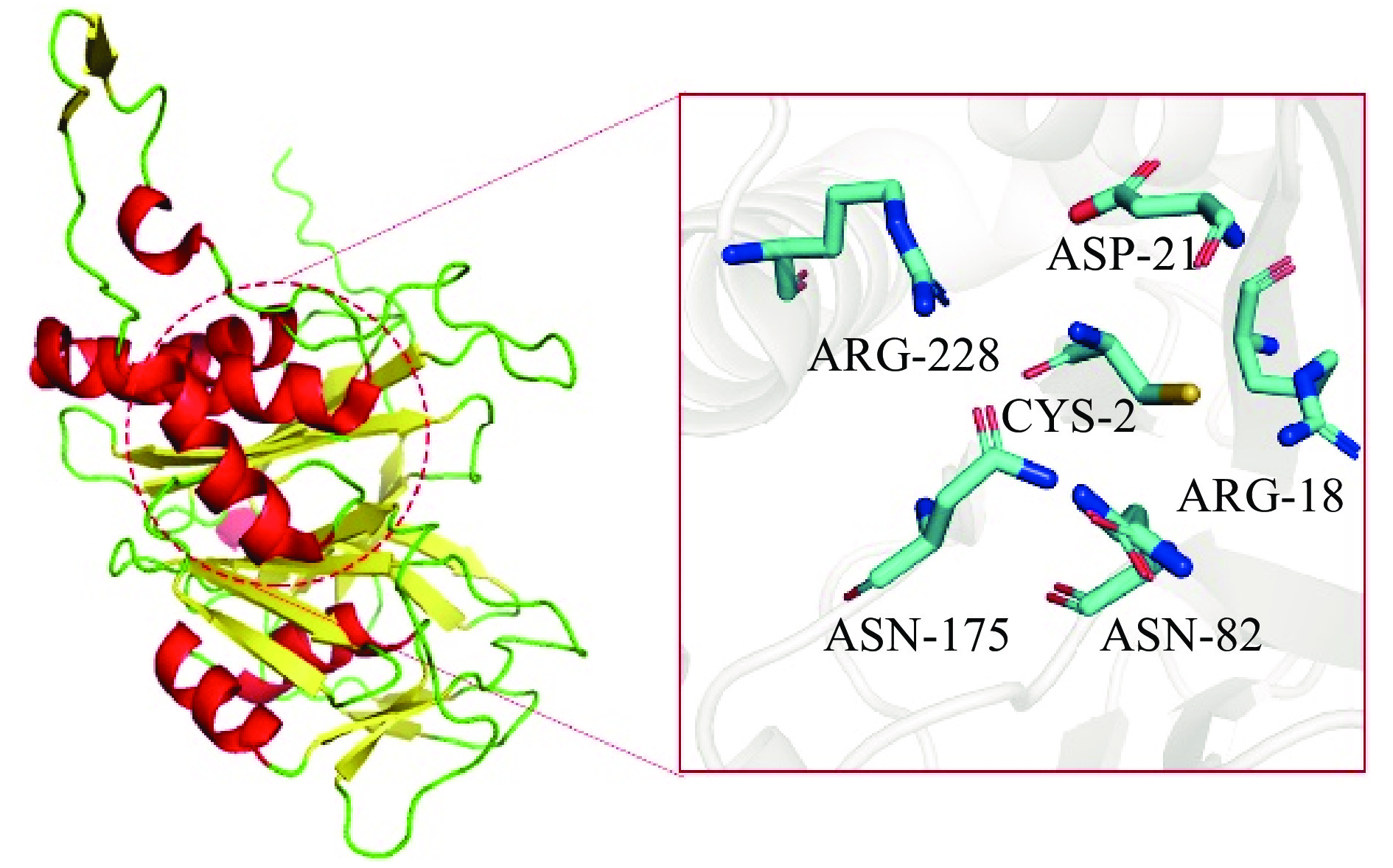
不同来源的BSH在氨基酸数量、最适pH、温度、底物偏好等方面存在差异,这些BSHS主要是由314~338个氨基酸编码的胞内酶[3],BSH的最适pH范围较广在3.8~7.0之间。除LjPF01_BSHC的最适温度为70 ℃外,大多数BSH酶的最适作用温度在30~55 ℃,底物特异性是BSH最大的酶学差异,不同来源的BSH对不同的胆汁酸有不同的水解能力[29],相对于牛磺型结合胆汁酸,大部分BSH水解甘氨型结合胆汁酸的能力更高,来源于乳酸菌属的BSH对甘氨脱氧胆酸的水解率超过90%,而对牛磺脱氧胆酸的水解率仅为33%~45%[14]。此外,同一菌株的多个BSH同系物可能表现出不同的优先活性,如LjPF01_BSHA和LjPF01_BSHC,分别对牛磺酸和甘氨酸结合胆汁酸表现出特异性亲和力[30]。BSH活性也与活性位点息息相关,胆盐水解酶的活性位点主要位于两个核心β折叠之间[31],通过对BSH和青霉素V酰化酶结构的比较,已经在BSH的活性位点上发现了几个保守的、重要的催化残基(Cys2、Arg16、Asp19、Asn79、Asn171和Arg224),这六个残基在三维结构中是封闭的,形成了BSH的核心活性位点,在这六种主要的催化残基中有5种都是保守的[32],根据产气荚膜梭菌的CpBSH编号,其几何结构如图3所示,其中Cys2是公认的活性位点,任何关于此位点的突变都会导致酶失活,重组酶s型曲线消失[33]。周晓玲等[34]在关于BSH的诱导表达研究中发现,在双歧杆菌来源BSH的N端添加组氨酸标签会导致N端催化反应中心Cys2结构改变,从而导致表达后失去BSH活性,而在C端添加组氨酸标签,诱导表达后可以检测到胞内活性。此外,Xu[35]等通过定点突变对六种结合胆汁酸的诱变和BSH活性分析,验证了几个活性位点残基在底物识别和催化中的重要性。其中,Cys2和Asn171对酶活性起关键作用,Tyr24、Phe65和Gln257对底物专一性有贡献[35]。Öztürk等对活性位点中R16和D19残基进行定点突变,R16和D19突变分别导致BSH活性的急剧下降和BSH蛋白产量的损失,证明了R16和D19残基在维持活性位点几何形状和过渡态稳定方面具有重要作用[36]。
3.3 底物识别机制
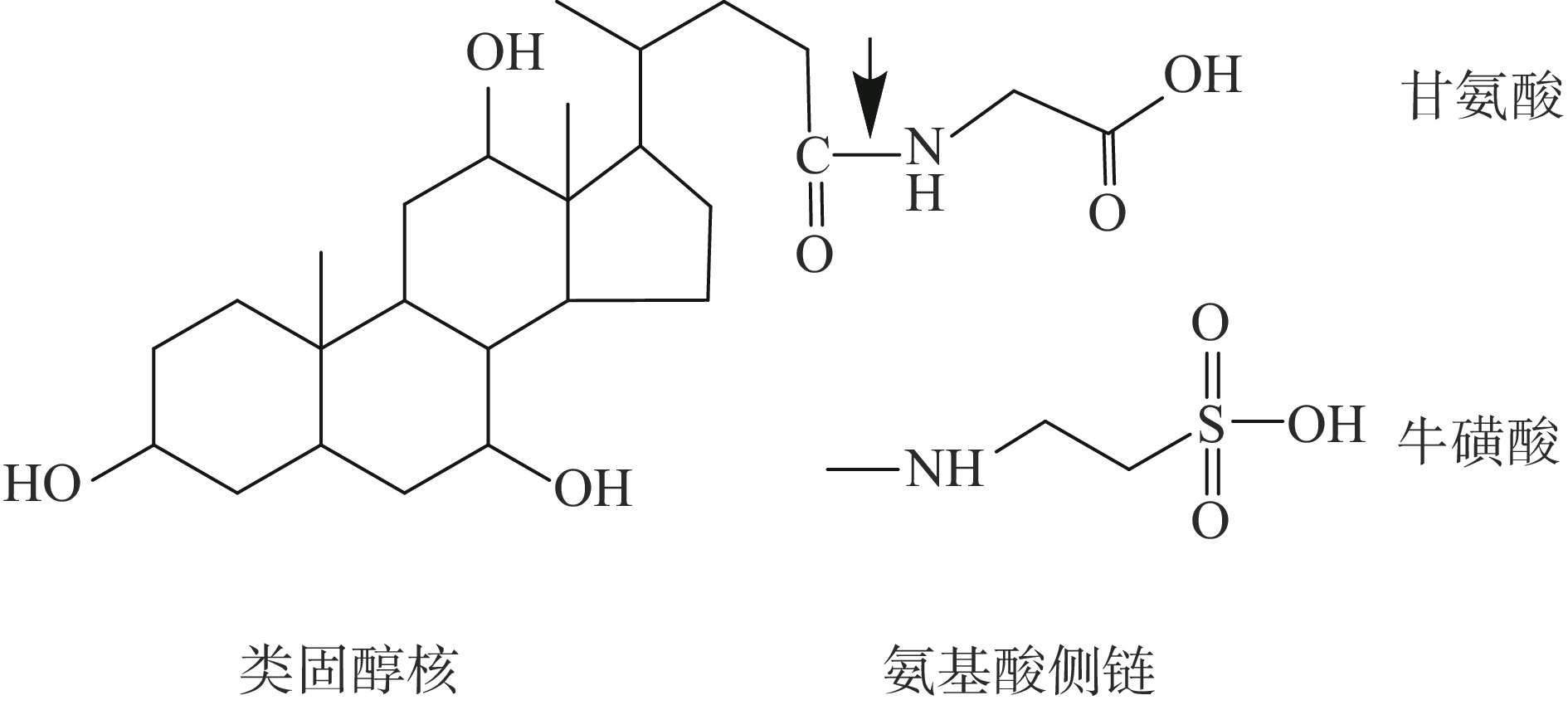
BSH的底物由胆酸盐类固醇核和氨基酸基团组成,根据氨基酸种类的不同分为甘氨型结合胆盐和牛磺型结合胆盐两大类(图4)[37],根据之前研究中动力学数据显示,BSH对甘氨型结合胆盐的水解能力普遍优于牛磺型结合胆盐,因而认为BSH优先识别氨基酸部分而不是胆酸部分,推测其主要原因是由于牛磺酸结合胆盐中硫原子引起的空间位阻(图4)[38]。也有观点认为宿主肠道中甘氨型结合胆汁酸的浓度高于牛磺型结合胆汁酸的浓度,且甘氨酸结合胆汁酸比牛磺酸结合胆汁酸对细菌的毒性大得多,这种底物偏好的选择性压力导致BSH对甘氨型结合胆汁酸的高酶活以削弱其对细菌的毒性[39]。最近研究者提出BSHS同时识别氨基酸和胆酸类固醇核上胆汁酸的假设,BSH能通过与类固醇核上的疏水作用识别底物[40]。通过结合位点相似性评分系统、晶体结构、动力学数据、分子对接和比较结构分析,在BSHS中发现了一些可能参与底物结合的关键氨基酸残基和二级结构元素,BSH活性的重要残基存在于环区1(20~26)、环区2(59~68)和环区3(131~142)中,而在环区4(263~275)中没有发现关键残基[23],Philem等研究发现植物乳杆菌B14胆盐水解酶第三环中的F129和L138是影响其催化活性和底物特异性的关键因素[41]。环区3中的残基Leu133可能通过浓缩底物结合口袋的入口,从而限制了空间构型[36]。LsBSH中的Gln257在丙氨酸突变时提供了不同的底物特异性,Tyr24和Phe65的突变也证实了这些残基对底物特异性的贡献[36],这种残基与口袋中的几个残基形成一个氢键网络,进而改变结合口袋的形状和亲水性,不同程度地影响甘氨酸和牛磺酸共轭底物的结合。虽然Gln257在LsBSH和EfBSH中保守,但在CpBSH和BlBSH中这一位置的残基却是蛋氨酸,进一步表明了该残基在底物特异性中的潜在重要性。也表明通过对活性位点和底物结合口袋进行微小的修饰,胆盐水解酶就能获得更广泛的底物特异性[41]。目前,关于BSH底物特异性决定因素的研究仍在初级阶段,未来对与特定底物反应的BSH的结构进行深入的分析将有助于发现底物偏好决定残基,了解底物特异性的结构基础,这对于BSH在益生菌或抑制剂等相关产品开发中具有重要作用。
4. 胆盐水解酶基因
BSH存在于与胃肠道环境相关的所有双歧杆菌菌株和乳酸杆菌菌株中,bsh基因在所有测试得双歧杆菌中高度保守,其G+C含量反射出了整个基因组的G+C含量,bsh基因可能是双歧杆菌基因组中的旁系同源基因,其它肠道微生物可能从这些菌株中获得bsh基因[42]。根据Sarah的研究,bsh基因在植物乳杆菌WCFS1、嗜酸乳杆菌NCFM、加色乳杆菌ATCC 33323和加色乳杆菌V-V03中均以单顺反子形式表达[43]。嗜酸乳杆菌PF01中BSH基因的核苷酸序列分析结果显示它是一个包含951个核苷酸的开放阅读框,编码316个氨基酸。BSH启动子位于起始密码子的上游,表达的蛋白表现出与其他宿主较高的同源性[44]。基因组学研究发现不同乳酸菌中含有1~4个编码BSH的基因,分散在基因组不同位置上,且氨基酸相似度较低,仅为21%~37% [45]。在某些菌株中,BSH基因的数量可以改变,其性质也可以改变。同时,对携带多个BSH基因的菌株进行分析,基因来源不同,其性质有所不同。嗜酸乳杆菌NCFM基因组序列中存在两个编码BSH的基因(BSHA和BSHB),两种酶的底物特异性不同,BSHA和BSHB活性分别受到胆盐类固醇核和氨基酸侧链诱导[46]。唾液乳杆菌LMG14476的两种BSH酶(BSH1和BSH2)在催化效率和底物选择性方面被发现具有显著不同的性质[27]。植物乳杆菌WCFS1携带四个BSH基因(bsh1、bsh2、bsh3、bsh4),研究者通过异源过表达以及构建植物乳杆菌WCFS1单敲除、双敲除、三敲除和四重bsh敲除突变体,以评估单个BSH基因对不同底物的水解贡献。结果证实仅bsh1与植物乳杆菌WCFS1 BSH活性有关,bsh2、bsh3和bsh4在植物乳杆菌菌株中高度保守,这些基因在植物乳杆菌的生理和生活方式中发挥的具体作用需要进一步研究[47]。
5. 胆盐水解酶应用
5.1 胆盐水解酶在畜禽业的应用
BSH具有调节动物生长和代谢的能力,调节BSH活性可显著影响宿主的脂质代谢、体重增加等[1],严敏等研究发现,在樱桃谷肉鸭饮水中添加具有特定BSH活性乳酸菌能改善樱桃谷肉鸭42日龄胸肌肌肉品质[48]。抗生素生长促进剂(AGP)主要添加在动物饲料中以提高畜禽的生产性能,然而,抗生素的使用所造成的抗生素耐药菌株的出现威胁食品安全和公共健康,限制抗生素的使用已成全球趋势,寻找安全有效的替代品保证食用动物的生产力是当前研究者广泛关注的热点[49]。常用的抗生素生长促进剂对BSH活性都具有抑制作用[50],研究发现,BSH抑制剂对肉鸡的日增重有明显促进作用 [51]。氯化铜和硫酸锌等金属离子可以有效地抑制BSH活性[52],但金属离子及抗生素等的安全隐患不适应畜禽工业的发展。BSH在非抗生素类生长添加剂的研发中具有重要作用,Dibamehr等研究发现绿茶和甜菜根提取物对BSH具有相对抑制作用,可作为潜在的AGP替代物[21]。益生菌、有机酸、酶和植物提取物等是目前在动物生长过程中常使用的生长促进剂[53],其促生长的作用与肠道微生物密切相关,通过BSH开发和评价这些替代品的促生长效果是未来的重要的研究方向。
5.2 胆盐水解酶在食品工业的应用
结合胆汁酸对细菌有毒性,由BSH水解引起的胆汁酸谱的改变可能会改变与代谢疾病相关的胃肠道微生物群,如糖尿病和高胆固醇血症以及人类炎症性肠病[38]。益生菌是重要的功能性食品原料,BSH活性与益生菌降低胆固醇能力相关,BSH可以提高益生菌在肠道中的定殖能力并赋予其胆盐抗性使其在肠道中最大程度发挥作用,能产生胆盐水解酶的益生菌能干预胆汁酸肝肠循环,连接宿主肠道和肝脏提供了治疗相关疾病可能性[54]。因此,BSH是将食品补充剂的调节与肠道微生物群联系起来的关键中介,通过膳食补充剂-肠道微生物群通路来合理化人类健康的饮食管理[55]。目前,具有BSH活性的益生菌已应用在保健品开发,蛋乳发酵饮料的研制方面,相对于可能会带来多种副作用的药物治疗,若能高效利用益生菌降低胆固醇的作用,对于解决高胆固醇及肝胆相关疾病具有重要意义[56]。
5.3 胆盐水解酶在医药方面应用
胆汁酸类成分通常作为医药被应用于临床,如鹅去氧胆酸和熊去氧胆酸均可作为临床常用药物,用于治疗胆囊结石或胆管结石等胆道系统结石性疾病,市场需求量较大。BSH将胆盐脱结合以后会生产游离胆盐和氨基酸,如图3所示,根据BSH的这种水解作用替代传统的强碱皂化方法将动物胆汁中结合型胆汁酸制备为游离型胆汁酸,提高胆汁酸在制药工业中的利用,满足市场需求[57-58]。此外,Jia等[59]通过宏基因组分析,将肠道菌群BSH重新分为不同簇,发现不同簇BSH与人类疾病之间存在一定相关性,推测BSH在未来疾病无创诊断和潜在治疗中可作为重要标志物从而发挥作用。Khodakivsky等[60]设计了一种荧光素探针,可以实现在体内体外不同情景及多不同生物学样本BSH活性测定,可用于活体动物的实时无创成像,评估小鼠和人胃肠道中的BSH活性,在临床诊断方面具有应用前景。
6. 结语与展望
目前关于BSH的分布、结构、酶学性质及底物特异性已经有了比较成熟的研究,关于BSH认识越来越全面,BSH活性与肠道菌群及许多肝胆疾病息息相关,BSH是治疗代谢性疾病的潜在靶点,是评价新的抗生素生长促进剂替代品和筛选特定功能益生菌的有效指标,同时,BSH在一定程度上可以作为评价益生菌功效的指标,但是BSH在胃肠道菌群中的确切作用及具体机制尚不明确,益生菌中不同BSH基因、蛋白结构和基因调控还有待深入探究。彻底阐明BSH结构和功能对于其应用于未来的疾病预防和工业应用等至关重要。未来,需要更多关于BSH功能、微生物组和代谢组的研究,更加明确BSH的天然功能,更深入地了解BSH底物偏好的结构基础,合理开发低成本、安全可持续的BSH抑制剂,设计合理定制的膳食补充剂,增强动物生长性能;更好地实现以胆盐水解酶为指标快速高效有针对性的筛选益生菌株;进一步揭示BSH降胆固醇机理,通过调节宿主代谢来控制胆固醇,缓解炎症性肠病等症状。
-
表 1 部分产胆盐水解酶菌株及来源
Table 1 Microbial sources of bile salt hydrolase
菌株 来源 参考文献 植物乳杆菌(L. plantarum DGIA1) 奶酪 [8] 嗜酸乳杆菌(L. acidophilus LA15) 开菲尔颗粒 [7] 植物乳杆菌(L. plantarum WCFS1) 人体唾液 [7] 副干酪乳杆菌(L. paracasei RN025880) 传统酵素 [10] 芽孢杆菌(Bacillus polyfermenticus) 朝鲜泡菜 [11] 枯草芽孢杆菌(B. subtilis Bn1) 牛奶 [7] 克里斯滕森菌(C. minuta DSM33407) 人体 [12] 粪肠球菌(E. faecalis UK873) 人体 [7] 酵母菌(Yeasts) 绿橄榄 [13] 片球菌(Pediococcus spp) 红酒 [14] -
[1] JOYCE S A, MACSHARRY J, CASEY P G, et al. Regulation of host weight gain and lipid metabolism by bacterial bile acid modification in the gut[J]. Proceedings of the National Academy of Sciences of the United States of America,2014,111:7421−7426. doi: 10.1073/pnas.1323599111
[2] DONG Z, LEE B H. Bile salt hydrolases: Structure and function, substrate preference, and inhibitor development[J]. Protein Science,2018,27(10):1742−1754. doi: 10.1002/pro.3484
[3] KIM G B, LEE B H. Biochemical and molecular insights into bile salt hydrolase in the gastrointestinal microflora-a review[J]. Asian Australasian Journal of Animal Sciences,2005,18(10):1505−1512. doi: 10.5713/ajas.2005.1505
[4] KORPELA K, SALONEN A, VIRTA L J, et al. Intestinal microbiome is related to lifetime antibiotic use in Finnish pre-school children[J]. Nature Communications,2016(7):10410. doi: 10.1038/ncomms10410
[5] SHIN D J, WANG L. Bile acid-activated receptors: A review on fxr and other nuclear receptors[J]. Handbook of Experimental Pharmacology,2019,256:51−72.
[6] JONES B, BEGLEY M, HILL C, et al. Functional and comparative metagenomic analysis of bile salt hydrolase activity in the human gut microbiome[J]. Proceedings of the National Academy of Sciences of the United States of America,2008,105(36):13580−13585. doi: 10.1073/pnas.0804437105
[7] REYESNAVA L A, RIVERAESPINOZA Y. Isolation sources of bile salt hydrolase microorganisms[J]. Herald Journal of Agriculture and Food Science Research,2014(3):49−54.
[8] HERNÁNDEZ-GÓMEZ J G, LÓPEZ-BONILLA A, TREJO-TAPIA G, et al. In vitro bile salt hydrolase (BSH) activity screening of different probiotic microorganisms[J]. Foods,2021,10(3):674. doi: 10.3390/foods10030674
[9] TANAKA H, DOESBURG T, IWASAKI T, et al. Screening of lactic acid bacteria for bile salt hydrolase activity[J]. Journal of Dairy Science,1999,82:2530−2535. doi: 10.3168/jds.S0022-0302(99)75506-2
[10] 玛丽娜·库尔曼, 包怡红. 传统酵素源高效降胆固醇菌株的筛选、鉴定及胆盐水解酶活性分析[J]. 中国食品学报,2021,21(1):266−275. [MALINA K E M, BAO Y H. Screening, identification and analysis of efficient cholesterol-lowering strains of traditional enzyme sources and bile salt hydrolase activity[J]. Journal of Chinese Institute of Food Science and Technology,2021,21(1):266−275. [11] LEE N K, SON S H, JEON E B, et al. The prophylactic effect of probiotic Bacillus polyfermenticus KU3 against cancer cells[J]. Journal of Functional Foods,2015(14):513−518. doi: 10.1016/j.jff.2015.02.019
[12] DÉJEAN G, TUDELA H, BRUNO L, et al. Identifying a novel bile salt hydrolase from the keystone gut bacterium Christensenella minuta[J]. Microorganisms,2021,9(6):1252. doi: 10.3390/microorganisms9061252
[13] OLIVEIRA T, RAMALHOSA E, NUNES L, et al. Probiotic potential of indigenous yeasts isolated during the fermentation of table olives from northeast of Portugal[J]. Innovative Food Science & Emerging Technologies,2017,44:167−172.
[14] GARCÍA-RUIZ A, LLANO D, ESTEBAN-FERNÁNDEZ A, et al. Assessment of probiotic properties in lactic acid bacteria isolated from wine[J]. Food Microbiology,2014,44:220−225. doi: 10.1016/j.fm.2014.06.015
[15] BUSTOS A Y, SAAVEDRA L, GRACIELA F V, et al. Relationship between bile salt hydrolase activity, changes in the internal pH and tolerance to bile acids in lactic acid bacteria[J]. Biotechnology letters,2012,34(8):1511−1518. doi: 10.1007/s10529-012-0932-5
[16] 夏永军, 王光强, 王巧惠, 等. HPLC分析植物乳杆菌胆盐水解酶降解特性[J]. 工业微生物,2015,45(6):16−20. [XIA Y J, WANG G Q, WANG Q H, et al. Characterization of bile salt hydrolase degradation in Lactobacillus plantarum by HPLC[J]. Industrial Microorganisms,2015,45(6):16−20. doi: 10.3969/j.issn.1001-6678.2015.06.003 [17] HA C G, CHO J K, CHAI Y G, et al. Purification and characterization of bile salt hydrolase from Lactobacillus plantarum CK 102[J]. Journal of Microbiology & Biotechnology,2006,16(7):1047−1052.
[18] RUIZ L, SÁNCHEZ B, MARGOLLES A. Determination of bile salt hydrolase activity in bifidobacteria[J]. Methods in molecular biology (Clifton, N J),2021,2278:149−155.
[19] MOSER S A, SAVAGE D C. Bile salt hydrolase activity and resistance to toxicity of conjugated bile salts are unrelated properties in lactobacilli[J]. Applied and Environmental Microbiology,2001,67(8):3476−3480. doi: 10.1128/AEM.67.8.3476-3480.2001
[20] ROHAWI N S, RAMASAMY K, AGATONOVIC K S, et al. A new high-performance thin-layer chromatographic method for determining bile salt hydrolase activity[J]. Journal of Chromatography,2018,1092:145−151.
[21] DIBAMEHR A, DANESHYAR M, TUKMECHI A, et al. The effects of different plant extracts on bile salt hydrolase activity of Lactobacillus strains isolated from the gastrointestinal tract of poultry[J]. Veterinarski Arhiv,2021,91(1):89−99. doi: 10.24099/vet.arhiv.0887
[22] 陈仪婷, 张红星, 谢远红, 等. 降胆固醇乳酸菌的筛选鉴定及其耐酸耐胆盐性能研究[J]. 食品与发酵工业,2018,44(5):29−33. [CHEN Y T, ZHANG H X, XIE Y H, et al. Selection of cholesterol-lowering lactic acid bacteria in vitro and study on it's tolerance of acid and bile salts[J]. Food and Fermentation Industries,2018,44(5):29−33. [23] KUMAR R S, BRANNIGAN J A, PRABHUNE, A A, et al. Structural and functional analysis of a conjugated bile salt hydrolase from Bifidobacterium longum reveals an evolutionary relationship with penicillin V acylase[J]. Journal of Biological Chemistry,2006,281:32516−32525. doi: 10.1074/jbc.M604172200
[24] ROSSOCHA M, SCHULTZ-HEIENBROK R, VON MOELLER H, et al. Conjugated bile acid hydrolase is a tetrameric N-terminal thiol hydrolase with specific recognition of its cholyl but not of its tauryl product[J]. Biochemistry,2005,44:5739−5748. doi: 10.1021/bi0473206
[25] CHAND D, PANIGRAHI P, VARSHNEY N, et al. Structure and function of a highly active Bile Salt Hydrolase (BSH) from Enterococcus faecalis and post-translational processing of BSH enzymes[J]. Biochimica et Biophysica Acta (BBA)-Proteins & Proteomics,2018,1866:507−518.
[26] XU F, HU X J, SINGH W, et al. The complex structure of bile salt hydrolase from Lactobacillus salivarius reveals the structural basis of substrate specificity[J]. Scientific Reports,2019,9(1):1−11. doi: 10.1038/s41598-018-37186-2
[27] AVINASH V S, PANIGRAHI P, CHAND D, et al. Structural analysis of a penicillin V acylase from Pectobacterium atrosepticum confirms the importance of two Trp residues for activity and specificity[J]. Journal of Structural Biology,2016,193(2):85−94. doi: 10.1016/j.jsb.2015.12.008
[28] STELLWAG E J, HYLEMON P B. Purification and characterization of bile salt hydrolase from Bacteroides fragilis subsp[J]. Biochimica Et Biophysica Acta,1976,452(1):165−176. doi: 10.1016/0005-2744(76)90068-1
[29] 雷霜江, 吕青遥, 王文杰, 等. 屎肠球菌R40产胆盐水解酶发酵动力学及底物特异性分析[J]. 中国酿造,2021,40(11):144−148. [LEI S J, LV Q Y, WANG W J, et al. Fermentation kinetic and substrate specificity of bile saline hydrolase produced by Enterococcus faecium R40[J]. China Brewing,2021,40(11):144−148. doi: 10.11882/j.issn.0254-5071.2021.11.026 [30] CHAE J P, VALERIANO V D, KIM G B, et al. Molecular cloning, characterization and comparison of bile salt hydrolases from Lactobacillus johnsonii PF01[J]. Journal of Applied Microbiology,2013,114(1):121−133. doi: 10.1111/jam.12027
[31] SONG Z, CAI Y, LAO X, et al. Taxonomic profiling and populational patterns of bacterial bile salt hydrolase (BSH) genes based on worldwide human gut microbiome[J]. Microbiome,2019,7(1):9. doi: 10.1186/s40168-019-0628-3
[32] OZTURK M, ONAL C. Asparagine 79 is an important amino acid for catalytic activity and substrate specificity of bile salt hydrolase (BSH)[J]. Molecular Biology Reports,2019,46(4):4361−4368. doi: 10.1007/s11033-019-04889-2
[33] 周晓玲, 张娟, 陈坚, 等. 双歧杆菌胆盐水解酶基因的重组表达、纯化与酶学性质[J]. 食品与生物技术学报,2016,35(8):9. [ZHOU X L, ZHANG J, CHEN J, et al. Expression, purification and enzymatic properties study of the bile salt hydrolase from bifidobacterium in Escherichia coli[J]. Journal of Food Science and Biotechnology,2016,35(8):9. doi: 10.3969/j.issn.1673-1689.2016.08.002 [34] 周晓玲. 双歧杆菌胆盐水解酶基因的重组表达、纯化与酶学性质研究[D]. 江南大学, 2015. ZHOU X L. Expression, purification and enzymatic analysis of bile salt hydrolase from Bifidobacterium in Escherichia coli[D]. Wuxi: Jiangnan University, 2015.
[35] XU F ZH, GUO F F, HU X J, et al. Crystal structure of bile salt hydrolase from Lactobacillus salivarius[J]. Acta Crystallographica Section F,2016,72:376−381. doi: 10.1107/S2053273316003867
[36] ÖZTÜRK M, HACIBEYOĞLU, K, ÖNAL C, et al. Construction of R16F and D19L mutations in the loop I of bile salt hydrolase (BSH) enzyme from Lactobacillus plantarum B14 and structural and functional analysis of the mutant BSHs[J]. Food Biotechnology,2019,33(2):125−141. doi: 10.1080/08905436.2019.1570853
[37] CHAND D, AVINASH V S, YADAV Y, et al. Molecular features of bile salt hydrolases and relevance in human health[J]. Biochimica et Biophysica Acta (BBA)-General Subjects,2017,1861(1):2981−2991. doi: 10.1016/j.bbagen.2016.09.024
[38] KIM G B, YI S H, LEE B H. Purification and characterization of three different types of bile salt hydrolases from Bifidobacterium strains[J]. Journal of Dairy Science,2004,87(2):258−266. doi: 10.3168/jds.S0022-0302(04)73164-1
[39] 王建琴. 麦芽糊精促进Lactobacillus plantarum Lp-115胆盐耐受性及相关酶学性质的研究[D]. 上海: 华东理工大学, 2018. WANG J J. Maltodextrin improves bile salt tolerance of Lactobacillus plantarum Lp-115 and related enzymatic studies[D]. Shanghai: East China University of Science and Technology, 2018.
[40] ÖZTÜRK M, ÖNAL C, BA N M. Critical F129 and L138 in loop III of bile salt hydrolase (BSH) in Lactobacillus plantarum B14 are essential for the catalytic activity and substrate specificity[J]. Food Biotechnology,2019,33(4):325−337. doi: 10.1080/08905436.2019.1673172
[41] PHILEM P D, YADAV Y, SUNDER A V, et al. Structural and enzymatic analysis of a dimeric cholylglycine hydrolase like acylase active on N-acyl homoserine lactones[J]. Biochimie,2020,177:108−116. doi: 10.1016/j.biochi.2020.07.017
[42] BEGLEY M, HILL C, GAHAN C. Bile salt hydrolase activity in probiotics[J]. Applied and Environmental Microbiology,2006,72(3):1729−1738. doi: 10.1128/AEM.72.3.1729-1738.2006
[43] SARAH O, ALEXANDRA B C, THERIOT C M, et al. The Lactobacillus bile salt hydrolase repertoire reveals niche-specific adaptation[J]. Msphere,2018,3(3):e00140−18.
[44] OH H K, LEE J Y, LIM S J, et al. Molecular cloning and characterization of a bile salt hydrolase from Lactobacillus acidophilus PF01[J]. Journal of Microbiology and Biotechnology,2008,18:449−456.
[45] 任婧, 吴正钧, 王荫榆. 益生菌中胆盐水解酶作用机理研究现状[J]. 中国乳品工业,2010,38(2):47−50, 53. [REN J, WU ZH J, WANG Y Y. Research progress on mechanism of bile salt hydrolase in probiotics[J]. China Dairy Industry,2010,38(2):47−50, 53. doi: 10.3969/j.issn.1001-2230.2010.02.013 [46] MCAULIFFE O, CANO R J, KLAENHAMMER T R. Genetic analysis of two bile salt hydrolase activities in Lactobacillus acidophilus NCFM[J]. Applied & Environmental Microbiology,2005,71(8):4925−4929.
[47] LAMBERT J M, BONGERS R S, DEVOS W M, et al. Functional analysis of four bile salt hydrolase and penicillin acylase family members in Lactobacillus plantarum WCFS1[J]. Applied & Environmental Microbiology,2008,74(15):4719−4726.
[48] 严敏, 林勇, 张文, 等. 不同植物乳杆菌对樱桃谷肉鸭生长性能、血浆生化指标、屠宰性能与肌肉品质的影响[J]. 中国家禽,2021,43(5):49−54. [YAN M, LIN Y, ZHANG W, et al. Effects of different Lactobacillus plantarum on growth performance, plasma biochemical indexes, slaughter performance and meat quality of cherry valley ducks[J]. China Poultry,2021,43(5):49−54. [49] MARON D, SMITH T J, NACHMAN K E. Restrictions on antimicrobial use in food animal production: An international regulatory and economic survey[J]. Globalization and Health,2013,9(1):48. doi: 10.1186/1744-8603-9-48
[50] SMITH K, ZENG X, LIN J. Discovery of bile salt hydrolase inhibitors using an efficient high-throughput screening system[J]. PLoS ONE,2014,9(1):e85344. doi: 10.1371/journal.pone.0085344
[51] GENG W, LONG S L, CHANG Y J, et al. Evaluation of bile salt hydrolase inhibitor efficacy for modulating host bile profile and physiology using a chicken model system[J]. Scientific Reports,2020,10(1):4941. doi: 10.1038/s41598-020-61723-7
[52] WANG Z, ZENG X, MO Y, et al. Identification and characterization of a bile salt hydrolase from Lactobacillus salivarius for development of novel alternatives to antibiotic growth promoters[J]. Applied and Environmental Microbiology,2012,78(24):8795−8802. doi: 10.1128/AEM.02519-12
[53] HUYGHEBAERT G, DUCATELLE R, IMMERSEEL F V. An update on alternatives to antimicrobial growth promoters for broilers[J]. The Veterinary Journal,2011,187(2):182−188. doi: 10.1016/j.tvjl.2010.03.003
[54] 唐洪玉, 宋馨, 夏永军, 等. 益生菌中胆盐水解酶作用机制研究进展[J]. 食品与发酵工业,2020,46(13):286−292. [TANG H Y, SONG X, XIA Y J, et al. Research progress on the mechanism of bile salt hydrolase in probiotics[J]. Food and Fermentation Industries,2020,46(13):286−292. [55] YANG Y A, WU C M. Targeting gut microbial bile salt hydrolase (BSH) by diet supplements: new insights into dietary modulation of human health[J]. Food & Function, 2022. doi: 10.1039/D2FO01252A.
[56] 牟志勇, 杨昳津, 王光强, 等. 酵母菌的益生功能及在食品中的应用[J]. 食品科学,2021,42(15):309−318. [MOU Z Y, YANG Y J, WAGN G Q, et al. Beneficial effects of yeasts and applications in foods[J]. Food Science,2021,42(15):309−318. doi: 10.7506/spkx1002-6630-20200609-123 [57] 刘雁红, 胡祥正. 鸭胆膏中鹅去氧胆酸的提取工艺[J]. 天津科技大学学报,2009,24(3):43−45. [LIU Y H, HU X Z. Extraction technique of chenodeoxycholic acid from duck bile paste[J]. Journal of Tianjin University of Science & Technology,2009,24(3):43−45. [58] NOVOZYMES A. Method for producing bile acids: WO 2017/089408 Al [P]. 2017-01-06.
[59] JIA B L, PARK D, HAHN Y, et al. Metagenomic analysis of the human microbiome reveals the association between the abundance of gut bile salt hydrolases and host health[J]. Gut Microbes,2020(5):1300−1313.
[60] KHODAKIVSKYI P V, LAUBER C L, YEVTODIYENKO A, et al. Noninvasive imaging and quantification of bile salt hydrolase activity: From bacteria to humans[J]. Science Advances,2021,7(6):eaaz9857. doi: 10.1126/sciadv.aaz9857
-
期刊类型引用(3)
1. 王玉净,都治香,张霞,王旭,王娜. 沙棘黄酮通过调控TLR4/NF-κB信号通路改善大鼠多囊卵巢综合征的作用. 食品工业科技. 2024(16): 340-347 .  本站查看
本站查看
2. 王杰,常晨城,杨彦达,郭丽丽,张景萍,付绍印,石彩霞,张文广. 黄酮在反刍动物生产中的应用研究进展. 饲料研究. 2023(12): 144-147 .  百度学术
百度学术
3. 陈美庆,朱卫丰,管咏梅,冯育林,张艳丽,景秀村,彭万钱,欧阳辉,李琼. 基于UPLC-Q-TOF-MS/MS技术分析葛根配方颗粒的化学成分. 中国实验方剂学杂志. 2023(19): 176-186 .  百度学术
百度学术
其他类型引用(4)





 下载:
下载:
 下载:
下载:



