Bacterial Diversity Analysis of Sour Porridge from Ordos and Isolation and Identification of Lactic Acid Bacteria
-
摘要: 以采集自内蒙古鄂尔多斯地区的8个酸粥样品为研究对象,利用Illumina MiSeq高通量测序技术,对样品进行细菌菌群结构及多样性研究,分离鉴定其优势菌株。结果表明:8个样品中的细菌主要来自厚壁菌门(Firmicutes)和变形菌门(Proteobacteria),乳酸杆菌属(Lactobacillus)和醋酸杆菌属(Acetobacter)为优势菌属。乳酸杆菌属在各样品中占比最高,为42.63%~97.49%。SZ1、SZ2样品与其他样品的菌群结构差异较大,可能与不同采集时间有关。对上述样品中的乳酸菌进行分离纯化,通过生理生化试验结合分子生物学方法,鉴定获得1株干酪乳杆菌(Lactobacillus casei),1株发酵乳杆菌(Lactobacillus fermentum),2株植物乳杆菌(Lactobacillus plantarum)和4株副干酪乳杆菌(Lactobacillus paracasei)。上述研究结果为酸粥工业化生产提供菌种资源和理论依据。Abstract: The bacterial community structure and diversity of eight sour porridge samples collected from Ordos, Inner Mongolia were studied by Illumina Miseq high-throughput sequencing technology, and the dominant strains were isolated and identified. The results showed that the bacteria in the eight samples mainly belonged to Firmicutes and Proteobacteria. Lactobacillus and Acetobacter were the dominant genera. Lactobacillus was the highest abundance genus in all samples accounting for 42.63% to 97.49%. The structures of bacterial community in SZ1 and SZ2 differed from those in other samples, which was probably related to different sampling time. One strain was identified as Lactobacillus casei, one strain was identified as Lactobacillus fermentum, two strains were identified as Lactobacillus plantarum and four strains were identified as Lactobacillus paracasei by physiological and biochemical experiments as well as molecular biological method. The above research results provide strain resources and theoretical basis for the industrialized production of sour porridge.
-
糜米在我国多地栽培并作为主粮食用,富含蛋白质、淀粉、脂肪、维生素、矿物质等多种营养成分[1-2],在高脂饮食大鼠模型试验中具有降血脂的作用[3]。糜米发酵液,又称酸粥,是一种民间传统的谷物发酵食品,在我国陕西、山西、内蒙古等地区有着悠久的制作和食用历史[4-5]。用糜米发酵后的酸浆制得的酸粥米粒晶莹剔透,味道酸糯可口,其风味不仅取决于制作原料和生产工艺,微生物代谢产生的有机酸、氨基酸等风味物质也会影响酸粥品质的形成[6]。目前酸粥多以家庭自制为主,制作环境相对开放,蕴含着对酸粥品质形成有积极意义或具有潜在益生特性的菌株资源。对酸粥中功能菌株进行挖掘,有利于推动酸粥的工业化生产。薛建岗等[5]对内蒙古西部地区自然发酵酸粥中微生物组成进行分析,发现乳酸菌含量在7.51±1.23 lg CFU/mL。王炜宏等[7]对内蒙古鄂尔多斯地区采集的酸粥样品中的乳酸菌进行分离鉴定,表明酸粥发酵液中干酪乳杆菌(Lactobacillus casei)为优势菌群。通过分离纯化的方法,王琪等[8]从晋西北酸粥发酵液中筛选获得5株具有潜在益生特性的短乳杆菌(Lactobacillus brevis)。虽然传统微生物检测方法操作简单且可直接得到结果,但检测效率低,不能准确揭示酸粥中的物种构成和微生物多样性。以Illumina MiSeq为代表的第二代测序技术逐渐广泛应用于传统发酵食品的微生物多样性解析。如 KAVITAKE 等[9]对印度天然发酵谷物食品 “Idil” 的微生物多样性进行了研究,发现厚壁菌门(Firmicutes)和变形菌门(Proteobacteria)为优势菌门。张青等[10]采用Illumina MiSeq高通量测序技术对内蒙古巴彦淖尔地区10个酸粥样品中的细菌多样性进行解析,结果表明样品间细菌多样性和丰度存在明显差异,所有样品中的细菌隶属于9个门的55个属。对河曲和五原酸粥的研究也表明这两个地区的酸粥在菌群结构和功能上存在差异[11]。因此,将高通量测序技术与传统微生物学手段相结合,对不同地域酸粥的细菌类群进行解析,有利于揭示传统发酵食品的发酵过程与产品品质的内在联系,挖掘潜在的优良微生物资源,为传统食品工业化奠定基础。
本研究采用Illumina MiSeq高通量测序技术对采集自内蒙古鄂尔多斯地区的8个酸粥样品中的细菌多样性进行解析并对优势乳酸菌进行分离鉴定,以期揭示自然发酵酸粥中细菌的菌群特征,为酸粥的安全生产提供理论指导,同时为筛选获得发酵酸粥的优良菌株提供基础。
1. 材料与方法
1.1 材料与仪器
酸粥 采集自内蒙古自治区鄂尔多斯市达拉特旗8户普通家庭的8个样品,分别编号为SZ1~SZ8,其中 SZ1 和 SZ2 采集时间为 2020 年 10 月,SZ3~SZ8 采集时间为 2021 年 2 月;1 U/μL KOD FX Neo酶(TOYOBO) 北京百灵克生物科技有限公司;TGuide S96磁珠法土壤/粪便基因组DNA提取试剂盒 天根生化科技(北京)有限公司;乳酸菌成套生化鉴定管 青岛高科技工业园海博生物技术有限公司;MRS肉汤 北京奥博星生物技术有限责任公司;2×Taq Master Mix、PCR产物纯化试剂盒 上海捷瑞生物工程有限公司;琼脂糖 北京鼎国昌盛生物技术有限责任公司;GoldView核酸染料 天津为科生物技术有限公司;DL 2000 DNA Marker 宝日医生物技术(北京)有限公司。
SW-CJ-1FD型超净工作台 上海苏净实业有限公司;LDZX-50KB型立式高压蒸汽灭菌锅 上海申安医疗器械厂;DHG-9245A型电热鼓风干燥箱 上海一恒科学仪器有限公司;AX224ZH型电子天平、ST3100型pH计 奥豪斯仪器(常州)有限公司;DYY-7C型水平电泳仪 北京市六一仪器厂;YLN-III型暗箱式紫外透射仪 北京亚力恩机电技术研究所;Veriti 96型梯度基因扩增仪 Applied Biosystems公司;YS100型光学显微镜 尼康仪器(中国)有限公司;GSP-9160MBE隔水式恒温培养箱 上海博讯实业有限公司。
1.2 实验方法
1.2.1 细菌基因组DNA提取
根据DNA提取试剂盒说明书提取酸粥发酵液细菌基因组DNA,采用1%琼脂糖凝胶电泳检测DNA的浓度及完整性。
1.2.2 PCR扩增及高通量测序
以16S rRNA的V3~V4区为目标DNA序列,采用通用上游引物338F(5'-ACTCCTACGGGAGGCAGCAG-3')和下游引物806R(5'-GGACTACHVGGGTWTCTAAT-3')进行扩增。PCR反应体系:基因组DNA 50 ng,上、下游引物(10 μmol/L)各0.3 μL,dNTP(各2 mmol/L)2 μL,KOD FX Neo酶0.2 μL,ddH2O补至10 μL。PCR反应条件:95 ℃预变性5 min;95 ℃变性30 s,50 ℃延伸30 s,72 ℃退火1 min,30个循环;72 ℃延伸5 min。回收纯化的产物由北京百迈客生物科技有限公司进行高通量测序。
1.2.3 测序数据分析
使用Trimmomatic 0.33软件,对原始序列(Raw Reads)进行过滤,使用Cutadapt 1.9.1软件进行引物序列的识别与去除,得到不包含引物序列的原始序列,质控后得到高质量序列(Clean Reads);使用Usearch 10软件,对每个样品的Clean Reads进行双端序列拼接和过滤;使用UCHIME 4.2软件鉴定并去除嵌合体序列,得到最终有效序列(Effective Reads)。用质量值≥20的碱基占总碱基数的百分比(Q20)和质量值≥30的碱基占总碱基数的百分比(Q30)评价测序的准确度。通过Usearch软件在97%的相似度水平下划分操作分类单元(OTU),根据OTU的序列组成得到其物种分类。以SILVA为参考数据库用Mothur方法进行分类学注释,利用QIIME软件计算ACE、Chao1、Shannon及Simpson指数,使用R软件绘制样品稀释曲线、香农曲线、进行Alpha多样性分析和Beta多样性分析,并绘制PCoA图。
1.2.4 pH的测定
采用ST3100型pH计进行测定。
1.2.5 乳酸菌菌落计数
参考GB 4789.35-2016《食品安全国家标准 食品微生物学检验 乳酸菌检验》[12]。
1.2.6 乳酸菌的分离和初步筛选
在含有1%碳酸钙的MRS固体培养基上,挑取形成透明圈的菌落,进行革兰氏染色、显微镜观察及过氧化氢酶反应,选取革兰氏阳性且过氧化氢酶反应阴性的菌株[13],初步筛选乳酸菌。
1.2.7 乳酸菌的生理生化鉴定
参考乳酸菌成套生化鉴定管使用说明,将MRS固体培养基上单一供试菌落分别接种于生化鉴定管中,将接种后的生化管置于37 ℃恒温培养箱中,48 h后观察结果并记录。
1.2.8 乳酸菌的16S rDNA鉴定
按照细菌基因组DNA快速提取试剂盒说明书,进行乳酸菌基因组提取。以提取的基因组DNA为模板,以27F(5'-AGAGTTTGATCMTGGCTCAG-3')和1492R(5'-ACCTTGTTACGACTT-3')为引物,对菌株进行PCR扩增,反应体系:DNA模板1 μL,上、下游引物(10 μmol/L)各1 μL,2×Taq Master Mix 12.5 μL,ddH2O补至25 μL。反应条件:95 ℃预变性5 min;95 ℃变性40 s,57 ℃退火30 s,72 ℃延伸2 min,35个循环;72 ℃延伸10 min。将纯化后的PCR产物送至生工生物工程(上海)股份有限公司测序。测序结果与NCBI数据库(National Center for Biotechnology Information)中的参考菌株基因序列进行同源性比对。
1.3 数据处理
数据平行测定3次,使用SPSS 22.0软件对数据进行统计学分析,以平均值±标准偏差(standard deviation,SD)表示,P<0.05表示存在显著差异。
2. 结果与分析
2.1 酸粥样品高通量测序数据分析
如表1所示,本研究中的8个样品平均产生79845条高质量序列数,每个样品至少产生79492条高质量序列。样品平均序列长度在416~429 bp之间,GC碱基占总碱基的百分比均大于50%;Q20在98.92%~99.40%之间,Q30在95.70%~97.23%之间,测序准确度高,可进行后续数据处理。
表 1 酸粥样品高通量测序结果Table 1. High-throughput sequencing data of sour porridge samples样品名称 原始序列数 高质量序列数 有效序列数 样品平均序列长度(bp) GC(%) Q20(%) Q30(%) SZ1 79915 79554 75123 423 52.76 98.92 95.70 SZ2 80121 79763 78951 416 52.77 99.00 95.89 SZ3 80129 79938 76016 429 51.70 99.29 96.80 SZ4 79925 79767 78243 422 53.00 99.38 97.23 SZ5 79652 79492 78233 428 52.14 99.31 96.95 SZ6 80224 80047 77922 419 53.40 99.39 97.21 SZ7 80359 80179 76030 428 51.99 99.30 96.90 SZ8 80173 80019 76593 422 51.97 99.40 97.20 2.2 稀释性曲线和香农曲线
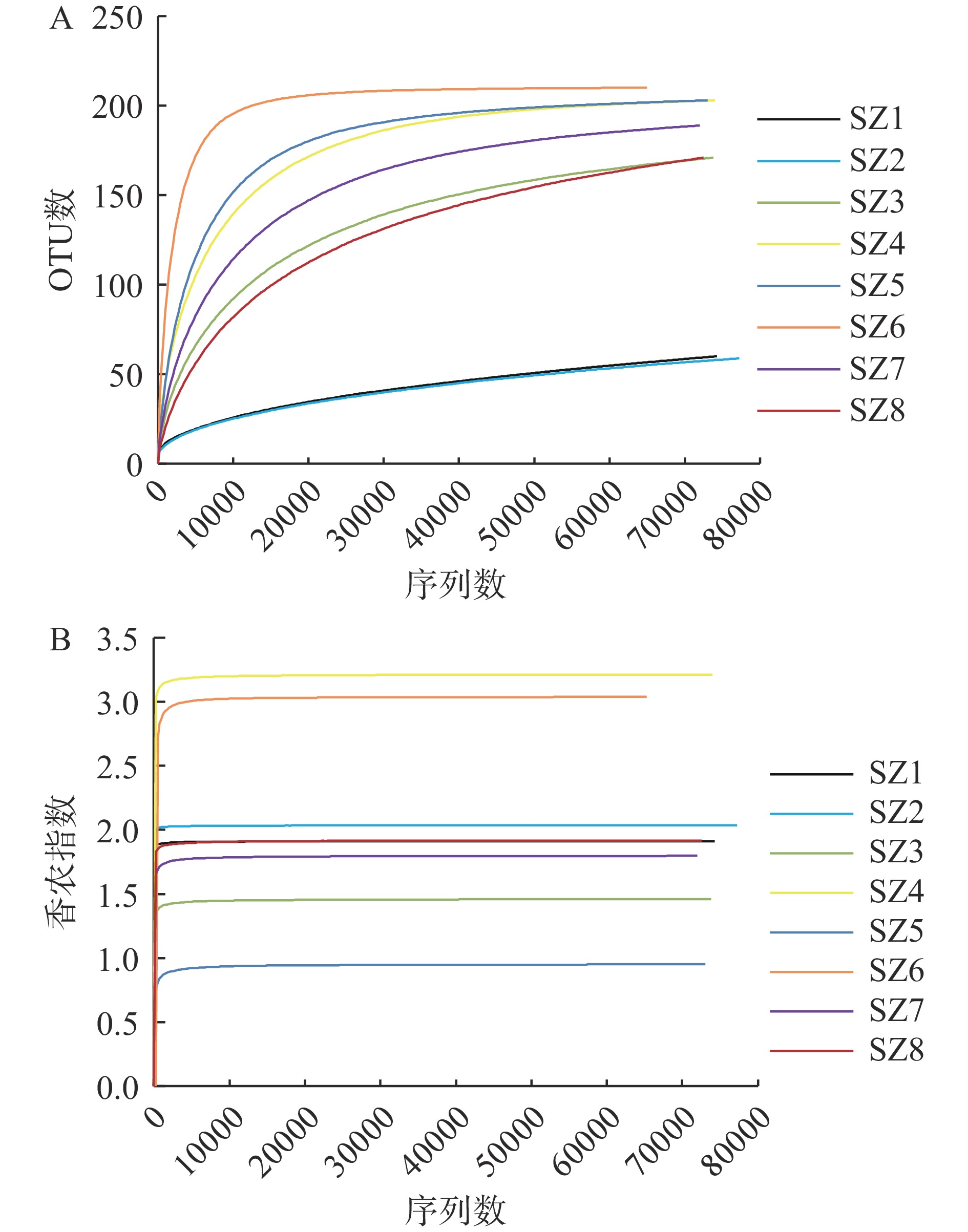
由图1A可知,酸粥发酵液样品中,序列数在0~10000时OTU数目增长最快,随着序列数的增加OTU数目趋于平缓,说明测序量充分反映样品中大部分的微生物多样性。由图1B可知,各样品随着测序量的增加,香农曲线均趋向平坦,菌群多样性不再发生明显变化,说明测序深度合适,可满足后续分析要求。其中SZ4趋于饱和时香农指数值最大,微生物多样性最高,其次为SZ6,SZ5趋于饱和时香农指数值最低。
2.3 Alpha多样性指数分析
表2中8个样品的文库覆盖率均接近于1,说明样品测序深度良好,覆盖率高,可以真实展示样品中的细菌种类[14]。在97%相似度条件下获得OTU,其中SZ1与SZ2样品的OTU数分别为60和59,明显低于其他样本,SZ3和SZ8的OTU数均为171,SZ7的OTU数为189,SZ4~SZ6样品中的OTU数均超过200,其中SZ6样品的OTU数最高,为210。上述结果说明,不同采样时间的酸粥样品中微生物多样性存在差异,可能与环境条件有关。通过Alpha多样性对样品的微生物群落进行丰度和多样性分析,ACE和Chao1指数的数值越高,表明菌群丰富度越高[15]。在覆盖率大于99%的条件下,SZ6的ACE和Chao1指数均高于其他样品,说明其物种数丰富度最高,而SZ1和SZ2的ACE和Chao1指数明显低于其他样品。Simpson和Shannon指数反映了菌群的多样性,呈正相关[16]。SZ4的Simpson和Shannon指数均高于其他样品,说明SZ4的物种多样性最高,而SZ5的Simpson和Shannon指数最低,物种多样性程度最低。
表 2 酸粥样品的Alpha多样性指数分析Table 2. Analysis of Alpha diversity indexes of sour porridge samples样品名称 OTU数 ACE Chao1 Simpson Shannon 覆盖率(%) SZ1 60 128.64 93.33 0.69 1.91 99.97 SZ2 59 123.70 84.30 0.70 2.04 99.97 SZ3 171 187.89 192.75 0.44 1.46 99.96 SZ4 203 205.35 205.40 0.83 3.21 99.99 SZ5 203 205.44 207.50 0.17 0.95 99.99 SZ6 210 210.16 210.00 0.75 3.04 100.00 SZ7 189 197.01 199.06 0.57 1.80 99.97 SZ8 171 204.95 202.89 0.69 1.92 99.94 2.4 物种分类与注释
图2A所示为门水平的细菌群落结构,厚壁菌门占绝对优势,各样品中的丰度在42.66%~98.16%之间,特别是在SZ3、SZ5和SZ7中的占比超过95%。其次为变形菌门,在SZ2、SZ4和SZ6中的占比超过40%。各样品中相对丰度大于1%的菌门还包括蓝细菌门(Cyanobacteria)、放线菌门(Actinobacteria)和拟杆菌门(Bacteroidetes)。在属水平的物种分布柱状图(图2B)中,SZ1~SZ8相对丰度大于1%的菌属分别有2、5、1、5、1、3、1和2个。乳酸杆菌属在各样品中占比最高,在42.63%~97.49%之间,特别是SZ3、SZ5和SZ7中乳酸杆菌属的丰度均在90%以上。醋酸杆菌属在SZ1、SZ2、SZ4、SZ6和SZ8中的占比均高于20%,但在SZ3、SZ5和SZ7中的占比不足1%。乳酸菌作为食品发酵剂能够代谢产生氨基酸、维生素、有机酸、芳香族化合物、酶类等[17],改善产品的质地和感官特征,提高保藏性和安全性[18]。醋酸菌除了生成乙酸外,还能产生多种有机酸[19],应用于食品、医药、化工等领域。研究表明,酸粥发酵过程中酒石酸、醋酸、乳酸含量均与乳酸杆菌和醋酸杆菌呈正相关[20]。国内对酸粥的研究中也有乳酸菌和醋酸菌的报道,例如张青等[10]和WANG等[21]发现乳酸杆菌属和醋酸杆菌属为内蒙古地区酸粥中的主要细菌,常健[22]发现晋西北酸粥中乳酸菌和醋酸菌的丰度可占细菌总量的91.74%。研究表明,山西和内蒙古地区的酸粥样品中醋酸杆菌属丰度显著高于广西样品,而芽孢杆菌属(Bacillus)丰度显著低于广西样品[21],说明菌群组成差异可能与采样地区、环境、制作方式等相关。
2.5 PCoA分析
基于非加权unifrac分析不同样品之间细菌群落的差异并用PCoA显示Beta多样性,样品物种组成越相似,反应在图中的距离越近[23]。由图3可知,基于第一主成分(PC1)的贡献率为78.97%,基于第二主成分(PC2)的贡献率为16.33%,两主成分之和为95.30%,表明这两个主成分很好地代表了样品中的细菌群落信息。由此可以推断8个样品中SZ1、SZ2相较于其他样品的菌落结构差异明显,可能来自于一些含量较低的特有菌群。上述结果说明,酸粥的采样时间可能会对菌群多样性产生影响。
2.6 乳酸菌菌株的分离
乳酸菌可以利用不同底物进行发酵,制成各类发酵食品,因此传统发酵食品中蕴含着丰富的乳酸菌资源。例如LI等[24]从新疆传统发酵乳制品中分离获得的瑞士乳杆菌(Lactobacillus helveticus)和嗜热链球菌(Stresptococcus thermophilus)可作为生产高粘度发酵乳的发酵剂;TIAN等[25]从西藏地区开菲尔粒中筛选获得产乙醛和双乙酰的植物乳杆菌(Lactobacillus plantarum)以改善发酵乳制品的风味;陈雨等[26]从西双版纳传统腌制品中筛选获得高产乳酸的植物乳杆菌,可为规模化生产乳酸提供优良菌种;孟宪刚等[27]从浆水中分离获得的乳酸菌具有降胆固醇和降亚硝酸盐的能力,可用于乳酸菌制剂的开发和利用。本研究以酸粥为研究对象,筛选获得优势乳酸菌。由表3可知,8个酸粥样品的pH在3.49~4.52之间,乳酸菌菌落总数在4.18~6.78 lg CFU/mL,其中SZ4的菌落总数最高,SZ5、SZ6和SZ8的菌落总数显著低于其他样品(P<0.05)。如图4所示,筛选获得8株有明显溶钙圈的乳酸菌菌株,观察其菌落隆起、表面光滑、边缘整齐、呈乳白色、不透明,经革兰氏染色后,菌株细胞均呈紫色杆状(图5)。
表 3 酸粥pH及乳酸菌的菌落总数Table 3. The pH of sour porridge samples and the total number of lactic acid bacteria colonies样品名称 pH 乳酸菌菌落总数(lg CFU/mL) SZ1 3.49±0.02c 6.02±0.56d SZ2 3.51±0.01c 6.08±0.47d SZ3 4.03±0.02b 6.65±0.38c SZ4 4.01±0.03b 6.78±0.26a SZ5 4.52±0.03a 4.18±0.71e SZ6 3.98±0.01b 5.20±0.64e SZ7 4.01±0.03b 6.70±0.39b SZ8 4.01±0.01b 5.51±0.46e 注:同列不同小写字母表示差异显著(P<0.05)。 2.7 乳酸菌菌株的生理生化鉴定
由表4可知,分离获得的8株菌株皆能水解七叶苷和水杨苷,并且皆可利用纤维二糖、麦芽糖、蔗糖、菊糖和乳糖。除L813的其他菌株可利用甘露醇和山梨醇,初步判断L813为嗜酸乳杆菌(Lactobacillus acidophilus)或发酵乳杆菌(Lactobacillus fermentum)[28];L24和L218菌株还可利用棉子糖,可初步判断为植物乳杆菌(Lactobacillus plantarum)[29];其余菌株为干酪乳杆菌或副干酪乳杆菌(Lactobacillus paracasei)[29-30]。
表 4 乳酸菌的生理生化鉴定结果Table 4. Physiological and biochemical identification results of lactic acid bacteria菌株
名称来源 七叶
苷纤维
二糖麦芽
糖甘露
醇水杨
苷山梨
醇蔗糖 棉子
糖菊糖 乳糖 L140 SZ1 + + + + + + + − + + L24 SZ2 + + + + + + + + + + L218 SZ2 + + + + + + + + + + L353 SZ3 + + + + + + + − + + L520 SZ5 + + + + + + + − + + L74 SZ7 + + + + + + + − + + L813 SZ8 + + + − + − + − + + A117 SZ1 + + + + + + + − + + 注:“+”表示结果呈阳性;“−”表示结果呈阴性。 2.8 乳酸菌菌株的16S rDNA鉴定
由表5可知,从采集的8个样品中分离获得的8株乳酸菌中,L140与参考菌株干酪乳杆菌的同源性为99.79%,L24和L218与参考菌株植物乳杆菌的同源性分别为99.79%和99.93%,L353、L520、L74和A117与参考菌株副干酪乳杆菌的同源性在99.7%以上,L813与参考菌株发酵乳杆菌的同源性为99.73%。
表 5 乳酸菌的16S rDNA序列同源性分析Table 5. 16S rDNA sequence homology analysis of lactic acid bacteria菌株名称 鉴定结果 同源性(%) NCBI登录号 L140 Lactobacillus casei 99.79 9W0ZB1H3016 L24 Lactobacillus plantarum 99.79 EHFNJHFC016 L218 Lactobacillus plantarum 99.93 9W1GPU97016 L353 Lactobacillus paracasei 99.79 EHG55D2H013 L520 Lactobacillus paracasei 99.97 9W1YJJPB01R L74 Lactobacillus paracasei 99.86 9W22CWAC016 L813 Lactobacillus fermentum 99.73 EHGAFDAN013 A117 Lactobacillus paracasei 100 9W2A3P4501R 3. 结论
本研究基于Illumina MiSeq高通量测序技术分析鄂尔多斯地区8个酸粥样品中细菌多样性,同时对酸粥中乳酸菌进行分离鉴定。研究发现SZ1、SZ2与其他样品的菌群结构差异较大,SZ4的物种多样性最高,SZ6的OTU数最高。酸粥中细菌主要来自厚壁菌门,各样品中的丰度在42.66%~98.16%之间,其次为变形菌门。乳酸杆菌属为优势菌属,占比在42.63%~97.49%之间,其次为醋酸杆菌属。通过分离鉴定,获得8株乳酸杆菌,其中1株为干酪乳杆菌,1株为发酵乳杆菌,2株为植物乳杆菌,4株为副干酪乳杆菌。上述研究结果为酸粥产品生产工艺优化提供菌株资源。
-
表 1 酸粥样品高通量测序结果
Table 1 High-throughput sequencing data of sour porridge samples
样品名称 原始序列数 高质量序列数 有效序列数 样品平均序列长度(bp) GC(%) Q20(%) Q30(%) SZ1 79915 79554 75123 423 52.76 98.92 95.70 SZ2 80121 79763 78951 416 52.77 99.00 95.89 SZ3 80129 79938 76016 429 51.70 99.29 96.80 SZ4 79925 79767 78243 422 53.00 99.38 97.23 SZ5 79652 79492 78233 428 52.14 99.31 96.95 SZ6 80224 80047 77922 419 53.40 99.39 97.21 SZ7 80359 80179 76030 428 51.99 99.30 96.90 SZ8 80173 80019 76593 422 51.97 99.40 97.20 表 2 酸粥样品的Alpha多样性指数分析
Table 2 Analysis of Alpha diversity indexes of sour porridge samples
样品名称 OTU数 ACE Chao1 Simpson Shannon 覆盖率(%) SZ1 60 128.64 93.33 0.69 1.91 99.97 SZ2 59 123.70 84.30 0.70 2.04 99.97 SZ3 171 187.89 192.75 0.44 1.46 99.96 SZ4 203 205.35 205.40 0.83 3.21 99.99 SZ5 203 205.44 207.50 0.17 0.95 99.99 SZ6 210 210.16 210.00 0.75 3.04 100.00 SZ7 189 197.01 199.06 0.57 1.80 99.97 SZ8 171 204.95 202.89 0.69 1.92 99.94 表 3 酸粥pH及乳酸菌的菌落总数
Table 3 The pH of sour porridge samples and the total number of lactic acid bacteria colonies
样品名称 pH 乳酸菌菌落总数(lg CFU/mL) SZ1 3.49±0.02c 6.02±0.56d SZ2 3.51±0.01c 6.08±0.47d SZ3 4.03±0.02b 6.65±0.38c SZ4 4.01±0.03b 6.78±0.26a SZ5 4.52±0.03a 4.18±0.71e SZ6 3.98±0.01b 5.20±0.64e SZ7 4.01±0.03b 6.70±0.39b SZ8 4.01±0.01b 5.51±0.46e 注:同列不同小写字母表示差异显著(P<0.05)。 表 4 乳酸菌的生理生化鉴定结果
Table 4 Physiological and biochemical identification results of lactic acid bacteria
菌株
名称来源 七叶
苷纤维
二糖麦芽
糖甘露
醇水杨
苷山梨
醇蔗糖 棉子
糖菊糖 乳糖 L140 SZ1 + + + + + + + − + + L24 SZ2 + + + + + + + + + + L218 SZ2 + + + + + + + + + + L353 SZ3 + + + + + + + − + + L520 SZ5 + + + + + + + − + + L74 SZ7 + + + + + + + − + + L813 SZ8 + + + − + − + − + + A117 SZ1 + + + + + + + − + + 注:“+”表示结果呈阳性;“−”表示结果呈阴性。 表 5 乳酸菌的16S rDNA序列同源性分析
Table 5 16S rDNA sequence homology analysis of lactic acid bacteria
菌株名称 鉴定结果 同源性(%) NCBI登录号 L140 Lactobacillus casei 99.79 9W0ZB1H3016 L24 Lactobacillus plantarum 99.79 EHFNJHFC016 L218 Lactobacillus plantarum 99.93 9W1GPU97016 L353 Lactobacillus paracasei 99.79 EHG55D2H013 L520 Lactobacillus paracasei 99.97 9W1YJJPB01R L74 Lactobacillus paracasei 99.86 9W22CWAC016 L813 Lactobacillus fermentum 99.73 EHGAFDAN013 A117 Lactobacillus paracasei 100 9W2A3P4501R -
[1] 王强. 糜米粉对面团特性及其面包品质影响研究[D]. 天津: 天津科技大学, 2019. WANG Q. Studies on the effect of proso millet flour on dough properties and bread qualities[D]. Tianjin: Tianjin University of Science and Technology, 2019.
[2] 马薇薇. 糜米馒头关键技术及蛋清粉对其品质影响研究[D]. 天津: 天津科技大学, 2020. MA W W. Studies on the key technology of proso millet steamed bread and the effect of egg white powder on its quality[D]. Tianjin: Tianjin University of Science and Technology, 2020.
[3] 王吉力特, 刘聪, 夏美茹, 等. 糜米对高脂饮食大鼠血脂的影响[J]. 粮食与油脂,2022,35(1):30−34. [WANG J L T, LIU C, XIA M R, et al. Effects of Panicum miliaceum L. on blood lipid in rats fed with high-fat diet[J]. Cereals & Oils,2022,35(1):30−34. doi: 10.3969/j.issn.1008-9578.2022.01.008 [4] 王凤玲, 刘爱国, 李娜, 等. 糜米发酵液中优势乳酸菌分离鉴定[J]. 食品科学,2007,28(11):342−345. [WANG F L, LIU A G, LI N, et a. Isolation and identification of properties of dominant biology lactic acid bacteria form mimi fermented liquid[J]. Food Science,2007,28(11):342−345. doi: 10.3321/j.issn:1002-6630.2007.11.078 [5] 薛建岗, 陈永福, 于海静, 等. 内蒙古西部地区自然发酵酸粥化学成分及微生物组成分析[J]. 食品科技,2013,38(7):10−14. [XUE J G, CHEN Y F, YU H J, et al. Analysis of chemical and microbiological composition in naturally fermented acidic-gruel from western areas of Inner Mongolia[J]. Food Science and Technology,2013,38(7):10−14. doi: 10.13684/j.cnki.spkj.2013.07.035 [6] 张悦, 张川, 李中媛, 等. 传统酸粥制备工艺的优化[J]. 食品工业,2020,41(1):128−132. [ZHANG Y, ZHANG C, LI Z Y, et al. Optimization of traditional suanzhou preparation process[J]. The Food Industry,2020,41(1):128−132. [7] 王炜宏, 杜晓华, 张家超, 等. 内蒙古鄂尔多斯地区酸粥发酵液中乳酸菌的分离鉴定[J]. 食品与生物技术学报,2010,29(2):266−270. [WANG W H, DU X H, ZHANG J C, et al. Isolation and identification of LAB from naturally fermented congee in Ordos area of Inner Mongolia[J]. Journal of Food Science and Biotechnology,2010,29(2):266−270. [8] 王琪, 刘聪, 景彦萍, 等. 晋西北酸粥发酵液中乳酸菌的潜在益生特性[J]. 食品与发酵工业,2019,45(2):60−66. [WANG Q, LIU C, JING Y P, et al. Probiotic properties of LAB isolated from fermentation broth of sour porridge in Northwestern Shanxi[J]. Food and Fermentation Industries,2019,45(2):60−66. [9] KAVITAKE D, SURYAVANSHI M V, KANDASAMY S, et al. Bacterial diversity of traditional fermented food, Idli by high thorough-put sequencing[J]. Journal of Food Science and Technology, 2022.
[10] 张青, 郭淑文, 苏靖, 等. 基于高通量测序技术巴彦淖尔市酸粥中细菌多样性分析[J]. 中国酿造,2020,39(9):101−105. [ZHANG Q, GUO S W, SU J, et al. Bacterial diversity in sour porridge from Bayannur based on high-throughput sequencing[J]. China Brewing,2020,39(9):101−105. doi: 10.11882/j.issn.0254-5071.2020.09.019 [11] 张青, 石晶红. 山西河曲与内蒙古五原酸粥细菌类群结构及其基因功能的比较分析[J/OL]. 食品与发酵工业, 2022. DOI: 10.13995/j.cnki.11-1802/ts.032385. ZHANG Q, SHI J H. Comparative analysis of bacterial group structure and gene function between Hequ sour porridge and Wuyuan sour porridge[J/OL]. Food and Fermentation Industries, 2022. DOI: 10.13995/j.cnki.11-1802/ts.032385.
[12] 中华人民共和国卫生部. GB 4789.35-2016食品微生物学检验 乳酸菌检验[S]. 北京: 中国标准出版社, 2016. Ministry of Health of the People's Republic of China. GB 4789.35-2016 Food microbiological examination Lactic acid bacteria[S]. Beijing: Standards Press of China, 2016.
[13] 王素华. 新平腌菜中乳酸菌的筛选、特性及应用研究[D]. 天津: 天津农学院, 2020. WANG S H. Screening, characterization and application of lactic acid bacteria from Xinping pickles[D]. Tianjin: Tianjin Agricultural University, 2020.
[14] 虞任莹, 彭思佳, 李艺, 等. 梅干菜和腌制雪菜细菌菌群多样性分析[J]. 食品工业科技,2021,42(18):134−141. [YU R Y, PENG S J, LI Y, et al. Analysis of bacterial diversity of the pickled and dried mustard and the fermented potherb mustard[J]. Science and Technology of Food Industry,2021,42(18):134−141. doi: 10.13386/j.issn1002-0306.2021010065 [15] ZHANG X M, DANG X J, WANG Y B, et al. Diversity and composition of microbiota during fermentation of traditional Nuodeng ham[J]. Journal of Microbiology,2021,59(1):20−28. doi: 10.1007/s12275-021-0219-4
[16] GRICE E A, KONG H H, CONLAN S, et al. Topographical and temporal diversity of the human skin microbiome[J]. Science,2009,324(5931):1190−1192. doi: 10.1126/science.1171700
[17] CHEN W. Lactic acid bacteria[M]. Singapore: Springer, 2019: 181−209.
[18] 吴燕. 特色自然发酵浆水中乳酸菌的筛选、表征及应用[D]. 无锡: 江南大学, 2019. WU Y. Screening, characterization and application of lactic acid bacteria in characteristic natural fermented Jiangshui[D]. Wuxi: Jiangnan University, 2019.
[19] 牟俊. 山西老陈醋醋酸发酵阶段乳酸菌和醋酸菌相互作用研究[D]. 天津: 天津科技大学, 2018. MOU J. Interaction between lactic acid bacteria and acetic acid bacteria in the acetic acid fermentation process of Shanxi aged vinegar[D]. Tianjin: Tianjin University of Science and Technology, 2018.
[20] GUO H, SUN Z, HAO Y, et al. Correlation between bacterial communities and organic acids in the fermentation stage of traditional Chinese sour porridge[J]. International Journal of Food Properties,2020,23(1):1430−1440. doi: 10.1080/10942912.2020.1801724
[21] WANG Y R, HOU Q C, GUO Z, et al. Bacterial community structure in acidic gruel from different regions and its influence on quality[J]. Food Research International,2021,141:110130. doi: 10.1016/j.foodres.2021.110130
[22] 常健. 晋西北酸粥发酵过程中细菌种群分析及乳酸菌、醋酸菌的分离鉴定[D]. 太原: 山西大学, 2016. CHANG J. Bacterial population analysis, isolation and identification of lactic acid bacteria and acetic acid bacteria during the fermentation of Northwestern Shanxi acid porridge[D]. Taiyuan: Shanxi University, 2016.
[23] LIU W J, ZHENG Y, KWOK L Y, et al. High-throughput sequencing for the detection of the bacterial and fungal diversity in Mongolian naturally fermented cow’s milk in Russia[J]. BMC Microbiology,2015,15:45. doi: 10.1186/s12866-015-0385-9
[24] LI W, MUTUVULLA M, CHEN X, et al. Isolation and identification of high viscosity-producing lactic acid bacteria from a traditional fermented milk in Xinjiang and its role in fermentation process[J]. European Food Research and Technology,2012,235:497−505. doi: 10.1007/s00217-012-1779-7
[25] TIAN H X, SHI Y H, ZHANG Y, et al. Screening of aroma-producing lactic acid bacteria and their application in improving the aromatic profile of yogurt[J]. Journal of Food Biochemistry,2019,43(10):12837.
[26] 陈雨, 吉巧琳, 李智昊, 等. 高产乳酸菌株的筛选、鉴定及发酵条件的研究[J/OL]. 云南民族大学学报(自然科学版), 2023(2): 1−14. https://kns.cnki.net/kcms/detail/53.1192.N.20220426.0827.002.html. CHEN Y, JI Q L, LI Z H, et al. Screening, identification and fermentation conditions of high yield kactic acid bacteria[J/OL]. Journal of Yunnan Minzu University (Natural Sciences Edition), 2023(2): 1−14. https://kns.cnki.net/kcms/detail/53.1192.N.20220426.0827.002.html.
[27] 孟宪刚, 李雪萍, 李建宏, 等. 浆水中乳酸菌分离鉴定及其代谢特性的初步研究[J]. 食品工业科技,2015,36(1):181−186, 210. [MENG X G, LI X P, LI J H, et al. Isolation and identification of lactic acid bacteria from Jiangshui and preliminary study on metabolic characteristics[J]. Science and Technology of Food Industry,2015,36(1):181−186, 210. [28] 王琳珺, 潘道东, 杨媛, 等. 鱼类肠道嗜酸乳杆菌的分离及功能特性分析[J]. 中国食品学报,2014,14(5):20−25. [WANG L J, PAN D D, YANG Y, et al. Isolation and functional analysis of Lactobacillus acidophilus from fish intestine[J]. Journal of Chinese Institute of Food Science and Technology,2014,14(5):20−25. [29] 张文平, 赵英杰, 罗晟, 等. 高产胞外多糖植物乳杆菌筛选及其发酵工艺优化[J]. 食品与发酵工业,2019,45(21):38−45. [ZHANG W P, ZHAO Y J, LUO S, et al. Screening of Lactobacillus plantarum with higher yield of exopolysaccharides and optimization of fermentation conditions[J]. Food and Fermentation Industries,2019,45(21):38−45. doi: 10.13995/j.cnki.11-1802/ts.021589 [30] 余萍, 曹蓝, 矫艳平, 等. 副干酪乳杆菌HCS17-040的筛选鉴定及其益生特性[J]. 食品与机械,2021,37(11):28−33, 129. [YU P, CAO L, JIAO Y P, et al. Screening of Lactobacillus paracasei HCS17-040 with triglyceride tunction and study on its probiotic characteristics[J]. Food & Machinery,2021,37(11):28−33, 129.





 下载:
下载:



 下载:
下载:



