Modification of Substrate Affinity of Nitrile Hydratase Based on Amino Acid Hotspot Mutation
-
摘要: 目的:以Rhodococcus erythropolis CCM2595来源的腈水合酶为研究对象,利用半理性设计以及定点突变以获取其对烟腈底物亲和力更高的突变体。方法:通过序列比对找到同源性很高的1AHJ蛋白并采用Swiss-Model和iTASSER等软件进行评估。利用Discovery Studio 2016(DS)将1AHJ与烟腈进行分子对接并进行虚拟氨基酸突变,预测并筛选出亲和力明显提升的氨基酸突变位点。构建突变重组质粒转化至大肠杆菌表达感受态中进行异源表达,并通过高效液相色谱检测分析对烟腈的生物转化情况。结果:通过虚拟氨基酸突变,得到腈水合酶α亚基CYS113突变为TYR(C113Y)、CYS115突变为ASN(C115N)、β亚基VAL52突变为ARG(V52R)后的三位点突变体,酶纯化后通过酶促反应常数分析发现,与野生型相比突变体Km值由突变前的16.78 mmol/L降低为12.69 mmol/L,酶活由12.14 U/mL提高到15.15 U/mL。结论:通过虚拟氨基酸突变得到的腈水合酶三突变体ReNHase C113Y /C115N/ V52R对烟腈的底物亲和力提高了24.37%,酶活提高了24.79%,以上结果为烟腈工业化应用的提高提供了新的理论依据。Abstract: Objective: A semi-rational design was used to increase the affinity of the nitrile hydratase (ReNHase) derived from Rhodococcus erythropolis CCM2595 with the substrate nicotinonitrile. Methods: The 1AHJ protein with high homology was found through sequence comparison and evaluated by software Swiss-Model and iTASSER. The molecular docking of nicotinonitrile with 1AHJ was then performed with Discovery Studio 2016 (DS), which aimed to obtain the virtual amino acid mutations with significantly improved affinity. The mutant recombinant plasmid was then constructed and transformed into E. coli expression competent cells for heterologous expression. After the purification of mutant ReNHase from the recombined E. coli, the biotransformation of nicotinonitrile was detected and analyzed by high performance liquid chromatography. Results: According to predicting the calculate mutation energy (Binding) of nicotinonitrile with ReNHase, CYS113 and CYS115 of αsubunit were mutated to TYR (C113Y) and ASN (C115N), VAL52 of βsubunit was mutated to ARG (V52R). According to the kinetic parameters of the reaction by the purified ReNHase followed the Michaelis–Menten model, the Km value of mutant ReNHase C113Y /C115N/ V52R decreased from 16.78 mmol/L to 12.69 mmol/L when compared with wild ReNHase, the enzyme activity increased from 12.14 U/mL to 15.15 U/mL. Conclusion: Compared with wild ReNHase, the substrate affinity of nicotinitrile with mutant ReNHase C113Y /C115N/ V52R increased by 24.37%, the enzyme activity increased by 24.79%. The above results provided a new theoretical basis for the industrial application of nicotinonitrile.
-
烟酰胺又称为尼克酰胺、维生素B3,是辅酶Ⅰ和辅酶Ⅱ结构的一部分[1],在人体中可以参与多种氧化还原反应、细胞的代谢过程以及蛋白质、DNA等生物大分子的合成过程[2]。烟酰胺在常温下为白色粉末,具有易溶于水和乙醇等有机溶剂的物理性质[3]。烟酰胺除了在食品及饲料添加剂方面有着广泛的应用外,在人体衰老、疾病时,对神经元的健康和存活也有一定影响,并且其作为神经退行性疾病的治疗剂也具有重要的作用和潜力[4-5]。虽然烟酰胺在国民生产应用中的作用越来越重要,但是传统工业生产中,烟酰胺的化学合成反应条件苛刻,反应过程中产生大量的副产物和污染物[6]。而利用腈水合酶进行生物催化具有高效、反应条件温和及绿色环保等显著优势,符合绿色化学发展的要求和方向[7]。腈水合酶(Nitrile Hydratase,NHase,EC 4.2.1.84) 是一种重要的金属酶,通常由α、β亚基构成四聚体,亚基分子量大多在20~30 kDa左右,不同的结构形式会造成腈水合酶的热稳定性、底物和产物耐受性的巨大差异[8]。腈水合酶可催化腈类物质水合(部分水解)为相应的酰胺,具有很高的工业应用价值。
随着我国市场的不断拓展,人们对于烟酰胺的需求在不断增长,但目前应用在工业生产烟酰胺的菌株的知识产权均被国外发达国家所掌握,我国仍需要大量进口[9-10]。此外,生产烟酰胺所用的第三代工业用菌的腈水合酶活性和转化率较低[11],所以筛选提高产烟酰胺的腈水合酶的催化活性具有必要性。
本研究在前期工作中已经成功构建了来自于野生菌R. erythropolis CCM2595的腈水合酶基因工程菌ReNHase,发现其对烟腈有一定的转化率 [12-13],为进一步提高该腈水合酶对烟腈的生物转化活性,本研究将通过基于氨基酸热点突变的半理性设计对烟腈的亲和力进行定向改造以获取底物亲和力更高的突变体。
1. 材料与方法
1.1 材料与仪器
重组腈水合酶(ReNHase)质粒 为本实验室保藏;烟腈(分析纯)、烟酰胺(分析纯)、甲醇(色谱纯)、乙腈(色谱纯) 上海麦克林生化科技有限公司;胰蛋白胨、酵母提取物 美国Thermo Fisher Scientific 公司;异丙基-β-D-硫代半乳糖苷(IPTG)、卡那霉素、NaCl 上海生工生物工程有限公司;LB培养基:胰蛋白胨10 g/L、酵母提取物5 g/L、NaCl 10 g/L(固体培养基再加入琼脂粉20 g/L),高压灭菌后需加入终浓度为50 μg/mL卡那霉素再使用。
BSA124S 分析天平 德国 Sartorius 集团;HNY-200B 恒温摇床 天津欧诺仪器有限公司;VD-850 超净工作台 上海尚道仪器制造有限公司;ST16R 低温冷冻离心机、933 超低温冰箱、EVO300 PC 紫外分光光度计 美国Thermo Fisher Scientific 公司;PHS-3C pH计 上海雷磁仪器厂;S6000 高效液相色谱仪 华谱科仪科技有限公司。
1.2 实验方法
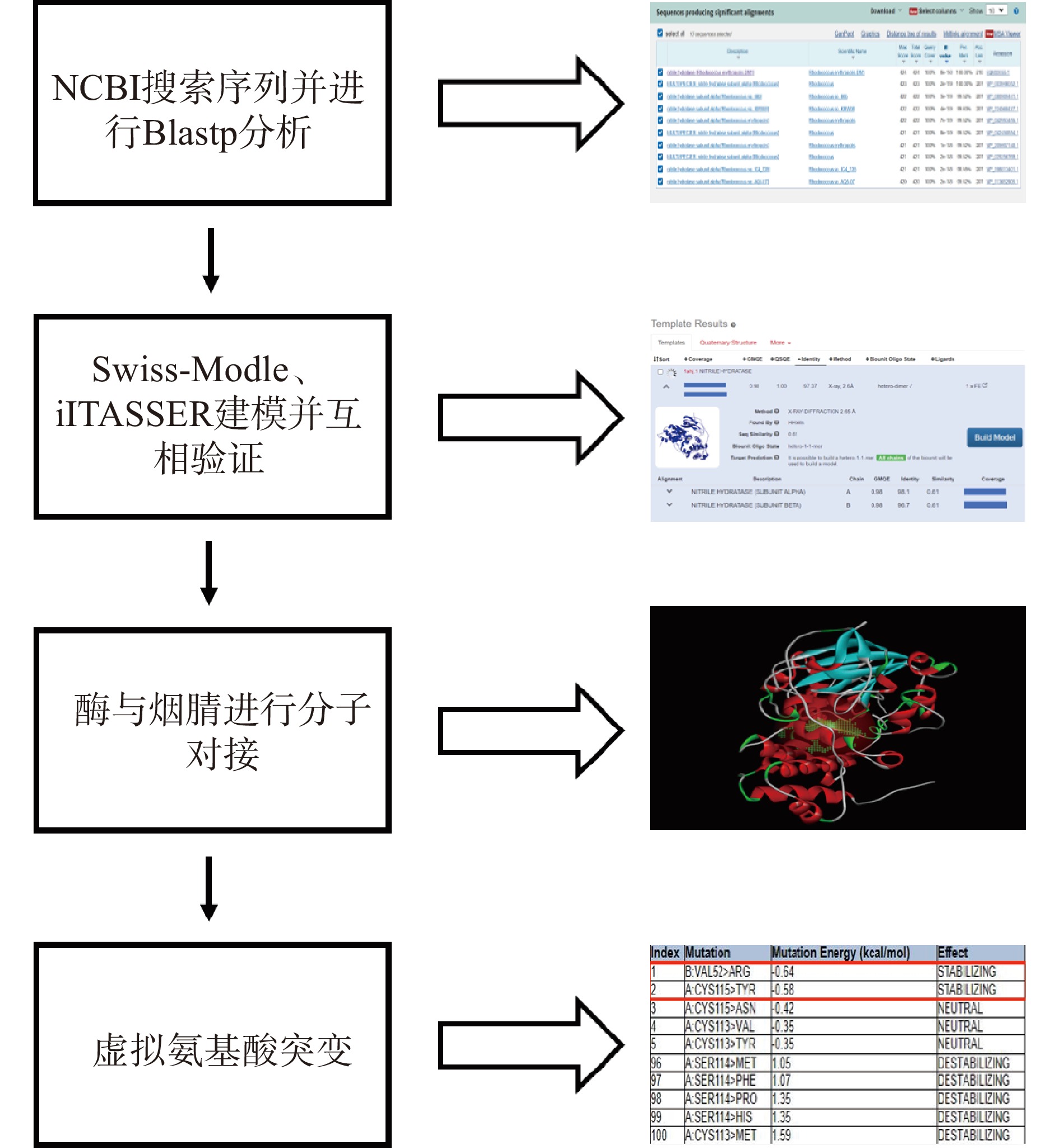
1.2.1 同源建模及虚拟氨基酸突变
本研究中的腈水合酶来源于R. erythropolis CCM2595,各亚基氨基酸序列下载自美国国家生物技术中心NCBI(https://www.ncbi.nlm.nih.gov)。利用在线工具Blastp 分析腈水合酶序列,得到多条与目标序列同源性大于30%的同源序列后使用Siwss-Modle (https://swissmodel.expasy.org)进行同源建模[14-15]。同时采用 iTASSER(Iterative Threading ASSEmbly Refinement)进行同源建模结果分析的验证[16],将以上结果结合Swiss-Modle同源建模的结果后确定可信度较高的蛋白结构。通过DS软件进行蛋白质活性位点预测和基于C-DOCER模块的烟腈和腈水合酶分子对接模拟[17]。通过丙氨酸扫描,找到计算突变能量(亲和力)(Calculate Mutation Energy(Bingding))大于0.5的氨基酸位点,表明这些突变位点对蛋白和小分子的结合起到关键的作用,可以推测这些氨基酸就是关键氨基酸。进一步对以上关键氨基酸进行单点或者多点突变,筛选出Calculate Mutation Energy(Bingding)小于−0.5的突变氨基酸并进行打分排序,流程如图1。将以上筛选出来的突变氨基酸进行突变体质粒的构建(委托江苏常州基宇生物科技有限公司)。
1.2.2 大肠杆菌感受态的热激转化
将5 μg质粒溶解于10 μL灭菌的去离子水中,混合均匀后取1 μL加入到100 μL Arctic Expression(DE3)感受态细胞中,使用移液枪使其混合均匀,放置在冰盒中30 min后,将EP管放置在42 ℃恒温水浴锅中进行热激90 s,热激过后迅速将其转移置冰中冷却3 min;再向其中加入600 μL 已灭菌的LB培养基;放置在37 ℃的摇床中,220 r/min振摇培养1 h,取100 μL和200 μL两种体积的菌液均匀涂布于含50 μg/mL 卡那霉素的LB平板上,37 ℃倒置培养9 h。
1.2.3 ReNHase的异源表达
挑取1个Arctic Expression(DE3)的单菌落接种于含50 μg/mL 卡那霉素的4 mL LB液体培养基中,37 ℃,220 r/min振摇培养约8 h;将0.5 mL种子液接种到50 mL LB液体培养基中,37 ℃,220 r/min振摇培养。当培养液OD600达到0.6~0.8时,加入终浓度为0.1 mmol/L的IPTG于摇菌管中,16 ℃,转速160 r/min诱导过夜。通过SDS-PAGE分析蛋白表达情况。
1.2.4 催化性能探究
1.2.4.1 静息细胞悬液制备
将诱导16 h的菌液4000 r/min,8 min离心去除上清,并使用磷酸缓冲液(50 mmol/L,pH7.4)重悬菌体,重复离心2次,去除培养基,最终用8 mL磷酸缓冲液将菌体重悬,得到均匀的菌体悬液,利用紫外分光光度计测定600 nm处吸光值,计算菌体浓度,将菌悬液密封保存4 ℃备用。
1.2.4.2 催化烟腈
催化反应在1.5 mL离心管中进行,反应总体积为 500 μL。包含20 mmol/L烟腈、均匀的菌体悬液(终浓度4.389 mg/mL(DCW))及磷酸缓冲液。放置在摇床中200 r/min,30 ℃下反应10 min,最后加入500 μL乙腈用来终止催化反应。
1.2.5 酶活测定
配制终浓度分别为0.5、1、1.5、2、2.5、3 mmol/L的烟酰胺标准溶液,用高效液相色谱测定其在220 nm处的吸收值。以峰面积为Y轴,烟酰胺浓度为X轴,制作标准曲线:Y=8802.58064×X,R2=0.99994。 R2(决定系数)>0.99,此标准曲线可用。
反应液经过13000 r/min离心10 min,取上清液经0.22 μm 滤膜过滤后,取一定量实验样品置于液相管中,利用高效液相色谱定量检测产物。高效液相色谱检测条件:Ultimate LP-C18柱(5 μm,4.6 mm×250 mm);流动相为水和乙腈(67:33,vol:vol),检测波长220 nm,柱温30 ℃,流速0.8 mL/min。
一个酶活力单位(IU)定义为: 在 30 ℃,pH=7.4 的条件下,每分钟生成1 μmol产物所需要的酶量[18]。
1.2.6 米氏常数测定
以烟腈为底物,设置梯度底物浓度,30 ℃下反应2 min,得到初始反应速率,通过软件 GraphPad Prism 5得到米氏常数[19]。
1.2.7 PredyFlexy柔性分析
进入PredyFlexy服务器(http://www.dsimb.inserm.fr/dsimb_tools/predyflexy),提交目标氨基酸序列或者直接上传FASTA文件进行预测分析[20]。
1.3 数据处理
液相数据经原始数据记录软件导出后经Excel软件计算处理,标准曲线和反应影响因素探究图像由Origin软件绘制得到。标准曲线每点数据由4组平行实验取平均值得到,其他数据均为3组平行实验得到。
2. 结果与分析
2.1 Siwss-Model 与iTASSER同源建模结果
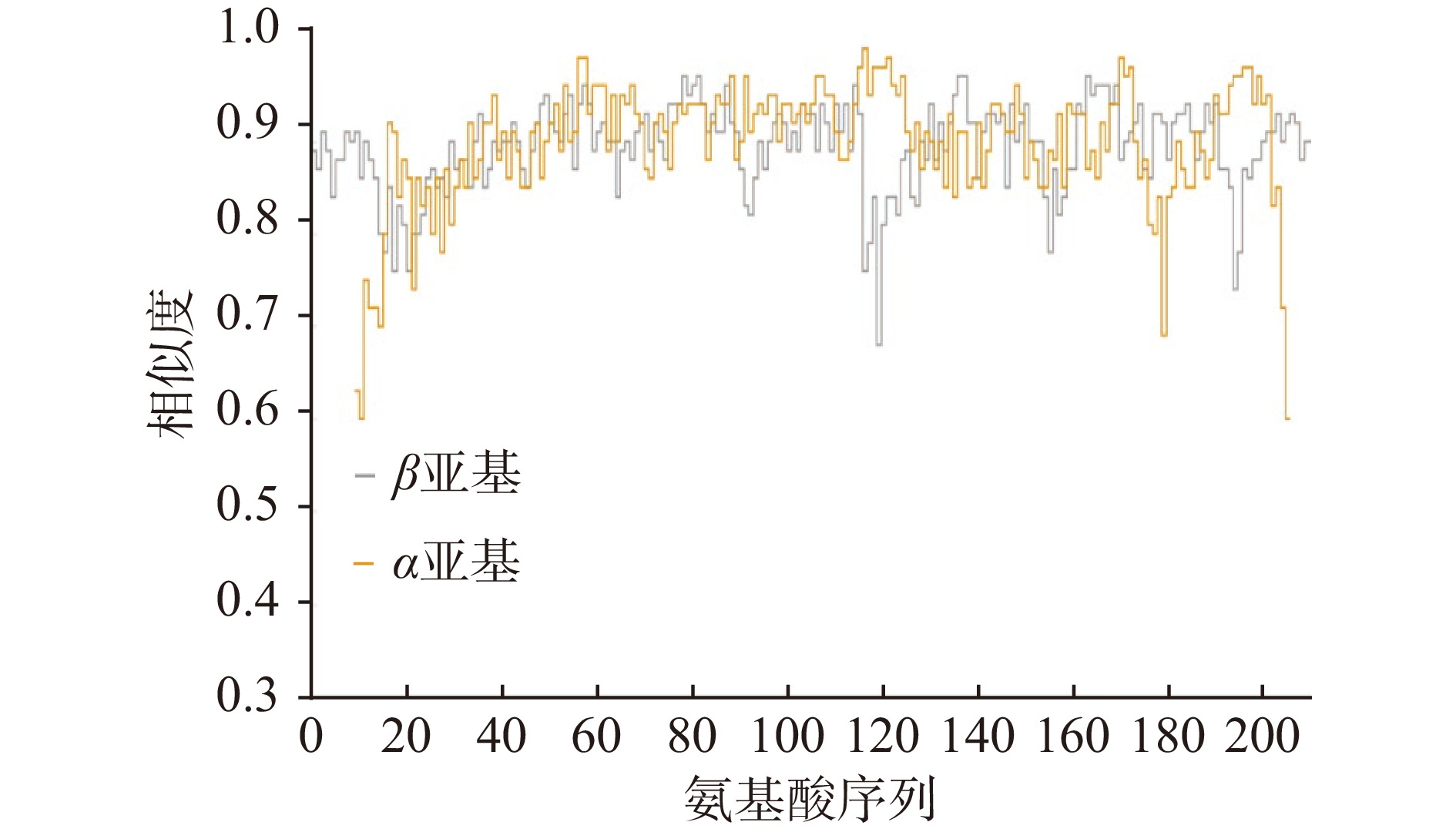
通过Siwss-Model首先选定同源性大于30%的同源序列作为待测蛋白质序列的模板,然后将目标蛋白质序列与筛选得到的模板序列进行序列比对, 使目标序列中各个位置的二级结构与模板的结构尽量对应,得到待测蛋白质空间结构模型,最后根据能量最小化原理,调整蛋白质模板的分子集团,得到折叠自由能处于最小值的结果。通过 Siwss-Model建模算法得到了有三个符合条件的蛋白分子模型,分别是PDB ID: 3A8M、1AHJ、2ZPH。其中1AHJ来源于红球菌R312,与目标序列的相似性达到61%(如图2),故选用1AHJ 来作为腈水合酶模板来代替红球菌CCM2595与底物烟腈进行分子对接和反应。
2.2 iTASSER同源建模结果
iTASSER是一种用于蛋白质结构和功能预测的方法[21]。首先通过多线程方法LOMETS从PDB数据库中识别结构模板,并通过基于迭代模板的碎片组装模拟构建完整的原子模型。接着通过蛋白质功能数据库BioLiP对3D模型进行重新线程化,从而预测靶标的功能。i-TASSER仅使用线程排列中显著性最高的模板,其显著性通过Z分数来衡量,即以标准偏差为单位的原始分数和平均分数之间的差异。本研究从LOMETS线程程序中选择10个最佳模板(表1)并进一步进行Gene Onotology(GO)terms分析(表2)。由Gene Onotology(GO)terms表可得,1AHJ 的CscoreGO、TM-score、IDENa、Cov得分较其他模板均较高,说明1AHJ作为模板与实际的输入序列蛋白结构相似性较高,结合通过Swiss-Model同源建模的结果,使用1AHJ 来当做目标序列的建模结果具有最高的可信度。
表 1 LOMETS线程程序中10个建模最佳模板Table 1. 10 Best modeling templates in LOMETS thread program顺序 PDB ID Iden1 Iden2 覆盖率 Z分数 1 1AHJA 0.98 0.46 0.47 1.70 2 2AHJB 0.95 0.49 0.50 2.71 3 3QYGA 0.49 0.24 0.47 3.58 4 3A8GB 0.97 0.49 0.51 4.94 5 3QXEA 0.49 0.24 0.47 1.99 6 4FM4B 0.41 0.21 0.49 2.62 7 1AHJ 0.98 0.46 0.47 3.40 8 2CZ7 0.97 0.49 0.50 3.28 9 1AHJ 0.98 0.46 0.47 2.51 10 1AHJA 0.97 0.49 0.51 3.36 注:Iden1:表示线程拟合区域模板序列与目标序列对比的比值;Iden2:是具有目标序列与整个模板链的比值;覆盖率:表示线程拟合区域的覆盖率,等于对应的残基数除以目标蛋白质的长度;Z分数:是多线程拟合的标准化Z分数,标准化Z分数大于1表示拟合良好。 表 2 10个最佳模板Gene Onotology(GO)terms分析Table 2. Analysis of top10 templates Gene Onotology (GO) terms顺序 CscoreGO TM-score RMSDa IDENa 覆盖率 PDB ID 1 0.32 0.4885 1.42 0.97 0.51 1ahjB 2 0.31 0.4449 1.51 0.94 0.46 1ahjA 3 0.21 0.41633 2.25 0.35 0.45 3qxeB 4 0.18 0.4209 2.55 0.30 0.46 1ireB 5 0.15 0.4202 2.62 0.33 0.46 1v29B 6 0.11 0.3780 7.51 0.04 0.66 1r8wA 7 0.11 0.4658 1.33 0.49 0.48 3qxeA 8 0.10 0.4490 1.79 0.43 0.47 1ugqA 9 0.10 0.4469 2.26 0.40 0.47 2dd4C 10 0.10 0.4426 1.66 0.49 0.46 1v29A 注: CscoreGO:一种用于评估查询和模板蛋白质之间的全局和局部相似性的组合度量。它的范围是[0-1],值越大表示预测越可靠;TM-score:查询和模板蛋白质之间的全局结构相似性的度量; RMSDa:由TM-align在结构上排列的残基之间的RMSD;IDENa:结构对齐区域的百分比序列同一性;覆盖率:全局结构比对的覆盖率,等于结构比对的残基数除以蛋白质的长度。 2.3 蛋白质活性位点预测和分子对接
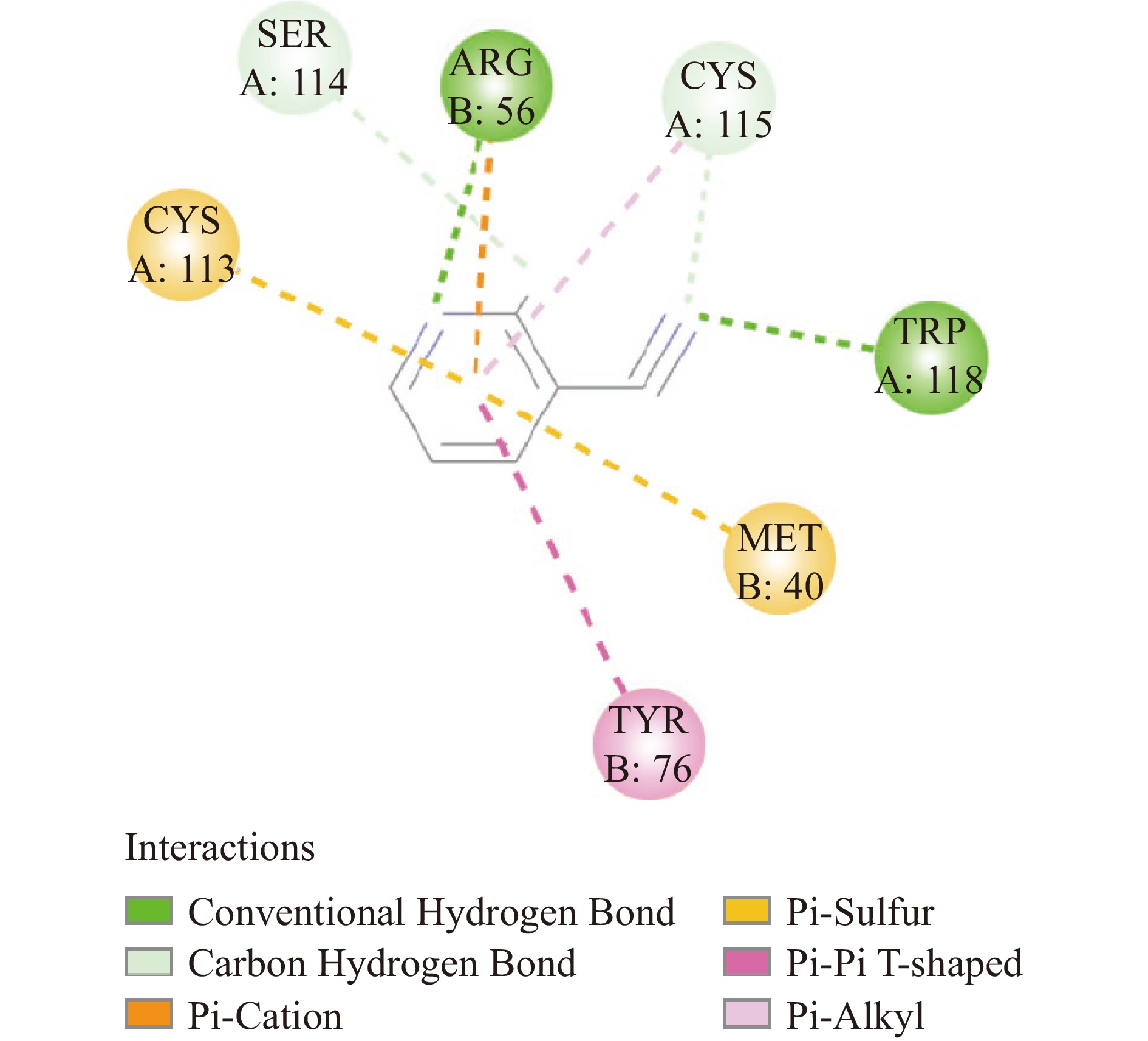
酶的活性结构是蛋白质表面的凹陷或缝隙,这些结构可以让底物结合并嵌入。底物一般通过氢键、范德华力、离子键等相互作用或反应与活性位点上的氨基酸的结合而形成[22]。本研究采用DS软件中的CDOCKER算法对蛋白和底物进行了对接,得到了两者的对接结果及相互作用氨基酸位点,通过受体和配体的相互作用2D(图3)可以看出1AHJ 腈水合酶可以和烟腈形成化学键的氨基酸主要为:A:CYS113、B:ARG56、B:MET40、B:TYR76、A:TRP118、A:SER114、A:CYS115。
2.4 虚拟氨基酸突变
丙氨酸扫描是一种研究蛋白质表面特定区域功能极其有效的方法[23]。丙氨酸扫描是把氨基酸残基置换成丙氨酸,将侧链上的活性基团改换成了体积较小、无其他复杂侧链基团的甲基,这种置换不会干扰蛋白核心结构的折叠,对蛋白质的立体结构几乎没有影响[24]。本研究通过对C-DOCKER中小分子结合能量最稳定的烟腈分子构象进行分析,将烟腈分子与1AHJ结合,进行丙氨酸扫描,结果如表3所示,在突变的8个位点中突变能在−0.5至+0.5之间的数据表明该位点对亲和力没有影响,Effect of Mutation为NETURAL;当突变能>0.5,则表明这些突变点会使蛋白之间的亲和力降低,相互作用能力减弱,表明这些突变位点对蛋白和小分子的结合起到关键的作用。
表 3 丙氨酸扫描结果Table 3. Alanine scan results顺序 突变 突变能(kcal/mol) 突变结果 1 A:CYS113>ALA 0.04 无影响 2 A:SER114>ALA 0.2 无影响 3 A:CYS115>ALA 0.1 无影响 4 A:TRP118>ALA 0.57 不稳定 5 B:MET40>ALA 0.3 无影响 6 B:VAL52>ALA 0.22 无影响 7 B:ARG56>ALA 0.87 不稳定 8 B:TYR76>ALA 0.76 不稳定 将上述关键氨基酸为基本位点,将每个氨基酸突变为除丙氨酸以外的19种氨基酸,进行单位点突变和多位点突变筛选排序得到表4结果。
表 4 关键氨基酸位点突变结果Table 4. Key amino acid mutation results突变 突变能(kcal/mol) 突变结果 A:CYS113>TYR.A:CYS115>ASN.B:VAL52>ARG −5.65 稳定 A:CYS113>TYR.A:CYS115>TYR −0.61 稳定 A:CYS113>TYR.A:CYS115>TYR.B:VAL52>ARG −5.57 稳定 A:CYS113>VAL.A:CYS115>TYR.B:VAL52>ARG −3.6 稳定 A:CYS113>VAL.B:VAL52>ARG −0.58 稳定 A:CYS115>TYR −0.58 稳定 B:VAL52>ARG −0.64 稳定 由上述图表可知,其中三位点突变 CYS113→TYR(C113Y)、CYS115→ASN(C115N)、VAL52→ARG(V52R)的突变能为:−5.65 kcal/mol分值最高,预测该三位点突变可以提高底物与酶的亲和能力。接下来在本研究中将上述突变氨基酸进行定点突变并进行基因质粒的构建和后续异源蛋白的表达。
2.5 重组菌突变体ReNHase C113Y/C115N/V52R的异源表达
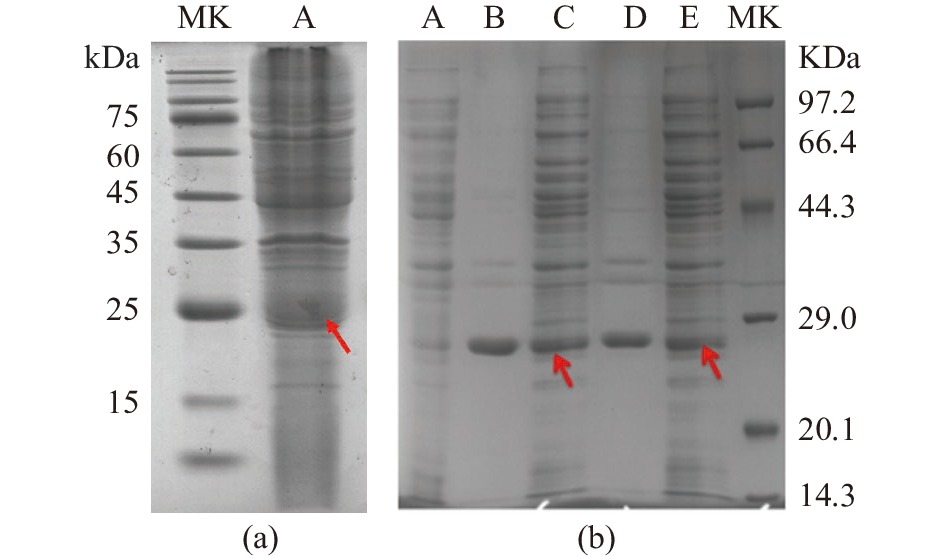
将ReNHase C113Y/C115N/V52R质粒转化入大肠杆菌感受态中,得到突变体重组菌 ReNHase C113Y/C115N/V52R,通过 SDS-PAGE 验证重组蛋白能否表达。腈水合酶通常是由α、β亚基构成的四聚体,完整蛋白分子量约96 kDa。α、β单亚基分子量约24 kDa,因此,蛋白电泳时α、β亚基条带发生重叠。如图4(a)所示,红色箭头指示的是得到成功异源表达的野生型腈水合酶。图4(b)显示突变体ReNHase C113Y/C115N/V52R在Arctic Expression(DE3)和BL21(DE3)两种菌的上清中均有大量表达。同时发现,在诱导过后的沉淀中也有大量目标蛋白,这可能是由于表达过程中形成一部分包涵体[25]。由于突变体只突变了三个氨基酸位点,因此分子量较野生型没有明显变化。
![]() 图 4 野生型与突变体的异源表达注:(a)野生型: Lane A: 16 ℃诱导破碎上清(Arctic Expression(DE3))。(b)突变体ReNHase C113Y/C115N/V52R: Lane A: 阴性对照,Lane B: 37 ℃诱导破碎沉淀(BL21(DE3)),Lane C: 37 ℃诱导破碎上清(BL21(DE3)),Lane D: 16 ℃诱导破碎沉淀(Arctic Expression(DE3)),Lane E: 16 ℃诱导破碎上清(Arctic Expression(DE3)。Figure 4. Heterologous expression of wild-type and mutants
图 4 野生型与突变体的异源表达注:(a)野生型: Lane A: 16 ℃诱导破碎上清(Arctic Expression(DE3))。(b)突变体ReNHase C113Y/C115N/V52R: Lane A: 阴性对照,Lane B: 37 ℃诱导破碎沉淀(BL21(DE3)),Lane C: 37 ℃诱导破碎上清(BL21(DE3)),Lane D: 16 ℃诱导破碎沉淀(Arctic Expression(DE3)),Lane E: 16 ℃诱导破碎上清(Arctic Expression(DE3)。Figure 4. Heterologous expression of wild-type and mutants2.6 突变体ReNHase C113Y /C115N/ V52R对烟腈的底物亲和力影响
将重组菌突变体ReNHase C113Y/C115N/V52R进行大肠杆菌异源表达后进行His标签分离纯化,然后检测酶和底物亲和力变化。采用外标法对对烟酰胺含量进行高效液相色谱检测, 20 mmol/L烟腈与突变体反应48 h后,对烟腈的转化率为36%±1.1%,较野生型酶的转化率提高了约50%(24%±0.8%)。酶促反应动力学常数(Km)可以反映酶与底物的亲和力,即Km值越小,酶与底物的亲和力越大。通过Mchaelis-Menton模型可以得到动力学参数Km,结果显示野生型酶Km为16.78 mmol/L,突变体酶Km为12.69 mmol/L。同时发现,相对于野生型ReNHase的酶活(12.14 U/mL),突变体的酶活(15.15 U/mL),相较于野生型,酶活提高了24.79%。以上结果说明突变体对烟腈的底物亲和力和酶活都有了明显的提高。
2.7 突变体ReNHase C113Y /C115N/ V52R 的柔性变化分析
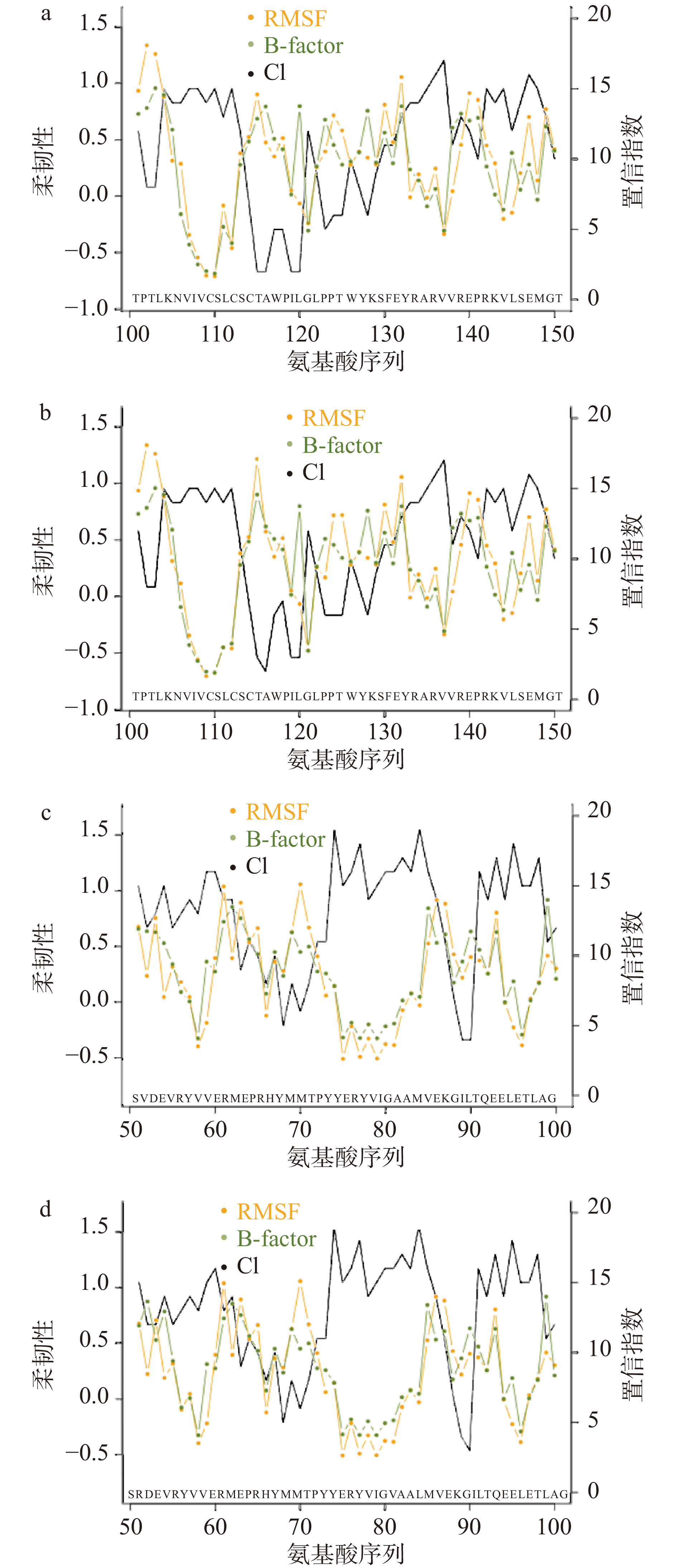
通过PredyFlexy服务器(http://www.dsimb.inserm.fr/dsimb_tools/predyflexy),提交突变体各亚基氨基酸序列进行蛋白柔性预测分析,图5中灵活度越高代表柔韧性越好,置信指数越高说明预测的越准确。根据预测结果发现α亚基的CYS113突变为TYR后柔韧性没有变化,而CYS115突变为ASN后,柔韧性从0.687上升至0.899(图5(a)、(b)),并且β亚基上VAL52突变为ARG后柔韧性从0.637上升至0.875(图5(c)、(d))。根据以上结果推测α亚基CYS115和β亚基VAL52位点的突变使蛋白质的柔性得到了提升,因此使蛋白质和配体的结合过程变得更加容易。
3. 结论
本研究前期发现了一种来源于R. erythropolis CCM2595的腈水合酶,发现其对烟腈具有一定的催化活性。在本研究中使用半理性设计对腈水合酶中烟腈的结合位点进行氨基酸热点突变,并分析突变位点对酶与底物之间亲和能力的影响,预测筛选出使酶对底物亲和力明显提升的突变体。结果发现当腈水合酶α亚基CYS113突变为TYR(C113Y)、CYS115突变为ASN(C115N)和β亚基VAL52突变为ARG(V52R)后,突变体 Km值由突变前的 16.78 mmol/L降低为12.69 mmol /L, 酶活由12.14 U/mL提高到15.15 U/mL,以上结果表明,通过虚拟氨基酸突变得到的腈水合酶三突变体ReNHase C113Y/C115N/V52R对烟腈的底物亲和力和比酶活均得到了明显的提高。本研究通过运用生物信息学软件对腈水合酶底物亲和力进行了半理性设计和改造,并通过定点突变的异源表达进行了验证,为腈水合酶在烟腈中的工业应用提供了新的理论依据。
-
图 4 野生型与突变体的异源表达
注:(a)野生型: Lane A: 16 ℃诱导破碎上清(Arctic Expression(DE3))。(b)突变体ReNHase C113Y/C115N/V52R: Lane A: 阴性对照,Lane B: 37 ℃诱导破碎沉淀(BL21(DE3)),Lane C: 37 ℃诱导破碎上清(BL21(DE3)),Lane D: 16 ℃诱导破碎沉淀(Arctic Expression(DE3)),Lane E: 16 ℃诱导破碎上清(Arctic Expression(DE3)。
Figure 4. Heterologous expression of wild-type and mutants
表 1 LOMETS线程程序中10个建模最佳模板
Table 1 10 Best modeling templates in LOMETS thread program
顺序 PDB ID Iden1 Iden2 覆盖率 Z分数 1 1AHJA 0.98 0.46 0.47 1.70 2 2AHJB 0.95 0.49 0.50 2.71 3 3QYGA 0.49 0.24 0.47 3.58 4 3A8GB 0.97 0.49 0.51 4.94 5 3QXEA 0.49 0.24 0.47 1.99 6 4FM4B 0.41 0.21 0.49 2.62 7 1AHJ 0.98 0.46 0.47 3.40 8 2CZ7 0.97 0.49 0.50 3.28 9 1AHJ 0.98 0.46 0.47 2.51 10 1AHJA 0.97 0.49 0.51 3.36 注:Iden1:表示线程拟合区域模板序列与目标序列对比的比值;Iden2:是具有目标序列与整个模板链的比值;覆盖率:表示线程拟合区域的覆盖率,等于对应的残基数除以目标蛋白质的长度;Z分数:是多线程拟合的标准化Z分数,标准化Z分数大于1表示拟合良好。 表 2 10个最佳模板Gene Onotology(GO)terms分析
Table 2 Analysis of top10 templates Gene Onotology (GO) terms
顺序 CscoreGO TM-score RMSDa IDENa 覆盖率 PDB ID 1 0.32 0.4885 1.42 0.97 0.51 1ahjB 2 0.31 0.4449 1.51 0.94 0.46 1ahjA 3 0.21 0.41633 2.25 0.35 0.45 3qxeB 4 0.18 0.4209 2.55 0.30 0.46 1ireB 5 0.15 0.4202 2.62 0.33 0.46 1v29B 6 0.11 0.3780 7.51 0.04 0.66 1r8wA 7 0.11 0.4658 1.33 0.49 0.48 3qxeA 8 0.10 0.4490 1.79 0.43 0.47 1ugqA 9 0.10 0.4469 2.26 0.40 0.47 2dd4C 10 0.10 0.4426 1.66 0.49 0.46 1v29A 注: CscoreGO:一种用于评估查询和模板蛋白质之间的全局和局部相似性的组合度量。它的范围是[0-1],值越大表示预测越可靠;TM-score:查询和模板蛋白质之间的全局结构相似性的度量; RMSDa:由TM-align在结构上排列的残基之间的RMSD;IDENa:结构对齐区域的百分比序列同一性;覆盖率:全局结构比对的覆盖率,等于结构比对的残基数除以蛋白质的长度。 表 3 丙氨酸扫描结果
Table 3 Alanine scan results
顺序 突变 突变能(kcal/mol) 突变结果 1 A:CYS113>ALA 0.04 无影响 2 A:SER114>ALA 0.2 无影响 3 A:CYS115>ALA 0.1 无影响 4 A:TRP118>ALA 0.57 不稳定 5 B:MET40>ALA 0.3 无影响 6 B:VAL52>ALA 0.22 无影响 7 B:ARG56>ALA 0.87 不稳定 8 B:TYR76>ALA 0.76 不稳定 表 4 关键氨基酸位点突变结果
Table 4 Key amino acid mutation results
突变 突变能(kcal/mol) 突变结果 A:CYS113>TYR.A:CYS115>ASN.B:VAL52>ARG −5.65 稳定 A:CYS113>TYR.A:CYS115>TYR −0.61 稳定 A:CYS113>TYR.A:CYS115>TYR.B:VAL52>ARG −5.57 稳定 A:CYS113>VAL.A:CYS115>TYR.B:VAL52>ARG −3.6 稳定 A:CYS113>VAL.B:VAL52>ARG −0.58 稳定 A:CYS115>TYR −0.58 稳定 B:VAL52>ARG −0.64 稳定 -
[1] 徐兆瑜. 烟酸和烟酰胺的合成和应用[J]. 精细化工原料及中间体,2003,9:6−9. [XU Z Y. Synthesis and application of niacin and nicotinamide[J]. Journal of Fine Chemical Materials and Intermediates,2003,9:6−9. [2] ZHANG Z J, PAN J, LI C X, et al. Efficient production of (R)-(-)-mandelic acid using glutaraldehyde cross-linked Escherichia coli cells expressing Alcaligenes sp. nitrilase[J]. Bioprocess Biosyst Eng,2014,37(7):1241−1248. doi: 10.1007/s00449-013-1096-y
[3] PRASAD S, MISRA A, JANGIR V P, et al. A propionitrile-induced nitrilase of Rhodococcus sp. NDB 1165 and its application in nicotinic acid synthesis[J]. World Journal of Microbiology and Biotechnology,2006,23(3):345−353.
[4] ULLAH A, ZHAO T, MUHAMMAD M T, et al. Synthesis of novel nicotinamide susbstituted phthalocyanine and photodynamic antomicrobial chemotherapy evaluation potentiated by potassium iodide against the gram positive S. aureus and gram negative E. coli[J]. Biotechnol Lett,2021,43(4):781−790. doi: 10.1007/s10529-020-03071-0
[5] BORLONGAN C V, GRIFFIN S M, PICKARD M R, et al. Nicotinamide restricts neural precursor proliferation to enhance catecholaminergic neuronal subtype differentiation from mouse embryonic stem cells[J]. Plos One,2020,15(9):e0233477. doi: 10.1371/journal.pone.0233477
[6] YANG Z, PEI X, XU G, et al. Efficient production of 2, 6-difluorobenzamide by recombinant Escherichia coli expressing the Aurantimonas manganoxydans nitrile hydratase[J]. Appl Biochem Biotechnol,2019,187(2):439−449. doi: 10.1007/s12010-018-2823-2
[7] BHALLA T C, KUMAR V, THAKUR N, et al. Nitrile metabolizing enzymes in biocatalysis and biotransformation[J]. Appl Biochem Biotechnol,2018,185(4):925−946. doi: 10.1007/s12010-018-2705-7
[8] MASHWEU A R, CHHIBA-GOVINDJEE V P, BODE M L, et al. Substrate profiling of the cobalt nitrile hydratase from Rhodococcus rhodochrous ATCC BAA870[J]. Molecules,2020,25(1):238. doi: 10.3390/molecules25010238
[9] VESELA A B, RUCKA L, KAPLAN O, et al. Bringing nitrilase sequences from databases to life: The search for novel substrate specificities with a focus on dinitriles[J]. Appl Microbiol Biotechnol,2016,100(5):2193−2202. doi: 10.1007/s00253-015-7023-1
[10] XIA Y, PEPLOWSKI L, CHENG Z, et al. Metallochaperone function of the self-subunit swapping chaperone involved in the maturation of subunit-fused cobalt-type nitrile hydratase[J]. Biotechnol Bioeng,2019,116(3):481−489. doi: 10.1002/bit.26865
[11] SUN J, YU H, CHEN J, et al. Ammonium acrylate biomanufacturing by an engineered Rhodococcus ruber with nitrilase overexpression and double-knockout of nitrile hydratase and amidase[J]. J Ind Microbiol Biotechnol,2016,43(12):1631−1639. doi: 10.1007/s10295-016-1840-9
[12] WANG L, LIU S X, DU W J, et al. High Regioselectivity production of 5-cyanovaleramide from adiponitrile by a novel nitrile hydratase derived from Rhodococcus erythropolis CCM2595[J]. ACS Omega,2020,5(29):18397−18402. doi: 10.1021/acsomega.0c02188
[13] DU W J, HUANG J, CUI B C, et al. Efficient biodegradation of nitriles by a novel nitrile hydratase derived from Rhodococcus erythropolis CCM2595[J]. Biotechnology, Biotechnological Equipment,2021,35(1):1127−1135. doi: 10.1080/13102818.2021.1941253
[14] CHEN Q M, XIAO Y Q, SHAKHNOVICH E I, et al. Semi-rational design and molecular dynamics simulations study of the thermostability enhancement of cellobiose 2-epimerases[J]. International Journal of Biological Macromolecules,2020,154:1356−1365. doi: 10.1016/j.ijbiomac.2019.11.015
[15] SCHWEDE T, KOPP J, GUEX N, et al. SWISS-MODEL: An automated protein homology-modeling server[J]. Nucleic Acids Res,2003,31(13):3381−3385. doi: 10.1093/nar/gkg520
[16] WU S, SKOLNICK J, ZHANG Y. Ab initio modeling of small proteins by iterative TASSER simulations[J]. BMC Biol,2007,5:17. doi: 10.1186/1741-7007-5-17
[17] ESWAR N, WEBB B, MARTI-RENOM M A, et al. Comparative protein structure modeling using Modeller [J]. Curr Protoc Bioinformatics, 2006, Chapter 5: Unit-5 6.
[18] PEIWEN F E I, DEIRY E L, WAFIK S. P53 and radiation responses[J]. Oncogene,2003,22(37):5774−5783. doi: 10.1038/sj.onc.1206677
[19] EISENTHAL R, DANSON M J, HOUGH DW. Catalytic efficiency and kcat/KM: A useful comparator?[J]. Trends Biotechnol,2007,25(6):247−249. doi: 10.1016/j.tibtech.2007.03.010
[20] BREVERN A, BORNOT A, CRAVEUR P, et al. PredyFlexy: Flexibility and local structure prediction from sequence[J]. Nucleic Acids Research,2012,40(W1):W317−W322. doi: 10.1093/nar/gks482
[21] YANG J, YAN R, ROY A, et al. The I-TASSER Suite: Protein structure and function prediction[J]. Nat Methods,2015,12(1):7−8. doi: 10.1038/nmeth.3213
[22] ROBUSTELLI P, PIANA S, SHAW D E. Developing a molecular dynamics force field for both folded and disordered protein states[J]. Proceedings of the National Academy of Sciences,2018,115(21):E4758−E4766. doi: 10.1073/pnas.1800690115
[23] ZONG Z Y, GAO L, CAI W S, et al. Computer-assisted rational modifications to improve the thermostability of β-glucosidase from Penicillium piceum H16[J]. BioEnergy Research,2015,8(3):1384−1390. doi: 10.1007/s12155-015-9603-4
[24] RAMOS R M, MOREIRA I S. Computational alanine scanning mutagenesis-an improved methodological approach for protein-DNA complexes[J]. J Chem Theory Comput,2013,9(9):4243−4256. doi: 10.1021/ct400387r
[25] GILEADI O. Recombinant protein expression in E. coli: A historical perspective[J]. Methods Mol Biol,2017,1586:3−10.
-
期刊类型引用(6)
1. 杨琳,江蓉倩,张杭颖,李树玉,武马璐,张君诚. 五指毛桃水提液制备泡腾片工艺优化. 食品工业科技. 2024(08): 200-207 .  本站查看
本站查看
2. 王江,马彩珍,李甜,霍彦雄,张亮亮,吴子健,徐建国. 野生接骨木果含片的制备及其抗氧化、抗运动疲劳作用研究. 保鲜与加工. 2024(05): 69-76 .  百度学术
百度学术
3. 许甜,吴萌,葛世辉,陈君,李赛,王智森. 沙棘果、叶的研究进展. 今日药学. 2024(06): 466-472 .  百度学术
百度学术
4. 冯思思,张喜瑞,于梦言,徐玉孟,李梓诺,李嘉慧,孙万萌,王璐,梁彬,孙婵婵. 黑蒜泡腾片的制备工艺优化及其对肠道微生态的影响. 食品与发酵工业. 2024(19): 49-55+86 .  百度学术
百度学术
5. 王媛媛,冯勇荣,孙青占,吕兆林. 不同产地沙棘叶茶品质分析及综合评价. 食品与发酵工业. 2024(24): 331-338 .  百度学术
百度学术
6. 王菲,沈卉芳,王瑶,沈鑫婷,郑先哲,姚鑫淼. 葡萄籽原花青素泡腾片的制备工艺优化及品质分析. 食品工业. 2024(12): 48-53 .  百度学术
百度学术
其他类型引用(3)





 下载:
下载:
 下载:
下载:



