Molecular Recognition and Threshold Prediction Model of Bitterness in Natural Compounds
-
摘要: 鉴定天然化合物中苦味物质和确定其苦味阈值对于食物中苦味分子的发掘和利用至关重要。基于构效关系识别苦味分子及预测苦味分子阈值是一种低成本快速的方法。本研究利用分子操作环境(Molecular Operating Environment, MOE)、Chemopy和Mordred生成2D描述符,利用支持向量机(Support Vector Machine, SVM)、随机森林(Random Forests, RF)算法建立苦味分子识别模型,利用偏最小二乘回归(Partial Least Squares Regression, PLSR)、随机森林回归(Random Forests Regression, RFR)、k-最近邻回归(k-Nearest Neighbor Regression, kNNR)、主成分回归(Principle Component Regression, PCR)算法建立苦味阈值预测模型。结果表明:MOE-RF模型能够较好地识别分子是否具有苦味,准确度为0.982;ChemoPy-PLSR模型的苦味阈值预测效果最好,决定系数为0.85,误差均方根为0.43,可将这两个模型联合使用来预测分子是否具有苦味及苦味阈值。Abstract: It is important to identify the bitter substances in natural compounds and determine their bitterness threshold for finding out the bitter molecules that affect the flavor of food and developing some foods with unique flavors. Identifying bitter molecules and predicting the threshold of bitter molecules based on the quantitative structure-activity relationship is a low-cost and rapid method. This research used Molecular Operating Environment (MOE), Chemopy and Mordred to generate 2D molecular descriptor to establish bitterness molecular recognition models with Support Vector Machine (SVM) and Random Forests (RF) algorithms. This study used above descriptors to establish bitterness threshold prediction models with Partial Least Squares Regression (PLSR), Random Forests Regression (RFR), k-Nearest Neighbor Regression (kNNR), and Principle Component Regression (PCR) algorithms. The results showed that the MOE-RF model had the highest accuracy of 0.982, the ChemoPy-PLSR model had the best bitterness prediction effect with a coefficient of determination of 0.85 and a root mean square error of 0.43. The two models would be combined to predict whether the molecule has bitterness and the threshold of bitterness or not.
-
Keywords:
- bitter compounds /
- recognition /
- threshold /
- prediction /
- verification
-
苦味是人能感知的6种味觉之一,既能导致食品的风味变差,也能给食品带来更加丰富的味感。啤酒、茶饮和咖啡等带苦味的产品深受人们欢迎。人们对食物中的苦味组分知之甚少,如蓝靛果、柚子、核桃等食物中苦味的呈味分子并不十分清楚[1]。这一方面给掩盖苦味、改善风味带来困难,也使这些苦味成分很少被食品研发人员用于开发新的产品。
目前鉴定苦味的方法都来源于药物研究。主要通过志愿者来品尝,配体验证和构效关系模型(Structure Activity Relationships, SAR)的方法进行苦味验证。志愿者品尝通常要进行志愿者筛选及苦味标准化培训,培训周期一般较长[2];配体验证一般通过将待鉴定物质与苦味受体作用靶点进行分子对接,需要消耗较大的计算机资源[3];而SAR的方法因其准确度高、简便成为应用最多的方法。Rodgers等[4]使用包括649个苦味分子和13530个从MDL药物数据仓库(MDL Drug Data Repository, MDDR)中随机选择的分子作为数据集,利用圆形指纹(MOLPRINT 2D)和朴素贝叶斯算法对苦味化合物进行分类,在五折交叉验证中,预测模型的准确度、精密度、特异性和灵敏度分别为88%、24%、89%和72%;Banerjee等使用从SuperSweet和BitterDB数据集中获得的包含517种人工和天然甜味剂、685种苦味化合物的数据集,利用分子指纹和随机森林分类器预测苦味,模型准确率为95%,ROC曲线下面积(Area Under Curve, AUC)为0.98[5-7];Margulis等[8]使用包括极致梯度提升(eXtreme Gradient Boosting, XGBoost)在由169个非常苦的化合物和324个表现不是非常苦的化合物组成的493个化合物的训练集上建立苦味程度识别模型,其准确率达到了87%。这些研究中,模型使用的苦味数据集包含较多非天然化合物,对食品中天然苦味分子的鉴定适用性尚未可知。到目前为止,还没有针对性预测食品中苦味分子的数据集和SAR研究。
本研究以苦味分子研究文献、专利及公开数据集为数据源,采用人工交叉验证的方法搜集清洗数据,在Mysql数据集中建立最大的人工修正的苦味、非苦味数据集和苦味分子苦味阈值公开数据集。利用机器学习算法,首先建立定性构效关系模型,鉴别食品中的分子是否呈现苦味,然后,建立定量构效关系模型,对分子的苦味阈值做出预测,最后联用上述模型预测FooDB数据集中潜在的苦味分子及其苦味阈值,并结合感官评价进行结果验证。本研究建立了一种食品中苦味分子的快速鉴定方法,对于改善食品风味和开发新的苦味回甘的食品具有实际意义,以及对解释苦味分子的结构特征、了解苦味受体及苦味信号传递奠定了数据基础。
1. 材料与方法
1.1 苦味-非苦味数据集及苦味阈值数据集建立
苦味和非苦味分子的数据都来自公开的数据集、文献及专利。其中苦味分子及阈值来自BitterDB[6]中的天然化合物,并在此基础上,扩展了从文献、专利中获得的天然苦味分子的阈值;非苦味分子来自FooDB[9]、FlavorDB[10]及Fenaroli的风味成分手册(第5版)[11]。
非苦味分子数据集的选择至关重要。非苦味分子数据集作为负集,既要有明确的文献证明没有苦味,也要在结构上尽量与苦味分子相似,这样才能让模型更加科学有效。根据苦味分子结构和物化特性,本文建立了如下规则来建立非苦味分子数据集:首先根据苦味分子的分子量范围,筛选分子量范围在85~1224之间的分子建立苦味分子数据集;其次苦味分子有较强的疏水性,根据苦味分子的疏水特性,选择疏水性范围与苦味分子相近的分子建立苦味分子数据集。
将不同来源的分子收集起来,通过Python语言,调用Pubchem API接口,查阅Pubchem数据集[12]获得所有分子的简化分子线性输入规范(SMILES, Simplified molecular input line entry specification)格式,通过开源软件(R语言(version 3.5.1),Python语言(version 3.6))和人工筛查方式去除重复、错误的分子,形成有意义的化学空间来训练和评估机器学习模型。因苦味分子阈值的取值范围较大,本研究进行适当缩放处理,将其取对数后,存入数据集。
1.2 分子描述符筛选
本研究利用分子操作环境(Molecular Operating Environment, MOE)[13]、ChemoPy[14]及Mordred[15]生成描述符。分别将分子的SMILES作为输入,加载到描述符生成软件中,输出分子描述符。剔除含有缺失值的描述符后,利用R语言进行描述符优化和选择。首先根据近零方差剔除描述符:设定频数比率(freqCut)=25和唯一值比例(uniqueCut)=20两个阈值,保留频数比率小于25、唯一值比例大于20的描述符,删除不具代表性的以及特殊的描述符,去除相关的变量,减少冗余;其次去除高共线性描述符变量:设定临界值(cutoff)为0.95,删除并只保留一个相似程度高于0.95的描述符,避免多个描述符描述同一特征;最后利用主成分分析(Principal Component Analysis, PCA)[16]进行描述符筛选:设定贡献率阈值为0.5,保留贡献率大于0.5的描述符。
1.3 苦味分子识别模型的建立和评价
建立苦味分子识别模型使用的工具是R语言[17]及其扩展包,包括:ggplot2[18]、RandomForest[19]、e1071[20]、kknn[21]、MASS[22]、sampling[23]。实验采用了两种算法:RF[24]和SVM[25]。RF算法是基于决策树的分类器集成算法,其中每一棵树都依赖于一个随机向量,这些向量都是独立分布的,通过生成多个分类树,最终将分类树结果进行汇总。通过十折交叉验证和网格搜索,设置结点值(mtry)为9设置决策树数目(ntree)为20构建随机森林模型。SVM算法是在线性可分的情况下,在原空间寻找两类样本的最优分类超平面,在线性不可分的情况下,通过使用非线性映射将低维属性空间的样本映射到高维属性空间使其变为线性情况,从而在该特征空间中寻找最优分类超平面。设置核函数为径向基核函数(RBF核函数),gamma参数为0.1,惩罚系数(cost)为10建支持向量机模型。
所建立的苦味-非苦味数据集由139个苦味分子以及139个非苦味分子共有278个分子组成用于建立苦味分子识别模型,实验采用无放回随机分层抽样,分为苦味与非苦味两层,抽取训练集和测试集,训练集占总数据的3/4(208个分子),测试集占总数据的1/4(70个分子),训练集用于模型训练,而测试集用于模型验证。使用准确度(Accuracy)和精确度(Precision)评估模型性能。
Accuracy=TPTP+FN (1) Precision=TNFP+TN (2) 式中:TP-真苦,即真的苦味分子被预测为苦味;FP-假苦,即真的非苦味分子被预测成苦味,TN-真非苦,即真的非苦味被预测成非苦味,FN-假非苦,即真的苦味被预测成非苦味。
1.4 苦味分子阈值预测模型的建立和评价
建立苦味分子阈值预测模型使用的工具是R语言及其扩展包:caret、FactoMineR、factoextra、tidyverse、pls。实验采用PLSR、RFR、kNNR、PCR四种算法进行苦味分子阈值预测。PLSR是常用的定量分析建模方法,它能有效地解决变量间的多重关联问题。PLSR可以通过降维提取因子,设置提取的因子成分数(nt)为2,并将其作为回归分析的目标。RFR是指通过集成学习的思想集成多棵树的算法。它的基本单位是决策树,基本上是一种集成学习方法,基于决策树之间的距离进行回归预测,设置结点值(mtry)为2,设置决策树数目(ntree)为500。kNNR根据最接近某一未知点的k个数据点对该未知点进行回归预测,设置邻居个数(k)为5。PCR通过将一组高度相关的变量转化为一组新的不相关的主成分变量来减少数据冗余来对未知点预测,设置主成分个数(ncomp)为5。实验首先采用无放回随机抽样抽取训练集和测试集,其中训练集占总数据集的3/4,测试集占总数据的1/4,接着对实验搜集到具有阈值的苦味分子经分子量和疏水性验证,将验证通过后的苦味分子用来建立苦味分子阈值预测模型。
苦味分子阈值预测模型建立之后,在苦味分子阈值预测模型中,使用五折交叉验证(cross-validation)的方法评价模型的稳健性[26]。将训练集划分成5个互补相交的子集,每次选取其中一个子集做测试集,其余4个数据子集做训练集构建模型,这个过程不断重复,直到每个数据集的样本都被用作测试集。通过计算模型的决定系数及误差均方根,来评价模型的拟合优度和预测能力。当模型经过评价和验证,证明其在统计学上具有稳健性和较好的预测能力后,该模型才可用于未知苦味分子的预测。
1.5 苦味分子及阈值验证
对感官小组由12名评估员(5名女性和7名男性,年龄25~40岁)组成,所有参与人员均签署感官评价知情同意书,并且没有已知的味觉障碍病史。对于苦味的训练和分类,MgSO4(166 mmol/L)溶液代表了一种短暂的金属苦味品质,主要在舌头的前部感知,水杨苷(1.4 mmol/L)则赋予了一种持久的苦味。主要在舌后部和喉咙中感知到的苦味感和在口腔中提供持久苦味的咖啡因(8.0 mmol/L)被用作参考。感官分析在22~25 °C下进行,为了最大限度地减少任何有毒化合物的摄入,通过使用吞-吐法[27]进行感官分析,通过这种方法,测试材料不是吞咽而是吐出。苦味识别阈值由12名小组成员根据 ISO 4120[28]中详述的方案通过三角测试确定。使用瓶装水(pH4.5)作为溶剂和5 min的刺激间隔长度,在三个不同时间节点,将待测定呈味物质加水1:1稀释成7个浓度梯度,每个浓度的样品和另外两个空白(水)组成一组,并用四位数字随机编号呈现给受过训练的感官小组,每人品评三次。要求小组成员将待测呈味物质在口腔中保持10 s后吐出。待7个梯度样品品评完后,按浓度由低到高的顺序整理数据,取不能感知到的浓度和上一个能感知到味觉的浓度的几何平均值为品评人员的个人识别阈值浓度,苦识别阈值由所有个体阈值浓度的几何平均值计算。
2. 结果与分析
2.1 数据集的建立
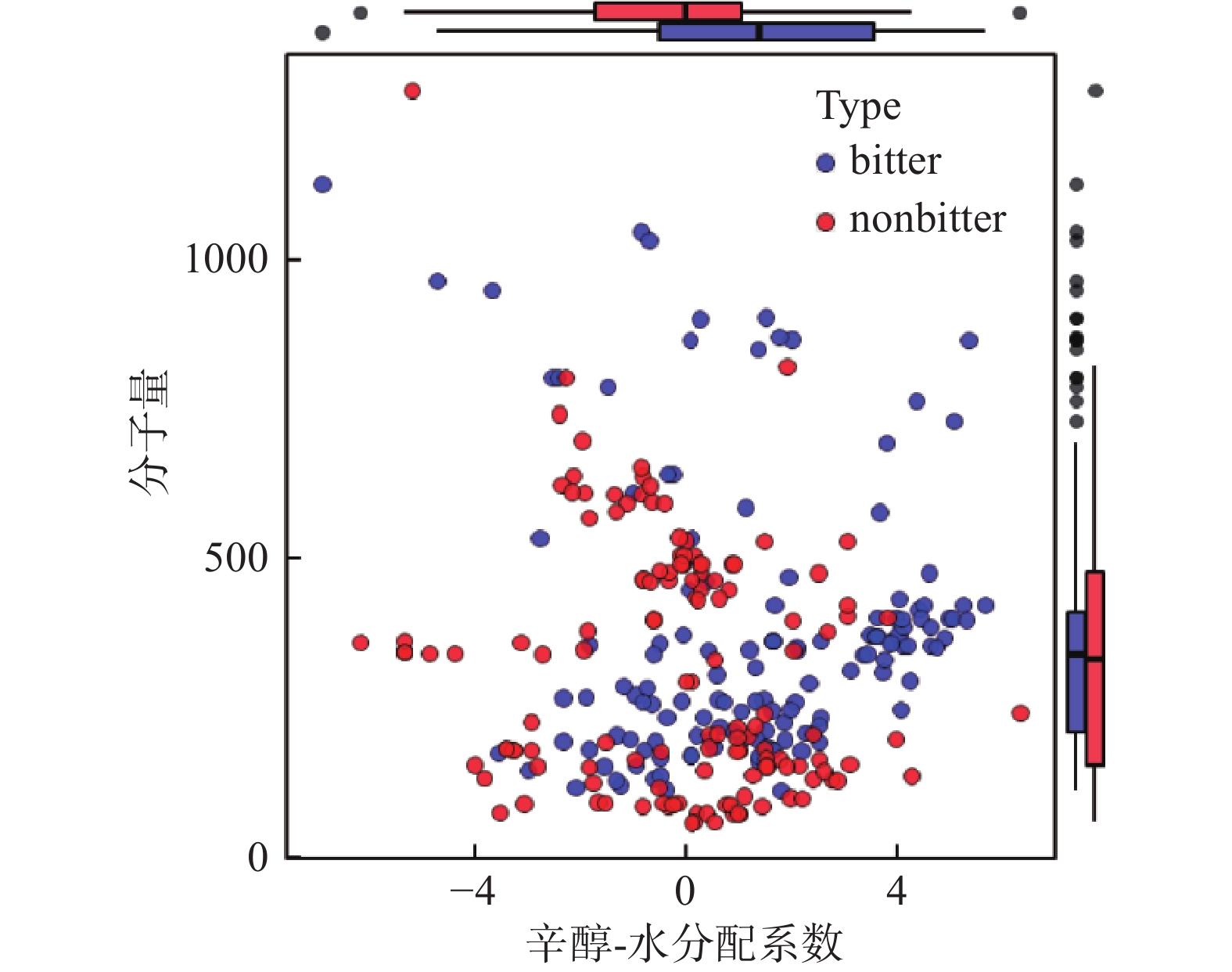
所建立的苦味-非苦味数据集由139个苦味分子以及139个非苦味分子共278个分子组成用于建立苦味分子识别模型,苦味阈值数据集共有139个分子用于建立苦味分子阈值预测模型。苦味分子的阈值范围在0.004~166之间,阈值分布近正态分布,说明用于建模的数据合理,适用于建立预测模型并采用显著性分析比较模型间差异。非苦味分子数据集有139个非苦味分子,数据来源于FooDB、FlavorDB及Fenaroli的风味成分手册(第5版)。如图1展示了分子量与辛醇-水分配系数(Log octanol/water partition coefficient,log10P(o/w))的特征分布,log10P(o/w)为分子疏水性的物理参数,其分布反映了分子的疏水特性。苦味与非苦味分子在log10P(o/w)和分子量上的分布范围基本一致,提高了模型的准确度。Rodgers等[4]使用的非苦味数据集是从MDL药物数据集(MDDR)中随机选择的13530个假设不具有苦味的分子作为非苦数据集,与此相比,本实验所用的非苦味数据集是经人工修正的数据集,经实验验证过的数据,降低了模型的噪声,提高模型的实际应用能力;Tuwani等[29]使用甜味剂作为非苦味数据集,虽然数据集采用的是经实验验证过的数据,但两种分子结构差异太大,对于结构相似的物质区分的准确性有待考证。本文搜集的数据集,其苦味、非苦味数据集贴近食品实际,增加了模型的实际应用价值。
2.2 分子描述符的选择
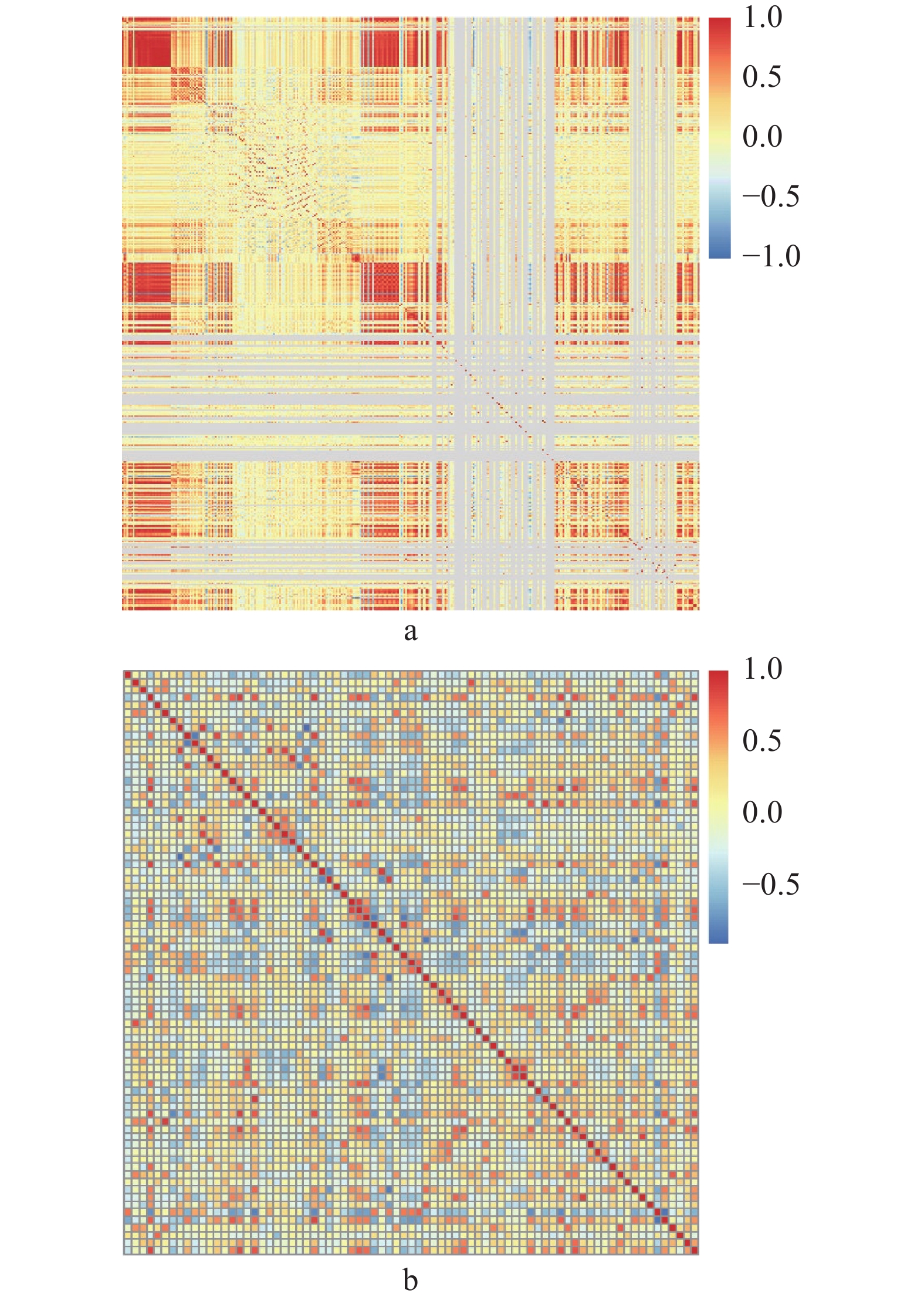
为了降低冗余特征的影响,本文使用了近零方差、描述符共线性和主成分分析(PCA)的方法来筛选分子描述符。近零方差去除了无代表性特征和不相关特征,很多学者研究认为酚类化合物的分子量越小,苦味强度越高,对于小分子肽,其苦味与含有的疏水性氨基酸的数量和种类有关,通常苦味会随着疏水性的增加而增强[30-31],所以像以上两种能够有效描述苦味分子的特征描述符予以保留,反之剔除;描述符共线性去除并只保留一个描述同一特征的描述符;PCA线性地组合属性,使得特征彼此正交,并捕获数据的最大方差。以上方法都适用于训练集分子对应的描述符。MOE 2D共生成206个描述符,有33个分子描述符被近零方差识别为与苦味分类预测无关,有89个分子描述符被共线性识别为彼此之间存在线性关系,不能区分各自对所要描述调整的具体作用,PCA优化后分子描述符集合中含有80个分子描述符。ChemoPy 2D共生成574个描述符,有230个分子描述符被近零方差识别为与苦味分类预测无关,有219个分子描述符被共线性识别为彼此之间存在线性关系,不能区分各自对所要描述调整的具体作用,PCA优化后分子描述符集合中含有97个分子描述符。Mordred 2D共生成1613个描述符,有415个分子描述符被近零方差识别为与苦味分类预测无关,有787个分子描述符被共线性识别为彼此之间存在线性关系,不能区分各自对所要描述调整的具体作用,PCA优化后分子描述符集合中含有34个分子描述符。如图2所示,图2a为Mordred 2D描述符筛选前热图,图2b为Mordred 2D描述符筛选后的热图。其中红色代表正相关,蓝色代表负相关,经过描述符筛选后,红色区域减少。结果显示经过筛选后的描述相关性较低,表明这些描述符提供的化学信息在某种程度上是独一无二的。
高度的多重共线性会导致说明变量对因变量的单独影响区分不开,变量间相互依存的强度和出现的共线性变量的重要性,会影响参数估计值[32]。PCA把多指标转化为几个综合指标,降低观测空间的维数,以获取最主要的信息,通过少数几个主成分最大限度地描述数据特点[16]。因此,通过以上优化可大大降低模型噪声,提高模型准确度。
2.3 苦味分子识别模型
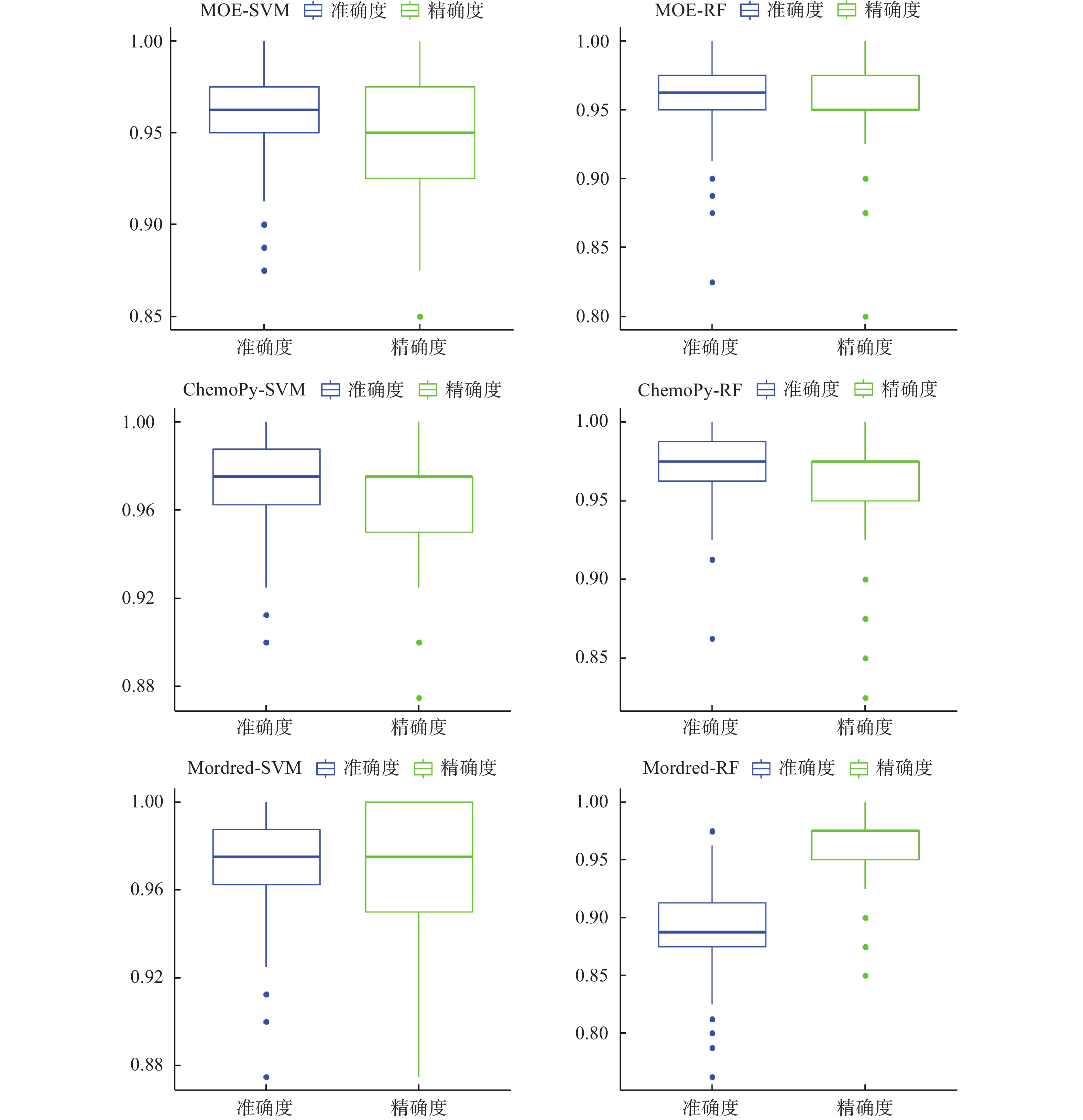
通过在苦味-非苦味数据集上进行分层抽样,抽取3/4的数据(208个分子)作为训练集,1/4的数据(70个分子)作为测试集建立苦味分子识别模型。如图3所示,基于MOE 2D、ChemoPy 2D及Mordred 2D转化的描述符数据建立的SVM苦味分子识别模型,以下分别简称MOE-SVM模型、ChemoPy-SVM模型及Mordred-SVM模型,其在测试集上的准确度(Accuracy)范围分别为:0.900~1.000、0.886~1.000及0.929~1.000,其在测试集上的准确度(Accuracy)平均值为:0.968、0.965及0.979,其在测试集上的精确度(Precision)范围分别为:0.857~1.000、0.800~1.000及0.943~1.000,其在测试集上的精确度(Precision)平均值分别为:0.963、0.944、0.985。基于MOE 2D、ChemoPy 2D及Mordred 2D转化的描述符数据建立的RF苦味分子识别模型,以下分别简称MOE-RF模型、ChemoPy-RF模型及Mordred-RF模型,其在测试集上的准确度(Accuracy)范围分别为:0.929~1.000、0.843~1.000及0.729~0.986,其在测试集上的准确度(Accuracy)平均值为:0.982、0.960及0.881,其在测试集上的精确度(Precision)范围分别为:0.943~1.000、0.771~1.000及0.943~1.000,其在测试集上的精确度(Precision)平均值分别为:0.987、0.942及0.985。MOE-RF模型的准确度和精确度为0.982和0.987,均高于其他模型,说明MOE-RF模型可以较好地进行苦味分子识别。
2.4 苦味分子阈值预测模型
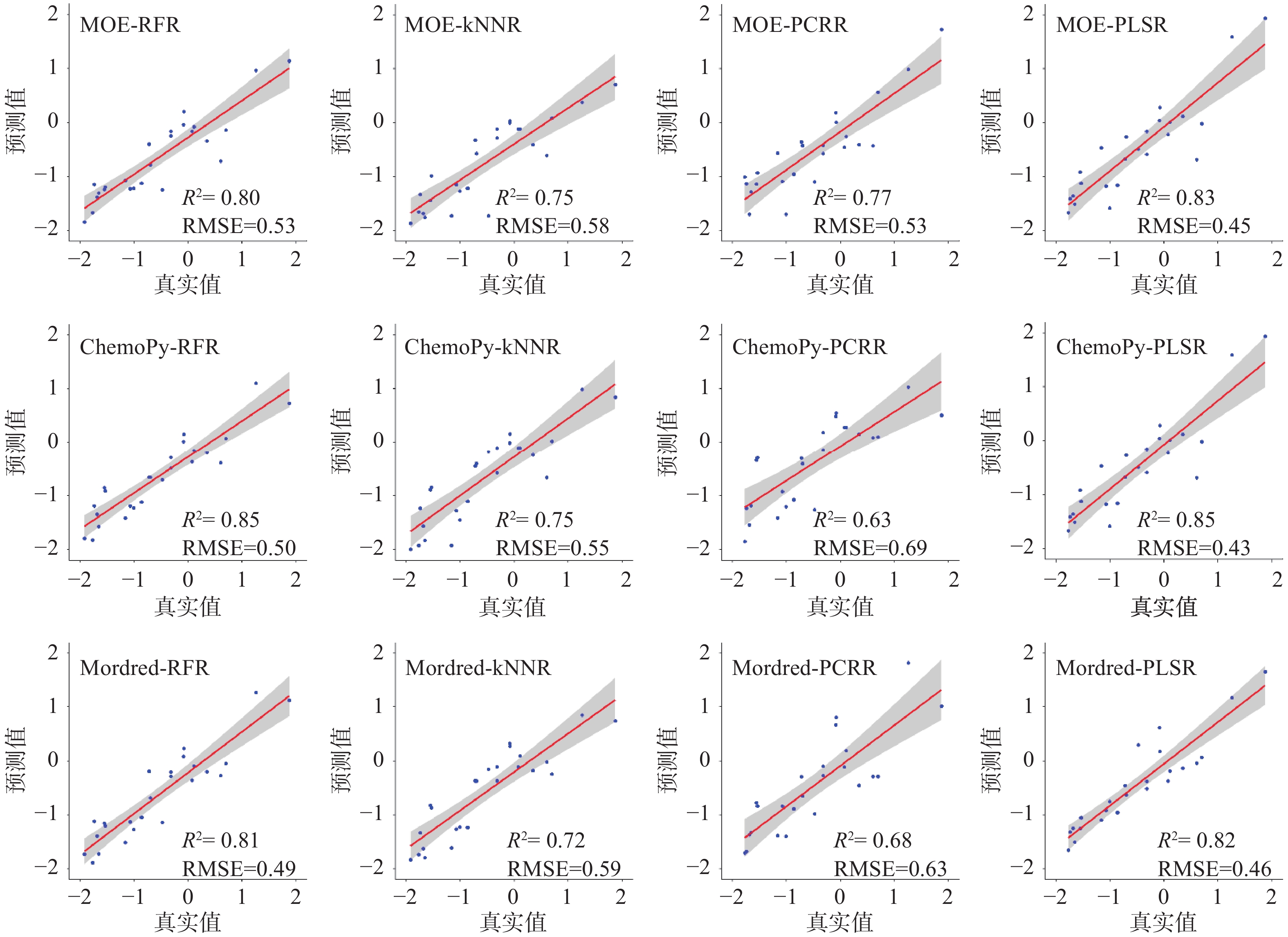
通过在苦味阈值数据集上进行无放回随机抽样,抽取3/4的数据(104个分子)作为训练集,1/4的数据(35个分子)作为测试集建立苦味分子阈值预测模型,模型在测试集上的性能评估如图4所示,采用MOE 2D描述符转化的数据和基于RFR、kNNR、PCR和PLSR四种算法从而建立的苦味分子阈值预测模型,以下分别简称MOE-RFR模型、MOE-kNNR模型、MOE-PCR模型及MOE-PLSR模型,其决定系数(R2)分别为:0.80、0.75、0.77、0.83,其均方根误差(RMSE)分别为0.53、0.58、0.53、0.45。采用ChemoPy 2D描述符转化的数据和基于RFR、kNNR、PCR和PLSR四种算法从而建立的苦味分子阈值预测模型,以下分别简称ChemoPy-RFR模型、ChemoPy-kNNR模型、ChemoPy-PCR模型及ChemoPy-PLSR模型,其决定系数(R2)分别为:0.85、0.75、0.63、0.85,其均方根误差(RMSE)分别为0.50、0.55、0.69、0.43。采用Mordred描述符转化的数据和基于RFR、kNNR、PCR和PLSR四种算法从而建立的苦味分子阈值预测模型,以下分别简称Mordred-RFR模型、Mordred-kNNR模型、Mordred-PCR模型及Mordred-PLSR模型,其决定系数(R2)分别为0.81、0.72、0.68、0.83,其均方根误差(RMSE)分别为:0.49、0.59、0.63、0.46,置信度为95%。
苦味分子在描述符转化后,利用RFR、kNNR、PCR和PLSR四种算法进行模型构建。在RFR模型中,使用ChemoPy 2D描述符转化数据建立的模型(ChemoPy-RFR)的拟合度最好,其决定系数(R2)为0.85,均方根误差(RMSE)为0.50;在kNNR模型中,使用ChemoPy 2D描述符转化数据建立的模型(ChemoPy-kNNR)的拟合度最好,其R2为0.75,RMSE为0.55;在PCR模型中,使用MOE 2D描述符转化数据建立的模型(MOE-PCR)的拟合度最好,其R2为0.77,RMSE为0.53;在PLSR模型中,使用ChemoPy 2D描述符转化数据从而建立的模型(ChemoPy-PLSR)的拟合度最高,其R2为0.85,RMSE为0.43。综合以上结果得出:基于ChemoPy 2D描述符建立的ChemoPy-PLSR模型的拟合度最好,预测效果最准确,均方根误差最低,模型稳定,其R2为0.85,RMSE为0.43,适合做苦味阈值预测模型。
2.5 苦味分子识别模型及苦味分子阈值预测模型应用
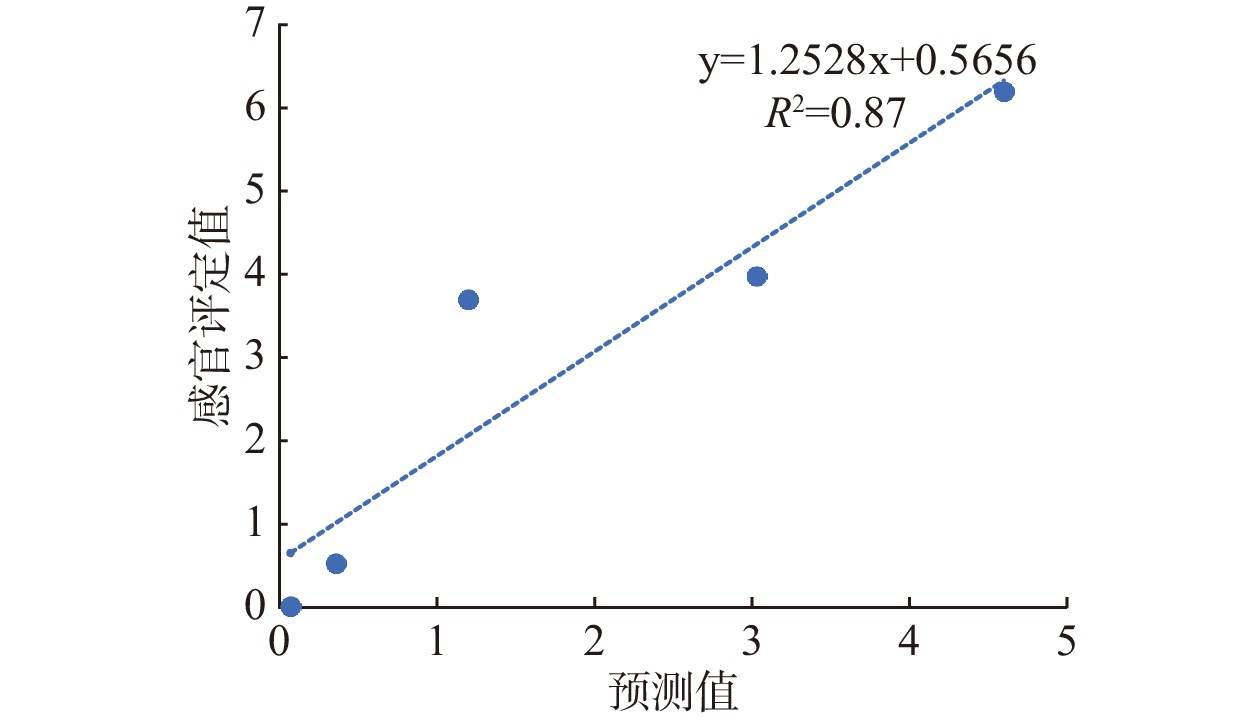
该研究为了预测潜在的苦味分子及其苦味阈值建立了苦味分子识别模型和苦味阈值预测模型,将上述模型应用于FooDB进行未知分子的苦味及其苦味阈值预测,并通过感官评价对对预测结果的苦味分子及阈值验证。经本研究的最优模型Mordred-SVM模型预测,并将预测为苦味的分子使用ChemoPy-PLSR模型进一步对阈值进行预测,预测结果见“FooDB_results.csv” (https://gitee.com/wang_lab/BRATP),其中有5417个分子被预测为苦味。随机挑选部分化合物的预测结果如表1所示:咖啡因、维生素B1、β-D-氨基葡萄糖、香橙素、叶酸。这些化合物的阈值分别为3.030、0.360、4.600、0.070、1.200 mmol/L。随机挑选的这5种物质用于验证模型的预测结果,虽然可能不是最具代表性的,结果如图5所示,模型预测阈值与感官评价值拟合曲线决定系数为0.87,取得了较好的验证结果。随着大量研究的进行,在除这5种之外的分子上也可能取得较好的验证结果。
表 1 化合物阈值预测结果Table 1. Prediction results of compound threshold化合物名字 分子结构 预测阈值(mmol/L) 感官评价值(mmol/L) 咖啡因 
3.030 3.980 维生素B1 
0.360 0.530 β-D-氨基葡萄糖 
4.600 6.200 香橙素 
0.070 0.0150 叶酸 
1.200 3.700 3. 讨论与结论
Luciana等[33]使用DRAGON plus v.5.0描述符和MobyDigs v.1.0 软件,在遗传算法(Genetic Algorithm, GA)方法下,使用多元线性回归(Multiple Linear Regression, MLR)搜索最佳模型,即可变子集选择-遗传算法(VSS-GA)方法,得出了苦味与分子极性非直接相关且羟基和酯片段可以降低苦味的结论,由此可见,苦味强度与结构有关。但是Luciana Scotti等并未训练苦味数据集,同时也未建立苦味分子结构与阈值的线性关系。苦味分子使用Mordred 2D转化生成描述符从而建立的SVM模型的性能最好,其准确度和精确度分别为0.979和0.985;使用MOE 2D转化生成描述符从而建立的RF模型的性能最好,其准确度和精确度分别为0.982和0.985。综合以上结果得出:基于MOE 2D描述符建立的RF模型(MOE-RF模型)性能最好,其准确度和精确度分别为:0.982、0.985,最适合做苦味识别模型。与公布的最佳表现模型相比,MOE-RF苦味识别模型在准确性方面优于Zheng等[34]的e-Bitter模型和Banerjee等[7]的BitterSweet Forest模型,其中e-Bitter模型使用“Phytochemical Dictionary”数据集和1024bit-ECFP4和2048bit-ECFP6描述符,运用RF算法得到模型的准确率为85%~92%,本研究苦味识别模型的准确度较其提高了6%~13%;BitterSweet Forest模型使用苦甜数据集和Morgan-feat分子描述符,运用RF算法得到模型的准确度为95%,本研究苦味识别模型的准确度较其提高了3%。由此可见,MOE-RF模型准确度高,稳定性强,更适用于苦味分子识别模型。且本研究的ChemoPy-PLSR苦味阈值预测模型对苦味分子结构及其阈值进行回归分析,得出了较好的结果,该模型更精确地预测了苦味分子的阈值。
本研究基于MOE 2D、ChemoPy 2D和Mordred 2D三种描述符的优化及选择,比较了由RF和SVM算法所建立的苦味分子识别模型,结果表明MOE-RF模型的准确度和精确度最高,其准确度和精确度分别为0.982、0.985。同时建立并比较了由RFR、PLSR、kNNR、PCR算法所建立的苦味阈值预测模型,结果表明,ChemoPy-PLSR模型的拟合度最好,均方根误差最低,其决定系数和均方根误差分别为0.85、0.43。由此可见,两个模型具有良好的预测能力,可以用于苦味分子的分类预测及阈值预测。苦味识别模型使用了经实验验证过的数据,且对非苦味数据集进行了规范,较以往的分类模型更有可信度,准确度更高;本研究进一步实现了基于苦味分子结构预测苦味阈值的方法,模型拟合度较好,为苦味阈值预测提供了可行方法,具有一定的实际意义。所有数据和代码存储在https://gitee.com/wang_lab/BRATP,其他研究人员既可以利用本研究的代码,继续发掘其他苦味剂,也可以设计新的算法,获得更为准确的预测结果。
-
表 1 化合物阈值预测结果
Table 1 Prediction results of compound threshold
化合物名字 分子结构 预测阈值(mmol/L) 感官评价值(mmol/L) 咖啡因 
3.030 3.980 维生素B1 
0.360 0.530 β-D-氨基葡萄糖 
4.600 6.200 香橙素 
0.070 0.0150 叶酸 
1.200 3.700 -
[1] 代丽凤, 罗理勇, 罗江琼, 等. 植物苦味物质概况及其在食品工业的应用[J]. 中国食品学报,2020,20(11):305−318. [DAI L F, LUO L Y, LUO J Q, et al. General situation of plant bitter substances and their application in food industry[J]. Chinese Journal of Food Science,2020,20(11):305−318. [2] 张璞, 张耀, 桂新景, 等. 基于经典人群口尝法和电子舌法的中药饮片水煎液苦度叠加规律研究[J]. 中草药,2021,52(3):653. [ZHANG P, ZHANG Y, GUI X J, et al. Study on the superimposition law of bitterness of Chinese herbal medicine decoction pieces based on the classic oral taste method and electronic tongue method[J]. Chinese Herbal Medicine,2021,52(3):653. doi: 10.7501/j.issn.0253-2670.2021.03.007 [3] ZHOU W, DAI Y, MENG J, et al. Network pharmacology integrated with molecular docking reveals the common experiment-validated antipyretic mechanism of bitter-cold herbs[J]. J Ethnopharmacol,2021,274:114042. doi: 10.1016/j.jep.2021.114042
[4] RODGERS S, GLEN R C, BENDER A. Characterizing bitterness: Identification of key structural features and development of a classification model[J]. Journal of Chemical Information & Modeling,2006,46(2):569−576.
[5] JESSICA A, SASKIA P, MATHIAS D, et al. Supersweet—a resource on natural and artificial sweetening agents[J]. Nucleic Acids Research,2010,39(Database):377−382.
[6] AYANA W, MARINA S, ANAT L, et al. BitterDB: A database of bitter compounds[J]. Nucleic Acids Research,2012,40(D1):D413−D419. doi: 10.1093/nar/gkr755
[7] BANERJEE P. Bitter Sweet forest: A random forest based binary classifier to predict bitterness and sweetness of chemical compounds[J]. Front Chem,2018,6:93. doi: 10.3389/fchem.2018.00093
[8] MARGULIS E, DAGAN A, IVES S, et al. Intense bitterness of molecules: Machine learning for expediting drug discovery[J]. Computational and Structural Biotechnology Journal,2021,19:568−576. doi: 10.1016/j.csbj.2020.12.030
[9] JESÚS J, RICO P, MEDINA L. Analysis of a large food chemical database: Chemical space, diversity, and complexity[J]. F1000 Research,2018,7:993. doi: 10.12688/f1000research.15440.2
[10] NEELANSH G, APUROOP S, RUDRAKSH T, et al. FlavorDB: A database of flavor molecules[J]. Nucleic Acids Research, 2018, 4(46): 1210-1216.
[11] PANESAR S, KENNEDY F. Burdock (Ed.), Fenaroli’s Handbook of Flavor Ingredients, 5th ed., CRC Press, Boca Raton, FL, USA, ISBN 0-8493-3034-3[J]. Carbohydrate Polymers,2006,65(3):386.
[12] YANLI W, BRYANT H, TIEJUN C, et al. PubChem BioAssay: 2017 update[J]. Nucleic Acids Research,2017,45:955−963. doi: 10.1093/nar/gkw1118
[13] SANTIAGO V. Medicinal chemistry and the molecular operating environment (MOE): Application of QSAR and molecular docking to drug discovery[J]. Current Topics in Medicinal Chemistry,2008,8(18):1555−1572. doi: 10.2174/156802608786786624
[14] DONG C, QING X, QIAN H, et al. ChemoPy: Freely available python package for computational biology and chemoinformatics[J]. Bioinformatics,2013,29(8):1092−1094. doi: 10.1093/bioinformatics/btt105
[15] MORIWAKI H, TIAN Y, KAWASHITA N, et al. Mordred: A molecular descriptor calculator[J]. Journal of Cheminformatics,2018,10(1):4. doi: 10.1186/s13321-018-0258-y
[16] VED A, DDDS F, PHGD D, et al. Scores selection via Fisher’s discriminant power in PCA-LDA to improve the classification of food data[J]. Food Chemistry,2021,363:130296. doi: 10.1016/j.foodchem.2021.130296
[17] TEAM R. R: A language and environment for statistical computing[Z]. 3.5. 1. R Foundation for Statistical Computing, 2020.
[18] WICKHAM H. Ggplot2: Elegant graphics for data analysis[Z]. Springer-Verlag New York, 2016.
[19] LIAW A, MATTHEW W. Classification and regression by randomForest[Z]. R News, 2002: 2, 18-22.
[20] MEYER D, DIMITRIADOU E, HORNIK K, et al. Misc functions of the department of statistics, probability theory group (Formerly: E1071), TU Wien[Z]. 2020: e1071.
[21] SCHLIEP K, HECHENBICHLER K. Kknn: Weighted k-nearest neighbors[Z]. 2016.
[22] VENABLES W, RIPLEY B. Modern applied statistics with s[Z]. New York: Springer, 2002.
[23] TILLÉ Y, MATEI A. Sampling: Survey sampling[Z]. 2016.
[24] FB S, WB N, RJ P. Random forest as one-class classifier and infrared spectroscopy for food adulteration detection[J]. Food Chemistry,2019,293:323−332. doi: 10.1016/j.foodchem.2019.04.073
[25] PHILLIPS T, ABDULLA W. Developing a new ensemble approach with multi-class SVMs for Manuka honey quality classification[J]. Applied Soft Computing,2021,111:107710. doi: 10.1016/j.asoc.2021.107710
[26] YONG L, LIAO S, JIANG S, et al. Fast cross-validation for kernel-based algorithms[J]. IEEE Transactions on Pattern Analysis and Machine Intelligence,2020,42(5):1083−1096.
[27] INTELMANN D, BATRAM C, KUHN C, et al. Three TAS2R bitter taste receptors mediate the psychophysical responses to bitter compounds of hops (Humulus lupulus L.) and beer[J]. Chemosensory Perception,2009,2(3):118−132. doi: 10.1007/s12078-009-9049-1
[28] ANONYMOUS D. ISO 4120—Sensory analysis—methodology—triangle test (ISO 4120: 2004). German Version (EN ISO 4120: 2007)[S].
[29] TUWANI R, WADHWA S, BAGLER G. BitterSweet: Building machine learning models for predicting the bitter and sweet taste of small molecules[J]. Scientific Reports,2019,9(1):7155. doi: 10.1038/s41598-019-43664-y
[30] 郭兴峰, 魏芳, 周祥山, 等. 苦味肽的形成机理及脱苦技术研究进展[J]. 食品研究与开发,2017,38(21):207−211. [GUO X F, WEI F, ZHOU X S, et al. The formation mechanism of bitter peptides and the research progress of debittering technology[J]. Food Research and Development,2017,38(21):207−211. doi: 10.3969/j.issn.1005-6521.2017.21.041 [31] 毕继才, 崔震昆, 张令文, 等. 苦味传递机制与苦味肽研究进展[J]. 食品工业科技,2018,39(11):333−338. [BI J C, CUI Z K, ZHANG L W, et al. Bitterness transmission mechanism and research progress of bitter peptides[J]. Food Industry Science and Technology,2018,39(11):333−338. [32] BAYMAN E O, DEXTER F. Multicollinearity in logistic regression models[J]. Anesthesia & Analgesia,2021,133(2):362−365.
[33] LUCIANA S, MARCUS T S, HAMILTON M I, et al. Quantitative elucidation of the structure–bitterness relationship of cynaropicrin and grosheimin derivatives[J]. Food Chemistry,2007,105(1):77−83. doi: 10.1016/j.foodchem.2007.03.038
[34] ZHENG S Q, JIANG M Y, ZHAO C W, et al. E-Bitter: Bitterant prediction by the consensus voting from the machine-learning methods[J]. Frontiers in Chemistry,2018,6:82. doi: 10.3389/fchem.2018.00082
-
期刊类型引用(4)
1. 刘一江,方芳. 高盐稀态酱油发酵过程褐变类型及影响褐变糖类的研究. 食品与发酵工业. 2025(02): 99-104 .  百度学术
百度学术
2. 魏玉磊,许明磊,崔宏伟,田雷,张忠锋,杜咏梅,周静,赵昆,侯小东. 烤烟烟花游离氨基酸组成及在发育过程中的变化研究. 中国烟草科学. 2025(01): 81-86 .  百度学术
百度学术
3. 阿丽耶·司马义,热伊汉古丽·萨地克,黄蓉,冯作山,阿衣古丽·阿力木. 羊肉蛋白酶解工艺优化及酶解液中氨基酸含量分析. 食品研究与开发. 2024(01): 92-98 .  百度学术
百度学术
4. 芦鑫,张丽霞,孙强,游静,黄纪念. 配料组成对高温芝麻饼粕蛋白酶解物制备肉味香精的影响. 食品工业科技. 2024(14): 50-61 .  本站查看
本站查看
其他类型引用(3)







 下载:
下载:
 下载:
下载:



