Investigation on the Effect of Temperature on the Activity of Lactate Dehydrogenase Based on Molecular Dynamics Simulation
-
摘要: 乳酸脱氢酶是糖无氧酵解及糖异生的重要酶系之一,它能够催化丙酮酸形成乳酸,在食品发酵工业中具有很高的应用价值,但该酶易受高温影响,导致乳酸制品产量下降。为了研究不同温度对乳酸脱氢酶的构象及活性的影响,采用分子动力学模拟的方法,针对4种不同温度条件下(37、55、70和85 ℃)的乳酸脱氢酶分别进行了80 ns的计算模拟,分析了构象变化及酶活性中心的差异。研究发现,在37 和55 ℃条件下,乳酸脱氢酶比较稳定;当温度升高至70和85 ℃,乳酸脱氢酶的均方根误差、均方根波动、回旋半径值和溶剂可及表面积显著增加,而85 ℃ 时的蛋白二级结构已发生较大改变,这表明高温会导致蛋白质构象不稳定。对比37和85 ℃条件下该酶的底物丙酮酸的结合能力,发现高温会导致丙酮酸的结合位点残基之间的距离增大,进而破坏底物分子结合的微环境。因此,乳酸脱氢酶在温度超过70 ℃时发生变性,并随着温度的升高变性程度增加,进而导致酶活性丧失,不利于其在食品发酵等方面的应用。本研究在原子水平上分析了4种不同温度对乳酸脱氢酶的影响,揭示了其酶活性及构象变化的关键信息,为乳酸制品在发酵过程中选择合适温度提供了理论支撑。Abstract: Lactate dehydrogenase (LDH) is one of the important enzymes for anaerobic glycolysis and gluconeogenesis. It could catalyze pyruvate to form lactic acid, which had high application value in food fermentation industry. However, LDH was easily affected by high temperature, which leaded to the decline of lactic acid production. To study the effects of different temperature on the conformation and activity of LDH, molecular dynamics simulations at four different temperatures (37, 55, 70 and 85 °C) were performed for 80 ns, respectively. The overall conformational changed and the differences of enzyme active centers were analyzed. The results showed that LDH was relatively stable at 37 and 55 °C, and the root mean square error, root mean square fluctuation, radius gyration and solvent accessible surface area of LDH increased significantly at 70 and 85 °C. Moreover, the secondary structure of protein had changed greatly at 85 °C, which indicated that high temperature would lead to protein conformational instability. Comparing the binding ability of pyruvate at 37 and 85 °C, which was the substrate of the enzyme, we found that high temperature would increase the distance between the residues of pyruvate binding sites, and then destroy the microenvironment of substrate molecular binding. Therefore, the denaturation of LDH occurred when the temperature exceeded 70 °C, and the denaturation degree was positively correlated with the increase of temperature. This would lead to the loss of its enzyme activity, which was not conducive to the application value in food industry. In this study, the effects of four different temperatures on LDH were analyzed at atomic level, and the key information of enzyme activity and conformation changes were revealed, which provided theoretical support for the selection of appropriate temperature in the fermentation process of lactic acid products.
-
Keywords:
- lactate dehydrogenase /
- molecular dynamics simulation /
- temperature /
- stability /
- enzyme activity
-
近年来,有益菌被用于食品相关的生产加工屡见不鲜,因为其除了能够提供给人体营养物质之外,也对肠道微生物菌群的平衡起到重要作用[1-3]。干酪乳杆菌是一类有益于人体健康的活性微生物食品原料,与嗜酸乳杆菌和双歧杆菌一起被称为“健康三益菌”[4-5]。近年来,干酪乳杆菌经常被用作豆奶、酸乳、牛奶、干酪和奶油等乳制品的发酵工艺中,这可能与该菌在发酵过程中拥有较高的乳酸脱氢酶(LDH)活性密不可分[6-9]。该菌的最适生长温度为37 ℃,但是发酵工程中,往往需要提高温度抑制杂菌的生长,甚至需要加热杀菌以保证奶制品的品质,这可能会导致发酵效率降低甚至口感改变,这也是相关食品生产过程中遭遇的难题[10-11]。
LDH广泛存在于动物、植物、细菌、真菌等几乎所有生命体的组织细胞中。该酶一般是以辅酶烟酰胺腺嘌呤二核苷酸NADH传递氢,在生物体内能够催化丙酮酸形成乳酸[12-16]。LDH是同源四聚体结构,一个酶分子含有4个相同的亚基,每个亚基的催化功能都是相对独立的,当催化底物分子为丙酮酸时,该酶首先和辅酶 NADH 发生结合,进而丙酮酸与该酶结合并接受 NADH 提供的[H],丙酮酸发生氧化反应,最终生成乳酸和NAD+的产物[17]。
通常情况下,LDH 可将手性的α-酮酸还原生成手性α-羟基酸,故该酶的底物非常广泛,除丙酮酸外,还有对羟基苯丙酮酸、草酰乙酸、苹果酸和苯甲酰甲酸等[18]。因此,研究不同温度梯度条件对LDH酶活性的影响,对生命体具有普遍性意义。然而,目前研究LDH热稳定性的方法主要体现在常规的实验方法上,该方法无法直观反映出温度引起氨基酸残基的波动性、回旋半径、溶剂可及表面积等微观原子水平的变化程度。随着计算生物学的发展,近年来,越来越多的研究者采用分子动力学模拟的技术手段来研究蛋白质分子的运动轨迹,从而分析原子水平的构象变化信息[19]。
本研究选择干酪乳杆菌的LDH为研究目标,采用分子动力学模拟的方法,研究LDH在不同温度梯度条件下(37、55、70和85 ℃)的热稳定性以及酶活性中心氨基酸残基的变化,将分析LDH的均方根误差、回旋半径、均方根波动值、溶剂可及表面积以及二级结构含量等原子水平的指标变化,以期为LDH参与的食品发酵过程中采取适宜加工温度提供有利的参考。
1. 材料与方法
1.1 分子模拟的条件
本研究利用Gromacs2020.4软件包,LDH的结构(PDB: 2zqy)来源于RCSB蛋白质数据库网站(https://www.rcsb.org/),首先对LDH结构进行预处理,即去除结晶水和杂质离子。分子模拟分为四个独立的体系,对应的温度设置分别为37、55、70和85 ℃,即在正式模拟的mdp文件中分别修改“ref_t和gen_temp”值为开尔文310.15、328.15、343.15和358.15。选择这四个温度的依据分别是,37 ℃为干酪乳杆菌的最适生长温度,可代表正常状态下的LDH;55 ℃为大多数蛋白比较常见的变性起始温度[20-21];70 ℃是经过预实验测试的大致变性温度;85 ℃是能够导致绝大多数蛋白变性的温度。采用Gromos43a1分子力场、SPC水模型进行模拟[22],模拟总时长为80 ns。模拟之前,将LDH的结构作为一个立方周期盒中的起始构象,并将蛋白质与盒边界的最小距离设为1.0 nm。模拟系统的周期性边界条件适用于X/Y/Z三个方向,在溶胶中加入0.15 mol/L NaCl盐的溶质对系统的电荷进行中和。在模拟系统中,用粒子网格法PME计算静电相互作用,用蛙跳算法计算原子运动。用最速下降能量法进行400步能量的最小化。接下来,共轭梯度法被用于执行25000步的能量最小化,随后对每个系统进行了50 ps的位置约束仿真。正式动力学模拟具有随机的起始速度。利用PyMOL和VMD软件作为可视化工具,用Origin 8.5软件生成数据结果。
1.2 模拟结果的分析
分别用gmx rmsf工具计算了每个氨基酸残基的α-C的均方根涨落,用gmx gyrate工具计算了原子演化随模拟时间的回转半径。利用VMD-1.9.1软件从分子动力学模拟轨迹上观察四个不同温度的模拟体系中的蛋白构象变化[23]。然后用gmx distance分别测量了LDH的典型丙酮酸结合位点残基的平均距离,如精氨酸-106(Arg106)和精氨酸-169(Arg169)、组氨酸-193(His193)和苏氨酸-247(Thr247)。并用PyMOL和origin 8.5软件绘制了结构图。
2. 结果与分析
2.1 LDH蛋白的三维结构概况
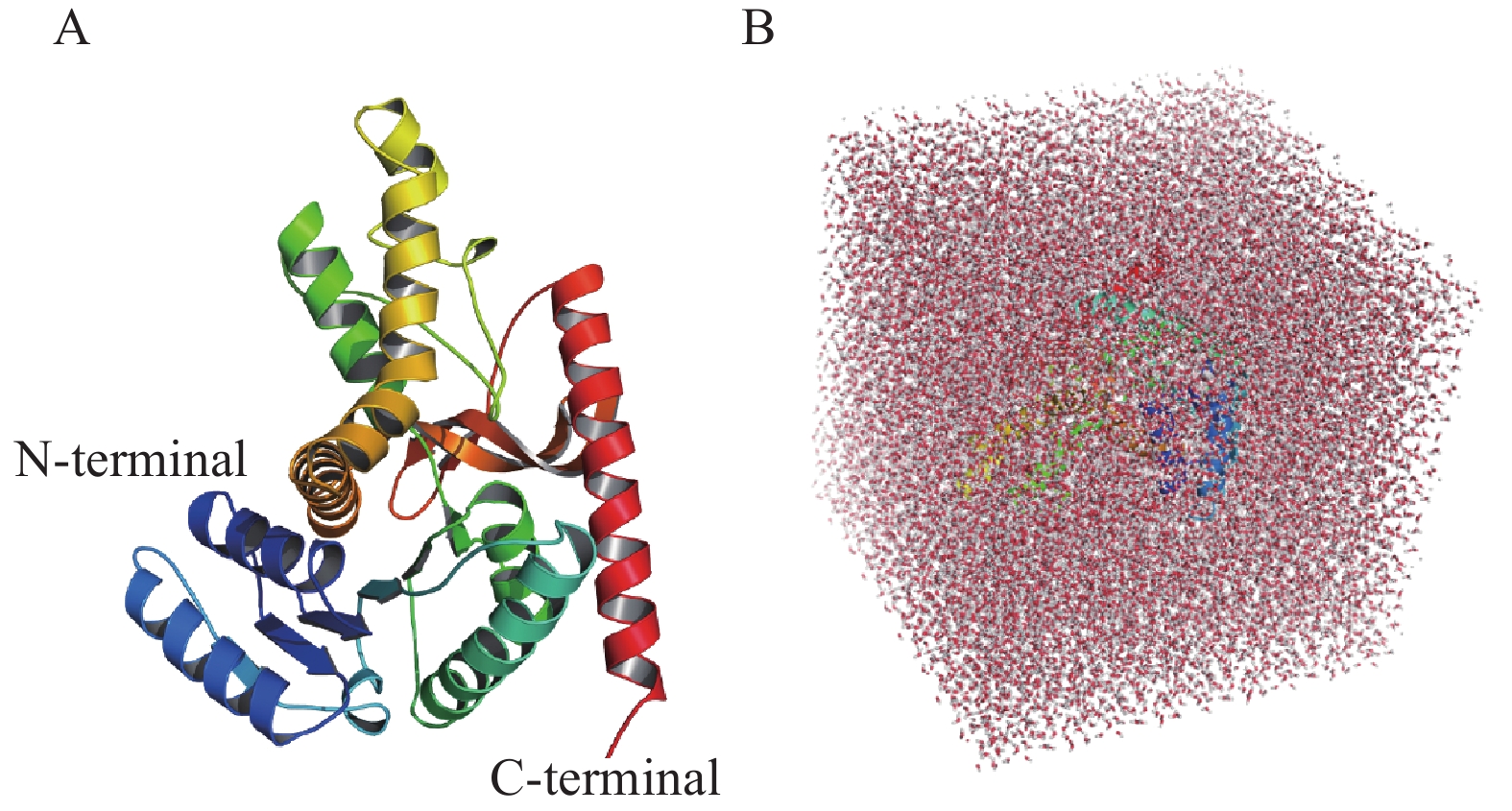
LDH广泛存在于动物、植物及微生物等生物体内,据报道,该酶的主要存在形式是含有四个相同亚基的同源四聚体结构,且每个亚基都能单独发挥催化活性[24-26]。每个亚基由326个氨基酸残基构成蛋白质一级结构,如图1A所示,其分子量约为35.5 kDa,它的二级结构主要由8个α-螺旋和9个β-折叠组成,但大多数β-折叠被包裹在内部,形成疏水基团。根据PDB数据库多种LDH的结构信息得知,其与底物分子的结合活性中心主要位于α-螺旋之间[27];而β-折叠则对蛋白整体构象的稳定起到重要作用。在模拟体系中添加了水分子和NaCl盐离子,并进行了能量最小化,能量优化后的晶胞参数x/y/z分别为8.82403、8.82403、8.82403 nm(图1B),展示了该蛋白的立方体模拟体系。
2.2 均方根误差的分析
首先针对37、55、70和85 ℃四个不同温度,分别构建了四个独立的模拟体系。为了评价蛋白质在80 ns总时长模拟过程中是否达到平衡,采用均方根误差(root mean square deviation, RMSD) 以度量蛋白质构象与原始结构之间的平均偏差,RSMD是衡量体系是否稳定的重要依据[28]。如图2所示,在37 ℃的模拟体系中,LDH最先达到平衡状态,且RMSD值是最小的(约为3.4 Å);55 ℃的条件下的RMSD值在0~55 ns上升到4.1 Å,55~80 ns进入平衡状态,说明该条件下的蛋白体系仍然趋于稳定;当蛋白处于70 ℃和85 ℃条件下,发现其RMSD值曲折上升,最大值高达6.0 Å以上,即在80 ns内仍无法达到平衡,这说明温度大于70 ℃的情况下蛋白质的构象不稳定,逐渐开始变性。根据文献报道,GREEN[29]等已证实LDH的熔解温度为(70.13±0.062)°C,因此,70 ℃的模拟条件大致能代表LDH熔解温度的临界值。
2.3 氨基酸残基的均方根波动分析
均方根波动值(root mean square fluctuation, RMSF)的计算,能够准确表示出每个原子相对于其平均位置的涨落,它表征了结构相对于时间的平均变化,可以从中分析获得蛋白柔性区域尤其是关键氨基酸残基的稳定性等信息,对应于晶体学中的b温度因子[30]。
通过观察残基的RMSF值的变化,可以获取不同温度下蛋白质的变性程度,如图3所示,37 ℃为干酪乳杆菌的最适生长温度,该温度下LDH具备正常工作的酶活力,对应的RMSF值最低(正常值),说明此时的蛋白构象最稳定;与37 ℃的正常值相比,55 ℃的RMSD值几乎没有出现剧烈波动,这表明该温度对蛋白结构没有造成本质的影响;然而,70 ℃对蛋白N端和C端的多数残基造成了波动性增大的影响,这说明随着温度上升,LDH蛋白碳骨架的波动性逐渐增大;85 ℃条件下,几乎所有残基的RMSF值都大于正常值,这说明此时LDH蛋白已经变性。因此,温度会导致LDH蛋白酶的残基波动性升高,进而造成蛋白整体构象的不稳定性,这不利于LDH与底物分子的结合,这些结果都证明温度越高对该蛋白酶的影响越大。
2.4 蛋白骨架α-C的回旋半径差异分析
每个氨基酸残基都有一个α-C,而α-C的回旋半径能够代表该蛋白在盐溶液中的结构致密性。因此,统计了LDH的回旋半径随80 ns模拟时间而变化的情况,其值越大表示蛋白构象越松散,其值越小则构象越致密和稳定[31]。如图4所示,37 ℃和55 ℃的两个体系中,LDH的α-C的回旋半径值是最低的,分别为1.85 nm和1.86 nm;而在LDH的熔解温度临界值70 ℃条件下,α-C的回旋半径值升高至1.90 nm,该数据表明此时LDH蛋白正处于膨胀体系、结构开始改变;85 ℃条件下的蛋白结构已经遭到剧烈的破坏,对应的α-C回旋半径值为1.92 nm。这一结果与RMSF的分析结果保持一致。结果说明,随着温度升高,LDH空间构象由致密变成松散状态,大于70 ℃会导致蛋白出现不同程度的变性。
2.5 蛋白构象的溶剂可及表面积分析
溶剂可及表面积(solvent accessible surface area, SASA)是描述蛋白质与水溶液接触的表面积的物理量,由于温度能够对蛋白质构象造成较大的影响,因此精确计算了四个不同温度条件下,LDH蛋白质整体构象残基的SASA随着时间的变化,并统计了在60~80 ns平衡时间段内SASA的平均值,如图5A-B所示,37 ℃和55 ℃的蛋白整体构象SASA值分别稳定在132.4 nm2和132.2 nm2,而70 ℃和85 ℃的SASA值则分别升高至135.6 nm2及136.7 nm2,这可能是由于埋在蛋白内部的疏水基团被暴露在蛋白表面所致。同时,采用gmx make_ndx将丙酮酸结合位点残基Arg106、Arg169、His193和Thr247归为一个组,进而统计了该组对应的数据,如图5C展示了丙酮酸的四个结合位点残基的SASA值,图5D则详细展示了四个温度条件下的SASA平均值分别为11.62、12.01、12.72及12.50 nm2。这些结果说明与正常状态下的LDH结构相比,高温促使丙酮酸结合位点残基的溶剂可及表面积变大,因此高温不利于丙酮酸的结合。
2.6 不同温度影响的蛋白质二级结构变化分析
蛋白质的二级结构会受到温度、电荷、氢键以及配体分子结合的影响,为了明确四种不同温度梯度对LDH二级结构变化的影响,采用do_dssp插件分析了蛋白模拟的轨迹。如图6A,37 ℃条件下平均有139个残基参与形成α−螺旋,56个残基形成无规卷曲,28个残基形成β−转角;与37 ℃相比,55 ℃条件下平均有135个残基参与形成α−螺旋,55个残基形成无规卷曲,25个残基形成β−转角(图6B),二级结构含量的变化均较小,因此55 ℃对LDH活性的影响较小。
与37 ℃体系形成鲜明对比的是70 ℃和85 ℃,二级结构在高温条件下受到较大的改变。主要体现在α−螺旋、无规卷曲、β−转角含量的改变,即在70 ℃仅有123个残基形成LDH的α−螺旋,28个残基形成β−转角,而无规卷曲的数量则升高到61个(图6C);在85 ℃体系中,仅有112个残基形成α−螺旋,32个残基形成β−转角,68个残基形成了无规卷曲(图6D)。
根据这些结果,可以得知85 ℃体系的α−螺旋含量是四个温度中最低的,无规卷曲含量是最高的。这说明高温促使LDH蛋白内部的二级结构出现根本性的转变,从而改变了空间构象,破坏了蛋白质结构稳定性,最终使酶丧失活性。
2.7 对比37 ℃和85 ℃条件下LDH蛋白构象差异以及丙酮酸的结合能力
上述的数据已证明温度能够影响LDH的空间结构变化,温度过高甚至会导致蛋白变性,这会导致LDH在食品发酵过程中酶活性降低,而丙酮酸作为该酶的底物分子之一,其与LDH的结合亦会受到抑制。计算37 ℃和85 ℃条件下的丙酮酸结合位点残基Arg106、Arg169、His193和Thr247的空间位置信息,并利用PymoL软件进行作图分析,如图7所示,结果显示丙酮酸分子在37 ℃时与LDH结合紧密,而85 ℃时的结合位点残基之间的空间距离较大,这不利于丙酮酸分子的结合,进而抑制了LDH催化的丙酮酸生成乳酸的过程。
3. 讨论与结论
本研究以干酪乳杆菌的LDH蛋白为切入点,采用分子动力学模拟的手段探讨了37、55、70和85 ℃温度梯度对LDH的稳定性及酶活性的影响。LDH在低于70 ℃时,结构比较稳定。但是当温度升高至70 ℃或85 ℃,蛋白质结构开始变得松散、不稳定,二级结构含量也被改变。主要表现在,RMSD和回旋半径值显著增加,RMSF值显示不稳定残基数变多,埋藏在蛋白内部的β-折叠疏水基团暴露,与37 ℃条件相比,70 ℃和85 ℃分别导致蛋白整体溶剂可及表面积增加了3.2 nm2和4.3 nm2。更重要的是,37 ℃条件下LDH的丙酮酸活性位点为Arg106、Arg169、His193和Thr247,但是70 ℃以上的温度导致蛋白变性之后,Arg106与Arg169、His193与Thr247的空间距离均不同程度增大,这意味着LDH活性中心微环境遭到破坏,无法结合丙酮酸;而且LDH的α−螺旋含量在高温条件下骤然下降、无规卷曲含量升高,说明高温是导致蛋白空间构象改变的重要原因,这提示在食品发酵各个环节需要严格控制温度低于70 ℃。总之,本研究揭示了温度影响LDH酶活性及构象变化的关键信息,为乳酸制品在发酵过程中选择合适温度提供了理论支撑。
-
-
[1] DELCENSERIE V, MARTEL D, LAMOUREUX M, et al. Immunomodulatory effects of probiotics in the intestinal tract[J]. Current Issues in Molecular Biology,2008,10(1-2):37−54.
[2] ZHU Z Y, CUI D, GAO H, et al. Efficient synthesis and activity of beneficial intestinal flora of two lactulose-derived oligosaccharides[J]. European Journal of Medicinal Chemistry,2016,114:8−13. doi: 10.1016/j.ejmech.2016.03.007
[3] WANG L, ZHANG Y, FAN G, et al. Effects of orange essential oil on intestinal microflora in mice[J]. Journal of the Science of Food and Agriculture,2019,99(8):4019−4028. doi: 10.1002/jsfa.9629
[4] 尤可言, 荒草. 自制酸奶和市售酸奶[J]. 少儿科技,2019,182,183(Z2):20−20. [YOU K Y, HUANG C. Homemade yogurt and commercially available yogurt[J]. Children's Science and Technology,2019,182,183(Z2):20−20. [5] 刘学云, 于新, 何嘉敏, 等. 九种益生菌之间的相互作用及协同共生机理[J]. 食品与发酵工业,2019,45(13):65−70. [LIU X Y, YU X, HE J M, et al. Interactions between nine probiotics and mechanisms of cooperative symbiosis[J]. Food and Fermentation Industry,2019,45(13):65−70. [6] 王希, 洪鲲, 赵玉丹, 等. 乳酸发酵第2阶段能量释放生物学教学研究[J]. 生物学通报,2019(2):43−45. [WANG X, HONG K, ZHAO Y D, et al. Teaching research on energy release biology in the second stage of lactic acid fermentation[J]. Biology Bulletin,2019(2):43−45. doi: 10.3969/j.issn.0006-3193.2019.02.018 [7] BUJNA E, NIKOLETTA A F, TRAN A M, et al. Lactic acid fermentation of apricot juice by mono- and mixed cultures of probiotic Lactobacillus and Bifidobacterium strains[J]. Food Science and Biotechnology,2018,27(2):547−554.
[8] GODERSKA K. The antioxidant and prebiotic properties of lactobionic acid[J]. Applied Microbiology & Biotechnology,2019,103(9):3737−3751.
[9] WANGA M, CHENA Y, WANGC Y, et al. Beneficial changes of gut microbiota and metabolism in weaned rats with Lactobacillus acidophilus NCFM and Bifidobacterium lactis Bi-07 supplementation[J]. Journal of Functional Foods,2018,48:252−265. doi: 10.1016/j.jff.2018.07.008
[10] 钟秀斌, 邓申彪, 沈洋. 一种乳酸菌发酵的温度控制装置: 中国, CN211199202U[P], 2020.08. ZHONG X B, DENG S B, SHEN Y. A temperature control device for lactic acid bacteria fermentation: China, CN21119902u [P]. 2020.08.
[11] 岳林芳, 王俊国, 萨如拉, 等. 培养条件对乳酸菌发酵剂抗冷冻干燥性能影响的研究进展[J]. 食品科学,2016,7(11):270−276. [YUE L F, WANG J G, SA R L, et al. Effects of culture time, temperature and medium composition on freeze drying resistance of lactic acid bacteria starter[J]. Food Science,2016,7(11):270−276. doi: 10.7506/spkx1002-6630-201611047 [12] 侯若冰, 陈志达, 卞江, 等. L-乳酸脱氢酶催化反应机理的理论研究进展[J]. 化学通报,2000(1):15−21. [HOU R B, CHEN Z D, BIAN J, et al. Progress in theoretical research on catalytic reaction mechanism of L-lactate dehydrogenase[J]. Chemical Bulletin,2000(1):15−21. doi: 10.3969/j.issn.0441-3776.2000.01.004 [13] DE BEER D, TOBIN J, WALCZAK B, et al. Phenolic composition of rooibos changes during simulated fermentation: Effect of endogenous enzymes and fermentation temperature on reaction kinetics[J]. Food Research International,2019,121(JUL.):185−196.
[14] 蔡沛蓉, 冯楠楠, 郑豪, 等. 玉米赤霉烯酮对大鼠睾丸支持细胞乳酸产生及相关蛋白表达的影响[J]. 南京农业大学学报,2019,42(5):911−916. [CAI P R, FENG N N, ZHENG H, et al. Effect of zearalenone on lactic acid production and expression of related proteins in Sertoli cells[J]. Journal of Nanjing Agricultural University,2019,42(5):911−916. doi: 10.7685/jnau.201812027 [15] 鲍志伟, 苏晓, 杨柳婷, 等. 重组大肠杆菌全细胞合成D-苯基乳酸[J]. 食品与发酵工业,2019,45(1):49−53. [BAO Z W, SU X, YANG L T, et al. Biocatalytic production of D-phenyllactic acid by using whole cells of recombinant Escherichia coli[J]. Food and Fermentation Industries,2019,45(1):49−53. [16] BLECKWEDEL J, MOHAMED F, MOZZI F, et al. Major role of lactate dehydrogenase D-LDH1 for the synthesis of lactic acid in Fructobacillus tropaeoli CRL 2034[J]. Applied Microbiology and Biotechnology,2020,104(3):7409−7426.
[17] ANDRES J, MOLINER V, KRECHL J, et al. A PM3 quantum chemical study of the pyruvate reduction mechanism catalyzed by lactate dehydrogenase[J]. Bioorganic Chemistry,1993,21(3):260−274. doi: 10.1006/bioo.1993.1022
[18] ASLAN A S, BIRMINGHAM W R, KARAGULER N G, et al. Semi-rational design of Geobacillus stearothermophilus l-lactate dehydrogenase to access various chiral alpha-Hydroxy acids[J]. Applied Biochemistry and Biotechnology,2016,179:474−484. doi: 10.1007/s12010-016-2007-x
[19] ZHANG Y, TAN H, ZHAO J X, et al. Structural change from homogenous structure to staging in benzoic acid intercalated LDH: Experimental and molecular dynamics simulation insights[J]. Physical Chemistry Chemical Physics,2012,14(25):9067. doi: 10.1039/c2cp40674h
[20] CAO K, LI N, WANG H, et al. Two zinc-binding domains in the transporter AdcA from Streptococcus pyogenes facilitate high-affinity binding and fast transport of zinc[J]. Journal of Biological Chemistry,2018:6075−6089.
[21] CAO K, ZHANG J, MIAO X Y, et al. Evolution and molecular mechanism of PitAs in iron transport ofStreptococcus species[J]. Journal of Inorganic Biochemistry,2018:113−123.
[22] 曹剑, 曹赞霞, 赵立岭, 等. 分子动力学模拟Cu2+对α-突触核蛋白(1-17)肽段构象变化的影响[J]. 物理化学学报,2012(2):479−488. [CAO J, CAO Z X, ZHAO L L, et al. Effect of Cu2+ on conformational changes of α-synuclein (1-17) peptide by molecular dynamics simulation[J]. Acta Physicochemical Sinica,2012(2):479−488. doi: 10.3866/PKU.WHXB201111231 [23] VERMAAS J V, HARDY D J, STONE J E, et al. TopoGromacs: Automated topology conversion from CHARMM to Gromacs within VMD[J]. Journal of Chemical Information and Modeling,2016,56(6):1112−1116. doi: 10.1021/acs.jcim.6b00103
[24] ADAMS M J, FORD G C, KOEKOEK R, et al. Structure of lactate dehydrogenase at 2.8 Å resolution[J]. Nature,1972,227(5263):1098−1103.
[25] ZHENG Y, GUO S, GUO Z, et al. Effects of N-terminal deletion mutation on rabbit muscle lactate dehydrogenase[J]. Biochemistry (00062979),2004,69(4):401−406.
[26] UCHIKOBA H, FUSHINOBU S, WAKAGI T, et al. Crystal structure of non-allosteric L-lactate dehydrogenase from Lactobacillus pentosus at 2.3 A resolution: specific interactions at subunit interfaces[J]. Proteins-structure Function & Bioinformatics,2010,46(2):206−214.
[27] ARAI K, ISHIMITSU T, FUSHINOBU S, et al. Active and inactive state structures of unliganded Lactobacillus casei allosteric L-lactate dehydrogenase[J]. Proteins-structure Function & Bioinformatics,2010,78(3):681−694.
[28] 冯涛, 刘芳芳, 荣志伟, 等. 基于分子动力学模拟的直链淀粉风味分子包合物形成机理的研究[J]. 现代食品科技,2015(3):126−132. [FENG T, LIU F F, RONG Z W, et al. Formation mechanism of amylose flavor molecular inclusion complex based on molecular dynamics simulation[J]. Modern Food Science and Technology,2015(3):126−132. [29] GREEN S R, STOREY K B. Regulation of crayfish, Orconectes virilis, tail muscle lactate dehydrogenase (LDH) in response to anoxic conditions is associated with alterations in phosphorylation patterns[J]. Comparative Biochemistry & Physiology Part B,2016,202:67−74.
[30] 陈娇, 王玉丽, 徐为人, 等. 分子动力学模拟法研究糖类衍生物与钠-葡萄糖协同转运蛋白2的相互作用[J]. 中草药,2013,44(10):1440−1447. [CHEN J, WANG Y L, XU W R, et al. Study on the interaction between carbohydrate derivatives and sodium glucose cotransporter 2 by molecular dynamics simulation[J]. Chinese Herbal Medicine,2013,44(10):1440−1447. [31] 丁伟, 刘国宇, 于涛, 等. 分子动力学模拟HEWL晶体在不同环境中的动力学行为[J]. 计算机与应用化学,2010,27(2):173−178. [DING W, LIU G Y, YU T, et al. The molecular dynamics simulation of HEWL in different conditions[J]. Computers and Applied Chemistry,2010,27(2):173−178. doi: 10.3969/j.issn.1001-4160.2010.02.009 -
期刊类型引用(6)
1. 焦艳娜,彭梦香,梁香,毛宪,肖中宁,曾亮,刘仲华. 茶叶中吡咯里西啶生物碱残留量检测方法及研究现状. 中国茶叶. 2024(03): 12-20 .  百度学术
百度学术
2. 佟晓波,刘广福,周政秀. 吡咯里西啶类生物碱的提取与定量分析研究. 粮食与油脂. 2024(04): 158-162 .  百度学术
百度学术
3. 张新娜,马丽艳. QuEChERS结合高效液相色谱-串联质谱法检测甘草中吡咯里西啶生物碱与风险分析. 食品安全质量检测学报. 2024(08): 314-321 .  百度学术
百度学术
4. 章豪,杨挺,吴银良,朱勇. 固相萃取-超高效液相色谱-串联质谱法测定菊花中15种吡咯里西啶生物碱毒素. 食品安全质量检测学报. 2024(21): 41-49 .  百度学术
百度学术
5. 陈言凯. 固相萃取结合超高效液相色谱-串联质谱法快速测定乳粉及液体乳中26种吡咯里西啶生物碱的含量. 食品科学. 2024(24): 266-272 .  百度学术
百度学术
6. 杨方,刘少明. 基于RASFF通报分析技术性贸易措施对商品茶出口贸易的影响. 食品安全质量检测学报. 2024(23): 192-199 .  百度学术
百度学术
其他类型引用(1)





 下载:
下载:






 下载:
下载:



